Pró-Reitoria Acadêmica
Escola de Saúde
Programa de Pós-Graduação Stricto Sensu em Ciências
Genômicas e Biotecnologia
Estratégia molecular aplicada ao controle do
bicudo-do-algodoeiro: superexpressão de toxina Cry e moléculas de
dsRNA
Autora: Stéfani Kárita Lima da Silva Martins
Orientadora: Profa. Dra. Maria Fátima Grossi de Sá
Co-orientador: Prof. Dr. Leonardo Lima Pepino Macedo
STÉFANI KÁRITA LIMA DA SILVA MARTINS
ESTRATÉGIA MOLECULAR APLICADA AO CONTROLE DO BICUDO-DO-ALGODOEIRO: SUPEREXPRESSÃO DE TOXINA CRY E MOLÉCULAS DE
dsRNA
Dissertação apresentada ao Programa de Pós- Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador (a): Profa. Dra. Maria Fátima Grossi de Sá
Co-orientador: Prof. Dr. Leonardo Lima Pepino Macedo
AGRADECIMENTO
Agradeço ao meu Deus, por me escolher para conquistar os sonhos que Ele sonhou para mim. Eu sou muito grata por tanto amor demonstrado em cada passo que eu dei. Passos estes, guiados na dependência Dele. Obrigada Pai, por acreditar em mim.
Agradeço ao meu esposo abençoado, pelo companheirismo durante esses 730 dias de fortes emoções!!! A certeza de poder abraçá-lo ao fim de todos os dias, me fortaleceu bastante, acredite! À minha família: Mãe e pai, amo vocês. Agradeço por tudo mesmo. Irmã, obrigada por me ajudar a ensaiar cada apresentação e me fazer repetir mil vezes a mesma frase, até que ficasse “bonito” (risos), obrigada!
Agradeço às minhas colegas, que se tornaram amigas, “psicólogas”, “professoras”, irmãs e muito mais: Liana, Uriele, Rayssa, Rayanne, Rayanne Luzia, Kellyane, Jéssica, Isis, Thuanne, Priscila, Vânia, Isabela, Janaína, Katiane, Fernanda, Alessandra, Sineide, Maria Eugênia, Regina e Janny. Por todos os momentos de estudo, conversa e pelo carinho durante esses anos.
Aos amigos que lutaram comigo até o fim: Laene, Ítalo, Amanda, Meiry, Victor Hugo, Aloísa, Fran, Cestari, Samara, Gabrielle, Carolina, Shaiene, Camila, Daniela, Wanda, Lorena. Obrigada!!!!
Agradeço aos meus professores e orientadores, Maria Fátima e Leonardo, por esta oportunidade única. Obrigada pela paciência e pela confiança que depositaram em mim.
RESUMO
MARTINS, Stéfani Kárita Lima da Silva. Estratégia molecular aplicada ao controle do bicudo-do-algodoeiro: superexpressão de toxinas Cry e silenciamento
gênico. 2014. 76 folhas. Mestrado em Ciências Genômicas e Biotecnologia – Universidade Católica de Brasília, 2014.
O Algodão (Gossypium hirsutum) é uma das principais commodities agrícolas, sendo o Brasil o quinto maior produtor mundial. Dentre os diversos insetos-praga que atacam a cultura, o bicudo-do-algodoeiro (Anthonomus grandis) é o mais destrutivo e, devido a seu hábito endofítico, a ação de agrotóxicos é dificultada. Além disso, não há no mercado, nenhum evento transgênico resistente a este inseto-praga. O presente estudo tem por objetivo validar, em plantas de algodão geneticamente modificadas, o potencial uso de genes envolvidos na toxicidade e no desenvolvimento do inseto, por meio de duas abordagens biotecnológicas piramidizadas: a superexpressão de moléculas inseticidas e o silenciamento gênico. A estratégia de piramidização de genes foi construída de modo que a expressão da toxina Cry8Ka5 está controlada pelo promotor específico de botão floral- (GhPGFS1) e a expressão do dsRNA para o silenciamento gênico, por meio do RNAi da Quitina sintase II (CHS2) e da vitelogenina do bicudo, que está sob controle do promotor constitutivo UCeA 1.7. Ademais, o gene de seleção ahas, que confere resistência ao herbicida Imazapir, faz parte do cassete gênico utilizado na transformação das plantas. Foram bombardeados 3.500 embriões, os quais originaram 19 eventos positivos detectados por PCR, conferindo uma eficiência de transformação de 0,54%. Destes, onze eventos mostraram expressão da proteína Cry8Ka5 quantificada pela técnica de ELISA, cujos níveis variaram entre 8-90 g/g de tecido fresco. Na geração T1, obtida por autofecundação, sete eventos mostraram a presença dos três transgenes e, dentre estes, foram selecionados os eventos T1 do parental número 7 (T0), que expressou 60 g de toxina Cry8Ka5/g de tecido floral. A caracterização molecular avaliada por análises de qRT-PCR mostrou que larvas/insetos que ingeriram tecidos dessas plantas tiveram uma redução da expressão quantitativa de CHS2 em larvas de A. grandis alimentadas com o evento 7.5 (T1), que foi 6 vezes menor do que no controle. Estes resultados demonstraram que a tecnologia de piramidização de genes pode ser usada para engenheirar cultivares de algodão resistentes a este inseto-praga.
ABSTRACT
Applied molecular strategy in the control of the cotton boll weevil: overexpression of Cry toxins and gene silencing.
Cotton (Gossypium hirsutum) is one of the main agricultural commodities and Brazil
is the fifth major producer worldwide. Among several insect pest that attacks the crops, the cotton boll weevil (Anthonomus grandis) is the most destructive and due its endophytic habit, the pesticide action is hampered. Moreover, there is no transgenic plant resistant to this particular insect pest current on the market. The present study aimed to validated, in genetic modified cotton plant, the potential use of genes involved in the insect’s toxicity and development, by using two biotechnological approaches: the overexpression of inseticides and gene silencing. The gene pyramiding strategy was built such as, a flower bud specific promoter (GhPGFS1) controlled the Cry8Ka5 toxin expression, and the constitutive promoter UCeA 1.7, controlled the dsRNA expression for the RNAi gene silencing of the quitin sintase II (CHS2) and vitelogenin of the cotton boll weevil. Furthermore, the ahas selection gene, which confers resistance to the herbicide Imazapir, is part of the gene cassett used in the processing plant. 3.500 embryos were bombarbed, which originated 19 positive events detected by PCR, granting a 0.54% of transformation efficiency. Of those, eleven events showed Cry8Ka5 protein expression quantified by ELISA, whose levels ranged from 8-90 g/g of fresh tissue. After obtaining the T1 generation by self-pollination, only seven events showed the presence of the tree transgenes, and, among them, the T1 events from the parental number 7 (T0) were selected, for expressing 60 g of Cry8Ka5 toxin per gram of bud tissue. The molecular characterization by qRT-PCR analysis showed that the ingestion of these plants reduced the CHS2 quantitative expression of A. grandis larvae feed by the 7.5 (T1) event, which was 6 times lower than the control. These results show that the gene pyramiding technology could be used to engineer cotton cultivars resistant to this insect pest.
LISTA DE ILUSTRAÇÕES
Figura 3. Estrutura tridimensional da toxina
Cry1Aa...8
Figura 4. Esquema ilustrando o modelo de formação de poros para a ação das toxinas Cry...9
Figura 5. Esquema ilustrando o modelo de transdução de sinal para a ação das toxinas Cry...10
Figura 6. Esquema do mecanismo molecular da via do RNAi...13
Figura 7. Representação esquemática do cassete de expressão contendo o gene de seleção Ahas, o gene para super-expressão do dsRNA-CHS2vit e Cry8Ka5...23
Figura 8. Placa para cultura de células contendo botões florais inoculados por larvas de bicudo-do-algodoeiro...31
Figura 9. Organograma metodológico da transformação, caracterização e desafio de plantas de algodão GM...34
Figura 10. Eventos T0 detectados por PCR para o gene dsRNA...37
Figura 11. Eventos T0 detectados por PCR para o gene Cry8Ka5...37
Figura 12. Eventos T0 detectados por PCR para o gene AHAS...37
Figura 13. Avaliação da expressão da entomotoxina Cry8Ka5 pela técnica de ELISA...38
Figura 14. Análise morfológica de larvas de instar III de insetos de A. grandis após serem alimentados com plantas de algodão GM contendo a construção pBSK-dsRNAvitCHS2Cry8Ka5...40
LISTA DE TABELAS
Tabela 1. Eventos de algodão GM aprovados no Brasil... 18
Tabela 2. Lista de oligonucleotídeos utilizados na análise de plantas de algodão por PCR... 28
Tabela 3. Oligonucleotídeos utilizados nas reações de PCR em tempo real para análise de CHS2... 33
LISTA DE ABREVIATURAS
µg Micrograma
µL Microlitro
Algodão GM Algodão Geneticamente Modificado Bt Bacillus thuringiensis
cDNA DNA complementar
CHS2 Quitina Sintase II
DNA Ácido desoxirribonucléico
dNTP Deoxinucleotídeo
dsRNA RNA dupla fita
GM Geneticamente modificado
kb Quilobase - 1000 pares de bases
miRNAs dsRNA com 21 a 26 nucleotídeos com bases despareadas
mRNA RNA mensageiro
ºC Graus Celsius
pb Pares de bases
PCR Reação em cadeia da polimerase ("polymerase chain reaction") PIB Produto Interno Bruto
Primers Oligonucleotídeos iniciadores
qRT-PCR RT-PCR quantitativa (em tempo real) RNA Ácido ribonucléico
RNAi RNA interferente
RT-PCR Transcrição reversa seguida de PCR
SUMÁRIO
1. INTRODUÇÃO ... 1
1.1 A biodiversidade de insetos ... 1
1.1.1 Os insetos-praga e os danos agrícolas ... 1
1.2 O bicudo-do-algodoeiro e a cotonicultura ... 2
1.3 Estratégias moleculares para a obtenção das plantas geneticamente modificadas para o controle de insetos-praga ... 4
1.3.1 Superexpressão de δ-endotoxinas de Bacillus thuringiensis (Bt) ... 6
1.3.2 O uso do RNA interferente (RNAi) em plantas para o controle de insetos-praga... ... 10
1.3.3 Silenciamento gênico por RNAi da Quitina Sintase II e da Vitelogenina do bicudo-do algodoeiro ...13
1.3.4 Expressão tecido-específica com a utilização de promotores...16
1.4 Métodos de transformação genética de plantas mediada por agrobacterium tumefaciens e por biobalística ... 16
1.5 Algodão GM ... 17
1.6 JUSTIFICATIVA ... 20
2. OBJETIVO ... 22
2.1 Objetivo geral ... 22
2.2 Objetivos específicos ... 22
3. METODOLOGIA ... 23
3.1. Construção gênica ... 23
3.2 Transformação genética via biobalística ... 23
3.3.2 Aplicação da técnica de bombardeio de DNA ... 24
3.4. CARACTERIZAÇÃO MOLECULAR DE PLANTAS POTENCIALMENTE
TRANSFORMADAS ... 26
3.4.1 Extração de DNA ... 26
3.4.2 Verificação da amplificabilidade do DNA extraído de folhas de plantas de algodão ...26
3.4.3 Extração e determinação de proteínas totais ... 28
3.4.4 Quantificação da expressão da entomotoxina Cry8Ka5 por meio de ensaio imunoenzimático do tipo ELISA ... 28
3.5 DESAFIO DAS PLANTAS T1 COM O INSETO-PRAGA ... 29
3.5.1 Extração de RNA total de larvas de A. grandis alimentadas por botão floral transgênico. ... 30
3.5.2 Síntese de cDNA... 31
3.5.3 Confirmação da síntese do DNA complementar (cDNA) ... 32
3.5.4 Análise do silenciamento do gene alvo do ds RNA por qPCR ... 32
4. RESULTADOS ... 35
4.1 Bombardeamento de embriões ... 35
4.3 Quantificação da expressão de entomotoxina Cry8Ka5 por meio do ensaio imunoenzimático do tipo elisa ... 38
4.4 Identificação de genes por pcr de geração T1 ... 38
4.5 Desafio das plantas gm com o inseto-alvo ... 39
5. DISCUSSÃO ... 43
6. CONCLUSÃO E PERSPECTIVAS ... 48
1. INTRODUÇÃO
1.1 A BIODIVERSIDADE DE INSETOS
Os insetos compõem um grupo de animais muito diversificado (TRIPLEHORN; JOHNSON, 2005), visto ser três vezes maior que os demais grupos de animais (RUPERT; BARNES, 1996). Estima-se que sejam descritos aproximadamente um milhão de espécies de insetos, classificados em 32 ordens e cerca de 762 famílias (DELVARE; ABERLENC, 1989; DALY et al., 1998; GALLO et al., 2002). Trata-se do grupo mais bem sucedido evolutivamente, devido às características adaptativas, incluindo, presença de exoesqueleto quitinoso, alta taxa reprodutiva em curto ciclo de vida, desenvolvimento indireto (metamorfose), dentre outros (GULLAN; CRANSTON, 1994; RUPERT; BARNES, 1996; ALMEIDA et al., 2002; GILLOTT, 2005).
Podendo interagir de modo benéfico ou maléfico com o homem e outros organismos, os insetos executam papéis importantes e intransferíveis. Dentre os insetos considerados benéficos, encontram-se os que são capazes de produzir mel, seda e laca, os capazes de polinizar plantas, os que atuam como agentes de controle biológico, além dos que auxiliam na decomposição de matéria orgânica. Contrariamente, além de atuarem como vetores de patógenos para diferentes organismos, existem também insetos que se tornaram os grandes inimigos dos agricultores pelas incontáveis perdas, ano após ano, em diversos cultivares, resultando em prejuízos significativos para a agricultura (GILLOTT, 2005).
1.1.1 Os insetos-praga e os danos agrícolas
para a agricultura é o nível dos danos e perdas que a população de determinado inseto será capaz de gerar, sem que haja controle, baseando-se na relação entre praga e dano (HILL, 1997). Os aspectos problemáticos, resultantes de pragas agrícolas, têm sua principal razão devido à prática de monoculturas, visto que, se reduz intensamente a variedade faunística e simplifica-se a flora do local (LUTZENBERGER, 1978).
Diversas são as plantações que sofrem com o descontrole de insetos que, por sua vez, já se tornaram pragas. A ação descontrolada dos insetos altera os processos fisiológicos da planta e reflete no custo de produção, causando danos diretos ao atacar o produto a ser comercializado; ou indiretos, quando atacam estruturas vegetais que não poderão ser comercializadas (BENTO, 1999; GALLO et
al., 2002; GILLOTT, 2005).
1.2 O BICUDO-DO-ALGODOEIRO E A COTONICULTURA
No Brasil, o cultivo do algodão é considerado uma das plantações mais susceptíveis ao desenvolvimento e ataque de insetos-praga, com mais de 20 espécies relatadas e conhecidas (GALLO et al., 2002). A planta de algodão possui inúmeras glândulas denominadas nectários, que, por sua vez, têm a capacidade de produzir uma secreção líquido-resinosa açucarada, tornando o algodoeiro bastante atrativo aos insetos (MONNERAT et al., 2000). Os insetos-praga que atacam o algodoeiro podem ser divididos em dois grupos: os que ocorrem no estabelecimento da cultura (broca-da-raiz, bronca-do-ponteiro, pulgão) e as que ocorrem no florescimento e na frutificação (curuquerê, mosca branca, bicudo-do-algodoeiro) (MAPA, 2007).
O bicudo-do-algodoeiro, da subfamília Anthonominae, Anthonomus grandis (Coleoptera: Curculionidae), é considerada uma das ameaças agrícolas mais prejudiciais para a cotonicultura do Brasil.
diretamente na desvalorização de propriedades, no desemprego e na emigração de produtores para regiões onde se concentram as grandes cidades (RAMALHO, et al. 2000).
O bicudo-do-algodoeiro, chega a medir aproximadamente 7 mm de comprimento na fase adulta. Uma fêmea adulta com uma vida média de 20-40 dias é capaz de ovipositar cerca de 300 ovos, que poderá produzir outros 10.000 insetos ao final de uma safra (BARBOSA et al., 1983). Seu hábito endofítico resulta em perdas exorbitantes da cultura de algodão, visto que estes insetos se desenvolvem no interior de botões florais e maçãs de plantas de algodão, destruindo-o totalmente (HAYNES; SMITH, 1992). Uma vez que a oviposição das fêmeas ocorre no interior destas estruturas vegetais, a progênie encontra-se relativamente protegida, até a sua emergência em fase adulta, devido à sua permanência no interior do botão floral (BRAGA SOBRINHO; LUKEFAHR, 1983).
A detecção de densidades populacionais deste inseto-praga acima dos níveis aceitáveis, quando não é seguido da medida de controle, pode inviabilizar o cultivo do algodoeiro (BASTOS et al., 2005), devido à sua alta capacidade de proliferação. Além disso, a migração e a dispersão deste inseto é bastante facilitada pelo vento, o que tem levado a atingir o status de praga chave da cotonicultura (STADLER; BUTELER, 2007).
Apesar de não ser totalmente eficaz, o controle do bicudo-do-algodoeiro é largamente voltado para a aplicação de produtos químicos, os quais resultam em danos significativos à entomofauna benéfica, além dos riscos toxicológicos ao homem e, elevação do custo de produção (DEGRANDE et al., 2003; RAMALHO; DIAS, 2003). Adicionalmente, também tem sido usada pelos agricultores, a catação manual de botões florais e maçãs caídos no solo, a eliminação de plantas nas margens de estradas, a limpeza severa de máquinas e veículos de transporte, além do intenso monitoramento e detecção de populações do bicudo (AMPASUL, 2014).
utilizados no Brasil e, ainda mais alarmante é que grande parte desses produtos são usados para o controle do bicudo-do-algodoeiro (JAMES, 2013).
1.3 ESTRATÉGIAS MOLECULARES PARA A OBTENÇÃO DAS PLANTAS
GENETICAMENTE MODIFICADAS PARA O CONTROLE DE
INSETOS-PRAGA
Controlar a população de A. grandis nas plantações de algodão tem sido considerado prioritário para a cotonicultura do Brasil. Devido ao hábito endofítico do inseto, mais especificamente em botão floral, as atuais formas de controle são ineficientes, demandando a utilização de meios alternativos (ALMEIDA; SILVA, 1999; DEGRANDE et. al, 2002). O uso de ferramentas biotecnológicas, visando o desenvolvimento de plantas geneticamente modificadas (GM) resistentes a insetos-praga tem se mostrado uma alternativa eficaz para evitar o intenso uso de agroquímicos.
Há quase duas décadas (de 1996 a 2013), a economia de diversos países tem desfrutado dos benefícios decorrentes da adoção das tecnologias GM em níveis, sem precedentes. No último ano, 175,2 milhões de hectares de variedades transgênicas foram cultivadas mundialmente, tornando a tecnologia agrícola mais adotada na história moderna. O Brasil tornou-se um forte líder global na produção de transgênicos, ocupando uma área de 40,3 milhões de hectares (ha), ou seja, 23% da área mundial, perdendo apenas para os Estados Unidos. O algodão é a terceira principal cultura transgênica produzida no mundo, sua adoção comercial por quase todos os cultivares é resultado dos diversos benefícios gerados após a utilização desta tecnologia, o que resultou num aumento considerável da produção, redução da exposição dos cultivares a inseticidas e, por conseqüência, diminuição dos custos de produção (JAMES, 2013).
Tabela 1. Eventos de algodão GM aprovados no Brasil.
(CTNBIO, 2014).
Evento Gene inserido Característica Companhia
Bollgard Cry1ac Resistência a insetos Monsanto
Bollgard II Cry1Ac +
Cry2Ab2 Resistência a insetos Monsanto
WideStrike Cry1ac + Cry1f Resistência a insetos/Tolerância a
herbicida Dow AgroSciences
GlyTol™ 2mepsps Tolerância a herbicida Bayer CropScience
GlyTol™ Liberty
Link™ 2mepsps + Bar Tolerância a herbicidas Bayer CropScience
Glytol™ x Twinlink™
2mepsps + Bar
+ Cry1Ab
+Cry2ae
Resistência a insetos/Tolerância ao
herbicida Bayer CropScience
Fibermax™ Liberty
Link™ Bar Tolerância a herbicidas Bayer CropScience
RoundUp Ready
(RR) CP4 epsps Tolerância a herbicida Monsanto
Roundup Ready™ Bollgard II™ Cotton
CP4 epsps+
Cry1Ac +
Cry2ab2
Resistência a insetos/Tolerância a
herbicida Monsanto
Roundup Ready™
Flex™ Cotton CP4 epsps Tolerância a herbicida Monsanto
Roundup Ready™ Flex™
Bollgard II™ Cotton
CP4 epsps+
Cry1Ac +
Cry2ab2
Resistência a insetos/Tolerância a
herbicida Monsanto
TwinLink™ Cotton
Bar + Cry1Ab
+Cry2ae
Resistência a insetos/Tolerância a
Apesar das inúmeras variedades transgênicas de algodão disponíveis no mercado, atualmente não existe nenhum evento disponível comercialmente que apresente resistência ao A. grandis. Dessa forma, há forte demanda para o desenvolvimento de estratégias específicas para o controle deste inseto-praga. A busca por moléculas que resultem em variedades transgênicas de algodão resistentes ao bicudo-do-algodoeiro tem sido o alvo de diversos investimentos.
Dentre as diferentes estratégias, visando à obtenção de plantas GM resistentes a insetos-praga, a equipe do Laboratório de Interação Planta Praga da Embrapa Recursos Genéticos e Biotecnologia (LIMPP/Cenargen) tem focado tanto em estratégias baseadas na superexpressão de proteínas entomotóxicas, como na interrupção da expressão de proteínas essenciais do inseto alvo, por meio do silenciamento genético, utilizando a tecnologia do RNA interferente (RNAi).
A manipulação de promotores específicos que promovam a expressão de proteínas heterólogas, em níveis adequados para a letalidade do inseto-praga, também tem sido utilizada no desenvolvimento de plantas transgênicas. Essa especificidade restringe a expressão da proteína somente (ou em maior quantidade) no tecido de interesse (ZHENG; MURAI, 1997; GREEN et al., 2002; NEUTEBOOM
et al., 2002).
1.3.1 Superexpressão de δ-endotoxinas de Bacillus thuringiensis (Bt)
O Bacillus thuringiensis (Bt) é uma bactéria anaeróbia facultativa, Gram-positiva, esporulante, que habita naturalmente o solo. Esse microrganismo possui atividade entomopatogênica devido à presença da inclusão cristalina produzida durante a esporulação. Esse cristal é composto por proteínas denominadas: entomotoxinas Cry. Tais proteínas possuem ampla atividade inseticida por serem extremamente tóxicas a insetos de diversas ordens. Dentre as estratégias de obtenção de plantas GM resistentes a insetos, destaca-se a inserção dessas proteínas Cry (SCHNEPF et al., 1998; MONNERAT; BRAVO, 2000; BRAVO et al., 2007).
células intestinais do inseto-alvo. Existem mais de 120 diferentes genes que codificam para proteínas Cry, distribuídas em 22 classes. Essas toxinas vêm sendo utilizadas como uma ferramenta biotecnológica para o controle de pragas, por meio de sua expressão em plantas GM (MONNERAT; BRAVO, 2000; BRAVO et al., 2005; HECKMANN et al., 2006). Conforme relacionado na Tabela 1, todos os eventos de algodão GM resistentes a insetos, cultivados no Brasil, expressam genes derivados da bactéria Bt.
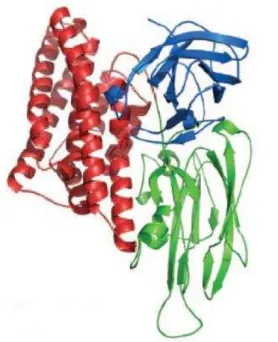
As estruturas tridimensionais de algumas entomotoxinas Cry (Cry1Aa,
Cry1Ac, Cry2Aa, Cry3A, Cry3Bb1, Cry4Aa, Cry4Ba, Cry8Ea1) já foram determinadas (BOONSERM et al., 2005; BOONSERM et al., 2006; DERBYSHIRE et al., 2001; GALITSKY et al., 2001; GROCHULSKI et al., 2005; GUO et al., 2009; LI et al., 1991; MORSE et al., 2001) e, são formadas por três domínios (Figura 1). O domínio I consiste em um feixe de sete hélices antiparalelas, com hélice central hidrofóbica (LI et al., 1991); o domínio II é formado por três folhas antiparalelas e duas hélices curtas, conferindo especificidade de ligação aos receptores (DEAN et al., 1996); e o domínio III consiste de duas folhas antiparalelas, altamente envelopadas, contendo a região C-terminal na maioria das proteínas, e está fortemente envolvido na estabilidade estrutural da molécula inteira, além da especificidade ou formação de poro juntamente com o domínio I (RUKMINI et al., 2000).
Figura 1. Estrutura tridimensional da toxina Cry1Aa, mostrando seus domínios. Adaptado de Piggot e
Ellar, 2007.
Domínio II
Uma vez que essas proteínas são ingeridas pelo inseto susceptível, estas são solubilizadas e clivadas por proteases intestinais capazes de localizar os sítios de clivagem da toxina, tornando-se ativas. Receptores específicos presentes no epitélio intestinal são reconhecidos pelas proteínas Cry, permitindo passagem de água e íons livremente nas células, desencadeando na turgidez, lise e morte do inseto-alvo (KNOWLES; ELLAR, 1987; GILL, 1995; GILL et al., 1992).
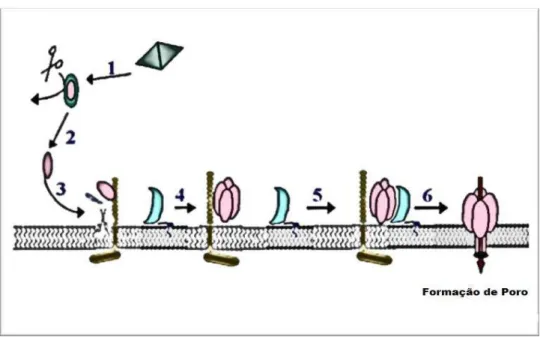
São descritos pelo menos 3 modelos para a ação das proteínas Cry. O modelo de formação de poros (Figura 2) é baseado nos experimentos de interação entre Cry1Ab e as vesículas da membrana apical do epitélio intestinal do inseto lepdóptero Manduca sexta (BRAVO et al., 2004; GÓMEZ et al., 2002; ZHUANG et
al., 2002).Este modelo sugere que, após a interação dos monômeros da toxina ao receptor de membrana, a mesma sofre uma mudança conformacional que facilita a clivagem da hélice por proteases ligadas à membrana. Esta forma da toxina se oligomeriza a fim de formar um pré-poro tetramérico que se liga a um receptor aminopeptidase N, dando início ao processo de formação do poro e morte do inseto.
Figura 2. Esquema ilustrando o modelo de formação de poros para a ação das toxinas Cry. (1)
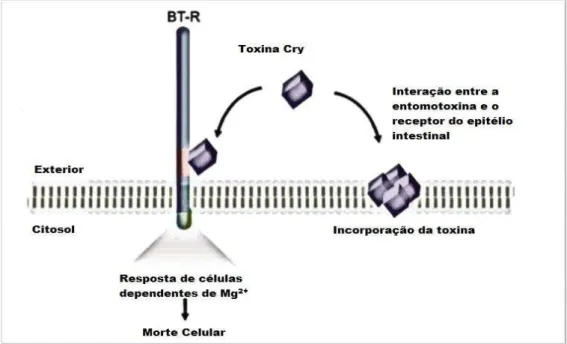
O modelo de transdução de sinal (Figura 3) baseia-se na ligação entre a Cry e o receptor BT-R, iniciando uma cascata de sinalizações dependente de Mg2+, promovendo a morte celular (ZHANG et al., 2005).
Devido a essa atividade entomotóxica, os genes Cry vêm sendo cada vez mais estudados para o desenvolvimento de plantas transgênicas resistentes a insetos-praga. Versões das proteínas Cry já se tornaram passíveis de comercialização e foram inseridas em diversos organismos transgênicos, destacando-se: Cry1Ac (Dekalb DBT418 – Bt XtraTM), Cry9C (Avenis ACS-ZM4-3, StarLink TM) e Cry1Ab (Syngenta Bt176, Bt11; Monsanto Mon80100, Mon802, Mon809, Mon810) para o milho GM. Para o algodão GM, destacam-se: Cry1Ac (Monsanto, Mon531-Bollgard®) e Cry1Ac associado ao Cry2Ab (BollgardII®, DOW 3006, Calgene 31807).
A fim de obter plantas de algodão resistentes ao A. grandis, diversosestudos têm auxiliado na descoberta de genes que codifiquem para proteínas nocivas ao
Figura 3. Esquema ilustrando o modelo de transdução de sinal para a ação das toxinas Cry. O
modelo prediz que a citoxicidade associada com as toxinas Cry depende estritamente de respostas
inseto, provenientes de B. thuringiensis. Dessa maneira, em trabalhos anteriores, o grupo de pesquisa do LIMPP/ Cenargen, isolou e caracterizou três diferentes genes Cry, cujas toxinas codificadas são eficientes no controle de A. grandis: Cry1Ba, Cry1Ia12 (MAGALHÃES, 2006) e Cry8K (OLIVEIRA, 2008). Em 2008, Oliveira e colaboradores construíram uma biblioteca combinatória de mutantes cujos variantes foram isolados do gene Cry8, proveniente da estirpe S811 de Bt, visando selecionar as variantes com atividade melhorada para pragas da cotonicultura, principalmente, o bicudo-do-algodoeiro. A partir disso, foi gerada uma biblioteca recombinada de variantes dessas toxinas, permitindo a seleção de toxinas mais eficazes para o controle de A. grandis.
1.3.2 O uso do RNA interferente (RNAi) em plantas para o controle de insetos-praga
A estratégia de silenciamento gênico, via RNAi, é uma das alternativas que vem sendo utilizada para o controle de pragas, baseando-se na interrupção da síntese protéica de genes vitais para o inseto, por meio da inserção de RNA dupla fita em organismos alvo, através de microinjeção ou ingestão (FIRE et al., 1998). Um dos primeiros estudos realizados para silenciamento gênico foi com o nematóide e organismo modelo Caenorhabditis elegans, onde se observou a capacidade de moléculas de dsRNA em interromper a expressão de determinado gene com sequência similar ao dsRNA (PRICE e GATEHOUSE, 2008; LILLEY et al., 2012). Este fenômeno de silenciamento de genes tem sido considerado como uma estratégia potencial para o controle de insetos-praga, pois a seleção do gene alvo e a síntese de dsRNA resultam o componente crucial da aplicação dessa tecnologia (KATOCH; THAKUR, 2012).
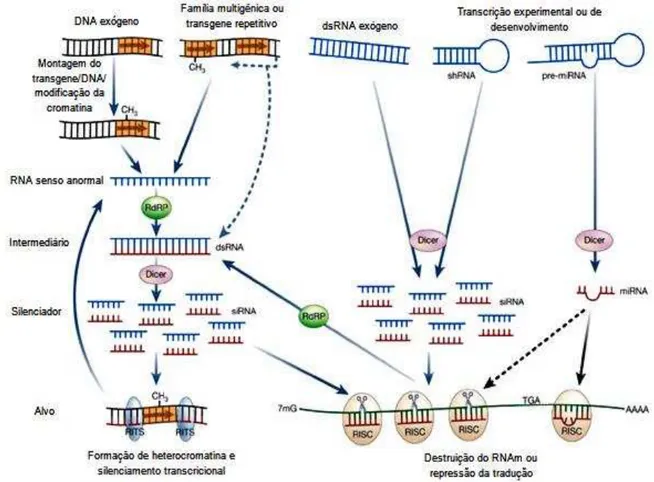
nucleotídeos, denominados siRNA (do inglês - short interfering RNA), por uma ribonuclease do tipo RNAse III, a Dicer. Dependendo do organismo pode existir uma ou mais DICER’s, cada uma responsável pela produção por um diferente tipo de dsRNA (MEISTER e TUSCHL, 2004). Por exemplo, em D. melanogaster, Dicer-1 está envolvida, principalmente, na produção de miRNA; enquanto, Dicer-2 é responsável pelo processamento de dsRNAs longos em siRNA (LEE et al., 2004). Durante a segunda etapa do mecanismo de RNAi, esses duplexs são transportados para o complexo efetor RISC (do inglês - RNA-induced silencing complex), onde ocorre a separação das fitas e uma delas, denominada de fita guia, é preferencialmente acoplada no complexo proteico RISC. Na última etapa, a fita guia é utilizada para reconhecer os RNAm alvos pelo pareamento de bases Watson-Crick. O silenciamento do gene é um resultado da degradação do RNAm alvo pela enzima Argonaute (Ago), uma RNaseH que faz parte do complexo RISC (GAYNOR
et al., 2010). Se o duplex siRNA/mRNA contém bases despareadas no sítio de clivagem, muitas vezes o caso de miRNAs, o RNAm não é clivado. Neste caso, o silenciamento do gene é um resultado da inibição da tradução (HAMMOND, 2005; GHILDIYAL e ZAMORE, 2009).
Figura 4. Esquema do mecanismo molecular da via do RNAi. A via do RNAi é iniciada pela presença de moléculas de dsRNA, as quais podem ser endógenas ou introduzidas experimentalmente. Moléculas de dsRNAs podem ser produzidas pela transcrição de RNA. Todos esses dsRNAs são digeridos pela enzima DICER, em siRNA ou miRNA. Uma das fitas desses pequenos dsRNAs são incorporados ao complexo RISC e induzem a destruição do mRNA alvo ou reprimem sua tradução. Reimpresso de Mello e Conte, 2004. Com permissão de Nature Publishing Group.
expressão do transcrito, resultando no silenciamento gênico pós-transcricional e, por fim, na morte do inseto-alvo pela interrupção de genes específicos e vitais para sua proliferação (BAUM, et al., 2007; MAO, et al., 2007).
A utilização da tecnologia do RNAi tem permitido a compreensão sobre a funcionalidade de genes, bem como sua função regulatória em rotas metabólicas que controlam importantes características, colaborando para a produção de plantas transgênicas que produzam transcritos de interesse (KATOCH; THAKUR, 2013). Diversos são os trabalhos que descrevem o processo de silenciamento gênico como participante da regulação da expressão gênica em alguns eucariotos, inclusive plantas (LILLEY, et al., 2007). Nestes, a interferência por RNA acontece por meio da inserção de vetores capazes de transcreverem o dsRNA (HORIGUCHI, 2004; BELLES, 2010; NOH et al., 2012).
Além do entendimento sobre a regulação da expressão gênica, o dinamismo dessa tecnologia tem surgido como uma poderosa ferramenta para aumento da produção agrícola, como por exemplo, o incremento dos níveis nutricionais em plantas de interesse comercial (KATOCH; THAKUR, 2013). Ademais, a metodologia da interferência por RNA tem sido bastante visada para o controle de insetos seja esta por meio de ingestão de dsRNA ou por microinjeção (ZHOU et al., 2008; WALSHE et al., 2009; CHEN et al., 2010; LI et al., 2011). Nos últimos anos, o silenciamento gênico tem provado seu potencial em inúmeras espécies de importância para o agronegócio, como resultado de um futuro promissor para a agricultura (ZHA et al., 2001; BAUM et al., 2007; MAO et al., 2007). Mao e colaboradores (2011) demonstraram que, plantas de algodão transgênicas expressando o dsRNA do gene do citocromo P450 (CYP6AE14) de H. armigera, resultaram em maior resistência a esta praga, que causa grandes prejuízos não apenas para o algodoeiro, mas também para diversas espécies de importância econômica.
1.3.3 Silenciamento gênico por RNAi da Quitina Sintase II e da Vitelogenina do bicudo-do-algodoeiro
possível identificar rotas metabólicas imprescindíveis ao desenvolvimento deste inseto-praga (FIRMINO et al., 2013). Estudos anteriores relataram a possibilidade de gerar danos no desenvolvimento de insetos, a partir de alterações no metabolismo da quitina, tornando-a excelente candidata para a estratégia de controle específico de insetos-praga (ALVES et al., 2010; AMPASALA et al., 2011; NOH et al., 2012). Outro evidente candidato, conforme dados experimentais, é a vitelogenina que está associada ao ciclo reprodutivo do inseto-alvo (COELHO, 2013).
A quitina é o segundo biopolímero mais abundante na natureza, sendo encontrado na parede celular de fungos, no exoesqueleto de vários crustáceos, dentre outros, principalmente no exoesqueleto de diversas espécies de artrópodes (MUZZARELLI, 1980; MERZENDORFER; ZIMOCH, 2003; MERZENDORFER, 2006). Esse polissacarídeo é formado por uma cadeia linear composta por unidades N-acetil-2-dioxi-D-glicopiranose, interligadas por ligações glicosídicas (1-4), sendo capaz de estruturar diversas porções anatômicas dos insetos. Encontra-se presente na constituição da cutícula, traquéia, na membrana peritrófica, a qual reveste o epitélio intestinal, nas glândulas salivares e nas estruturas bucais, tanto em larvas quanto em adultos de inúmeros insetos, incluindo A. grandis (MUTHUKRISHNAN et
al., 2012; MACEDO, 2012).
Conforme análises realizadas por difração de raios- X, a quitina pode apresentar de três formas cristalinas diferentes denominadas e O que caracteriza as diferenças de seu polimorfismo é o grau de hidratação resultando na diferenciação de suas propriedades físicas. É previsto que haja diferença funcional decorrente dessa diferenciação. A forma é a forma mais dominante e estável, podendo ser encontrada em estruturas que exigem maior resistência, como as cutículas dos artrópodes. No entanto, as formas e encontram-se onde há maior necessidade de flexibilidade, devido à sua estrutura mais relaxada, e essas são encontradas na membrana peritrófica de insetos (RUDALL; KENCHINGTON, 1973; PETERS, 1992).
SNIGIREVSKAYA et al., 1997). Sendo uma das principais reservas de aminoácidos de vertebrados e invertebrados no período inicial de sua formação (TREWITT et al., 1992), a interrupção da produção de vitelogenina no interior dos ovos de A. grandis é uma estratégia promissora para o controle desse inseto-alvo (COELHO, 2013).
1.3.4 Expressão tecido-específica com a utilização de promotores
A manipulação de promotores é uma ferramenta importante para a biotecnologia a fim de garantir que a expressão do gene de interesse seja efetiva. Os promotores mais utilizados na produção de plantas GM são o promotor 35S do Vírus do Mosaico da Couve Flor (CaMv 35S), nopalina sintetase (NOS) e octopina sintetase (OCS), ambos promotores de genes de Agrobacterium tumefaciens, além do promotor de gene presente no milho que codifica para ubiquitina (Ubi-1). Apesar da utilização desses promotores, a expressão de transgenes sob o domínio dos mesmos, é indefinida, podendo ser baixa em certos casos, não havendo garantia de expressão no tecido de interesse. A utilização de promotores de genes constitutivos, como os que codificam a ubiquitina, por exemplo, não é desejável em todos os casos, já que ativam a expressão gênica do transgene em todos os tecidos da planta (ZHENG; MURAI, 1997; GREEN et al., 2002; NEUTEBOOM et al., 2002). Portanto, a expressão tecido-específica de transgenes é de grande importância para insetos de hábitos endofíticos, como A. grandis, por exemplo, visto que é possível direcionar a expressão dos transgenes para os botões florais, local de oviposição e desenvolvimento do inseto-praga (BRAGA SOBRINHO; LUKEFAHR, 1983).
Em busca de novas sequências regulatórias, foi isolado e, posteriormente caracterizado pelo grupo do LIMPP/Cenargen, o promotor constitutivo proveniente do gene de conjugação à ubiquitina (UceA 1.7) em algodoeiro (GROSSI-DE-SA et
al., 2008). Plantas modelo de A. thaliana transformadas com este promotor mostraram expressão desse gene em diversos tecidos, principalmente em raiz e botão floral, quando comparado ao promotor 35S (GROSSI-DE-SA et al., 2008; VIANA, 2011). Além deste, outro promotor, denominado GhPGFS1, foi isolado de G.
A partir desse conjunto de informações será possível viabilizar o controle de insetos-praga, principalmente visando a obtenção de eventos transgênicos expressando altos níveis de RNAi e/ou entomotoxinas Cry direcionados ao botão floral.
1.4 MÉTODOS DE TRANSFORMAÇÃO GENÉTICA DE PLANTAS MEDIADA
POR AGROBACTERIUM TUMEFACIENS E POR BIOBALÍSTICA
A. tumefaciens é uma bactéria fitopatogênica, encontrada no solo, pertencente à família Rhizobiaceae (LIPP-NISSINEN, 1993). Essa bactéria causa a doença conhecida como galha da coroa e a síndrome da raiz em cabeleira (VAN SLUYS, 1999) e tem sido utilizada para a introdução de genes em células vegetais, mediada por suas cepas manipuladas (RIVA et al., 1998). A transformação genética via A. tumefaciens permite a inserção de transgenes em plantas de interesse (ALIMOHAMMADI; BAGHERIEH-NAJJAR, 2009). Entretanto, a transformação via A.
tumefaciens se limita a espécies que não apresentam características vegetais recalcitrantes. De outro modo, a metodologia de aceleração de partículas até o meristema alvo tem sido utilizada para transformar plantas que não apresentam esse potencial (CHENG et al., 2009).
região do meristema. O mecanismo de ação do Imazapyr se dá através da inibição da enzima aceto-hidroxi-acido-sintase, que catalisa, na região apical meristemática, os passos iniciais da biossíntese de três aminoácidos essenciais: isoleucina, leucina e valina, impedindo o crescimento apical da futura planta. Como consequência, após o bombardeamento das micropartículas, contendo a construção de interesse, juntamente com gene conferindo resistência ao Imazapyr, as possíveis células transgênicas da região apical dos eixos embrionários podem ser efetivamente selecionadas entre as células não transgênicas. Tal seleção se dá pela capacidade natural de que as células transgênicas têm de crescer quando expressando o gene
ahas, mesmo em presença de Imazapyr (RECH et al., 2008).
Um dos aspectos negativos da utilização da biobalística é a integração indeterminada de cópias do transgene de interesse (BARAMPURUM; ZHANG, 2011). Entretanto, a biobalística é uma ferramenta biotecnológica bastante eficiente, uma vez que facilita a transferência de genes para o interior de células vegetais e além do mais, é o único método de transformação aproveitável para algumas espécies (TAYLOR; FAUQUET, 2002; ZIOLKOWSKI, 2007).
1.5 ALGODÃO GM
A agricultura no Brasil é um forte contribuinte econômico direta, seja relacionado à capacidade de gerar empregos ou à comercialização de produtos (AVELAR; VILELA, 2006), o que tem providenciado destaque brasileiro para o agronegócio. Dentre as culturas que mais se destacam no Brasil (cana-de-açúcar, soja, milho, feijão, café e algodão), a cotonicultura foi uma das primeiras atividades agrícolas desenvolvidas no País e tem contribuído de forma significativa no aumento do PIB (CEPEA/USP, 2011; MAPA, 2012).
No Brasil, os índios utilizavam para fiar e tecer o algodão no início do século XVI. Em 1576, deu-se início aos relatos de suas utilizações: o português Gandavo relatou que as camas eram feitas de teias de fios de algodão, entretanto para os indígenas o algodão tinha muitas utilidades, inclusive se tratando de alimentação (caroço esmagado e cozido) e tratamento de feridas (sumo das folhas) (FREIRE, 1999).
De 1960 a 1970, estima-se que a cotonicultura no Brasil atingiu mais de três milhões de hectares plantados nas regiões centro-sul e nordeste, conhecida como agricultura de pequenas áreas e tecnologia remota. No entanto, na década de 80 deu-se início à grande dificuldade de produção desencadeada pela entrada de uma nova praga, o bicudo-do-algodoeiro (A. grandis), o que reduziu a produtividade nacional e tornou o Brasil de maior exportador de algodão para o maior importador mundial (CONAB, 2006). A partir de então, a cotonicultura migrou para a região centro-oeste, traçando novas metas para a agricultura nacional. No entanto, para o melhor desenvolvimento e estabilidade da cultura, tornava-se necessário o aprimoramento de tecnologias voltadas para as novas condições oferecidas pela região promissora para a cotonicultura (BELTRÃO, 1999).
O surgimento de problemas financeiros e de manejo fitossanitário resultou na mudança de local de produção para uma nova região com ausência da praga, o que promoveu um progresso econômico inesperado: o estado do Mato Grosso conseguiu alcançar um crescimento de aproximadamente 536, 25%, passando de 42,26 mil hectares para 268,87 mil hectares, entre 1998 e 2000, gerando um aumento de produção de 82,21%. Tamanho sucesso pôde ser justificado pela utilização dos cultivares adaptados, bem como de novas técnicas de cultivo, promovendo o controle temporário dos danos causados por pragas (RICHETTI; MELO FILHO, 2002; CONAB, 2006).
total dessa área. Atualmente, somando-se a produção do centro-oeste com a produção da Bahia e do Maranhão, o algodão representa mais de 80% da produção
nacional (AMPASUL, 2014).
O algodão é uma das culturas que mais tem se beneficiado dos avanços da biotecnologia, sendo o terceiro principal cultivo transgênico no mundo (JAMES, 2013). Atualmente, a resistência a inseto e tolerância a herbicida são as duas características agronômicas aprovadas e amplamente comercializadas que foram introduzidas no algodão através de engenharia genética (MEHBOOB-UR-RAHMAN, et al., 2012). Nas variedades resistentes a insetos foram incorporados ao genoma do algodoeiro genes que codificam proteínas entomotóxicas provenientes da bactéria Bt. Esses eventos GM foram genericamente denominados algodoeiros Bt (MOREIRA et al., 1999). Já as variedades resistentes a herbicida visam aumentar a facilidade de manejo de plantas invasoras, nas quais o transgene inserido é um homólogo do gene alvo insensível ao herbicida ou codifica uma enzima que degrada o herbicida (SAROHA et al., 1998; BECKIE, 2011; ICAC, 2012).
O avanço tecnológico e o aumento da produtividade do algodão permitiram ao Brasil passar de maior importador de algodão do mundo para o terceiro maior exportador do produto em apenas 12 anos. Destinada à indústria têxtil, a produção nacional de algodão no Mato Grosso e na Bahia é responsável por, cerca de, 82% da produção nacional e se destaca pelo investimento em biotecnologia, gerenciamento do setor e novas técnicas de manejo. Apenas a indústria têxtil do Brasil é capaz de consumir mais de um milhão de toneladas do algodão produzido no Brasil, totalizando 70% da produção total de 1,5 milhões de toneladas (MAPA, 2012).
2010; AMPASALA et al., 2011; ARAKANE etal., 2011). Quando a expressão desses genes foi inibida no coleóptero modelo, Tribolium castaneum, fenótipos letais foram observados (ARAKANE et al., 2008). Ademais, a validação da vitelogina, proteína precursora do vitelo presente no interior dos ovos de insetos, evidenciou a importância de se estudar o possível silenciamento desse gene em progênies de A. grandis, resultando na inviabilidade de parte dos ovos decorrentes da diminuição significativa da síntese dessa proteína (COELHO, 2013). Portanto, uma vez que a quitina está ausente em plantas e vertebrados, e o desenvolvimento e crescimento dos insetos dependem da biossíntese deste polímero, a identificação, a caracterização e o uso do silenciamento de genes envolvidos na síntese de quitina, assim como na síntese de vitelogenina, apresentam-se como estratégia promissora para desenvolvimento de novos métodos mais seguros e eficazes de controle desta praga.
1.6 JUSTIFICATIVA
2 OBJETIVO
2.1 OBJETIVO GERAL
Validar genes essenciais de A. grandis, o bicudo-do-algodoeiro, e a entomotoxina Cry8Ka5, a fim de obter plantas de algodão GM resistentes ao inseto-praga.
2.2 OBJETIVOS ESPECÍFICOS
1. Sintetizar a construção pBSKdsCH2vitCry8Ka5, contendo dsRNA para os genes Quitina Sintase II e Vitelogenina, sob controle do promotor constitutivo UCeA 1.7 e, sequência do gene que expressa a proteína Cry8Ka5, sob domínio do promotor tecido-específico GhPGFS1;
2. Transformar embriões de sementes de algodão da variedade BRS-372, através da técnica de biobalística com a construção pBSK-dsCH2vitCry8Ka5, visando a superexpressão da toxina Cry e o silenciamento gênico de quitina e vitelogenina;
3. Caracterizar molecularmente as plantas de algodão GM por meio das técnicas PCR, ELISA e qRT-PCR;
3. METODOLOGIA
3.1. CONSTRUÇÃO GÊNICA
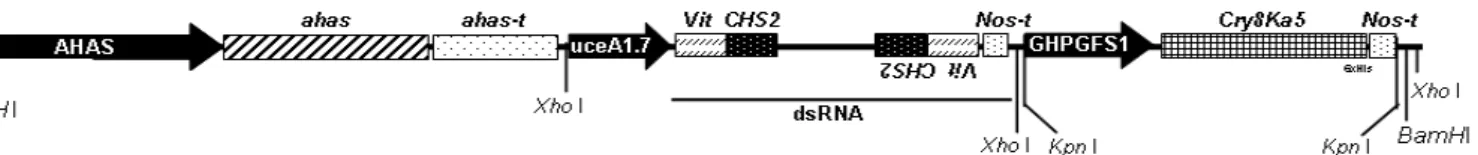
Foi utilizado o cassete pBSK-dsCHSvitCry8Ka5 para transformação de plantas de algodão, contendo dsRNA para os genes CHS2 e vitelogenina, sob domínio do promotor constitutivo, UceA 1.7, visando o silenciamento dos dois genes alvo de A. grandis. Agrupados in tandem (figura 5), na construção pBSK-dsCHSvitCry8Ka5, os transcritos dos genes alvos contêm 200pb específicos para cada um dos genes, cujos agrupamentos possuem direção invertida interrompida por intron, o que promove a formação de alça após a obtenção do transcrito, auto anelamento e dsRNA resultante. Além disso, o cassete contém a sequência que codifica para a proteína Cry8Ka5 sob domínio do promotor botão floral-específico GhPGFS1 para expressão direcionada para botão-floral. Como marca de seleção, o cassete possui o gene AHAS que confere resistência ao herbicida, Imazapyr.
Figura 5. Representação esquemática do cassete de expressão contendo o gene de seleção AHAS,
que confere resistência ao herbicida Imazapyr e o gene para super-expressão do dsRNA-CHS2vit, sob domínio do promotor UCeA 1.7 e o gene para a proteína Cry8Ka5 controlado pelo promotor tecido-específico GhPGFS1.
3.2 TRANSFORMAÇÃO GENÉTICA VIA BIOBALÍSTICA
semente) por 1 minuto, em constante homogeneização com bastão de vidro. Em seguida, as sementes foram transferidas para 5 litros de água destilada, lavadas por três vezes com água destilada e secas em papel toalha.
As sementes foram desinfestadas com etanol 70%, por 10 minutos, lavadas com água destilada estéril, por três vezes. Em seguida, foram colocadas em banho de hipoclorito de sódio 2,5% por 30 minutos e novamente, lavadas por três vezes com água destilada estéril. O recipiente contendo as sementes submersas em água destiladas estéril foi vedado e encoberto por papel alumínio, afim de permitir a germinação no escuro, durante 16 a 18 horas.
Após esse período, as sementes foram lavadas por três vezes com água estéril, secadas e vedadas em recipiente estéril e ambiente escuro por mais 16 a 18 horas. Posteriormente, os cotilédones de cada semente foram pressionados até que os eixos embrionários germinados fossem expulsos da casca da semente. Os eixos embrionários foram excisados, onde o meristema apical foi seccionado na região das primeiras folíolas, utilizando-se bisturi completamente estéril. Dez eixos embrionários foram posicionados em círculo para placas de Petri de 5 cm de diâmetro, a uma distância de 6 a 12 mm do centro da placa com a região meristêmica apical voltada para cima. O meio de cultura para combardeamento foi composto de sais basais para meio MS, suplementado com 3% de glicose, 0,8 % de Phytagel e pH ajustado para 5,7 antes da autoclavagem).
3.3.2 Aplicação da técnica de bombardeio de DNA
O cassete de transformação foi inserido no genoma da planta por meio de microparticulas de tungstênio, pela técnica de biobalística que com consiste na aceleração de microprojéteis envoltos de material genético de interesse de forma não letal à célula. Para a técnica atribuída, seguiu-se o protocolo subseqüente.
centrifugação a 3000g por 5 minutos. Este procedimento foi repetido por mais 2 vezes. Finalizadas as lavagens, o sobrenadante foi descartado e as partículas foram ressuspendidas em 1 mL de glicerol 50% autoclavado. Posteriormente, as micropartículas foram distribuídas em tubos de microcentrífuga em 50 µL e agitadas vigorosamente como o auxílio do vortex por aproximados 30 segundos, anteriormente sonicadas por 5 minutos, a fim de garantir total desagregação das partículas. No momento de cada bombardeamento, as partículas receberam o DNA de interesse numa proporção de 8 µL de DNA para cada 50 µL de partículas de tungstênio. O DNA de interesse foi previamente pesado e dosado a 1 µg/ µL. Após 2 segundos de rápida agitação em vortex, foram adicionados 50 µL de CaCl2 2.5 M e
mais uma vez homogeneizadas. Em seguida, 20 µL de espermidina a 0,1 M, mais uma vez homogeneizados. Deixou-se agitar por 10 minutos em velocidade mínima no vortex, seguida de centrifugação por 10 segundos a 3000 x g. Descartou-se sobrenadante, sem que o pellet fosse tocado. Foram adicionados 150 µL de etanol absoluto e rapidamente homogeneizado e centrifugado a 3000 x g. O sobrenadante foi descartado e finalmente, adicionou-se 24 µL de etanol absoluto e homogeneizou-se mais uma vez. Sonicou-homogeneizou-se por 3 homogeneizou-segundos cada conteúdo preparado.
Este conteúdo foi distribuído em alíquotas de 3,2 µL para cada membrana carreadora em sua região central, membrana esta, específica para a realização da técnica de biobalística. Essas membranas foram encaixadas em seu suporte e assim, transferidos para placas de sílica gel a fim de evitar umidade. Após aproximadamente 5 minutos, as placas contendo os suportes e suas membranas foram retiradas e utilizadas para bombardeamento. Por meio do equipamento de biobalística, as micropartículas envoltas por DNA foram projetadas sobre as placas contendo os eixos embrionários, conforme relatado no item 3.3.1.
Após o bombardeamento, as placas contendo os eixos embrionários pós- microprojetados foram armazenadas por 48 horas em ambiente escuro. Em seguida, os embriões foram transferidos para caixas tipo Magenta® contendo meio de cultura e seleção (sais basais para meio MS, glicose 3%, BAP (benzylaminopurina) 13.3µM, Imazapyr 300nM, carvão ativado 0,1%, Agar 0,6 e pH ajustado para 5.7).
3.3.3 Cultivo de plantas selecionadas por herbicida
Após o período de 30 dias, foram descartadas as plantas que apresentaram-se necrosadas por ação do Imazapyr. As plantas que demonstraram desenvolvimento diferenciado perceptível alcançando uma média de 10 cm de comprimento, foram transferidas e mantidas por 15 dias em copo plástico envolvido por saco plástico assegurado por elástico, contendo vermiculita, em sala de cultivo in vitro, sob temperatura (24-28 ºC) e luminosidade (16/8h), controladas. Após este período, as plantas foram aclimatadas em casa de vegetação, onde permaneceram por mais 15 dias. Posteriormente, o saco plástico foi retirado e, em mais 15 dias as plantas foram transferidas para sacos plásticos contendo solo, onde permaneceram por mais 30 dias até que fossem definitivamente transferidas para vasos. As análises de caracterização das plantas foram iniciadas assim que suas raízes foram aprofundadas resultando na garantia de sua adaptação ao solo a que foram submetidas.
3.4. CARACTERIZAÇÃO MOLECULAR DE PLANTAS POTENCIALMENTE TRANSFORMADAS
3.4.1 Extração de DNA
Neste trabalho, para a extração de DNA genômico foi utilizado o kit DNeasy Plant (QIAGEN). A extração seguiu-se conforme instruções do fabricante. As amostras foram tratadas com 1 µl de solução RNase (10 µg/µl) e aquecidas a 37º C por 45 minutos. Posteriormente, foram armazenadas em -80ºC.
3.4.2 Verificação da amplificabilidade do DNA extraído de folhas de plantas de algodão
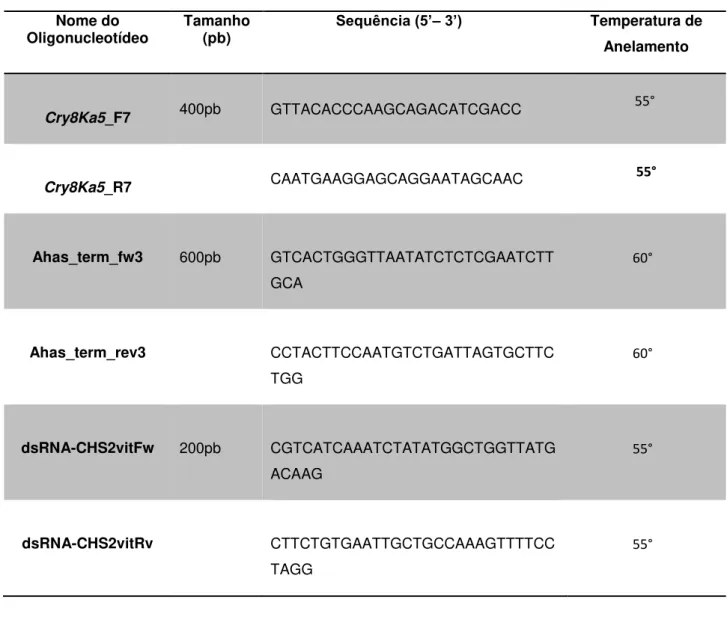
separados por eletroforese em gel de agarose 1% (p/v) contendo 0,3 µg/ml de brometo de etídio imerso por tampão TBE 1X (0,1M Trizma base; 90mM ácido bórico; 2,5mM EDTA ). A PCR para todos os genes foi realizada em um volume final de 10 µl contendo 5 µl de PCR ReadyMix, 0,4 µl de cada primer 10 µM, 1 µl de extrato de DNA. As temperaturas de anelamento específicas para cada primer, foram: 65ºC para AHAS, 55ºC Cry8Ka5 e 55ºC para RNAi .
Para todos os genes obedeceram-se os seguintes passos, alterando-se apenas a temperatura específica do anelamento de cada primer: 1- desnaturação inicial a 94ºC por 5 minutos; 2- 40 ciclos de 94ºC por 45 segundos, 65ºC por 45 segundos e 72º C por 1 minuto; 3- extensão final a 72ºC por 10 minutos, para o gene AHAS.
Tabela 2. Lista de oligonucleotídeos utilizados nas análises por PCR.
Nome do
Oligonucleotídeo Tamanho (pb) Sequência (5’–3’) Temperatura de Anelamento
Cry8Ka5_F7
400pb GTTACACCCAAGCAGACATCGACC
55°
Cry8Ka5_R7 CAATGAAGGAGCAGGAATAGCAAC
55°
Ahas_term_fw3
600pb GTCACTGGGTTAATATCTCTCGAATCTT GCA
60°
Ahas_term_rev3 CCTACTTCCAATGTCTGATTAGTGCTTC
TGG
60°
dsRNA-CHS2vitFw
200pb CGTCATCAAATCTATATGGCTGGTTATG ACAAG
55°
dsRNA-CHS2vitRv CTTCTGTGAATTGCTGCCAAAGTTTTCC
TAGG
3.4.3 Extração e determinação de proteínas totais
Para extração de proteínas totais de plantas de algodão foram utilizados 60mg de macerado de botão floral congelado. Adicionou-se ao material macerado 1ml de tampão de extração de proteínas (Tris-HCl 0,5 M, EDTA 50 mM, sacarose 0,7 M, KCl 0,1 M, Triton 0,1%, PMSF 10mM, DTT 1mM e PVPP 0,5%) e inverteu-se as amostras à 4ºC por 60 minutos, seguida de centrifugação por 45 minutos a 13200 x g. Após isso, coletou-se sobrenadante e transferiu-se para novos tubos de microcentrífuga.
A concentração das proteínas totais foi estabelecida segundo a metodologia de Bradford (1976), onde utilizou-se como padrão o BSA (Bovine Serum Albumin), com leitura em espectrofotômetro a 595 nm.
3.4.4 Quantificação da expressão da entomotoxina Cry8Ka5 por meio de
ensaio imunoenzimático do tipo ELISA
por 3 vezes. Foi adicionado o segundo anticorpo conjugado com peroxidase, anti-rabbit, na proporção de 1:3000 diluído em PBS 1X e distribuídos em 100 µl por poço e homogeneizando por 1 hora. Posteriormente, descartou-se todo o conteúdo da placa, lavou-se novamente com solução PBS 1X e tween 0,05% por 3 vezes.
A solução de revelação foi preparada, calculando-se o volume a ser preparado de acordo com o número de cavidades (100 µl/cavidade). Foram adicionados ao tampão de revelação (ácido cítrico 0,1M e fosfato de sódio dibásico 0,2M, pH 5,0) 10 µl de peróxido de hidrogênio e 1 pastilha de substrato enzimático para cada 10 ml de solução. Distribuiu-se 100 µl de solução reveladora por cavidade e esperou-se que a reação acontecesse em ambiente escuro por 30 minutos. Em seguida, foram distribuídas alíquotas a 50 µl de ácido sulfúrico 2M por cavidade.
A leitura foi realizada em um aparelho de espectrometria de luz UV/visível, utilizando um filtro específico de 450nm.
3.5 DESAFIO DAS PLANTAS T1 COM O INSETO-PRAGA
Sementes de plantas T1 GM, obtidas por auto-fecundação de indivíduos T0, foram plantadas e analisadas por PCR utilizando as mesmas condições do item 3.4.2.
3.5.1 Extração de RNA total de larvas de A. grandis alimentadas por
botão floral transgênico.
Figura 6. Placa para cultura de células contendo botões florais inoculados com larvas de bicudo-do-algodoeiro.
3.5.2 Síntese de cDNA
A reação de síntese de DNA complementar (cDNA) ocorre a partir de um molde de RNA utilizando-se a enzima transcriptase reversa, na realização desse papel.
O cDNA foi sintetizado utilizando-se kit Superscript First-Strans Synthesis System for RT-PCR (Invitrogen) a partir de 2µg de RNA total extraído de larvas de A.
grandis alimentadas por botões florais de plantas de algodão potencialmente transgênicas, 2 µL de oligo dT a 10µM, completando-se com água estéril para 11 µL de volume final. As amostras foram incubadas a 70ºC por 3 minutos, colocadas rapidamente em gelo por mais 3 minutos, adicionando-se, em seguida, mix contendo 4 µL de tampão 5x, 2 µL de ditiotreitol (DTT), 1 µL de dNTP e 1 µL de RNase out. Prosseguiu-se em 2 minutos de incubação a 42ºC. Em seguida, foram acrescentadas 40 unidades de transcriptase reversa - Superscript First-Strans Synthesis System for RT-PCR (Invitrogen). O restante da reação ocorreu em termociclador obedecendo os passos: 90 minutos a 42ºC, 15 minutos 70ºC e 4ºC. O
3.5.3 Confirmação da síntese do DNA complementar (cDNA)
Foi realizada reação de PCR para o gene constitutivo que transcreve para Actina, proteína normalmente sintetizada em A. grandis, a fim de confirmar síntese do cDNA.
A PCR foi realizada em um volume final de 20 µL contendo 2 µL de tampão 10X para Taq DNA polimerase, 0,8 µL de MgCl 50mM, 0,4 µL de dNTP 10 mM, 0,4 µL de cada iniciador (senso e anti-senso) 10 mM, 0,4 µL de Taq DNA Polimerase 5 u/µL e 1 µl de DNA complementar, diluído em 1:20. A reação de PCR foi realizada em termociclador obedecendo os seguintes passos: 1- desnaturação inicial a 94ºC por 2 minutos; 2- nova desnaturação a 94ºC por 20 segundos; 3- anelamento de iniciadores a 55ºC por 20 segundos; 4- extensão a 72ºC por 45 segundos; 5- voltando ao passo 2 (repetindo por 30 ciclos sob as mesmas condições anteriormente descritas). Por fim, foi realizado um último ciclo de extensão a 72ºC, por 5 minutos. Como controle negativo foi utilizado para a PCR amostra que não continha cDNA.
O produto amplificado foi analisado através de uma eletroforese em gel de agarose 1% acrescido de brometo de etídio (0,5 µL/ml), utilizando como padrões de peso molecular de DNA 1 Kb plus DNA ladder.
3.5.4 Análise do silenciamento do gene alvo do ds RNA por qPCR
30 segundos. Para a análise das amplificações, a Ct (do inglês - cycle of the threshold) e a eficiência de amplificação de cada oligonucleotídeo (variando de 90% a 100%) foram determinadas pelo programa Real-time PCR Miner. RNA total extraído de larvas de A. grandis alimentadas por plantas botões florais de plantas de algodão potencialmente transgênicas, 2 µL de oligo dT a 10µM, completando-se com água estéril para 11 µL de volume final. Foram incubadas as amostras a 70ºC por 3 minutos, colocadas rapidamente em gelo por mais 3 minutos e adicionou-se, em seguida, mix contendo 4 µL de tampão 5x, 2 µL de ditiotreitol (DTT), 1 µL de dNTP e 1 µL de RNase out. Prosseguiu-se em 2 minutos de incubação a 42ºC. Após isso, foram acrescentadas 40 unidades de transcriptase reversa - Superscript First-Strans Synthesis System for RT-PCR (Invitrogen). O restante da reação ocorreu em termociclador obedecendo os passos: 90 minutos a 42ºC, 15 minutos 70ºC e 4ºC.
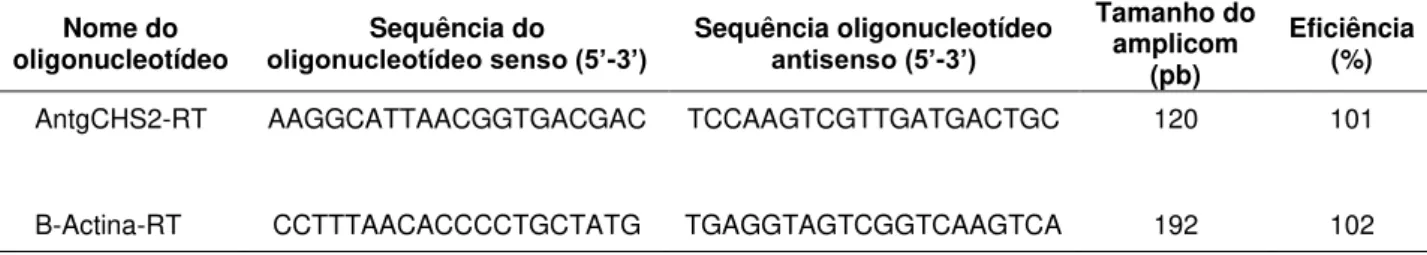
Tabela 3. Oligonucleotídeos utilizados nas reações de PCR em tempo real para análise de CHS2.
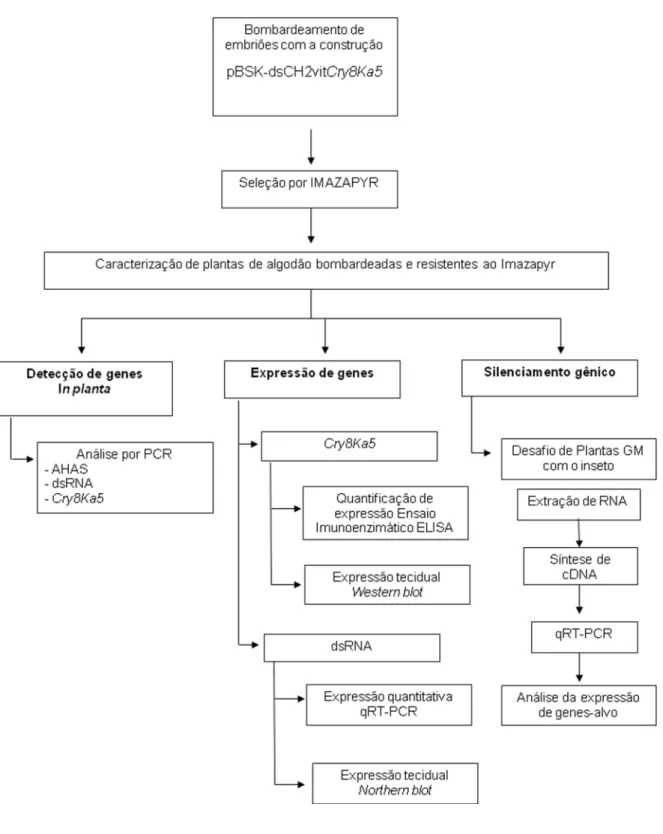
Uma visão geral da metodologia empregada na clonagem, caracterização e avaliação do efeito funcional da estratégia de transformação é mostrada na figura 7.
Nome do
oligonucleotídeo oligonucleotídeo senso (5’Sequência do -3’) Sequência oligonucleotídeo antisenso (5’-3’)
Tamanho do amplicom
(pb)
Eficiência (%)
AntgCHS2-RT AAGGCATTAACGGTGACGAC TCCAAGTCGTTGATGACTGC 120 101
4. RESULTADOS
4.1 BOMBARDEAMENTO DE EMBRIÕES
A técnica de biobalística foi utilizada no intuito de inserir em plantas de algodão os genes que codificassem para a entomotoxina Cry8Ka5 e para o dsRNA de sequência homóloga aos genes alvos de A. grandis, cujo silenciamento resultaria na interrupção da produção da CHS2 e da vitelogenina, resultando em fenótipos letais. Foram bombardeados 3500 embriões de algodão, variedade BRS-372, dos quais foram obtidas 186 plântulas contendo o gene de seleção ahas para resistência ao herbicida Imazapyr.
4.2 CARACTERIZAÇÃO
Tabela 4. Plantas T0 positivas resultantes da análise por PCR para os genes AHAS, Cry8Ka5 e
dsRNA.
Plantas ahas Cry8Ka5 dsRNA
2 + + +
3 + + +
7 + + +
9 + + +
11 + + +
12 + + +
13 + + +
15 + + +
32 + + +
44 51 52 53 55 58 63 120 144 156 + + + + + + + + + + + + + + + + + + + + + + + + + + + + + +
Figura 8. Eventos T0 detectados por PCR para o gene dsRNA. Gel de agarose 1% demonstrando reação de PCR. Poços 3, 7, 9, 11, 15, 32, 44, 51 e 120 amplificação do fragmento de dsRNA - Vitelogenina e CHS2, presença do gene para as nove plantas analisadas; C+: controle positivo (plasmídeo utilizado no bombardeamento); PM: Padrão de peso molecular em pares de bases (pb).
200pb
NT H20 3 7 9 11 15 32 44 51 120 C+ PM
Figura 9. Eventos T0 detectados por PCR para o gene Cry8Ka5. Gel de agarose 1% demonstrando
reação de PCR. Poços 3, 7, 9, 11, 15, 32 e 44 amplificação do fragmento de Cry8Ka5 - presença do
gene para as sete plantas analisadas; C+: controle positivo (plasmídeo utilizado no bombardeamento); PM: Padrão de peso molecular em pares de bases (pb).
400pb
Figura 10. Eventos T0 detectados por PCR para o gene ahas. Gel de agarose 1% demonstrando
reação de PCR. Poços 3, 7, 9, 11, 15 e 32 amplificação do fragmento de AHAS - presença do gene para as seis plantas analisadas; C+: controle positivo (plasmídeo utilizado no bombardeamento); PM: Padrão de peso molecular em pares de bases (pb).
600pb
3 7 9 11 15 32 44 NT H2O C+ PM
0,0 10,0 20,0 30,0 40,0 50,0 60,0 70,0 80,0 90,0 100,0
1 2 3 7 9 11 12 13 32 44 51 52 53 55 58 120 156 NT8
u g d e to xi n a C ry 8K a5 / g d e te ci d o fr es co
4.3 QUANTIFICAÇÃO DA EXPRESSÃO DE ENTOMOTOXINA CRY8KA5 POR MEIO DO ENSAIO IMUNOENZIMÁTICO DO TIPO ELISA
A concentração de proteína Cry8Ka5 expressa nas plantas de algodão consideradas positivas por PCR foi medida por ELISA em extrato de tecido do botão floral. Os níveis de proteína Cry8Ka5 foram detectados utilizando-se plantas de algodão não transgênico como controle (Figura 11).
Figura 11. Avaliação da expressão da entomotoxina Cry8Ka5 em amostras de botão floral macerado
de plantas potencialmente transgênicas. Das 17 plantas avaliadas pela técnica de ELISA, 11 plantas (amostras: 3, 7, 9, 11, 13, 44, 52, 3, 55 e 156) demonstraram significativa expressão da entomotoxina avaliada da construção pBSKdsCH2vitCry8Ka5.
.
4.4 IDENTIFICAÇÃO DE GENES POR PCR DE GERAÇÃO T1
genes de interesse (ahas, RNAi e Cry8Ka5), sendo duas oriundas do evento 7 (7.1 e 7.5), duas do evento 11 (11.1 e 11.9), uma do evento 13 (13.10), uma do evento 55 (55.4) e uma do evento 156 (156.1).
Tabela 5. Plantas de algodão geração T1 positivas resultantes da análise por PCR para os genes
ahas, Cry8Ka5 e dsRNA.
Plantas ahas Cry8Ka5 dsRNA
7.1 + + +
7.5 + + +
11.1 + + +
11.9 + + +
13.10 + + +
55.4 + + +
156.1 + + +
Total de 7 plantas
4.5 DESAFIO DAS PLANTAS GM COM O INSETO-ALVO
Para analisar o silenciamento de CHS2 nas plantas GM foi determinada a expressão quantitativa desse transcrito pela técnica de qRT-PCR. O RNA total foi extraído de larvas de A. grandis alimentadas por botões florais de plantas GM e seu controle (Figura 12) e o cDNA obtido foi utilizado para a realização do experimento de qRT-PCR.
figura 14 são apresentadas as etapas para obtenção e seleção do evento transgênico.
Figura 12. Análise morfológica de larvas de terceiro instar de insetos de A. grandis após serem
alimentados com plantas de algodão GM contendo a construção pBSKvitCHS2Cry8Ka5. As larvas
foram maceradas após 7 dias de inoculação em botão floral.
LARVA CONTROLE 7.1 7.2A 7.2B 7.5
Figura 13. Análise do silenciamento gênico por qPCR de CHS2 em larvas de terceiro instar de A.
grandis após ingestão de botão floral de plantas de algodão GM com a construção
pBSKdsvitCHS2Cry8Ka5 por um período de sete dias. -actina foi utilizado como gene de referência.