UNIVERSIDADE
CATÓLICA DE
BRASÍLIA
PROGRAMA DE PÓS-GRADUAÇÃO STRICTO
SENSU EM CIÊNCIAS GENÔMICAS E
BIOTECNOLOGIA
Mestrado
EXPRESSÃO DE PROTEÍNAS NÃO ESTRUTURAIS DO VÍRUS DO DENGUE EBUSCA DE GENES CANDIDATOS A SUPRESSÃO DE RNA INTERFERENTE
Autora: Aline dos Santos Ribeiro
Orientador: Prof. Dr. Tatsuya Nagata
Aline dos Santos Ribeiro
EXPRESSÃO DE PROTEÍNAS NÃO ESTRUTURAIS DO
VÍRUS DO DENGUE E BUSCA DE GENES CANDIDATOS
A SUPRESSÃO DE RNA INTERFERENTE
Dissertação apresentada ao Programa de Pós-Graduação “Stricto Sensu” em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito para obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador: Prof. Dr. Tatsuya
Nagata
Brasília
Dissertação defendida e aprovada como requisito parcial para obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia, defendida e aprovada em 12 de maio de 2008, pela banca examinadora constituída por:
Prof. Dr. Renato de Oliveira Resende
Universidade de Brasília - UNB
Profa. Dra. Eliane Ferreira de Noronha
Universidade Católica de Brasília
Prof. Dr. Tatsuya Nagata
Universidade Católica de Brasília
Brasília
Ofereço a Deus esse trabalho, reconhecendo que Ele é a fonte de toda sabedoria e conhecimento, minha força e motivação. Quando duvidei que pudesse chegar
ao fim, então pude respirar por saber que Ele é o começo e que dele, por meio dele e para Ele são todas as coisas.
Dedico à família Ochsendorf por ser a maior expressão de amor e milagre que eu vivi nessa trajetória. Olhando para vocês entendo que amor é mais do que um sentimento,
AGRADECIMENTOS
Agradeço a Deus por ter criado esse intervalo de tempo na minha história para provar que nele não há impossíveis.
Aos meus pais por me ensinarem que grandes conquistas não são feitas por meio de força,
mas com atitudes de fé.
Ao professor Tatsuya pela orientação e oportunidade de realizar esse projeto.
A professora Eliane Noronha pela forma carinhosa com que me ajudou na etapas de expressão.
A professora Danielle Cordeiro pela orientação durante a monitoria TA. Agradeço muito por tudo que me ensinou.
Aos queridos Jurandir, Vera, Érica e Karine por serem o abraço, o sorriso e o aconchego
quando não havia mais ninguém.
A minha irmã Alana por realizar todos os dias o meu sonho de não andar só.
A minha avó por sempre acreditar e investir em tudo que faço.
Aos primos Jurandir e Eduarlinda por caminharem junto durante toda a trajetória.
Aos pastores Marco Antônio e Juçara Peixoto pelas palavras que trouxeram vida a esse tempo.
Aos líderes Tony e Alcina por todo cuidado e amor.
A minha amiga Giselle por fazer do meu projeto seu próprio alvo de conquista e por me fazer acreditar todos os dias que andar junto é possível mesmo quando não estamos perto.
Agradeço a minha Mani Elisa o melhor ouvido
e a primeira voz de encorajamento que recebi quando tudo parecia impossível.
Às amigas Simone,Thaís e Michele por serem a minha família durante esse tempo. Amo muito vocês meninas!
Agradeço a Fernanda pelo companheirismo e cuidado sincero.
À Bruna por sempre ser a voz no meu ouvido dizendo: Agüenta só mais um pouquinho.
Você não é uma amiga, tornou-se uma irmã.
À Dione por todo apoio e orações.
À Keilly por ser uma excelente companheira de quarto e tornar muito agradável
o tempo da nossa convivência.
Agradeço a toda equipe do laboratório, Sandra, Karoline, Layssa, Natália, Fernanda, Ana, Grazzy,Camila, Patrícia, Francisca Aline e Francielle. Quero que saibam o quanto foram importantes nessa trajetória
e que reconheço que esse trabalho tem as digitais de vocês.
Agradeço ao Fábio, Secretário do curso de Biotecnologia, por ter feito sempre mais do que o seu trabalho, sendo um mediador em todas as coisas desde o princípio.
À Ida por facilitar meus experimentos, auxiliando sempre perto e longe dos olhos.
“A fé é a certeza de que vamos receber as coisas que esperamos e a prova de que existem coisas que não podemos ver”. Hebreus 11:1
ÍNDICE
RESUMO ...VIII ABSTRACT...IX LISTA DE FIGURAS... X LISTA DE TABELAS ... XIV
INTRODUÇÃO...1
Histórico do Dengue... 1
Dengue no Brasil ... 3
Dengue ... 7
Vírus do Dengue... 8
A importância de proteínas não-estruturais do vírus... 10
Silenciamento de genes ... 14
Supressores do silenciamento de genes ... 19
Expressão de proteínas... 26
JUSTIFICATIVA ...28
OBJETIVO GERAL ...29
OBJETIVOS ESPECÍFICOS...29
MATERIAL E MÉTODOS ...30
Clonagem de cDNA de genes do vírus da Dengue... 30
Extração de RNA viral ... 30
Amplificação de genes candidatos a supressores de RNAi... 31
Clonagem dos genes de proteínas não estruturais do Dengue ... 35
Digestão de insertos e vetor de clonagem... 35
Cálculo de proporção inserto: vetor... 36
Transformação de bactérias E.coli... 38
Subclonagem dos genes correspondentes as proteínas não-estruturais ... 39
Agroinfecção de Nicotiana benthamiana (Agrobacterium tumefaciens transient Assay) ... 42
Análise de fluorescência em folhas de N. bethamiana agroinculadas... 45
Extração de Proteínas totais de N. bethamiana... 46
Transferência de proteínas para membrana de nitrocelulose ... 46
Análises de Western Blot ... 47
Dot Imunobinding Assay (DIBA) ... 48
Expressão da proteína NS2A em bactéria ... 48
Digestão das construções em vetor de clonagem ... 52
Clonagem e Subclonagem dos genes das proteínas não estruturais do Dengue... 55
Agroinoculação... 55
Detecção das proteínas expressas em plantas... 61
Detecção de proteína expressa em bactérias... 65
CONSIDERAÇÕES FINAIS ...68
PERSPECTIVAS...71
REFERÊNCIAS BIBLIOGRÁFICAS ...72
APÊNCICE I ...81
Meios de Cultura e Soluções ... 81
Meio LB com ampicilina (Sambrook & Russell, 2001)... 81
Meio LB com ágar e ampicilina (Sambrook & Russell, 2001) ... 82
Meio SOC (Sambrook & Russell, 2001)... 82
Meio de cultura LBman ... 83
Tampão de Indução... 83
Tampão de indução completo ... 83
MES ... 84
MS Completo... 84
Tampão Alcalino... 84
Tampão de Transferência... 84
PBS 10x (pH 7,4)... 85
Tampão de Lise (pH 8,0) ... 85
Tampão de Amostra... 85
APÊNDICE II...87
A- Alinhamento da proteína NS1 com seqüências de proteína do Dengue depositadas no banco de dados do NCBI. Query: seqüência de NS1 obtida no seqüenciamento automático. Subjct: seqüência de proteína do Dengue proveniente do banco de dados do NCBI. ... 87
B- Alinhamento da proteína NS2A com seqüências de proteína do Dengue depositadas no banco de dados do NCBI. Query: seqüência de NS2A obtida no seqüenciamento automático. Subjct: seqüência de proteína do Dengue proveniente do banco de dados do NCBI. ... 88
C- Alinhamento da proteína NS2B com seqüências de proteína do Dengue depositadas no banco de dados do NCBI. Query: seqüência de NS2B obtida no seqüenciamento automático. Subjct: seqüência de proteína do Dengue proveniente do banco de dados do NCBI. ... 89
D- Alinhamento da proteína NS3 com seqüências de proteína do Dengue depositadas no banco de dados do NCBI. Query: seqüência de NS3 obtida no seqüenciamento automático. Subjct: seqüência de proteína do Dengue proveniente do banco de dados do NCBI. ... 90
G- Alinhamento da proteína NS5 com seqüências de proteína do Dengue depositadas no banco de dados do NCBI. Query: seqüência de NS5 obtida no seqüenciamento automático. Subjct: seqüência de proteína do Dengue
RESUMO
O genoma do vírus do Dengue é constituído de uma fita simples positiva
de RNA, capaz de codificar sete proteínas não-estruturais que assumem papel
importante na replicação, transmissão por mosquito, caráter de virulência e
outras funções essenciais à manutenção do vírus dentro do hospedeiro.
Devido a funções conhecidas, essas proteínas são alvos para o
desenvolvimento de controle do vírus e com a descoberta do envolvimento de
proteínas não estruturais na supressão do silenciamento de genes, tornou-se
ainda mais interessante o estudo de suas funções. Nesse trabalho, a atividade
supressora desse mecanismo nas sete proteínas não estruturais do Dengue foi
verificada, utilizando o sistema de Agrobacterium tumefaciens transient assay
(ATTA) em Nicotiana benthamiana. O silenciamento gênico na planta foi
confirmado através do baixo nível de expressão de “Green Fluorescent
protein”, comparando com o controle inoculado com a proteína supressora
HcPro de potyvirus, que após o período de seis dias pós inoculação foi capaz
de resgatar a fluorescência de GFP. Nenhuma das proteínas não estruturais do
Dengue apresentou capacidade de resgatar a fluorescência de GFP nas folhas,
o que parece indicar ausência de atividade supressora de interferência de
RNA identificada pelo sistema utilizado (ATTA). Paralelamente aos testes
realizados com as proteínas não estruturais foi realizada a expressão da
proteína NS2A em sistema procariótico, resultando nos primeiros indícios de
detecção da mesma através de ensaio imunológico a partir de proteínas totais
ABSTRACT
The genome of the Dengue virus is constituted of a simple positive strand of RNA,
capable of encode seven nonstructural proteins that assume important function in the
replication process, transmission by mosquito, character of virulence and other essential
functions to the maintenance of the virus inside the host. Due to known functions, those
proteins are aim for the development of control of the virus and with the discovery of the
involvement of nonstructural proteins in the suppression of genes silencing, became still more
interesting the study of its functions. In that work, the suppressive activity of that mechanism
in the seven nonstructural proteins of Dengue was noticed, utilizing the system of
Agrobacterium tumefaciens transient assay (ATTA) with Nicotiana benthamiana. The genes
silencing in the plant was confirmed through short level of expression of "Green Fluorescent
Protein", comparing with the control inoculated with the suppressive protein HcPro of
Potyvirus, which after the period of six days of inoculation was capable of rescue the
fluorescence of GFP. None of the nonstructural proteins of Dengue showed capacity of
rescue the fluorescence of GFP in the leaves, what seem to indicate absence of suppressive
activity of interference of RNA identified by the system utilized (ATTA). Concomitantly was
carried out the expression of the protein NS2A in prokaryotic system, resulting first sign of
detection of the protein through immunological assay from total proteins of Escherichia coli
LISTA DE FIGURAS
F i g u r a 1 : Q u a d r o d a e v o l u ç ã o d o D e n g u e n o p e r í o d o d e 1 9 8 0 - 2 0 0 7 ( O r g a n i z a ç ã o P a n A me r i c a n a d e S a ú d e , 2 0 0 7 ) . ... 4 F i g u r a 2 : M a p a i l u s t r a n d o a c o - c i r c u l a ç ã o d o s s o r o t i p o s d o D e n g u e e n t r e o s p a í s e s d e ma i o r i n c i d ê n c i a d a d o e n ç a n o p e r í o d o d e 2 0 0 6 - 2 0 0 7 ( O r g a n i z a ç ã o P a n
A me r i c a n a d e S a ú d e ) . ... 5 F i g u r a 3 : N ú me r o d e c a s o s d e D e n g u e r e g i s t r a d o e n t r e o s p a í s e s d o C o n e S u l , e m 2 0 0 7 ( O P A S , 2 0 0 8 ) . ... 6 F i g u r a 4 : V í r i o n d o D e n g u e . ( F o n t e : V í r u s T a x o n o my , 2 0 0 5 ) . A - E s q u e m a d o v i r i o n ma d u r o e i ma t u r o : E - E n v e l o p e , p r M - p r é - M e mb r a n a , C - C a p s í d e o , M - M e mb r a n a ; B - I ma g e m d a s u p e r f í c i e d o v í r u s . ... 9 Fi g u r a 5 : E s t r u t u r a e s q u e má t i c a d o g e n o ma d o v í r u s d o d e n g u e . ( F o n t e : V í r u s T a x o n o my , 2 0 0 5 ) . ... 11 F i g u r a 6 : E s q u e ma d o p r o c e s s o d e s i l e n c i a me n t o me d i a d o p o r R N A . A d u p l a f i t a d e R N A ( d s R N A ) é d e g r a d a p e l a r i b o n u c l e a s e D I C E R , g e r a n d o p e q u e n o s R N A s
( s i R N A s ) q u e s ã o i n c o r p o r a d o s n o c o mp l e x o e n z i má t i c o R I S C , p o d e n d o s e r u t i l i z a d o s c o mo p r i me r p e l a R N A p o l i me r a s e d e p e n d e n t e d e R N A ( R d R p ) .
r e s t r i ç ã o u t i l i z a d o s p a r a c l o n a g e m; A s c I e P a c I s ã o o s s í t i o s d e r e s t r i ç ã o u t i l i z a d o s p a r a a s u b c l o n a g e m e m v e t o r d e e x p r e s s ã o p B I N p l u s . ... 37 Figura 11: M a p a g e n é t i c o d o v e t o r d e e x p r e s s ã o p B I N p l u s l i g a d o à p a r t e d o v e r t o r p R A P s u t i l i z a d o p a r a c l o n a g e m d a s p r o t e í n a s n ã o e s t r u t u r a i s . I n s e r t o N S 1 , A s c I e P a c I r e p r e s e n t a m o s s í t i o s d e r e s t r i ç ã o p a r a a s u b c l o n a g e m. ... 40 F i g u r a 1 2 : E s q u e ma d e a g r o i n o c u l a ç ã o d a s c o n s t r u ç õ e s b a s e a d a s e m p B I N p l u s . A - C o n t r o l e n e g a t i v o : D u p l i c a t a d e N i c o t i a n a b e n t h a m i a n a i n o c u l a d a c o m
A g r o b a c t e r i u m t u ma f a c i e n s t r a n s f o r ma d a p e l a c o n s t r u ç ã o p B I N G F P . B - N i c o t i a n a b e n t h a m i a n a i n o c u l a d a c o m a s c o n s t r u ç õ e s p B I N G F P e p B I N H c P r o , p r o t e í n a
s u p r e s s o r a d e s i l e n c i a me n t o . C - D u p l i c a t a s d e N i c o t i a n a b e n t h a m i a n a i n o c u l a d a s c o m a s c o n s t r u ç õ e s p B I N G F P e o v e t o r p B I N p l u s l i g a d o a c a d a u m d o s g e n e s c a n d i d a t o s a s u p r e s s ã o d o s i l e n c i a me n t o d e R N A i . . . 4 4 F i g u r a 1 3 : V e t o r d e e x p r e s s ã o p E T - 1 7 b . T a ma n h o d e 3 3 0 6 b a s e s , s í t i o d e
t a ma n h o s e s p e r a d o s p a r a a d i g e s t ã o d e N S 4 B e N S 5 f o i d e 2 1 4 6 p b e 4 0 9 9 p b , r e s p e c t i v a me n t e . ... 53 F i g u r a 1 7 : D i g e s t ã o d o v e t o r p R A P s c o n t e n d o g e n e N S 1 c o m a s e n z i ma s A s c I e P a c I . 1 : 1 k b p l u s D N A L a d d e r ( I n v i t r o g e n ) , ma r c a d o r d e ma s s a mo l e c u l a r ; 2 : V e t o r p R A P s N S 1 d i g e r i d o c o m A s c I e P a c I . O p r o d u t o d a d i g e s t ã o p u r i f i c a d o p a r a c l o n a g e m t e m t a ma n h o e s p e r a d o d e 2 4 5 5 p b . ... 54 F i g u r a 1 8 : D i g e s t ã o d o v e t o r p R A P S c o n t e n d o g e n e N S 3 c o m a s e n z i ma s A s c I e P a c I . 1 : M a r c a d o r d e ma s s a mo l e c u l a r 1 k b p l u s D N A L a d d e r ( I n v i t r o g e n ) ; 2 : V e t o r p R A P s N S 3 d i g e r i d o c o m A s c I e P a c I . O p r o d u t o d a d i g e s t ã o p u r i f i c a d o p a r a
c l o n a g e m t e m t a ma n h o e s p e r a d o d e 3 2 5 6 p b . ... 54 F i g u r a 1 9 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m a s c o n s t r u ç õ e s p B I N G F P e p B I N N S 1 . B - C o n t r o l e p o s i t i v o : f o l h a i n o c u l a d a c o m a c o n s t r u ç ã o p B I N H c P r o e p B I N G F P . F o l h a s i r r a d i a d a s c o m l u z u l t r a v i o l e t a s e i s d i a s a p ó s a i n o c u l a ç ã o . ... 57 F i g u r a 2 0 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m a s c o n s t r u ç õ e s p B I N G F P e p B I N N S 2 A . B - C o n t r o l e p o s i t i v o : f o l h a i n o c u l a d a c o m a p r o t e í n a H c P r o e G F P . F o l h a s i r r a d i a d a s c o m l u z u l t r a v i o l e t a s e i s d i a s a p ó s a i n o c u l a ç ã o . ... 57 F i g u r a 2 1 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m a s c o n s t r u ç õ e s p B I N G F P e p B I N N S 2 B . B - C o n t r o l e p o s i t i v o : f o l h a i n o c u l a d a c o m a s c o n s t r u ç õ e s p B I N H c P r o e p B I N G F P . F o l h a s i r r a d i a d a s c o m l u z u l t r a v i o l e t a s e i s d i a s a p ó s a i n o c u l a ç ã o . . 58 F i g u r a 2 2 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m a s c o n s t r u ç õ e s G F P e p B I N N S 3 . B - C o n t r o l e p o s i t i v o : f o l h a i n o c u l a d a c o m a s c o n s t r u ç õ e s p B I N H c P r o e p B I N G F P . F o l h a s i r r a d i a d a s c o m l u z u l t r a v i o l e t a s e i s d i a s a p ó s a i n o c u l a ç ã o . ... 58 F i g u r a 2 3 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m a s c o n s t r u ç õ e s p B I N G F P e p B I N N S 4 A . B - C o n t r o l e p o s i t i v o : f o l h a i n o c u l a d a c o m a s c o n s t r u ç õ e s
F i g u r a 2 5 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m a s c o n s t r u ç õ e s p B I N G F P e p B I N N S 5 . B - C o n t r o l e p o s i t i v o : f o l h a i n o c u l a d a c o m a s c o n s t r u ç õ e s p B I N H c P r o e p B I N G F P . F o l h a s i r r a d i a d a s c o m l u z u l t r a v i o l e t a s e i s d i a s a p ó s a i n o c u l a ç ã o . ... 60 F i g u r a 2 6 : A - F o l h a d e N . b e t h a m i a n a a g r o i n f e c t a d a c o m A g r o b a c t e r i u m
t u m e f a c i e n s L B A 4 4 0 4 e a c o n s t r u ç ã o p B I N G F P . B - C o n t r o l e p o s i t i v o : f o l h a
i n o c u l a d a c o m a s c o n s t r u ç õ e s p B I N H c P r o e p B I N G F P . F o l h a s i r r a d i a d a s c o m l u z u l t r a v i o l e t a s e i s d i a s a p ó s a i n o c u l a ç ã o . ... 60 F i g u r a 2 7 : E l e t r o f o r e s e e m g e l d e p o l i a c r i l a mi d a , c o m a s p r o t e í n a s r e s u l t a n t e s d a e x t r a ç ã o d a s f o l h a s i n o c u l a d a s c o m a s p r o t e í n a s t e s t a d a s . M M : M a r c a d o r d e m a s s a mo l e c u l a r B e n c h M a r k p r o t e i n L a d d e r ( I n v i t r o g e n ) . ... 62 F i g u r a 2 9 : P r o t e í n a s e x p r e s s a s n o s p e r í o d o s d e 4 , 6 , 1 8 e 2 4 h o r a s p ó s i n d u ç ã o . P o s s í v e l p r o d u t o d a e x p r e s s ã o c o m ma s s a e q u i v a l e n t e a 2 5 K D a . C o n t r o l e n e g a t i v o : P r o d u t o d a e x p r e s s ã o d e p r o t e í n a s t o t a i s d e E . c o l i t r a n s f o r ma d a c o m p E T 1 7 b s e m a p r o t e í n a N S 2 A . M M : M a r c a d o r d e ma s s a mo l e c u l a r B e n c h M a r k p r o t e i n L a d d e r ( I n v i t r o g e n ) . ... 66 F i g u r a 3 0 : D o t I mu n o b i n d i n g A s s a y . D e t e c ç ã o d a p r o t e í n a N S 2 A f u s i o n a d a a u ma c a u d a d e h i s t i d i n a , e m e n s a i o i mu n o l ó g i c o u t i l i z a n d o a n t i c o r p o a n t i - H i s t a g .
LISTA DE TABELAS
INTRODUÇÃO
Histórico do Dengue
O primeiro marco epidêmico do Dengue ocorreu em 1779 no Cairo e na
Batavia citado por Siler et al. (1926). Registros posteriores foram
encontrados na Filadélfia, Zanzibar, Calcutá, Índi e Hong Kong (1901).
Embora os primeiros registros epidêmicos da doença tenham ocorrido em três
continentes (Ásia, África e América do Norte) em 1779 e 1780, foram
encontrados registros anteriores na enciclopédia chinesa publicada durante a
Dinastia Chinesa (265-420 A.D). A doença foi chamada pelos chineses de
“água envenenada” e foi pensado estar associada aos insetos que sobrevoavam
essas águas (HENCHAL & PUTNAK, 1990)
Foi citado por Henchal (1990), que a doença foi considerada um dos
maiores problemas de saúde pública nas regiões tropicais e subtropicais,
contudo somente a partir de 1906 foi demonstrado que o mosquito Aedes
aegypti era o agente transmissor do vírus.
As condições ambientais geradas após a II guerra mundial criaram um
cenário propício à propagação de doenças transmitidas por mosquitos e esse
contexto deu início a uma pandemia global do Dengue. Até 1997, o vírus já
havia alcançado uma ampla distribuição no mundo e atualmente estima-se a
ocorrência de 50-100 milhões de casos por ano em regiões tropicais e
subtropicais, dentre os quais 500.000 resultam em febre hemorrágica e choque
do Dengue, com mais de 20.000 mortes (GUZMAN & KOURI, 2003)
O vírus do Dengue apresenta quatro sorotipos conhecidos como DENV-1,
tipos sorológicos de um mesmo vírus, geralmente são designados como
espécies diferentes que são antigenicamente relacionadas (KURANE, 2007).
A relação sorológica entre o vírus do Dengue e outros Flavivirus foi
demonstrada por Sabin (1950), que observou a existência de reações
sorológicas cruzadas entre soro humano com anticorpos contra Dengue e
antígenos do vírus de Febre Amarela, Encefalite Japonesa e West Nile virus.
O compartilhamento de epítopos comuns, presentes na proteína do envelope
desses vírus, ocasiona que os testes sorológicos feitos para diagnosticar a
doença sejam positivos para diferentes vírus da mesma família (GUBLER,
1998).
A infecção com um dos sorotipos apresenta manifestações clínicas
restritas, cujos sintomas são brandos e na maioria dos casos não são
facilmente distinguidas de outras infecções virais (SETIATI et al., 2007).
Essa primeira exposição a um dos sorotipos não é capaz de conferir
imunidade contra a doença, podendo o indivíduo ser reinfectado por um
diferente sorotipo do vírus e durante a infecção secundária desenvolver um
quadro ainda mais grave. Temos como exemplo, registros de que a
co-circulação dos sorotipos 1, 2 e 3 tem sido apontada como a causa do aumento
das formas severas da doença no Brasil (GUZMAN et al., 1991).
O contexto atual aponta que a existência dessas variações sorológicas do
vírus constitui-se na maior dificuldade para o desenvolvimento de uma vacina
contra o vírus do Dengue, cuja eficiência é restrita a um caráter tetravalente
(WHITEHEAD et al., 2007). Tentativas recentemente direcionadas ao
combate da doença, utilizando proteínas como elicitores da resposta imune do
conhecido pelo aumento da infecção dependente de anticorpos, que além dos
efeitos desejáveis também induziram a produção de anticorpos
não-neutralizantes, aumentando o surgimento de febre hemorrágica como um
efeito de infecção secundária (COSTA et al., 2007). Portanto, até os dias
atuais, as medidas de controle e prevenção da doença permanecem focadas no
agente transmissor Aedes aegypti, evidenciando a necessidade de
desenvolvimento de novas estratégias co ntra o vírus e tratamento da doença.
Dengue no Brasil
O histórico do Dengue no Brasil teve início em 1846, com os primeiros
registros de surtos do Dengue ocorridos na cidade do Rio de Janeiro. O país
permaneceu livre do vetor Ae. aegypti até 1976 através de um programa de
erradicação contra Febre Amarela (RIGAU-PÉREZ et al., 1998). Contudo, as
áreas urbanas sofreram uma reinfestação do vetor nos anos 80, resultando
numa explosão da doença, segundo a Organização Pan-americana de Saúde
Figura 1: Q u a d r o d a e v o l u ç ã o d o D e n g u e n o p e r í o d o d e 1 9 8 0 - 2 0 0 7 ( Or g a n i z a ç ão
P a n A me r i c a n a d e S aú d e, 2 0 0 7 ) .
Observa-se que a doença tornou-se um problema de saúde pública com a
introdução do sorotipo DENV-1 em 1986, DENV-2 em 1990 e DENV-3 em
2000, registrados na região sudeste (MIAGOSTOVICH et al., 2002). Em 2003, o
vírus foi isolado em 16% dos casos analisados e os levantamentos estatísticos
feitos pela Organização Pan-americana de Saúde (OPAS), indicavam que no
Brasil estava estabelecida a predominância do sorotipo DENV-3,
representando 76,8% dos casos, seguido por DENV-1 (17,4%) e DENV-2
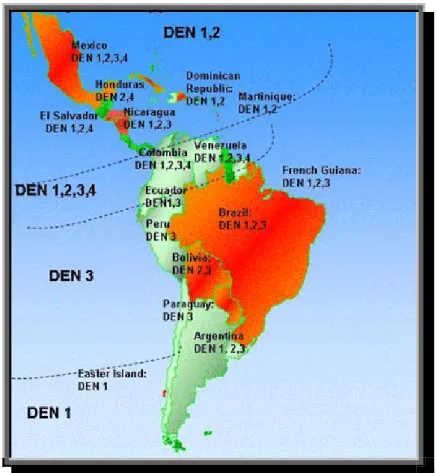
(5,8%). Atualmente, o Brasil permanece livre do sorotipo DENV-4 (Figura 2),
mas os riscos de uma repentina introdução tornam-se cada vez maiores,
devido ao índice de turismo no país e a existência de uma frágil fronteira com
Figura 2: M a p a i l u s t r a n d o a c o - c i r cu l a ç ão d o s s o r o t i p o s d o D en g u e e n t r e o s p a í s e s
d e ma i o r i n c i d ên c i a d a d o en ç a n o p e r í o d o d e 2 0 0 6 - 2 0 0 7 ( O r g a n i z a ç ã o P an
A me r i c a n a d e S a ú d e ) .
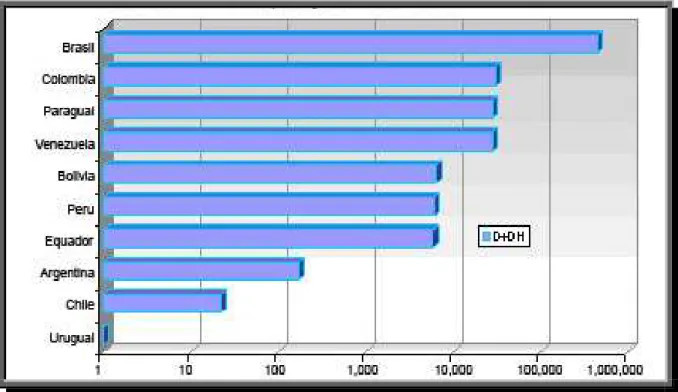
O Brasil tem sido apontado como o país de maior índice de casos da
doença entre os países da América do Sul (Figura 3). Em 2007, foram
reportados 76.626 casos do Dengue na região que compreende a Bolívia,
Colômbia, Equador, Peru e Venezuela. Dentre esses casos, 5.821 foram
identificados como Dengue hemorrágica e houve um registro de 25 mortes.
Contudo a região do cone Sul (Argentina, Brasil, Chile, Paraguai e Uruguai) é
responsável por 63% dos casos de mo rte pelo Dengue, com 94% dos casos
136.488 casos comparado a 2006 (Dados fornecidos pela Organização
Mundial de Saúde, 2008).
Figura 3: N ú me r o d e c a s o s d e D e n g u e r e g i s t r a d o ent r e o s p a í se s d o C o n e S u l , e m
Dengue
A transmissão da doença é dada quando a fêmea da espécie Aedes aegypti
alimenta-se do sangue de um indivíduo infectado, tornando-se hospedeira
intermediária do vírus, o qual se replica no seu mesentério e depois se espalha
para outros órgãos até alcançar as glândulas salivares. Então, o vírus passa a
ser transmitido através da saliva, enquanto a fêmea alimenta-se do sangue de
um novo hospedeiro (BANCROFT, 1906).
A infecção causada pelo vírus do Dengue pode ser assintomática, mas
costuma ser conhecida por manifestações de febre, dores de cabeça, dor
retro-orbital, mialgia, leucopenia e dores nas articulações. Esses sintomas
costumam aparecer no indivíduo infectado após um período de incubação de
2-7 dias e normalmente a recuperação é alcançada uma semana após essas
manifestações. Ainda que a maioria dos casos não represente um grande risco
para população, alguns pacientes desenvolvem a forma severa da doença,
conhecida como Febre Hemorrágica do Dengue (FHD). Podendo evoluir para
Síndrome de Choque do Dengue que é capaz de levar um indivíduo a morte
em 24 horas (Organização Mundial de Saúde, 1997).
A Febre Hemorrágica é caracterizada por um quadro de febre aguda,
tendências hemorrágicas seguidas de perda de plasma, devido ao aumento da
permeabilidade vascular que também pode acarretar em efusão pleural,
ascites, hipoproteinemia e aumento do hematócrito. Quando o quadro descrito
evolui para o choque, todos os sintomas são mantidos além de uma grave
hipotensão, levando a falência circulatória (GUBLER, 1998).
virulentas causando Febre do Dengue e cepas avir ulentas provocando Febre
Hemorrágica (COLOGNA et al., 2005), outra, considera a própria resposta
imune do hospedeiro como o agente intensificador da infecção. Foi reportada
por Mady (1991), a formação de um complexo vírus-anticorpo não
neutralizante, que se liga a receptores celulares aumentando a infecção
causada pelo vírus do Dengue. Esse fenômeno é conhecido como Aumento
Dependente de Anticorpo, ocorrendo durante uma infecção secundária.
Devido ao caráter infeccioso e a ampla disseminação anteriormente
citada, Dengue já havia sido considerada a mais importante doença viral
transmitida por artrópodes desde 1928, quando 650.000 moradores de Atenas
contraíram Febre do Dengue e 1061 pessoas foram mortas pelo agente
causador da doença (HALSTEAD & PAPAEVANGELOU, 1980)
Vírus do Dengue
O agente causal do Dengue é um vírus da família Flaviviridae e tal
como outros Flavivirus, são pequenas partículas (50 nm em diâmetro)
envelopadas, contendo o genoma de fita simples positiva de RNA com
aproximadamente 11 Kb (HAHN et al., 1988). Esse genoma codifica uma
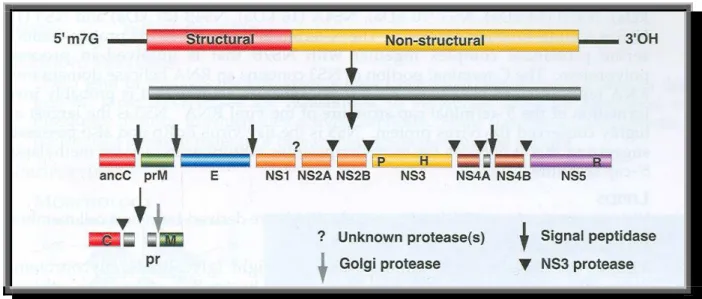
poliproteína precursora com cerca de 3.000 aminoácidos, que é processada
durante e após a tradução por prot eases virais, gerando as proteínas
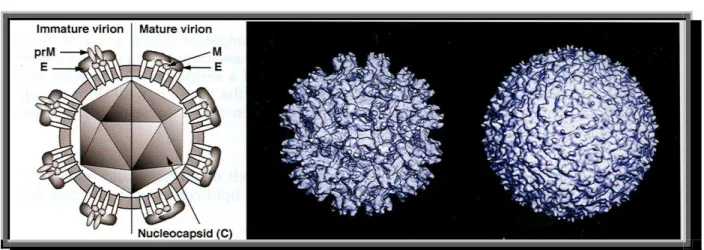
estruturais e não estruturais do vírus (Figura 5). Como característica
capisídio (C), premembrana (prM) e envelope (E), formadoras do vírion
(Figura 4), além de sete proteínas não-estruturais, NS1, NS2A, NS2B, NS3,
NS4A, NS4B e NS5 (Rice et al., 1985).
A B
Figura 4: V í r io n do D e n gu e . ( Fo nt e : V ír u s T a xo no my , 20 05 ). A- E s qu e ma d o v i ri on
ma d u r o e ima t u r o : E - E nv e lo p e, p r M- pr é -M e mb r a n a , C - C a p sí de o , M - M emb r a n a ;
B- I ma g e m d a s u p e r f í c i e d o v í r u s .
A glicoproteína E, presente na superfície do vírus media a fusão celular, apresentando
três domínios: o domínio central, o domínio de dimerização e o domnio de ligação, citado por
Hung (1999) O vírion entra na célula por endocitose mediada por receptor, processo no qual o
domínio de ligação da proteína E atua efetivamente. Dentro da vesícula endossomal,
mudanças conformacionais do vírion induzidas por pH, fusão entre a membrana do vírus e da
célula hospedeira e dissociação de partículas virais ocorrem dando início ao ciclo de vida do
vírus dentro célula. O genoma é liberado no citoplasma, contudo a replicação ocorre dentro de
uma membrana intracelular. Após a replicação, ocorre reunião das partículas virais na
RE através do Golgi, onde é promovida a maturação por clivagem de furinas do hospedeiro e
logo após a maturação, o vírion é liberado por exocitose (MUKHOPADHYAY et al., 2005).
A importância de proteínas não-estruturais do vírus
Proteínas não-estruturais de Flavivirus são conhecidas por desempenhar
papéis importantes na replicação do vírus e por serem determinantes de
patogenicidade. A atividade de polimerase, realizada por proteínas não
estruturais, foi detectada pela primeira vez em Flavivirus pelos pesquisadores
Chu e Westaway (1985), que observaram sua existência na me mbrana
perinuclear do Retículo Endoplasmático, onde foram co-localizadas NS3 e
NS5 (CHAMBERS et al., 1990). Estudos feitos com antisoro de coelho para
essas proteínas foram capazes de inibir a atividade da polimerase,
confirmando que essas proteínas estão envolvidas na replicação de RNA
(BARTHOLOMEUSZ & WRIGHT, 1993).
NS5 é a maior proteína codificada pelo genoma do Dengue virus,
possuindo 104 kDa, com a região amino terminal apresentando similaridade
com metiltransferases de inúmeras espécies e oito motivos conservados
encontrados em muitas RNA polimerases dependentes de RNA (RdRP). A
proteína NS5 foi encontrada formando um complexo com NS3 em células de
macaco infectadas com DENV-2 (KAPOOR et al., 1995).
A proteína NS3 é a segunda maior proteína encontrada em Flavivirus
com cerca de 70 kDa, apresenta 1/3 da porção N-terminal com características
um co-fator para a clivagem das junções NS2A-NS2B, NS2B-NS3, NS3-NS4A
e NS4B-NS5 da poliproteína (Figura 5). A porção restante de NS3 forma um
domínio helicase que age em conjunto NS5, durante a polimerização
(JOHANSSON et al., 2001).
Figura 5: E s t ru tu r a e s qu e má t i c a do g e no ma d o v ír u s do d en gu e . ( Font e : V ír u s
Taxo no my , 200 5 ).
P r o t e í n a s es t r u t u r a i s : C a p s í d e o , M e mb r a n a , E n v el o p e
Estudos realizados na FIOCRUZ do Rio de Janeiro demonstraram que a
proteína NS1 também está envolvida na replicação do vírus, através da
construção de um DNA complementar infeccioso, que continha mutações em
regiões conservadas de NS1 (SUZUKI et al., 2007). Quando essa proteína
mutante foi introduzida em células Vero, resultou na redução da replicação do
RNA. A presença dessa proteína em altos níveis no soro de pacientes tem sido
correlacionada com a severidade da doença em casos de infecções pelo DENV
(ALCON et al., 2002). Além do seu envolvimento na replicação, muitos
estudos têm relatado que NS1 é uma proteína secretada, encontrada em
células de mamíferos associada à membrana e na circulação (SCHLESINGER
et al., 1987). Por ser secretada, NS1 entra em contato com células de defesa
circulantes e é alvo da resposta imune promovida pelo hospedeiro.
Considerando essa afirmativa, anticorpos produzidos contra NS1 foram
analisados e demonstraram conferir proteção contra doenças induzidas por
Flavivirus e foram usadas profilaticamente e terapeuticamente em modelo
animal (FALGOUT et al., 1990).
Pequenas proteínas hidrofóbicas compõem a parte restante do genoma,
essas proteínas são NS2A, NS2B, NS4A e NS4B. As funções dessas proteínas
ainda não estão bem elucidadas, mas alguns esforços têm sido feitos
identificando o envolvimento delas na formação do complexo de replicação ou
atuando como co-fatores. O processamento eficiente da proteína NS1 requer a
participação de NS2A e análises mutacionais indicaram que a própria NS2A
apresenta atividade proteolítica (FALGOUT et al., 1989). Ambas, NS2A e
NS4A foram localizadas dentro de uma vesícula, formando um suposto
Muitas funções como as que foram aqui destacadas têm sido atribuídas às
proteínas não estruturais, contudo ainda existem inúmeras perguntas que
encontraram lugar no cenário das pesqui sas realizadas nos últimos anos. Um
desses questionamentos está centrado no envolvimento de proteínas não
estruturais em mecanismos contra defesa antivirais.
O comportamento dos vírus e seu caráter de virulência têm sido alvos de
pesquisa desde as primeiras manifestações infecciosas e têm sido bem
descrito através de estudos utilizando patógenos de plantas (REAVY et al.,
2004). Um exemplo bem-sucedido na utilização de sistemas de plantas para
avaliação da indução e supressão do silenciamento de RNA por vírus que
infectam células humanas foi descrito recentemente, utilizando uma proteína
não estrutural do vírus da influenz a humana. Comparado aos padrões de
supressão do silenciamento de genes encontrados em vírus de plantas, a
proteína NS1 da influenza humana apresentou capacidade de proteger RNA
viral contra degradação e aumentar significativamente a expressão de
proteínas cujo gene havia sido suprimido (BUCHER et al., 2004). A
viabilidade desses estudos rendeu, nos últimos tempos, grande esclarecimento
quanto aos mecanismos de defesa e escape, como uma das maiores expressões
da co-evolução estabelecida entre vírus e hospedeiro (PRUSS et al., 1997).
Muitos outros estudos feitos com proteínas não-estruturais têm
consolidado a idéia de que além das funções relacionadas à virulência, essas
proteínas podem fazer parte do mecanismo de escape dos vírus contra
estratégias de defesa do hospedeiro (BRIGNETI et al., 1998). Portanto,
considerando o caráter essencial de cada uma delas para a sobrevivência do
desenvolvimento de novos medicamentos e a identificação de atividade
supressora de silenciame nto de genes oferecem possibilidades, ainda mais
relevantes no tratamento à doença causada pelo vírus do Dengue.
Silenciamento de genes
RNA interferente é um mecanismo que atua na indução do silenciamento
de genes. Foi descrito originalmente em plantas e designado como
co-supressão (NAPOLI et al., 1990) e depois como silenciamento
pós-transcricional de genes, em animais, conhecido como interferência de RNA
mediada por uma dupla fita de RNA. Foi observado em fungos e nematóides,
quando seqüências de oligonucleotídeos foram inseridas em células resultando
na redução da expressão de genes (FIRE et al., 1998).
Em experimentos de super expressão de genes de Chalcona sintase,
relacionados à síntese de pigmentos, pela primeira vez foi observado o
fenômeno de silenciamento mediado por RNA, resultando na hipopigmentação
de petúnias. Surpreendentemente, a introdução desses genes em plantas
pigmentadas levou ao bloqueio da síntese de antocianina e esse fenômeno foi
relacionado aos níveis de mRNA que demonstraram ser 50 vezes menores nas
plantas sem pigmentos comparadas ao tipo selvagem (NAPOLI et al., 1990).
Fire e colaboradores (1998) descobriram que a introdução de uma dupla
fita de RNA dentro de Caenorhabditis elegans, diferente de injeções contendo
efeitos de manipulação da expressão nesses experimentos não foram
observados apenas nos animais injetados, mas também na sua progênie.
Esses primeiros estudos revolucionaram a pesquisa devido a
possibilidade de manipulação do controle da expressão gênica pela introdução
de uma seqüência nucleotídica exógena e após a descoberta desse fenômeno
em plantas, Tuschl e coloboradores (1999) mostraram que RNAi também
ocorre em mamíferos, corroborando com a idéia de conservação desse
mecanismo em células eucarióticas (ELBASHIR et al., 2001).
Tem sido afirmado que a interferência de RNA é usada naturalmente
como forma de imunidade primitiva para proteger o genoma contra invasões
de ácidos nucléicos exógenos, introduzidos por vírus ou retrotransposons. A
maior evidência de que o silenciamento mediado por RNA é um mecanismo
natural de defesa contra vírus, reside no fato de que mutações em genes que
codificam componentes dessa maquinaria resultam em aume nto da
susceptibilidade ao vírus (GITLING & ANDINO, 2003).
Além do papel antiviral, diversas funções têm sido apontadas como
conseqüência do silenciamento de RNA em muitos organismos. Isso inclui a
defesa do genoma contra transposons (VOINNET, 2002) estabilidade do
genoma por regulação da formação de heterocromatina (HAMILTON et al.,
2002), controle do desenvolvimento em plantas e animais, e baixa regulação
da expressão de genes por clivagem específica e repressão traducional de
RNAs mensageiros complementares por pequenos e micro RNAs (LI & DING,
2005).
Na busca pela elucidação de como o RNA interferente atua, foi
das seqüências exógenas (Figura 6), iniciando um processo de degradação de
RNA mensageiro com seqüência específica (TUSCHL et al., 1999). A dupla
fita de RNA é clivada, em seqüências que variam entre 21-24 nucleotídeos,
por uma RNase chamada Dicer (KETTING et al., 2001). Esses pequenos
RNAs são incorporados dentro de um complexo efetor do silenciamento
chamado RISC e guiam a maquinaria em direção a seqüências complementares
a fim de promover a supressão (ZAMORE et al., 2000).
Tem sido proposto para invertebra dos que esses pequenos RNAs
(siRNAs) funcionem como primers, sendo estendidos ao longo do RNA alvo
por uma RNA polimerase dependente de RNA (RdRp), contudo isso não foi
observado em animais (LIPARDI et al., 2001). A presença da RNA
polimerase evidencia uma das formas existente para formação da dupla fita de
RNA que inicia a maquinaria do silenciamento. Outro caminho é trilhado por
vírus de RNA em sua fase replicativa, onde uma dupla fita de RNA també m é
formada e é proposto para vírus de DNA que uma transcrição bidirecional do
Replicação viral
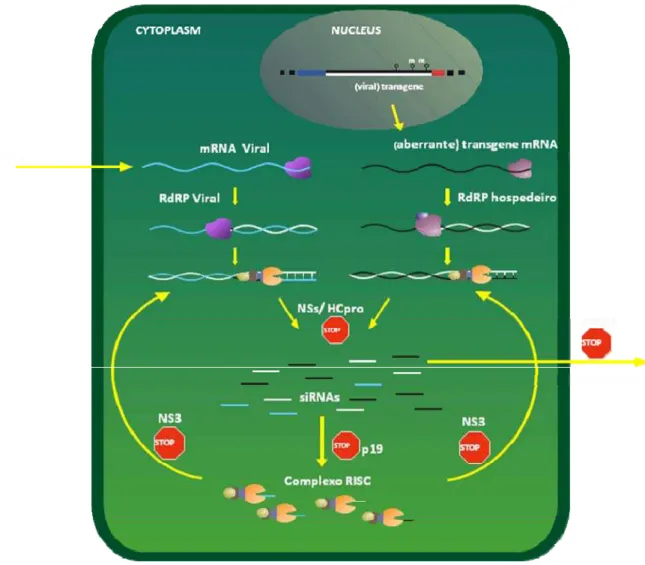
RNAs Aberrantes Transposon
dsRNA
DICER
RdRp
siRNA
RISC
Degradação
Figura 6: E s q u e ma d o p r o c e s so d e s i l en c i ame n t o me d i a d o p o r R N A . A d u p l a f i t a d e
R N A ( d s R N A ) é d eg r ad a p el a r i b o n u cl ea s e D I C E R , g er a n d o p eq u e n o s R N A s
( s i R N A s ) q u e s ã o in c or po r ados no co mp l ex o e n zimá t i c o R IS C , po d end o s e r
u t i l i z ado s c o mo p r i me r p e l a R NA p o l i me r a s e d e p en d en t e d e R NA ( R d R p ) . A d a p t a d o
Além da conservação do mecanismo observada em muitos organismos, o
esclarecimento de como se dá o processo de silenciamento utilizando sistemas
de plantas, tem favorecido novas aplicações biotecnológicas. Utilizando esses
recursos, vírus que infectam plantas têm sido muito bem estudados e grande
parte tem apresentado capacidade de recrutar os componentes do
silenciamento de genes em seus hospedeiros (ROTH et al., 2004). Contudo,
atualmente vírus que infectam outros organismos, além das plantas, têm sido
testados quanto à capacidade de indução e supressão desse mecanismo
(BUCHER et al., 2004).
Como resultado desse novo direcionamento, foi demonstrado que é
possível inibir a produção do vírus do Dengue por transfecção de uma dupla
fita de RNA em células do vetor (CAPLEN et al., 2001). Tem sido até mesmo
proposta uma nova estratégia baseada em silenciamento de RNA para o
controle da transmissão da doença por Aedes aegypti, visto que a interferência
mediada pelo RNA pode servir como mecanismo de resistência em células do
mosquito (ADELMAN et al., 2002). Além disso, a transdução das fêmeas de
Aedes aegypti com um vetor de expressão contendo a seqüência de uma
proteína do vírus do Dengue, conteve a replicação do vírus na glândula
salivar, tornando a fêmea incompetente para a transmissão do vírus
(TRAVANTY et al., 2004). Todos esses avanços têm sugerido que o
silenciamento mediado por RNA é um processo que pode ser naturalmente
induzido, mas não prevalente devido à existência de um possível mecanismo
Supressores do silenciamento de genes
Muitos supressores de interferência de RNA têm sido identificados em
resposta a estratégias de defesa de pl antas infectadas por vírus. A primeira
proteína a apresentar capacidade de suprimir o silenciamento mediado por
RNA foi uma proteinase (Hc-Pro) de Potyvirus, essa proteína demonstrou
capacidade de reversão do silenciamento de genes já estabelecido no
hospedeiro (BRIGNETI et al., 1998).
A proteína 2b de Cucumber mosaic virus também foi encontrada
assumindo atividades supr essoras do silenciamento e constitui-se num dos
exemplos mais estudados desse mecanismo. Diferente da proteína Hc-Pro, 2b
promove a inibição de etapas iniciais do silenciamento e não se demonstrou
capaz de suprimir etapas já estabelecidas do processo, evidenciando que
supressores podem assumir caminhos distintos para bloquear a interferência
de RNA (BRIGNETI et al., 1998).
Alguns supressores reduzem o acúmulo de pequenos RNAs interferentes
(siRNAs), sugerindo que a supressão ocor re durante o processamento da dupla
fita de RNA pela enzima Dicer (BUCHER et al., 2003). Outras proteínas não
interferem no acúmulo de siRNAs, mas interceptam sua incorporação no
complexo enzimático RISC, bloqueando uma etapa distinta do processo
(LAKATOS et al., 2004).
Inicialmente, dois grupos de supressores foram identificados. Aqueles
que degradam um componente requerido para a manutenção do silenciamento,
e/ou a ativação do componente requerido para o silenciamento, são restritos
as folhas novas (VOINNET et al., 1999).
A existência de um mecanismo de supressão do silenciamento de genes
tem sido bem elucidada, utilizando vírus que infectam plantas como modelo
experimental. Contudo já tem sido feita a aplicação desse mesmo modelo na
busca de supressores produzidos por vírus que infectam humanos. O vírus da
influenza humana foi recentemente testado quanto à existência de atividade
supressora pela proteína não-estrutural NS1 (BUCHER et al., 2004) os
resultados positivos desses estudos abriram caminho para novos
questionamentos, gerando a necessidade de novos testes capazes de identificar
o potencial de supressão em proteínas de outros vírus.
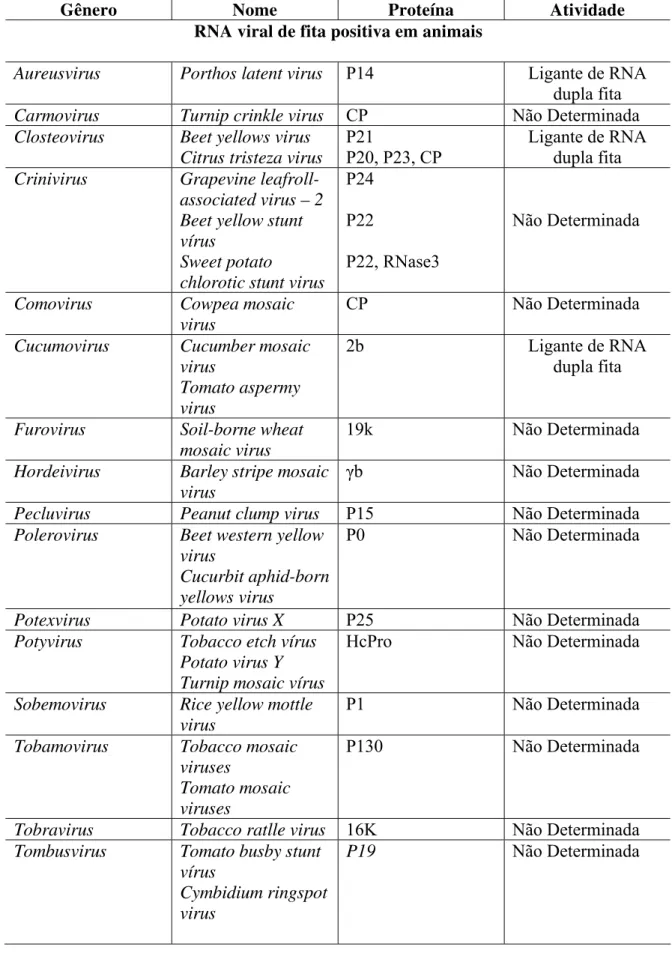
Consistente com a hipótese de que determinantes de patogenicidade são
candidatos a supressores, diversas proteínas foram encontradas exercendo
papéis de supressão no silenciamento e atuando como fatores essenciais na
patogênese (Tabela 1). Alguns mecanismos só foram esclarecidos através da
manipulação de determinadas proteínas, apontando pelo menos três supostos
caminhos pelos quais esses supressores têm interferido no silenciamento
Figura 7: A s p r ot e ína s H C p ro de P o ti v i ru s, N S s d e To s po v i ru s N S 3 d e T en ui v i ru s e
p 19 d e Tom b u s vi r u s i l u st r a m o s t r ê s p o s s ív e i s c a mi n h o s u t il i z ad o s p or s up r e s so r e s
d e s i l en c i ame n t o : I n at i v a ç ão d e D I C E R , d eg r ad a ç ã o d e p eq u en o s R N A s ( si R N A s ) e
b l o q u e i o d e s u a i n c o r p o r a ç ã o n o c o mp l ex o e n zi má t i c o R I S C . ( C e d i d o e au t o r i z ad o
Através do estudo de função da proteína p19 de tombusvirus foi proposto
que o silenciamento suprimido durante a infecção com esses vírus, é efetuado
pela ligação da proteína p19 a dupla fita de siRNA, impedindo que esta seja
incorporada ao complexo enzimático RISC (SILHAVY et al., 2002). Durante
a infecção com tombusvirus, as folhas superiores são completamente
invadidas por este, indicando que o seqüestramento de siRNAs previne o
desenvolvimento do silenciamento sistêmico, atuando como o próprio sinal
móvel (LAKATOS et al., 2004). É possível que esse sinal seja gerado pela
RNA polimerase dependente de RNA, sendo guiada pelos siRNAs ao
silencimento de RNAs e dessa forma a depleção de siRNAs por p19 resulta
na inativação da geração do sinal (VOINNET, 2001).
Supressão de silenciamento por alguns vírus de ma míferos foi
primariamente demonstrada com a descoberta das proteínas supressoras B2
Flock House virus, NS1 de Influenza virus, e E3L de Vaccinia virus, testadas
em sistemas heterólogos como células de insetos e plantas. Todos esses
supressores demonstraram ser requeridos para a infecção do vírus nas células
de mamíferos e alguns deles também são conhecidos por funcionar no
hospedeiro como inibidores da imunidade antiviral inata regulada por
interferon (GARCIA et al., 2002).
Concomitante com as evidências de que existe uma estratégia de defesa
consistente na presença de supressores, alguns vírus demonstraram ter
desenvolvido outros caminhos, como evitar o silenciamento executando a
replicação dentro de vesículas no Retículo Endoplasmático. Dessa forma a
dupla fita de RNA é protegida do processo de clivagem pela ribonuclease
A neutralização desse mecanismo de escape tem sido considerada um
alvo promissor para o desenvolvimento de novos controles para vírus
(DYKXHOORN & LIEBERMAN, 2005). Além do esclarecimento da
dinâmica do vírus frente ao silenciamento, o qual tem sido provado estar
presente tanto em humanos, quanto nos artrópodes (FRANZ et al., 2006;
Tabela 1. Proteínas supressoras de RNA interferente identificadas em diferentes gêneros de vírus. Adaptada de Li e Ding (2006).
Gênero Nome Proteína Atividade
RNA viral de fita positiva em animais
Aureusvirus Porthos latent virus P14 Ligante de RNA dupla fita
Carmovirus Turnip crinkle virus CP Não Determinada
Closteovirus Beet yellows virus Citrus tristeza virus
P21
P20, P23, CP
Ligante de RNA dupla fita
Crinivirus Grapevine leafroll-associated virus – 2 Beet yellow stunt vírus
Sweet potato
chlorotic stunt virus
P24
P22
P22, RNase3
Não Determinada
Comovirus Cowpea mosaic virus
CP Não Determinada
Cucumovirus Cucumber mosaic virus
Tomato aspermy virus
2b Ligante de RNA
dupla fita
Furovirus Soil-borne wheat mosaic virus
19k Não Determinada
Hordeivirus Barley stripe mosaic virus
b Não Determinada
Pecluvirus Peanut clump virus P15 Não Determinada
Polerovirus Beet western yellow virus
Cucurbit aphid-born yellows virus
P0 Não Determinada
Potexvirus Potato virus X P25 Não Determinada
Potyvirus Tobacco etch vírus Potato virus Y Turnip mosaic vírus
HcPro Não Determinada
Sobemovirus Rice yellow mottle virus
P1 Não Determinada
Tobamovirus Tobacco mosaic viruses
Tomato mosaic viruses
P130 Não Determinada
Tobravirus Tobacco ratlle virus 16K Não Determinada
Tombusvirus Tomato busby stunt vírus
Cymbidium ringspot virus
Tymovirus Turnip yellow mosaic virus
P69 Não Determinada
Vitiviruses
Grapevine vírus A
P10 Não Determinada
RNA viral de fita negativa em plantas Tenuivirus Rice hoja blanca
vírus
NS3 Não Determinada
Tospovirus Tomato spotted wilt vírus
NSs Não Determinada
Dupla fita de RNA viral em plantas
Phytoreovirus Rice dwar virus Pns10 Não Determinada
Virus de DNA em plantas Begomovirus
Curtovirus
Tomato leaf curl virus
Tomato yellow leaf curl virus
Mungbean yellow mosaic virus
Tomato golden musaic virus
Beet curly top virus
C2
C1
AC2
AC4 L2
Ligante de DNA
Ligante de DNA
Ligante de DNA
Ligante de miRNA Ligante de Proteína
Fita positiva de virus de RNA em animais Nodavirus Flock house virus,
Nodamura virus, Stripd Jack nervous nerosis virus,Greasy grouper ne rvous necrosis virus
B2 Ligante de RNA
dupla fita
Fita negativa de vírus de RNA em animais
Orthomyxovirus Infuenza virus A NS1 Ligante de RNA dupla fita
Orthobunyavirus La Crosse virus NSs Não Determinada
Dupla fita de de Rna em animais
Orthoreovirus σ3 Ligante de RNA
dupla fita Retrovirus em animais
Lentivirus HIV-1 Tat Não Determinada
Spumavirus PFV-1 Tas Não Determinada
Vírus de DNA em animais
Adenovirus Adenovius VA1 RNA Ligante de Dicer
Poxvirus Vaccinia virus E3L Ligante de RNA
Expressão de proteínas
O sistema procariótico é um sistema amplamente utilizado para produção de proteínas
heterólogas, possuindo algumas vantagens como o requerimento de uma fonte de baixo custo
de carbono e uma rápida acumulação de biomassa por cultura (SAHDEV et al., 2008). Esse
sistema dispõe de grande quantidade de cepas comercializadas, apresentando diferentes
vantagens e aplicabilidades segundo o interesse em produção de proteínas recombinantes. Um
exemplo dessa diversidade é a bactéria Escherichia coli BL21, uma cepa não patogênica
capaz de crescer vigorosamente em meio mínimo e deficiente em proteases capazes de
interferir no isolamento de proteínas recombinantes intactas (CHART et al., 2000). Diferentes
padrões de fusão são utilizados para facilitar a purificação dessas proteínas expressas, sendo
comum a inserção de caudas de histidina (His-tag) para cromatografia de afinidade a níquel
ou purificação em resina baseado em glutationa, para proteínas fusionadas com caudas de
glutationa S-transferase (GST) (TERPE, 2003).
A expressão de proteínas em sistemas heterólogos possui diversos atributos e a
finalidade de gerar anticorpos tornou-se uma das resultantes desse processo após uma intensa
busca para disponibilização de ferramentas terapêuticas, em alguns casos culminando no
desenvolvimento de vacinas. Recentemente, o domínio III da proteína E do sorotipo 2 do
DENV, que representa um alvo promissor para o desenvolvimento de vacinas devido a sua
capacidade de elicitar anticorpos neutralizantes e papel de interação com a célula hospedeira,
foi expressa em Escherichia coli BL21. A forma purificada da proteína expressa foi
empregada na imunização de camundongos, resultando na indução de anticorpos e proteção
de camundongos desafiados com o sorotipo 2 do DENV (ZHANG et al., 2007)
Um resultado semelhante foi obtido mediante a imunização de coelhos com uma
histidina. Foi observada a geração de um anti-soro específico que reconhece a proteína não
estrutural de células infectadas com o vírus. Mais de 1mg de proteína purificada pôde ser
isolado de 1L de cultura de E. coli com mais de 98% de pureza (LING et al., 2008). Pode
então ser observado que embora a produção de anticorpos possa ser feita em células de
mamíferos, outros sistemas são preferidos, podendo oferecer um alto rendimento do produto
em baixo custo comparado a culturas de células de mamíferos (CHADD & CHAMOW,
JUSTIFICATIVA
O desenvolvimento de novas estratégias de controle capazes de promover
a prevenção e contenção da severidade da doença provocada pelo vírus do
Dengue, ainda é um desafio, considerando os altos índices de morbidade e
mortalidade provocados pelo vírus. Proteínas não-estruturais são conhecidas
por seu papel determinante da patogenicidade e sua importância na replicação
do vírus dentro da célula hospedeira. Esse caráter faz com que essas proteínas
sejam possíveis alvos para o desenvolvimento de antivirais, tendo essa visão
reforçada pelo conhecimento de que algumas proteínas virais atuam como
supressores do silenciamento mediado por RNA, mecanismo conhecido por
desempenhar um papel natural de defesa contra vírus de RNA.
O mecanismo de interferência de RNA observado em plantas demonstrou
ser conservado em ma míferos e já foi observada a indução de silenciamento
de genes em células do vetor Ae. albopictus, corroborando com os indícios de
que esse mecanismo pode ocorrer naturalmente durante uma infecção
provocada pelo Flavivirus. Algumas proteínas produzidas por vírus que
infectam humanos foram identificadas como supressores de RNA interferente,
contudo ainda não foi determinado o papel das proteínas não-estruturais do
vírus do Dengue como supressores do silenciamento de genes.
Visando a oportuna neutralização do mecanismo de escape do vírus,
favorecida pela estratégia de interferência de RNA, esse trabalho propõe a
identificação de proteínas não-estruturais como possíveis supressores do
silenciamento de genes, bem como a expressão de proteínas do Dengue
OBJETIVO GERAL
Estudos recentes têm identificado que vírus de RNA fita simples positiva
produzem proteínas supressoras do silenciamento mediado por RNA, contudo,
ainda não foi identificada a existência desses supressores no genoma do vírus
do Dengue.
Esse trabalho propõe-se a expressar proteínas do Dengue em sistema de
bactéria e de plantas, investigando o papel das proteínas não-estruturais como
agentes supressores de interferência de RNA e identificando o possível gene
candidato ao conhecido mecanismo de supressão.
OBJETIVOS ESPECÍFICOS
• Clonar sete genes (NS1, NS2A, NS2B, NS3, NS4A, NS4B e NS5) de
proteínas não estruturais do vírus do Dengue em vetor de expressão em
plantas;
• Testar a expressão das proteínas não-estruturais em plantas;
• Monitorar o efeito de supressão de genes em plantas expressando
proteínas não-estruturais do vírus do Dengue;
MATERIAL E MÉTODOS
Clonagem de cDNA de genes do vírus da Dengue
Extração de RNA viral
O RNA foi extraído de células C6/36 de Aedes albopictus infectadas com o vírus do
Dengue sorotipo1 (Figura 8), autóctone do Distrito Federal (Isolado HUB 01021093), sendo
utilizado como molde para síntese de um DNA complementar através de transcrição reversa.
As amostras foram preparadas no Laboratório Central de Saúde Pública do Distrito Federal.
Figura 8: C é l u l a s C6/ 3 6 d e A e d e s a l b o p i c t u s i n f e c t a d a s c o m v í r u s d o D en g u e
Uma alíquota de 125μL de células infectadas com o vírus do Dengue em meio de
cultura foi transferida para um microtubo de 1,5 ml contendo 375 μL de Trizol LS, onde o
volume foi homogeneizado e o RNA total foi extraído, seguindo as instruções do fabricante
(Invitrogen). Após a secagem da amostra na etapa final, o pellet foi ressuspendido em 20 μL
Amplificação de genes candidatos a supressores de RNAi
Os fragmentos não-estruturais do vírus foram amplificados através de transcrição
reversa e reação em cadeia da polimerase. A transcrição reversa foi feita com o auxílio da
Transcriptase Reversa SuperScript III (Invitrogen) por 1 hora à 42°C em condições livre de
RNase, seguindo as instruções do fabricante.
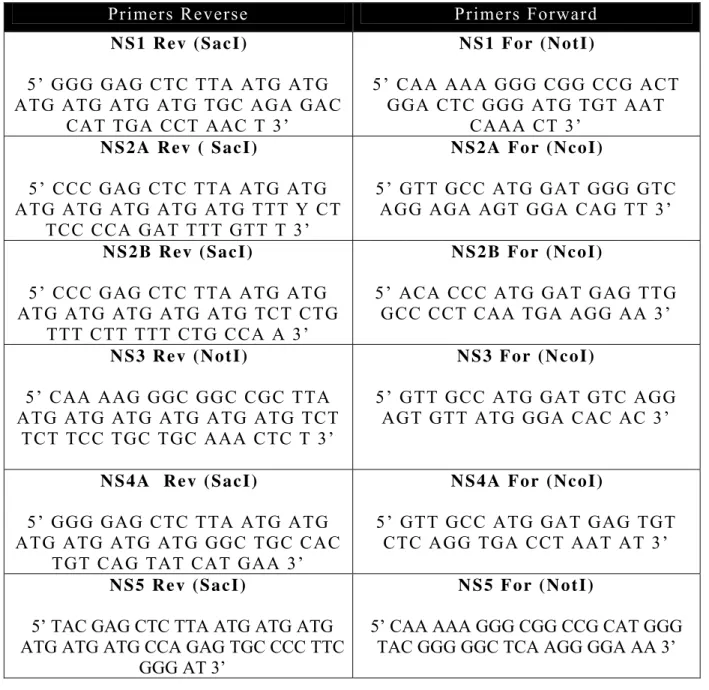
Foram construídos primers específicos para regiões não estruturais de interesse no
genoma do vírus, com os quais foram feitas amplificações dos fragmentos NS1, NS2A,
NS2B, NS3, NS4A, NS4B e NS5, a partir do cDNA resultante da transcrição reversa e a
confirmação do tamanho dos fragmentos feita por eletroforese em gel de agarose 1%, segundo
o protocolo descrito por Sambrook e colaboradores (1989). Foi inserida uma cauda de
pepitídeo de hexa-histidina nas seqüências de primers utilizados para amplificar cada gene
correspondente as proteínas não estruturais do vírus (Tabela 2), com a finalidade de favorecer
Tabela 2: Primers para amplificação de genes que codificam as proteínas não estruturais do Dengue.
Primers Reverse Primers Forward NS1 Rev (SacI)
5’ GGG GAG CTC TTA ATG ATG ATG ATG ATG ATG TGC AGA GAC
CAT TGA CCT AAC T 3’
NS1 For (NotI)
5’ CAA AAA GGG CGG CCG ACT GGA CTC GGG ATG TGT AAT
CAAA CT 3’
NS2A Rev ( SacI)
5’ CCC GAG CTC TTA ATG ATG ATG ATG ATG ATG ATG TTT Y CT
TCC CCA GAT TTT GTT T 3’
NS2A For (NcoI)
5’ GTT GCC ATG GAT GGG GTC AGG AGA AGT GGA CAG TT 3’
NS2B Rev (SacI)
5’ CCC GAG CTC TTA ATG ATG ATG ATG ATG ATG ATG TCT CTG
TTT CTT TTT CTG CCA A 3’
NS2B For (NcoI)
5’ ACA CCC ATG GAT GAG TTG GCC CCT CAA TGA AGG AA 3’
NS3 Rev (NotI)
5’ CAA AAG GGC GGC CGC TTA ATG ATG ATG ATG ATG ATG TCT
TCT TCC TGC TGC AAA CTC T 3’
NS3 For (NcoI)
5’ GTT GCC ATG GAT GTC AGG AGT GTT ATG GGA CAC AC 3’
NS4A Rev (SacI)
5’ GGG GAG CTC TTA ATG ATG ATG ATG ATG ATG GGC TGC CAC
TGT CAG TAT CAT GAA 3’
NS4A For (NcoI)
5’ GTT GCC ATG GAT GAG TGT CTC AGG TGA CCT AAT AT 3’
NS5 Rev (SacI)
5’ TAC GAG CTC TTA ATG ATG ATG ATG ATG ATG CCA GAG TGC CCC TTC
GGG AT 3’
NS5 For (NotI)
5’ CAA AAA GGG CGG CCG CAT GGG TAC GGG GGC TCA AGG GGA AA 3’
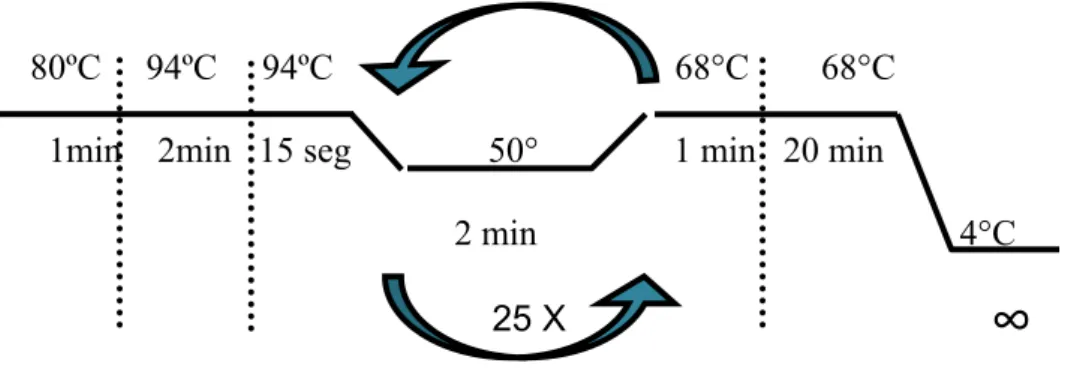
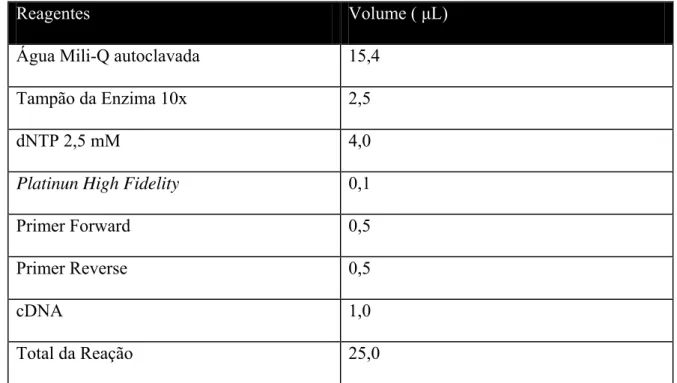
A reação em cadeia da polimerase (Tabela 3) foi feita utilizando a
enzima “Platinum Taq DNA Polimerase High Fidelity” (Invitrogen), que
apresenta atividade de auto-correção aumentando em seis vezes a fidelidade
das seqüências amplificadas, comparada à Taq DNA polimerase. Foi utilizado
um programa de 25 ciclos (Figura 9), realizado no termociclador Master
cycler (Eppendorf). O DNA dos produtos amplificados foram eluídos do gel
utilizando-se o kit Quick Gel Extration (Invitrogen), segundo o protocolo
oferecido pelo fabricante e o conteúdo resultante foi submetido a uma etapa
de digestão por enzimas de restrição específicas para cada seqüência
amplificada (Tabela 4).
80ºC 94ºC 94ºC 68°C 68°C
1min 2min 15 seg 50° 1 min 20 min
2 min 4°C
Figura 9: E s q u e ma d a s e t a p a s p a r a r e a l i z aç ã o d a a mp l i f i c a ç ão d o s g e n e s c an d i d at o s a s u p r e s so r e s d e R N A i n t er f e r en t e .
Tabela 3. Componentes da solução de PCR para amplificação das proteínas não-estruturais.
Reagentes Volume ( μL)
Água Mili-Q autoclavada 15,4
Tampão da Enzima 10x 2,5
dNTP 2,5 mM 4,0
Platinun High Fidelity 0,1
Primer Forward 0,5
Primer Reverse 0,5
cDNA 1,0
Clonagem dos genes de proteínas não estruturais do Dengue
Digestão de insertos e vetor de clonagem
Os produtos da PCR e o vetor de clonagem pRAPs foram submetidos a uma etapa de
digestão, na qual cada fragmento amplificado foi digerido com um par de enzimas de
restrição, de acordo com o sítio de restrição determinado pela seqüência de primers utilizados
na etapa de amplificação (Tabela 4). As enzimas foram utilizadas de acordo com o protocolo
fornecido pela New England Biolabs. Após a digestão, os fragmentos foram submetidos à
eletroforese em gel de agarose 1% (SAMBROOK et al., 1989) para confirmar a eficiência de
corte de cada par de enzimas. Tendo produzido fragmentos de tamanhos esperados e tendo
sido confirmados, foram eluídos do gel, utilizando o “Purelink DNA Gel extraction kit”
(Invitrogen), com a finalidade de eliminar os resíduos da digestão.
Tabela 4: Enzimas de restrição utilizadas para digestão dos fragmentos
Gene 5’fosfato 3’hidroxila
NS1 NotI SacI
NS2A NcoI SacI
NS2B NcoI SacI
NS3 NcoI NotI
NS4A NcoI SacI
NS4B NotI SacI
Ligação dos genes candidatos a atividade supressora em vetor de clonagem
Os fragmentos digeridos foram ligados com T4 DNA ligase (New England Biolabs) ao
vetor de clonagem pRAPs (Figura 10) que havia sido digerido com as mesmas enzimas
utilizadas para os insertos (Tabela 4). Portanto, foram feitas sete ligações distintas do vetor a
cada proteína não estrutural com a finalidade de avaliar a função de cada uma delas
separadamente, quando inoculadas na planta. A reação de ligação foi feita segundo o
protocolo oferecido pelo fabricante e a quantidade de inserto e vetor utilizada foi determinada
utilizando uma fórmula que considera o tamanho do vetor e do inserto, resultando na
quantidade em nano gramas, cuja proporção inserto: vetor é 3:1. Em cada transformação
utilizou-se 100 ng do vetor pRAPs que possui 4107 pb.
Cálculo de proporção inserto: vetor
Quantidade do vetor (100ng) X Tamanho do inserto X 3/1 = Quantidade de inserto (ng)
4107 pb
pRAPs
Figura 10: M a p a g e n ét i c o do v et o r p R A P s. 3 5S - Pr omo t o r d e ca ul if lo wer mo sai c
v i r u s, T n o s - T e r mi n ad o r d e N o p a l i n a si n t a s e d e Ag ro b a c t e ri u m t u m ef a ci en s, A mp -
g e n e d e r e s i s t ê n ci a à a mp i c i l i n a . N co I ,N o t I e S ac I r e p r e s en t a m o s s í t io s d e
r e s t r i ç ão u t i l i z a d o s p a r a c l o n a g e m; A s c I e Pa c I s ã o o s s í t i o s d e r e s t r i ç ão u t i l i z a d o s
Transformação de bactérias E.coli
Tendo sido efetuada a ligação dos genes candidatos ao vetor pRAPs, essas construções
foram utilizadas separadamente para transformação de bactérias. Para a realização deste
procedimento, fez-se o uso de células de E. coli cepa DH5α competentes para eletroporação.
Estas células foram previamente preparadas seguindo o protocolo de Sambrook e Roussel
(2001) e estocadas a -80°C. O plasmídeo pRAPs ligado ao inserto foi desalinizado em
membrana de nitrocelulose 0,025μm (Millipore) por 15 minutos.
O eletroporador Bio-Rad Gene Pulser II foi utilizado nas seguintes condições:
capacitância de 25 μF; resistência de 200Ω; voltagem de 1.80 KV. Foi aliquotado 1 μl da
reação de ligação desalinizada em 50 μl de células DH5 α eletro-competentes e após a
eletroporação realizada na curveta (Gene Pulser Curvette (Bio-Rad), foi adicionado 1 ml de
meio de cultura SOC (Apêndice I), misturado delicadamente e transferido para um microtubo
estéril. O tubo contendo o meio SOC e células competentes foi incubado com agitação de 240
rpm por 1 hora a 37°C.
As bactérias foram plaqueadas em meio LB contendo ampicilina (100μg/mL) e
mantidas vertidas em estufa a 37° por cerca de 12 horas. Este processo foi realizado para cada
uma das construções feitas com as proteínas não-estruturais em vetor pRAPs. Logo, uma
grande quantidade de colônias foi selecionada e submetida a um processo de extração do
DNA plasmidial de acordo com Sambrook & Maniats (1989), para confirmação de suas
seqüências através de seqüenciamento automático (Perkin Elmer ABI-modelo 377).