Stricto Sensu em Educação Física
Tese de Doutorado
Variabilidade Genética na População Brasileira:
ancestralidade
genômica e fenótipos de capacidade cardiovascular.
Brasília - DF
2012
TAILCE KALEY MOURA LEITE
VARIABILIDADE GENÉTICA NA POPULAÇÃO BRASILEIRA: ANCESTRALIDADE GENÔMICA E FENÓTIPOS DE CAPACIDADE
CARDIOVASCULAR.
Tese de Doutorado apresentada ao programa de Doutorado Strictu Sensu em Educação Física da Universidade Católica de Brasília, como requisito para obtenção de título de doutor.
Orientador: Dr. Rinaldo Wellerson Pereira
L533v Leite, Tailce Moura.
Variabilidade genética na população brasileira: ancestralidade genômica e fenótipos de capacidade cardiovascular – 2012.
152f. : il.; 30 cm
Tese (doutorado) – Universidade Católica de Brasília, 2012. Orientação: Rinaldo Wellerson Pereira
1. Genética humana. 2. Miscigenação. 3. Pigmentação. 4. Polimorfismo. I. Pereira, Rinaldo Wellerson, orient. II. Título.
RESUMO
O processo de miscigenação recente entre diferentes populações ancestrais pode levar a obtenção de resultados espúrios em estudos de associação genética. A estratificação pela classificação de cor de pele é uma ferramenta útil para controle de estrutura populacional, mas, para a população brasileira, alguns estudos demonstraram que este método de estratificação por classificação de cor de pele não é uma estratégia funcional, pois há uma dissociação entre a cor de pele autorrelatada e a ancestralidade genômica. No primeiro capítulo desta pesquisa, foram reportados resultados de um estudo em um grupo de irmãs germanas brasileiras, no qual a pigmentação de pele constitutiva foi mensurada utilizando-se um refratômetro, a ancestralidade genômica foi estimada pela genotipagem de 21 marcadores informativos de ancestralidade (AIMs) e a cor de pele autorrelatada foi avaliada de acordo com classificação adotada pelo censo demográfico brasileiro em cada participante. Foi explorada a associação entre cor de pele e pigmentação constitutiva, cor de pele e ancestralidade genômica, e pigmentação constitutiva e ancestralidade genômica, sendo observado que, apesar de haver diferenças significativas da ancestralidade genômica e da pigmentação entre as três classificações de cor de pele, há uma considerável dispersão dentro de cada grupo e uma grande sobreposição destas variáveis entre os grupos. Foi também observado que não houve uma boa concordância para as três categorias de cor de pele nos pares de irmãs e que o nível socioeconômico associou-se significativamente com a cor de pele autorrelatada e com a ancestralidade genômica. Adicionalmente, foi observada forte associação entre o rs1426654 (localizado no gene SLC24A5 e presente no painel de AIMs utilizado) e a pigmentação de pele constitutiva na presente amostra. Sendo assim, o primeiro capítulo mostrou que classificações subjetivas baseadas na cor de pele autorrelatada são inadequadas para descrever a estrutura populacional presente em populações recentemente miscigenadas. O segundo capítulo reporta a investigação do pico de potência aeróbia (VO2pico), mensurado por ergoespirometria em um teste incremental, com 12 SNPs do sistema
renina angiotensina aldosterona (SRAA) em mulheres idosas posmenopausadas, ajustando-se a análise para idade, percentual de gordura e massa livre de gordura, mensuradas por absortometria de raios X de dupla energia. Apesar da não observação de associações estatisticamente significativas após correção para comparações múltiplas pelo índice de falsas descobertas (FDR, do inglês false discovery rate), foram detectadas tendências de associações para os fenótipos cardiovasculares na análise de cada marcador genético individualmente. Também foram observadas tendências de associação na análise de efeitos epistáticos entre combinações genotípicas de 3 SNPs com o VO2pico e com o componente principal calculado a
partir do teste cardiorrespiratório. Em síntese, apesar das dificuldades em explicar a herdabilidade para fenótipos complexos atualmente demostrada pelos estudos de genoma total, o segundo capítulo desta pesquisa assinalou algumas possíveis contribuições genéticas do SRAA para a capacidade cardiovascular.
ABSTRACT
The process of recent admixture between ancestral populations can lead to spurious results in genetic association studies. Stratifying by “skin color” is a useful method to control for population structure, but, in the Brazilian population, several studies have demonstrated that stratifying according to “color” is not a suitable strategy, due to the dissociation between self-reported “skin color” and genomic ancestry. In the first chapter of this research we report the results of a study in a group of Brazilian siblings in which we measured skin pigmentation using a reflectometer, and estimated genomic ancestry using 21 Ancestry Informative Markers (AIMs). Self-reported “color”, according to the Brazilian census, was also available for each participant to explore the relationship between self-reported “color” and skin pigmentation, self-reported “color” and genomic ancestry, and skin pigmentation and genomic ancestry. We observed that, although there were significant differences between the three “color” groups in genomic ancestry and skin pigmentation, there was considerable dispersion within each group and substantial overlap between groups. We also saw that there was no good agreement between the “color” categories reported by each member of the sibling pair and that socioeconomic status was significantly associated with self-reported “color” and genomic ancestry. Also, we observed that rs1426654 (gene SLC24A5 from the AIM panel) was strongly associated with constitutive skin pigmentation in this sample. Hence, this chapter shows that subjective classifications based on self-reported “color” are inadequate to describe the population structure present in recently admixed populations. The second chapter report the investigation of peak oxygen uptake (VO2peak) measured by
ergoespirometric graded test with 12 SNPs markers of the renin angiotensin aldosterone system (RAAS) in postmenopausal elderly, adjusting for age, fat percentage and fat free mass measured by dual-energy X-ray absorptiometry. In despite of no observation of significant associations after correction for multiple comparisons by the false discovery rate (FDR) method, there was detected some borderline associations with the cardiovascular phenotypes in the analysis of each marker individually and some borderline associations of epistatic analysis of three SNPs genotype combinations with a principal component calculated from the cardiorespiratory test and with the VO2peak. In summary, even with the difficulties to explain
heritability for complex phenotypes, demonstrated by genome wide studies, the second chapter highlighted some possible genetic contributors to the cardiovascular fitness in the RAAS.
LISTA DE FIGURAS
Figura 01: Modelos da evolução humana. (a) Origem Africana Recente. (b) Evolução Multirregional. ...13
Figura 02: Rotas de migração do homem moderno. ...14
Figura 03: Diagrama das forças evolutivas moldando a estrutura genética de variabilidade do cromossomo Y na América do Sul. ...15
Figura 04: Modelo evolucionário da arquitetura genética da pigmentação humana em 3 populações: Africana (West Africa), Asiática (East Asian) e Europeia (Europe). ...17
Figura 05: Exemplo de fotografia da quantificação do DNA de alto peso molecular extraído das amostras sanguíneas realizada em gel de agarose. ...28
Figura 06: Análise gráfica de Bland-Altman: concordância das proporções de ancestralidade e do índice de melanina entre os pares de irmãs. ...41
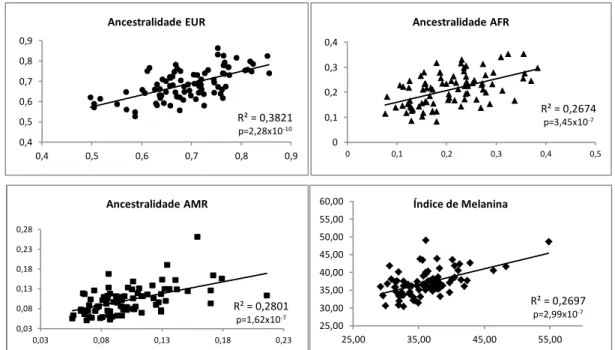
Figura 07: Diagrama de dispersão: ancestralidade genômica e índice de melanina entre os pares de irmãs. ...41
Figura 08: Proporções de Ancestralidade Genômica x Cor de Pele. ...44
Figura 09: Pigmentação (índice de melanina) x Cor de Pele. ...45
Figura 10: Esquema representativo do SRAA, incluindo os SNPs genotipados em cada componente. ...73
Figura 11: Gráfico representativo da variância explicada por cada componente principal (autovalores) - Scree Plot. ...89
LISTA DE TABELAS
Tabela 01: Descrição dos 21 AIMs: posições citogenéticas e físicas; alelos e frequências do alelo 1 em cada população parental; descrição do conteúdo informativo de ancestralidade pelos métodos de diferença de frequência alélica ( ), valor f e escore In entre as três populações parentais arranjadas par
a par...29
Tabela 02: Descrição das sequências e dos tamanhos dos iniciadores de PCR (F e R) e dos iniciadores de extensão de única base (S). ...31
Tabela 03: Concentração dos iniciadores de extensão de única base (S). ...34
Tabela 04: Caracterização amostral. ...38
Tabela 05: Descrição dos AIMs – Frequência Alélica e Teste de Hardy-Weinberg. ...39
Tabela 06: Ancestralidade Genômica ...40
Tabela 07: Tabela de contingência: cor de pele autodenominada. ...42
Tabela 08: Ancestralidade genômica (ANOVA com correção de Bonferroni). ...43
Tabela 09: Índice de Melanina x Cor de pele (ANOVA com correção de Bonferroni). ...45
Tabela 10: Nível socioeconômico x Cor de Pele (Teste de Qui-quadrado). ...46
Tabela 11: Ancestralidade genômica e Índice de Melanina X Nível Socioeconômico (ANOVA com correção de Bonferroni). ...47
Tabela 12: Teste de Associação Alélica (Ancestralidade x Pigmentação). ...48
Tabela 13: Estudos prévios que avaliaram a ancestralidade genômica na população brasileira. ...51
Tabela 14: Estudos prévios que avaliaram a ancestralidade genômica x cor de pele na população brasileira. ...56
Tabela 15: Herdabilidade de fenótipos antropométricos. Fonte: Adaptado de Clark (1956). ...68
Tabela 17: Estudos de herdabilidade para potência aeróbia e composição corporal em populações não
brasileiras. ...70
Tabela 18: Descrição dos 12 locos de acordo com o gene. ...82
Tabela 19: Descrição das sequências e tamanhos dos iniciadores da PCR (F e R) e da extensão de única base (S). ...84
Tabela 20: Concentração dos iniciadores de extensão de única base (S). ...86
Tabela 21: Especificações do teste de associação com os polimorfismos do SRAA...91
Tabela 22: Características amostrais. ...92
Tabela 23: Nível de atividade física e cor de pele autorrelatada (n=148). ...93
Tabela 24: Teste Ergoespirométrico (n=148). ...94
Tabela 25: Frequência alélica e Teste de Hardy-Weinberg. ...95
Tabela 26: Teste ergoespirométrico máximo x SRAA. ...96
Tabela 27: Pré-teste (repouso) e pós-teste (recuperação) x SRAA. ...97
Tabela 28: Teste MB-MDR para associação dos polimorfismos do SRAA (3 SNPs). ...98
SUMÁRIO
Introdução Geral ...11
Capítulo 1 – POPULAÇÃO BRASILEIRA: ANCESTRALIDADE GENÔMICA E SUAS IMPLICAÇÕES FENOTÍPICAS ....12
1 Introdução ...12
2 Revisão de literatura ...13
2.1 Origem, Evolução e Variabilidade Fenotípica em Humanos ...13
2.2 Ancestralidade Genética na População Brasileira ...19
3 Objetivo ...24
3.1 Geral: ...24
3.2 Específicos: ...24
4 Materiais e métodos ...25
4.1 Amostra ...25
4.2 Avaliação da cor de pele ...25
4.3 Pigmentação (Índice de Melanina) ...26
4.4 Classificação Socioeconômica ...26
4.5 Genotipagem ...26
4.5.1 Extração de DNA ...26
4.5.2 Quantificação do DNA ...28
4.5.3 Genotipagem dos Marcadores Informativos de Ancestralidade...29
4.5.3.1 Desenho de iniciadores para PCR e Genotipagem ...30
4.5.3.2 Protocolos das reações de PCR e Genotipagem ...33
4.6 Determinação da ancestralidade genética ...35
4.7 Análise Estatística ...35
5 Resultados ...38
5.1 Caracterização amostral ...38
5.2 Ancestralidade Genômica ...39
5.3 Análise de concordância entre os pares de irmãs ...40
5.4 Ancestralidade Genômica, Cor de Pele e Índice de Melanina...42
5.5 Nível Socioeconômico ...46
5.6 Associação de AIMs com Índice de Melanina (Pigmentação) ...47
6 Discussão ...49
6.1 Ancestralidade Genômica ...49
6.2 Análise de concordância entre os pares de irmãs ...52
6.4 Nível Socioeconômico ...58
6.5 Associação de AIMs com Índice de Melanina (Pigmentação) ...60
7 Conclusão ...61
Capítulo 2 – POLIMORFISMOS DO SISTEMA RENINA ANGIOTENSINA ALDOSTERONA E APTIDÃO CARDIOVASCULAR ...62
1 Introdução ...62
2 Revisão de literatura ...64
2.1 Aptidão cardiorrespiratória e envelhecimento ...64
2.2 Herdabilidade e Fenótipos Relacionados à Potência Aeróbia ...67
2.3 Sistema Renina Angiotensina Aldosterona (SNPs x Potência aeróbia) ...71
3 Objetivos ...75
3.1 Geral ...75
3.2 Específicos ...75
4 Materiais e métodos ...76
4.1 Amostra ...76
4.2 Caracterização amostral ...76
4.2.1 Nível de atividade física das participantes ...76
4.2.2 Composição Corporal ...77
4.2.3 Genotipagem dos Marcadores Informativos de Ancestralidade...78
4.3 Teste de Potência Aeróbia (VO2pico) ...79
4.4 Genotipagem ...81
4.4.1 Extração de DNA ...81
4.4.2 Quantificação do DNA ...81
4.4.3 Genotipagem dos Polimorfismos em Genes do SRAA ...81
4.4.3.1 Desenho de iniciadores para PCR e Genotipagem ...82
4.4.3.2 Protocolos das reações de PCR e Genotipagem ...85
4.5 Análise Estatística ...86
5 Resultados ...92
5.1 Caracterização amostral ...92
5.2 Teste ergoespirométrico ...93
5.3 Potência aeróbia e ancestralidade genômica (19 AIMs)...94
5.4 Marcadores genéticos do SRAA ...94
5.5 Aptidão cardiovascular e marcadores genéticos do SRAA ...96
5.5.1 Efeitos principais ...96
6 Discussão ... 100
6.1 Caracterização amostral ... 100
6.2 Teste ergoespirométrico ... 102
6.3 Potência aeróbia e ancestralidade genômica (19 AIMs)... 103
6.4 Aptidão cardiovascular e marcadores genéticos do SRAA ... 105
6.4.1 Efeitos principais ... 105
6.4.2 Efeitos epistáticos ... 111
7 Conclusão ... 118
Referências Bibliográficas ... 119
ANEXOS ... 130
ANEXOA. QUESTIONÁRIO DE CARACTERIZAÇÃO DA AMOSTRA (ADOLESCENTES) ... 130
ANEXOB. QUESTIONÁRIO DE CARACTERIZAÇÃO DA AMOSTRA (IDOSAS) ... 131
ANEXOC. PARECER DO COMITÊ DE ÉTICA EM PESQUISA ... 132
ANEXOD. PARECER DO COMITÊ DE ÉTICA EM PESQUISA ... 133
ANEXOE. TERMO DE CONSENTIMENTO LIVRE E ESCLARECIDO (ADOLESCENTES) ... 134
ANEXOF. TERMO DE CONSENTIMENTO LIVRE E ESCLARECIDO (IDOSAS) ... 137
ANEXOG. IPAQ(VERSÃO LONGA) ... 140
ANEXOH. CLASSIFICAÇÃO DO IPAQ ... 144
ANEXOI. QUESTIONÁRIO DE CLASSIFICAÇÃO SOCIOECONÔMICA ... 145
INTRODUÇÃO GERAL
O cenário ideal para os estudos em genética humana seria conhecer em nível molecular cada indivíduo, bem como quais os determinantes genéticos de cada fenótipo, incluindo no rol dos fenótipos variadas doenças e possíveis respostas a tratamentos medicinais. Entretanto esta abordagem individual é um quadro utópico e inviável para fenótipos complexos, os quais dependem de vários genes em conjunto, sendo escassa a associação genética acurada a cada trato fenotípico. Sendo assim, busca-se caracterizar e agrupar indivíduos semelhantes, e então desenvolver abordagens específicas para cada um destes grupos, sendo que a classificação de acordo com a etnia é uma das metodologias utilizadas.
Entretanto, existem populações, como é o caso da população brasileira, que sofreram recentemente um processo de miscigenação, sendo atualmente compostas por uma nova formação genética, decorrente da mistura dos genes herdados das populações ancestrais. Neste contexto, o presente estudo buscou, abordar a miscigenação genética da população brasileira e os efeitos da miscigenação em fenótipos complexos relacionados à aptidão física, mais especificamente na potência aeróbia (VO2max), tanto utilizando-a como covariante de
ajuste para controle de efeitos de estratificação populacional quanto como fator direto de associação com o fenótipo.
Sendo assim, o presente documento apresenta-se dividido em 2 capítulos, cada um abordando uma destas fases do estudo. O primeiro capítulo trata da ancestralidade genômica na população brasileira, associada às variáveis cor de pele autorrelatada, índice de melanina e nível socioeconômico. Já o segundo capítulo traz um estudo que utilizou os conhecimentos da ancestralidade genética empregando esta variável como covariante para o teste de associação entre 12 polimorfismos do sistema renina angiotensina com o fenótipo de aptidão cardiovascular. Além disso, o segundo capítulo aborda uma possível associação direta entre o fenótipo de aptidão cardiorrespiratória e a ancestralidade genômica determinada pelos marcadores informativos de ancestralidade, com exploração das proporções ancestrais de cada população parental no grupo amostral de brasileiras idosas, explorando possíveis relações entre estas duas variáveis.
CAPÍTULO 1 – POPULAÇÃO BRASILEIRA: ANCESTRALIDADE
GENÔMICA E SUAS IMPLICAÇÕES FENOTÍPICAS
1 INTRODUÇÃO
A população brasileira descende de ancestrais africanos, europeus e ameríndios, e, com respaldo da genética molecular e da genética de populações, sabe-se que a miscigenação existente é maior do que inicialmente se supunha com base apenas em critérios de natureza morfológica (ALVES-SILVA et al., 2000; PENA, 2000; CARVALHO-SILVA et al., 2001; CALLEGARI-JACQUES et al., 2003; PENA e BORTOLINI, 2004; LINS et al., 2010).
Estudos de associação genética são estratégias em potencial para identificação de genes de efeito modesto em fenótipos complexos, tais como ocorrem em algumas doenças (MORTON e COLLINS, 1998). Entretanto, o desenvolvimento deste tipo de estudo em populações recentemente miscigenadas, como a brasileira, merece atenção especial, pois a miscigenação pode levar à obtenção de resultados espúrios quando há um fenótipo com incidência diferenciada entre os subgrupos populacionais amostrados, correlacionado com diferença na frequência alélica entre estes grupos (KNOWLER et al., 1988; ROSENBERG e NORDBORG, 2006).
Portanto, identificar se os fenótipos estudados realmente apresentam risco relativo desigual em indivíduos com diferentes ancestralidades genéticas é um aspecto importante, seja para prevenção de resultados espúrios em estudos de associação genética (PRITCHARD e ROSENBERG, 1999; PRITCHARD et al., 2000) ou para utilização de mapeamento por desequilíbrio de ligação gerado por miscigenação (STEPHENS et al., 1994; MCKEIGUE, 1998). Sendo assim, a presente investigação se justifica ao buscar estas informações, ao determinar, descrever e comparar a ancestralidade genética em um grupo de irmãs brasileiras, bem como pela apresentação de fenótipos possivelmente relacionados à composição genética parental. Além disso, o delineamento se justifica pela busca de implicações metodológicas a serem seguidas no desenvolvimento e na aplicação de estudos de associação genética na população brasileira, as quais poderão ser desenvolvidas com diversos fenótipos complexos, seja por estudos de associação utilizando genes candidatos, seja por pesquisas de associação por desequilíbrio de ligação gerado pela miscigenação.
2 REVISÃO DE LITERATURA
2.1 Origem, Evolução e Variabilidade Fenotípica em Humanos
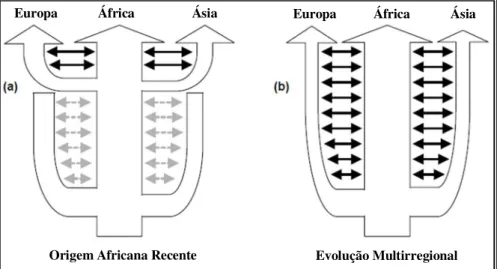
Existem duas teorias predominantes que explicam a origem do homem moderno (Homo sapiens sapiens): a teoria de uma única saída da África (Out of Africa), também chamada de Origem Africana Recente, e a teoria de origem multirregional. De acordo com a teoria Out of Africa, o homem moderno surgiu a partir de uma pequena população africana e então povoou o mundo (STRINGER e ANDREWS, 1988; HORAI et al., 1995). Já a teoria multirregional postula que o homem moderno surgiu gradual e simultaneamente em diferentes continentes: Europa, África e Ásia (THORNE e WOLPOFF, 1992). Estes dois modelos evolutivos estão representados abaixo na Figura 01.
Figura 01: Modelos da evolução humana. (a) Origem Africana Recente. (b) Evolução Multirregional.
FONTE: Adaptado de Excoffier (2002).
Estas teorias foram formuladas baseando-se em estudos em diferentes áreas do conhecimento, com grandes contribuições provenientes dos estudos antropológicos. No entanto, a biologia molecular atualmente possui grande destaque no estudo da evolução do homem moderno, buscando reconstituir a história da evolução a partir do estudo do DNA. Dados moleculares baseados nas análises de DNA mitocondrial, Y-não recombinante e genes autossômicos, suportam, em sua maioria, a teoria Out of Africa, tendo como argumento principal a maior diversidade genética encontrada em africanos quando estes são comparados com não africanos (HORAI et al., 1995; ARMOUR et al., 1996; SANTOS-LOPES et al., 2007; LI et al., 2008). Portanto, acredita-se que o homem seja descendente de um grupo de africanos, que gradualmente se disseminou, há cerca de 100.000 anos, para o continente Europeu e o continente Asiático e que a povoação da Oceania e da América ocorreu
Europa África Ásia Europa África Ásia
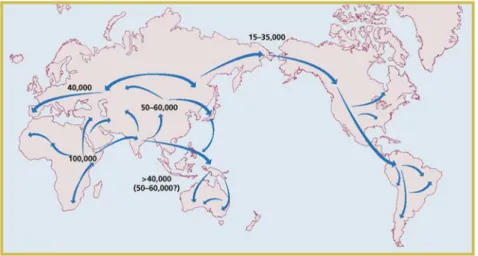
posteriormente, há aproximadamente 60.000 e 15.000 anos, respectivamente (CAVALLI-SFORZA e FELDMAN, 2003). A Figura 02 traz um esquema das rotas de migração do homem moderno, saindo da África em direção à Ásia e posteriormente para Europa, Oceania e América, incluindo o tempo aproximado em anos para cada movimentação.
Figura 02: Rotas de migração do homem moderno. FONTE: Cavalli-Sforza e Feldman (2003).
Alguns estudos trazem ainda evidências de um processo arcaico de miscigenação entre o homem moderno (Homo sapiens sapiens) em migração da África e outros hominídeos já existentes fora da África, os Neandertais (STONEKING e KRAUSE, 2011; YOTOVA et al., 2011), sendo a contribuição do homem Neandertal para o homem moderno não africano estimada em 2 a 4% da composição do genoma (STONEKING e KRAUSE, 2011) e 9% no cromossomo X (YOTOVA et al., 2011). Estes autores sugerem que o processo teria ocorrido entre 50.000 a 80.000 anos atrás, momento da saída do homem moderno da África coincidindo com a presença dos homens de Neandertal no Oriente Médio, antes de se dispersarem para outras regiões. Também foi observada miscigenação subsequente do homem moderno com os Denisovans na população habitante de Nova Guiné – Oceania (STONEKING e KRAUSE, 2011).
O povoamento das Américas continua em discussão. Uma das teorias mais aceitas é de que o continente americano foi primeiramente habitado por migração de três populações distintas há cerca de 15.000 anos, provenientes da Sibéria (Nordeste Asiático), teoria chamada
de “three-wave” (GREENBERG et al., 1986). Entretanto, Bonatto e Salzano (1997)
teve um papel importante na origem dos povos nativo americanos. Segundo estes autores, os habitantes da Beringia entraram nas Américas ao sul da América do Norte passando pelo corredor livre de gelo de Alberta, o qual foi posteriormente bloqueado isolando a Beringia do resto do continente americano, fato que desencadeou o desenvolvimento da identidade genética dos ameríndios, os quais comparados às populações que permaneceram em Beringia (Na-Dene e Eskimo) apresentam maior diversidade de mtDNA (BONATTO e SALZANO, 1997).
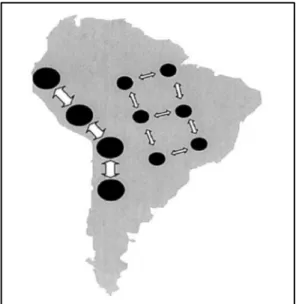
Em relação à população da América do Sul, Tarazona-Santos et al. (2001) propuseram um modelo evolutivo baseado em estudos de linhagens masculinas (cromossomo Y), no qual as populações do oeste sul-americano (área dos Andes) apresentam tamanhos efetivos e taxas de fluxo gênico maiores quando comparadas às populações do leste (região amazônica e Brasil), resultando em uma maior diferenciação genética entre estas últimas. A Figura 03 representa as forças evolutivas na América do Sul. Nesta figura, o tamanho dos círculos representa proporcionalmente de forma aproximada o tamanho das populações e o tamanho das flechas, a quantidade de troca genética.
Figura 03: Diagrama das forças evolutivas moldando a estrutura genética de variabilidade do cromossomo Y na América do Sul.
FONTE: Tarazona-Santos et al. (2001).
antigos, que antecedem a divergência das populações humanas (TISHKOFF e VERRELLI, 2003). Por exemplo, populações africanas apresentam maior prevalência de certas doenças complexas tais como hipertensão, diabetes e câncer de próstata (TISHKOFF e WILLIAMS, 2002). Sendo a população africana o berço genético do homem moderno, estas variações genéticas em africanos podem trazer implicações e tornarem-se fatores de risco para variados grupos étnicos da atualidade.
Outro fator importante é que uma mesma variante genética pode trazer uma diferente resposta fenotípica em dois ambientes diversos. Laland e colaboradores (2010) apresentaram uma revisão recente na qual abordam como aos hábitos culturais podem, juntamente com a genética, influenciar a evolução humana. Um grande exemplo de ação dos mecanismos de seleção natural é o descrito para o loco da lactase (LCT) em populações europeias, cujas variantes influenciam na produção de lactase na idade adulta, determinando a capacidade de digestão do leite (BURGER et al., 2007). Segundo Itan et al. (2009), a persistência da lactase sofreu processo de seleção natural positiva há cerca de 7.500 anos em decorrência da mudança de hábitos culturais. Em virtude do desenvolvimento do pastoreio e da disponibilidade de leite para alimentação, o alelo responsável pela persistência da lactase na vida adulta passou de baixa para alta frequência dentro da população da Europa central e do norte da África.
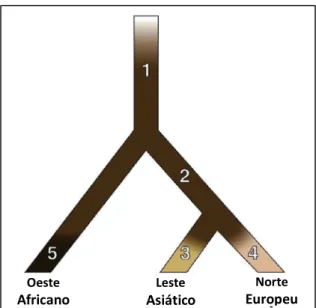
Figura 04: Modelo evolucionário da arquitetura genética da pigmentação humana em 3 populações:
Africana (West Africa), Asiática (East Asian) e Europeia
(Europe).
FONTE: Adaptado de McEvoy et al. (2006).
Este modelo simula uma árvore populacional, com a pigmentação de pele representada pela cor em cada segmento, que parte de um ancestral hominídeo comum, provavelmente de pele clara em decorrência da camada de pelo que cobria todo o corpo, substituído pelos indivíduos de pele escura devido à forte seleção natural da pigmentação de pele escura em decorrência da perda da pelagem, paralelamente ao surgimento do Homo sapiens sapiens, representado na Figura 04 pelo segmento 1. O modelo apresentado por estes autores (segmentos 3 e 4, Figura 04) propõe ainda que a diferenciação da cor de pele entre asiáticos e europeus provavelmente resultou de mecanismos genéticos diferentes e, portanto, não evoluiu antes da separação destas duas populações, identificando inclusive alguns genes como fortes indicativos da evolução da pigmentação de pele na população asiática (segmento 3) e na população europeia (segmento 4): genes ADAM17 e ADAMTS20 e genes SLC24A5 e MAPT, respectivamente (MCEVOY et al., 2006).
O grande ponto a ser ressaltado é que adaptações evolutivas que no passado foram benéficas ao homem, hoje podem representar fatores de risco em um ambiente modificado. Por exemplo, com as mudanças recentes nos hábitos de ingestão alimentar, grupos populacionais que passavam por períodos de restrição alimentar e sofreram seleção natural para maximização da eficiência metabólica, atualmente apresentam alelos comuns relacionados a doenças metabólicas, tal como diabetes (NEEL, 1999; BALARESQUE et al., 2007). Sendo assim, variantes genéticas presentes em populações ancestrais podem, ainda hoje, expressar fenótipos positivos ou negativos para o homem. Foi demonstrado em africanos
Oeste Africano
Leste Asiático
que mutações no gene da glicose-6-fostato-desidrogenase (G6PD), que resultam em deficiência enzimática e protegem indivíduos contra infecção pelo parasita da malária, causam também hematopatologias e, ainda assim, permanecem em frequências relativamente altas na população africana sub-Saara (TISHKOFF et al., 2001). Sendo assim, aos portadores desta mutação que vivem em locais onde a malária foi erradicada, resta apenas o ônus da maior probabilidade de apresentar doenças sanguíneas.
Colilla et al. (2000) desenvolveram uma comparação entre o índice de massa corporal (IMC) de africanos e afro-americanos, basicamente provenientes de ancestrais africanos, e verificaram que os primeiros possuem valores significativamente menores que os últimos. A explicação proposta é de que africanos passaram por longos períodos de escassez de alimento e carregam então “genes conservadores de energia”, compartilhados pelos afro-americanos, e que a obesidade predominante nos afro-americanos é uma consequência natural da união deste aparato genético com o estilo alimentar ocidental. Esta predisposição genética também explica porque americanos ou descendentes de europeus têm menor prevalência de obesidade que africanos e afro-americanos (COLILLA et al., 2000).
As principais forças evolutivas que determinam o destino de uma mutação inserida na bagagem genética de uma população são a deriva, que é um processo aleatório atuando no destino de alelos em uma população, e a seleção natural, que representa a seleção das mutações adaptativas e eliminação dos alelos desfavoráveis (WRIGHT, 1931). Estas forças atuam em conjunto no decorrer dos anos, modificando o conjunto de alelos em uma população e alterando as características das espécies ao longo do tempo. Entretanto, determinar como a variabilidade fenotípica em humanos é determinada a partir das estruturas genéticas não é tarefa fácil, pois este é um cenário extremamente complexo influenciado por vários fatores, por vezes entendidos como várias dimensões. Neste sentido, Hindorff et al. (2011) propuseram 3 dimensões para tentar explicar a variabilidade fenotípica: a própria heterogeneidade fenotípica entre indivíduos e entre populações, a heterogeneidade alélica (associação de uma característica fenotípica com múltiplas variantes dentro do mesmo gene ou loco) e a pleitropia (variantes genéticas associadas com múltiplos fenótipos diferentes, incluindo fenótipos aparentemente nada relacionados). Além disso, juntamente com a heterogeneidade alélica dentro do mesmo gene supracitada, figura neste cenário o efeito das interações gene-gene, conceituado por Moore et al. (2009) como epistasia.
hipótese doença comum variante comum (CD/CV, do inglês common disease/ common
variant) a qual postula que algumas poucas variantes comuns de alelos (frequência que >5% na população) determinam a variabilidade genética de risco para o fenótipo ou doença; e a hipótese doença comum variante raro (CD/RV, do inglês common disease / rare variant) assevera que variações raras na sequência de DNA são as principais causadoras de doenças comuns encontradas na população (SCHORK et al., 2009).
Mas, além de focar em variantes raros ou comuns, os estudos têm incluído, juntamente com a sequência herdada de DNA, variações genéticas estruturais, variações epigenéticas e interações entre genes e meio ambiente na tentativa de entender a presente estrutura fenotípica moldada ao longo do processo de evolução, tal como nos estudos de câncer revisados por Hindorff et al (2011).
2.2 Ancestralidade Genética na População Brasileira
Há aproximadamente 500 anos, em 1492, a América começou a ser colonizada pelos europeus, o que trouxe um novo cenário à composição genética do continente e, mais especificamente, do Brasil. Historicamente, sabe-se que a atual população brasileira é uma mistura de três populações parentais: europeus, ameríndios e africanos; e sabe-se também que este perfil é decorrente do processo de colonização do país. Estudos que investigaram a linhagem dos indivíduos brasileiros por meio de polimorfismos em mtDNA e cromossomo Y corroboram a história de união predominante entre homens europeus, principalmente portugueses, com mulheres ameríndias ou africanas (ALVES-SILVA et al., 2000; CARVALHO-SILVA et al., 2001). Callegari-Jacques e colaboradores (2003) genotiparam 12 microssatélites em uma amostra de 1.037 brasileiros provenientes de diferentes regiões do Brasil e mais uma vez ficou evidente a contribuição das populações parentais europeia, africana e ameríndia na formação da população brasileira, com predominância de europeus.
Portanto, a identidade genética das três populações ancestrais brasileiras, modelada ao longo de milhares de anos de evolução, vem sendo modificada há cerca de 500 anos em decorrência do novo fluxo gênico gerado pela mistura de ameríndios, europeus e africanos no Brasil.
entre grupos populacionais (LI, 1969; KNOWLER et al., 1988; DEVLIN e ROEDER, 1999; DEVLIN et al., 2004; ROSENBERG e NORDBORG, 2006; ZHENG et al., 2006). Portanto, se o fenótipo está correlacionado à ancestralidade e nos grupos de estudo existe uma maior representação de indivíduos com determinada ancestralidade, os alelos mais frequentes neste grupo populacional podem apresentar-se equivocadamente associados ao fenótipo sob investigação.
Para pesquisas desta natureza junto à população brasileira, mesmo agrupando-se os indivíduos por cor de pele ou características físicas que possam inferir a origem africana ou europeia, o risco dos grupos apresentarem heterogeneidade genética é alto (PARRA et al., 2003; SUAREZ-KURTZ et al., 2007; SANTOS et al., 2009), fazendo-se necessária a utilização de variáveis contínuas tal como a ancestralidade estimada a partir de dados genéticos (SORTICA et al., 2011). Pimenta et al. (2006) analisaram 12 microssatélites de uso comum na genética forense em 752 indivíduos da cidade de São Paulo e também verificaram dissociação entre a cor de pele dos 3 principais grupos raciais brasileiros (brancos, pretos e intermediários) e a ancestralidade genética.
Apesar deste aspecto negativo, populações geradas pela miscigenação recente representam uma possibilidade para a identificação de genes responsáveis pela expressão de determinados fenótipos, quando estes genes se manifestam de maneira diferenciada nos grupos populacionais ancestrais contribuintes para a formação destas populações (STEPHENS et al., 1994; MCKEIGUE, 1998). Esta metodologia é conhecida como mapeamento por MALD (do inglês, Mapping by Admixture Linkage Disequilibrium), ou seja, desequilíbrio de ligação gerado por miscigenação. As bases do MALD estão no desequilíbrio de ligação gerado pela miscigenação entre indivíduos de diferentes grupos populacionais e na possibilidade de identificar regiões genômicas que tenham sua origem em um determinado grupo populacional. Sendo assim, em um grupo de indivíduos miscigenados com determinado fenótipo, tem-se um maior compartilhamento de regiões genômicas com a mesma origem ancestral. Este desequilíbrio de ligação é particularmente alto entre marcadores polimórficos que apresentam alelos com grandes diferenças de frequência entre as populações parentais (HALDER e SHRIVER, 2003).
marcador molecular pode ser considerado um AIM, contanto que satisfaça a condição de diferenças de frequência alélica entre populações.
Campbell et al. (2005) avaliaram a estratificação dentro de uma população de norte-americanos descendentes de europeus, e, após a genotipagem de 67 AIMs específicos para diferenciação de regiões genéticas de ancestralidade descendente de africanos e europeus, não encontraram evidências de estratificação. No entanto, quando foi avaliado o polimorfismo LCT (rs4988235), cuja frequência alélica varia entre o norte e o sul europeu, foi possível identificar a estratificação, inclusive com este marcador fugindo ao equilíbrio de Hardy-Weinberg. Portanto, a qualidade dos AIMs, em termos de diferença de frequência alélica entre as populações a serem discriminadas, é fator indispensável na análise da ancestralidade genética, sendo inclusive preponderante sobre quantidade de marcadores.
Basicamente, a diferença de frequência alélica (δ) entre as populações ancestrais
indicada para um AIM de grande conteúdo de informação e correta atribuição é tal que δ ≥
0,60 (HOGGART et al., 2003; ROSENBERG et al., 2003). As diferenças na acurácia de predição da ancestralidade em função do tipo de marcador (SNPs em íntrons, éxons, regiões regulatórias ou codificantes de RNA mensageiro) são extremamente pequenas e SNPs que apresentam diferença de frequência entre os grupos são os mais eficazes (ALLOCCO et al., 2007).
Mas, além da diferença de frequência alélica, há na literatura formas mais complexas e acuradas de inferir o poder de discriminação ancestral de um painel de AIMs. McKeigue (1998) estabeleceu que o poder de um painel de AIMs depende de quão distante o processo de miscigenação está do presente (número de gerações) e do conteúdo informativo ancestral de cada marcador (f), o qual é derivado em função das frequências alélicas do marcador nas duas populações ancestrais. O cálculo do f para marcadores bialélicos está descrito na Equação 01.
= ( − )4 (1 − ) , = 12 ( + )
ux = frequência do alelo 1 na população X
uy = frequência do alelo 1 na população Y
(Equação 01)
10 ou menos gerações atrás. É importante ressaltar que quanto mais distante no tempo está o processo de miscigenação, maior o número de marcadores necessários para capturar a estrutura dentro da população (TIAN et al., 2007) e, consequentemente, quanto mais informativos os marcadores, melhor a estimativa.
Há ainda o método proposto por Rosenberg et al. (2003) para estimar o conteúdo informativo de cada AIM, denominado de informativeness (In). Este cálculo, assim como descrito de forma simplificada pela Equação 02, abaixo, é resultado de funções logarítmicas das probabilidades de cada alelo estar presente em cada uma das populações. O cálculo do somatório da equação abaixo para cada alelo resulta na In total para cada marcador.
= − og + og
In = conteúdo informativo;
pj= média da frequência do alelo entre as populações;
p i j = frequência do alelo na população i;
K = número de populações; N= número de alelos; i = populações (1, 2, ...., K); j = alelos (1, 2, ...N).
(Equação 02)
Estes métodos de avaliação dos painéis de AIMs são importantes, pois, de acordo com Rosenberg et al. (2003) marcadores altamente informativos reduzem a demanda de genotipagem, em relação ao número de marcadores, necessária para inferência da ancestralidade.
Uma das formas de utilização de marcadores informativos de ancestralidade é pela genotipagem de polimorfismos de base única (SNPs, do inglês single nucleotide
Cavalcante et al. (2011) apresentaram um estudo com 222 indivíduos da população brasileira no qual houve uma contribuição média especialmente alta de ancestralidade africana (45%), seguida da ancestralidade europeia (33%) e da ameríndia (27%), esta última também maior que o usual. As possíveis causas desta disparidade não podem ser afirmadas, mas especula-se que a amostra composta por indivíduos portadores da doença Hepatite C pode não ser representativa da população brasileira. Outra suposição é de que a ancestralidade estimada a partir de um painel com apenas 7 AIMs pode não ser suficiente para determinar as proporções de ancestralidade genética, ainda que em nível de média amostral.
Recentemente foi publicada uma pesquisa com genotipagem em larga escala de 365.116 SNPs, dos quais pela análise de componentes principais (PCA) foram determinados os 5 principais fatores explicativos da ancestralidade e escolhidos os top SNPs de cada um destes componentes para formar painéis de AIMs (50, 100 e 150 SNPs para cada componente, resultando em painéis com 250, 500 e 750), utilizando ainda dois modelos com populações ancestrais de referência diferentes, sendo o modelo 1 composto por europeus da região norte e oeste (CEU), africanos Youruba da Nigéria (YRI) e Mexicanos (MEX) e o modelo 2 composto por europeus da Itália (TSI), afro-americanos do Sul dos Estados Unidos (ASW) e mexicanos (MEX), além da população brasileira a ser testada (GIOLO et al., 2012). Este estudo mostrou que a ancestralidade da população brasileira (Região Sudeste) é formada por um contínuo entre Europeus e Africanos, com pequeno componente Mexicano. Não foram observadas diferenças significativas entre os modelos 1 e 2, sendo as proporções de ancestralidade 15% ameríndia, 24% africana e 61% europeia (modelo 1) e 17% ameríndia, 27% africana e 56% europeia (modelo 2).
3 OBJETIVO
3.1 Geral:
O presente estudo objetivou investigar a ancestralidade genética em uma amostra de pares de irmãs da população brasileira.
3.2 Específicos:
• Determinar a ancestralidade genética em uma amostra de pares de irmãs
brasileiras utilizando marcadores informativos de ancestralidade (AIMs).
• Verificar a associação entre a pigmentação de pele mensurada quantitativamente e
a ancestralidade genética.
• Verificar a associação entre a cor de pele autorrelatada e a ancestralidade genética. • Verificar a associação entre a cor de pele autorrelatada e a pigmentação de pele
mensurada quantitativamente.
• Verificar a associação entre a classificação socioeconômica e a ancestralidade
genética.
• Verificar a associação entre a classificação socioeconômica e a cor de pele
autorrelatada.
• Verificar a associação entre a classificação socioeconômica e a pigmentação de
pele mensurada quantitativamente.
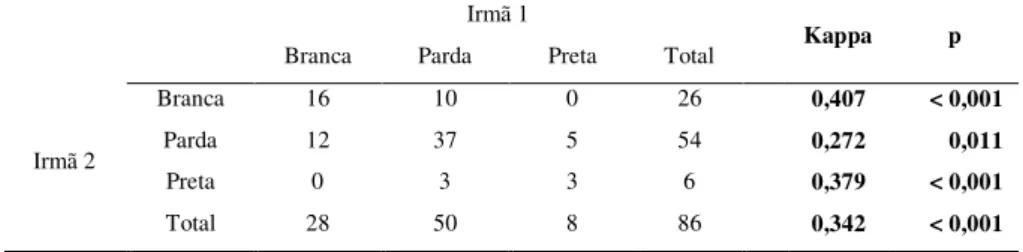
• Comparar a concordância da ancestralidade genética entre os pares de irmãs.
• Comparar a concordância da cor de pele autorrelatada entre os pares de irmãs. • Comparar a concordância da pigmentação de pele mensurada quantitativamente
entre os pares de irmãs.
• Testar a associação entre os marcadores genéticos de ancestralidade (AIMs) e a
4 MATERIAIS E MÉTODOS
4.1 Amostra
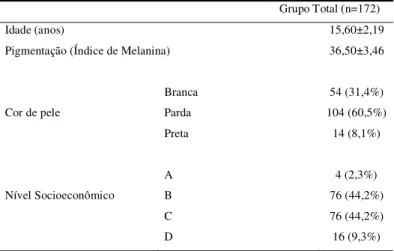
O estudo foi constituído por 86 pares (n=172) de irmãs germanas adolescentes pós-menarca com idade entre 10 e 20 anos. As amostras foram selecionadas em escolas do ensino fundamental e médio da rede pública de Brasília.
Foram adotados os critérios de inclusão seguintes:
a) Irmãs germanas (ou seja, filhas de mesmo pai e mesma mãe) com faixa etária entre 10 e 20 anos, em estágio pós-menarca;
b) Não possuir doença crônico-degenerativa.
O grau de parentesco de irmãs germanas foi previamente confirmado na amostra pela genotipagem de 17 marcadores microssatélites (FONSECA, 2011), utilizando o pacote estatístico Merlin (ABECASIS et al., 2002). Foi aplicado um questionário de caracterização amostral (Anexo A) em todas as voluntárias para discriminação daquelas que seriam mantidas nas análises, partindo de um número inicial de 161 pares de irmãs e finalizando com 86 pares. O grupo amostral de mulheres adolescentes foi obtido mediante parceria com os pesquisadores Dr. Rômulo Maia Carlos Fonseca e Dra. Nancí Maria de França, e sob aprovação pelo Comitê de Ética em Pesquisa da Universidade Católica de Brasília (CEP/UCB Nº 078/2006), de acordo com a Resolução 196/96 (Anexo C). Os dados obtidos no presente estudo, ao serem publicados e divulgados em revistas científicas, têm resguardada a identidade de cada participante.
Adicionalmente, todas as participantes da amostra (ou os responsáveis legais em caso de menores de 18 anos) receberam um Termo de Consentimento Livre e Esclarecido (Anexo E), pelo qual foram explicadas dos objetivos, dos procedimentos a serem realizados, dos possíveis desconfortos, riscos e benefícios decorrentes da participação na pesquisa. A assinatura do termo firmou a concordância em participar do estudo, com a garantia da manutenção do anonimato dos dados coletados e a possibilidade de desistência em participar da pesquisa a qualquer momento.
4.2 Avaliação da cor de pele
branca, preta, parda, amarela ou indígena (IBGE, 2000). A autodenominação da cor de pele de cada voluntária foi avaliada sem interferência de terceiros e sem conhecimento do resultado assinalado pela respectiva irmã.
4.3 Pigmentação (Índice de Melanina)
O índice de melanina é uma medida quantitativa da pigmentação da pele mensurada por um refratômetro. Este aparelho estima a quantidade de melanina a partir da quantidade de luz refletida de volta à máquina nos comprimentos de onda verde e vermelho, sendo que indivíduos com pele mais clara têm valores mais baixos de índice de melanina e indivíduos mais escuros têm valores mais altos, variando geralmente entre 20 até 100 (SHRIVER e PARRA, 2000). A pigmentação em áreas da pele que não ficam expostas à luz é denominada de pigmentação de pele constitutiva, fenótipo complexo e relativamente pouco afetado por fatores ambientais (PARRA et al., 2004b).
No presente estudo, a pigmentação de pele constitutiva foi avaliada utilizando o aparelho DermaSpectrometer (Cortex Technology, Hadsund, Denmark) na parte interna do braço, logo abaixo da axila. O aparelho foi calibrado de acordo com instruções do fabricante e foram realizadas 3 medidas em cada braço, sendo a média destes valores tomada como escore final da pigmentação de pele.
4.4 Classificação Socioeconômica
A classificação socioeconômica das participantes foi avaliada por meio de questionário Critério de Classificação Econômica Brasil (CCEB), elaborado pela Associação Brasileira de Empresas de Estatística (ABEP, 2008), disponível em www.abep.org, o qual é baseado na avaliação da posse de itens domésticos e cômodos da casa, bem como no grau de instrução educacional do chefe da família (Anexo I). A quantidade de itens corresponde a uma pontuação e o somatório dos pontos possibilita a classificação simplificada nas categorias: A, B, C, D e E (em ordem decrescente de renda e nível socioeconômico).
4.5 Genotipagem
4.5.1 Extração de DNA
armazenado em tubos estéreis com vácuo contendo anticoagulante EDTA. O DNA genômico de alto peso molecular foi extraído dos leucócitos periféricos utilizando-se o método salting
out (MILLER et al., 1988).
Os procedimentos de extração envolveram basicamente três passos:
a) Remoção de hemácias por meio da adição de água deionizada ultrapura. Após a homogeneização do sangue, um volume inicial de 300 µl foi depositado em um microtubo de 1,5 mL. Adicionou-se 1000 µl de água deionizada ultrapura, sendo o material centrifugado durante 5 minutos a 5000 rotações por minuto (rpm) para separação dos núcleos e sobrenadante. O sobrenadante foi descartado. Então, foi adicionado em cada microtubo mais 300 µl de sangue e 1000 µl de água deionizada ultrapura e repetido o processo.
b) Lise das células da linhagem branca por meio da adição de um tampão com detergente. A solução usada no procedimento foi denominada Tampão A, composto por sacarose (0,32 M), Tris-HCl (10 mM, pH 7,6), MgCl2 (5 mM) e o detergente não iônico
Triton X 100 (1 %). Suspendeu-se o pellet em 700 µl do Tampão A, sendo o material centrifugado a 5000 rpm por 15 minutos para condensação do pellet, com posterior descarte do sobrenadante.
c) Remoção das proteínas celulares e histonas ligadas ao DNA por meio da adição de uma protease e precipitação com sódio. O pellet foi suspenso em 500µl de um composto denominado Tampão B (25mM de EDTA com pH 8.0 e 75mM de NaCl), sendo adicionados 5µl de SDS (do inglês, sodium dodecyl sulfate) a 10% e 5µl proteinase K (na concentração de 10 mg/mL). Em seguida, os microtubos foram incubados a 55ºC durante uma hora para ação da enzima. Após a incubação foram adicionados 200µl NaCl (em concentração de 6 M) para precipitar restos celulares e proteicos, seguido de uma nova centrifugação da mistura a 3000 rpm durante 15 minutos, com a finalidade de precipitar as impurezas no fundo dos tubos. O
pellet, juntamente com o microtubo usado, foi descartado após a transferência do sobrenadante para um novo microtubo devidamente identificado.
completa do etanol, foram adicionados 100 µl de tampão TE (Tris-HCl 10mM e EDTA 0,1mM, pH 8,0) para conservação do DNA, e o material foi encubado durante 1 hora a 37ºC.
4.5.2 Quantificação do DNA
A concentração do DNA extraído previamente foi estimada por meio de eletroforese em gel de agarose a 1%, corado com 3 µl de brometo de etídio. Em cada poço do gel foi aplicado um volume de 7 µl, dos quais eram 4 µl de tampão de carregamento (2,5 mg/mL de azul de bromofenol; 400 mg/mL de sacarose; 0,02 mg/mL de brometo de etídio; 12,1mg/mL de Tris-HCl pH 8,0; 1 mM de EDTA pH 8,0) e 3 µl do DNA extraído e conservado em tampão Tris-EDTA (TE).
Foram utilizados como padrões para quantificação amostras de DNA do fago lambda em concentrações de 20, 50, 100 e 200ng/µl. Após aproximadamente 15 minutos de eletroforese a 80 V, o gel foi fotografado em luz ultravioleta, sendo as “bandas” formadas pelo DNA comparadas com aquelas dos padrões, e, através da inspeção visual, foi realizada a quantificação do DNA (Figura 05).
Figura 05: Exemplo de fotografia da quantificação do DNA de alto peso
molecular extraído das amostras
sanguíneas realizada em gel de agarose.
4.5.3 Genotipagem dos Marcadores Informativos de Ancestralidade
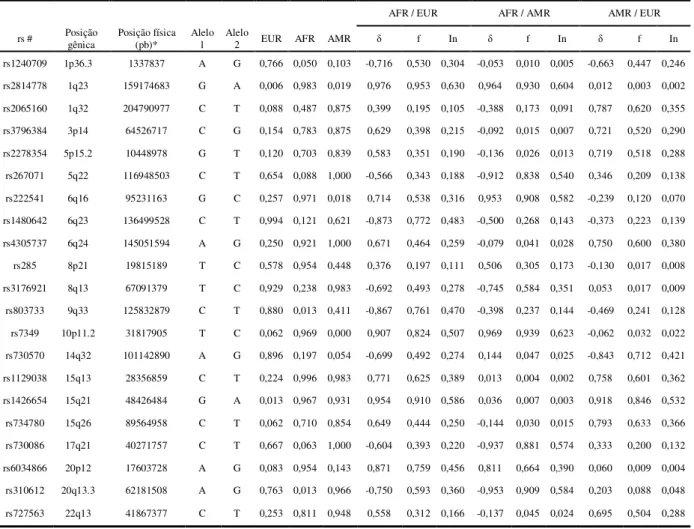
Foram selecionados na literatura marcadores informativos de ancestralidade a partir das diferenças em frequências alélicas ( ) das populações parentais europeia, africana e ameríndia (SHRIVER et al., 2003; SMITH, 2004), já utilizados em publicações prévias (LINS et al., 2010). O conjunto de marcadores foi composto por 21 SNPs informativos de ancestralidade, os quais foram divididos em 3 sistemas, para a genotipagem utilizando o método de minissequenciamento. A Tabela 01 descreve as frequências alélicas para cada um destes 21 locos nas três populações parentais, bem como estimativas do poder informativo de ancestralidade para cada marcador, obtidos pelo valor f e pela In, além da . Alguns destes marcadores são conhecidos por nomes específicos, referindo-se ao loco ou ao polimorfismo em questão.
Tabela 01: Descrição dos 21 AIMs: posições citogenéticas e físicas; alelos e frequências do alelo 1 em cada população parental; descrição do conteúdo informativo de ancestralidade pelos métodos de diferença de frequência alélica ( ), valor f e escore In entre as três populações parentais arranjadas par a par.
AFR / EUR AFR / AMR AMR / EUR
rs # Posição gênica Posição física (pb)* Alelo 1 Alelo 2 EUR AFR AMR f In f In f In
rs1240709 1p36.3 1337837 A G 0,766 0,050 0,103 -0,716 0,530 0,304 -0,053 0,010 0,005 -0,663 0,447 0,246
rs2814778 1q23 159174683 G A 0,006 0,983 0,019 0,976 0,953 0,630 0,964 0,930 0,604 0,012 0,003 0,002
rs2065160 1q32 204790977 C T 0,088 0,487 0,875 0,399 0,195 0,105 -0,388 0,173 0,091 0,787 0,620 0,355
rs3796384 3p14 64526717 C G 0,154 0,783 0,875 0,629 0,398 0,215 -0,092 0,015 0,007 0,721 0,520 0,290
rs2278354 5p15.2 10448978 G T 0,120 0,703 0,839 0,583 0,351 0,190 -0,136 0,026 0,013 0,719 0,518 0,288
rs267071 5q22 116948503 C T 0,654 0,088 1,000 -0,566 0,343 0,188 -0,912 0,838 0,540 0,346 0,209 0,138
rs222541 6q16 95231163 G C 0,257 0,971 0,018 0,714 0,538 0,316 0,953 0,908 0,582 -0,239 0,120 0,070
rs1480642 6q23 136499528 C T 0,994 0,121 0,621 -0,873 0,772 0,483 -0,500 0,268 0,143 -0,373 0,223 0,139
rs4305737 6q24 145051594 A G 0,250 0,921 1,000 0,671 0,464 0,259 -0,079 0,041 0,028 0,750 0,600 0,380
rs285 8p21 19815189 T C 0,578 0,954 0,448 0,376 0,197 0,111 0,506 0,305 0,173 -0,130 0,017 0,008
rs3176921 8q13 67091379 T C 0,929 0,238 0,983 -0,692 0,493 0,278 -0,745 0,584 0,351 0,053 0,017 0,009
rs803733 9q33 125832879 C T 0,880 0,013 0,411 -0,867 0,761 0,470 -0,398 0,237 0,144 -0,469 0,241 0,128
rs7349 10p11.2 31817905 T C 0,062 0,969 0,000 0,907 0,824 0,507 0,969 0,939 0,623 -0,062 0,032 0,022
rs730570 14q32 101142890 A G 0,896 0,197 0,054 -0,699 0,492 0,274 0,144 0,047 0,025 -0,843 0,712 0,421
rs1129038 15q13 28356859 C T 0,224 0,996 0,983 0,771 0,625 0,389 0,013 0,004 0,002 0,758 0,601 0,362
rs1426654 15q21 48426484 G A 0,013 0,967 0,931 0,954 0,910 0,586 0,036 0,007 0,003 0,918 0,846 0,532
rs734780 15q26 89564958 C T 0,062 0,710 0,854 0,649 0,444 0,250 -0,144 0,030 0,015 0,793 0,633 0,366
rs730086 17q21 40271757 C T 0,667 0,063 1,000 -0,604 0,393 0,220 -0,937 0,881 0,574 0,333 0,200 0,132
rs6034866 20p12 17603728 A G 0,083 0,954 0,143 0,871 0,759 0,456 0,811 0,664 0,390 0,060 0,009 0,004
rs310612 20q13.3 62181508 A G 0,763 0,013 0,966 -0,750 0,593 0,360 -0,953 0,909 0,584 0,203 0,088 0,048
rs727563 22q13 41867377 C T 0,253 0,811 0,948 0,558 0,312 0,166 -0,137 0,045 0,024 0,695 0,504 0,288
Os SNPs rs2065160 (TSC1102055), rs2814778 (FYNULL), rs285 (LPL) e rs3176921 (CRH) são eventualmente conhecidos por este nome funcional (PARRA et al., 2004a; LINS et al., 2010), mas no presente estudo serão tratados pelo número de referência da sequência (rs#) no banco de dados dbSNP (http://www.ncbi.nlm.nih.gov/snp/) com o intuito de facilitar sua identificação e compartilhamento do resultados.
A genotipagem por minissequenciamento envolve a amplificação do fragmento de DNA no qual se localiza a mutação de única base a ser investigada, também chamada de SNP (do inglês, single nucleotide polymorphism) em reação de PCR (do inglês, polimerase chain
reaction). Para genotipagem, um iniciador, também chamado de primer, exatamente adjacente ao SNP é utilizado, ocorrendo então a adição de um ddNTP (do inglês, dideoxy-nucleotide-tri
phosphate), permitindo a incorporação de um único nucleotídeo. Sendo assim, a genotipagem
por minissequenciamento é denominada também SBE (do inglês, single base extension), que significa extensão de um único nucleotídeo (SYVANEN et al., 1990; SOBRINO et al., 2005). A genotipagem por minissequenciamento foi realizada utilizando o sistema comercial SNaPshot Multiplex (ABI Prism® SNaPshot® Multiplex Kit, Applied Biosystems, Foster City,
CA, USA), o qual possibilita a genotipagem em multiplex com ddNTPs marcados com moléculas fluorescentes (ddATP, ddGTP, ddTTP, ddCTP). Diferentes cores ao final da eletroforese correspondem a alelos específicos, sendo: vermelho (alelo T), amarelo (alelo C), verde (alelo A) e azul (alelo G).
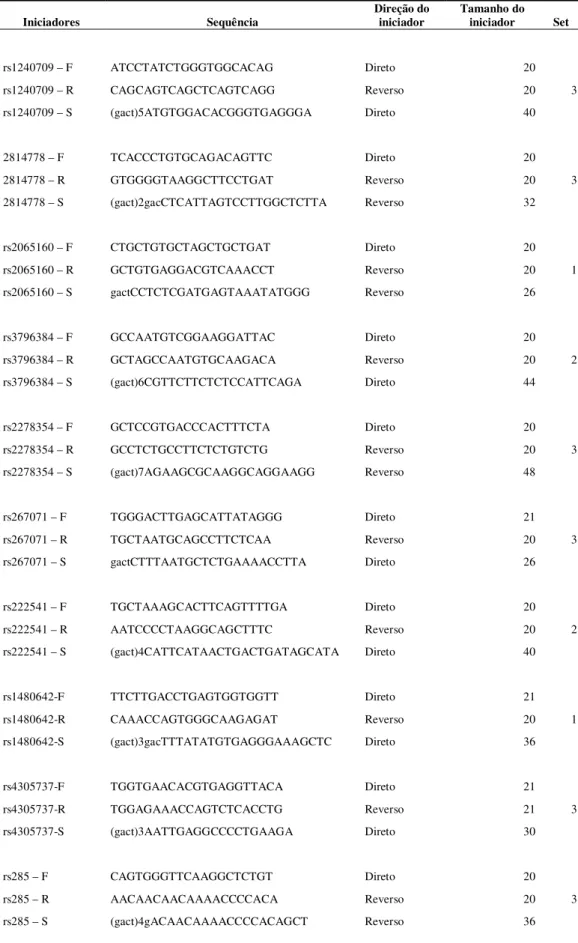
4.5.3.1 Desenho de iniciadores para PCR e Genotipagem
Tabela 02: Descrição das sequências e dos tamanhos dos iniciadores de PCR (F e R) e dos iniciadores de extensão de única base (S).
Iniciadores Sequência Direção do iniciador Tamanho do iniciador Set
rs1240709 – F ATCCTATCTGGGTGGCACAG Direto 20 rs1240709 – R CAGCAGTCAGCTCAGTCAGG Reverso 20 3 rs1240709 – S (gact)5ATGTGGACACGGGTGAGGGA Direto 40
2814778 – F TCACCCTGTGCAGACAGTTC Direto 20 2814778 – R GTGGGGTAAGGCTTCCTGAT Reverso 20 3 2814778 – S (gact)2gacCTCATTAGTCCTTGGCTCTTA Reverso 32
rs2065160 – F CTGCTGTGCTAGCTGCTGAT Direto 20 rs2065160 – R GCTGTGAGGACGTCAAACCT Reverso 20 1 rs2065160 – S gactCCTCTCGATGAGTAAATATGGG Reverso 26
rs3796384 – F GCCAATGTCGGAAGGATTAC Direto 20 rs3796384 – R GCTAGCCAATGTGCAAGACA Reverso 20 2 rs3796384 – S (gact)6CGTTCTTCTCTCCATTCAGA Direto 44
rs2278354 – F GCTCCGTGACCCACTTTCTA Direto 20 rs2278354 – R GCCTCTGCCTTCTCTGTCTG Reverso 20 3 rs2278354 – S (gact)7AGAAGCGCAAGGCAGGAAGG Reverso 48
rs267071 – F TGGGACTTGAGCATTATAGGG Direto 21 rs267071 – R TGCTAATGCAGCCTTCTCAA Reverso 20 3 rs267071 – S gactCTTTAATGCTCTGAAAACCTTA Direto 26
rs222541 – F TGCTAAAGCACTTCAGTTTTGA Direto 20 rs222541 – R AATCCCCTAAGGCAGCTTTC Reverso 20 2 rs222541 – S (gact)4CATTCATAACTGACTGATAGCATA Direto 40
rs1480642-F TTCTTGACCTGAGTGGTGGTT Direto 21 rs1480642-R CAAACCAGTGGGCAAGAGAT Reverso 20 1 rs1480642-S (gact)3gacTTTATATGTGAGGGAAAGCTC Direto 36
rs4305737-F TGGTGAACACGTGAGGTTACA Direto 21 rs4305737-R TGGAGAAACCAGTCTCACCTG Reverso 21 3 rs4305737-S (gact)3AATTGAGGCCCCTGAAGA Direto 30
rs285 – F CAGTGGGTTCAAGGCTCTGT Direto 20
rs285 – R AACAACAACAAAACCCCACA Reverso 20 3
Continuação – Tabela 02.
Iniciadores Sequência Direção do iniciador Tamanho do iniciador Set
rs3176921 – F TTTGTGCCCCTTCACTATGG Direto 20 rs3176921 – R CCATCTTTCTGCCTGGAAAA Reverso 20 2 rs3176921 – S (gact)3TGCAGAAGCAAGGCCAATAA Reverso 32
rs803733 – F TCCCCAAGAGTTCAACCAAC Direto 20 rs803733 – R AACCTTAGGCTTGAGCATGG Reverso 20 1 rs803733 – S (gact)7ATGTCATTGTGGAGGAGATA Reverso 48
rs7349 – F GCAATTGGTTCTCCTGCATT Direto 20 rs7349 – R GAAATGAGAGTTGTATGGTTAGGC Reverso 24 3 rs7349 – S (gact)5AAATGAGAGTTGTATGGTTAGGCT Reverso 44
rs730570 – F GCCTTCCATGGTTTCTCTGA Direto 20 rs730570 – R AGATTGTGGGGACTGTGAGC Reverso 20 2 rs730570 – S (gact)4TCACCTGCATCTCACACTGC Direto 36
rs1129038 – F CAGCAGCGACGATTCAGATA Direto 20 rs1129038 – R ATCACGGCCAGTCAGTCTCT Reverso 20 3 rs1129038 – S (gact)5ACAGTCTACACAGCAGCGAG Reverso 40
rs1426654 – F TTCAGCCCTTGGATTGTCTC Direto 20 rs1426654 – R AATTGCAGATCCAAGGATGG Reverso 20 2 rs1426654 – S gactgaCCGCTGCCATGAAAGTTG Reverso 24
rs734780 – F GATGGCACTGACCTTCCTTC Direto 20 rs734780 – R AGGTTGCAGTGAGCCAAGAT Reverso 20 1 rs734780 – S (gact)4CCCAGCAGTGGGTATCAC Direto 34
rs730086 – F GAGAACACTGGGGAGGTTCA Direto 20 rs730086 – R CAAAGTTCAGCACACCCTGA Reverso 20 2 rs730086 – S (gact)3CACCTCATTCCTGGTTTTAT Direto 32
rs6034866 – F TTGTGAGTCAAGGCAAGCTG Direto 20 rs6034866 – R TAGCTAGGGCAGGAGGTGAA Reverso 20 2 rs6034866 – S (gact)7TGTGAGTCAAGGCAAGCTGG Direto 48
rs310612 – F CACCCCTTCCTCACCTCAC Direto 19 rs310612 – R TTTCCTCACTCCCCTCTGTG Reverso 20 1 rs310612 – S (gact)4CAGGCGCAACACAGCCCAGC Direto 36
4.5.3.2 Protocolos das reações de PCR e Genotipagem
Previamente ao início da genotipagem em multiplex, todos os marcadores foram amplificados individualmente para conferência dos alelos e ajuste da concentração dos iniciadores. Quando amplificados em multiplex, ajustes adicionais de concentração foram realizados para otimizar a reação.
A reação de amplificação dos fragmentos de DNA através de PCR em multiplex foi realizada em volume final de 5µl utilizando o Qiagen Multiplex PCR Kit (QIAGEN®), o qual contém HotStarTaq® DNA polimerase, mistura de dNTPs, MgCl2 e tampão. Foi adotado o
seguinte protocolo: 2,5 µl do PCR Master Mix 2x, 0,5 µl de Solução Q 5x, 1,0 µl de uma mistura de iniciadores direto e reverso 10µM e 1,0 µl de DNA (5ng/ µl). Para cada amostra, foram realizadas 3 reações de PCR, uma para cada set de marcadores e esta separação foi mantida nas fases seguintes, até a obtenção dos genótipos. Após o preparo, a reação foi colocada em um termociclador (Veriti® 96-Well Thermal Cycler, Applied Biosystems, Foster City, CA, USA), alternando as temperaturas de 95, 57 e 72 ºC com o intuito de promover, respectivamente, a desnaturação do DNA (separação das fitas devido ao rompimento das pontes de hidrogênio), o anelamento dos iniciadores às fitas simples de DNA, e a incorporação dos dNTPs às novas fitas de DNA. Especificamente, o programa utilizado adotou a seguinte variação de temperatura: 15 minutos a 95ºC; 39 ciclos consistidos de 30 segundos a 94ºC, 1 minuto e 30 segundos a 57ºC e 1 minuto a 72ºC; 10 minutos a 72º; e manutenção da temperatura em 4ºC até a reação ser retirada do termociclador.
Após a amplificação, foi realizada uma purificação para a degradação enzimática do excesso de dNTPs e iniciadores, utilizando as enzimas exonuclease I (ExoI) e fosfatase alcalina de camarão (SAP, do inglês shrimp alkaline phosphatase), ambas produzidas pela USB (Cleveland, OH, USA). O protocolo de purificação foi: 0,5 L de SAP 1U/ L, 0,1 L de ExoI 10U/ L, e 0,9 L de tampão 10x da enzima SAP adicionados a 3 L de produto de PCR, com subsequente incubação em termociclador a 37oC durante 60 minutos, acrescidos 75ºC durante 15 minutos para inativação das enzimas. Os produtos da PCR purificados eram mantidos a -20oC, caso a reação de minissequenciamento não fosse realizada imediatamente.
e 0,34 L de água destilada ultrapura para completar o volume final da reação de 5 L. O programa de termociclagem utilizou as seguintes variações de temperatura: 2 minutos a 96ºC; 30 ciclos compostos de 10 segundos a 96ºC, 10 segundos a 58ºC e 20 segundos a 60ºC, com manutenção da temperatura em 4ºC até a reação ser retirada do termociclador.
Tabela 03: Concentração dos iniciadores de extensão de única base (S).
Iniciador (rs #) Tamanho do
iniciador (µM na reação) Concentração Set
rs1240709-S 40 0,60 3
rs2814778-S 32 1,00 3
rs2065160-S 26 0,55 1
rs3796384-S 44 0,55 2
rs2278354-S 48 0,60 3
rs267071-S 26 0,60 3
rs222541-S 40 0,55 2
rs1480642-S 36 0,80 1
rs4305737-S 30 0,60 3
rs285-S 36 0,60 3
rs3176921-S 32 1,00 2
rs803733-S 48 0,80 1
rs7349-S 44 0,60 3
rs730570-S 36 0,70 2
rs1129038-S 40 1,00 3
rs1426654-S 24 0,55 2
rs734780-S 34 0,80 1
rs730086-S 32 0,80 2
rs6034866-S 48 2,00 2
rs310612-S 36 0,55 1
rs727563-S 32 0,55 1
Set = Grupo no qual o marcador foi genotipado em multiplex.
Como último passo antes da eletroforese dos produtos de minissequenciamento, foi realizada uma segunda purificação das amostras, para a remoção dos ddNTPs fluorescentes não incorporados. Estes, caso não fossem removidos, comigrariam com os produtos de minissequenciamento durante a eletroforese causando dificuldades de interpretação dos resultados. Para purificação dos 5 L de produto da reação de minissequenciamento foram utilizados 0,5 L de SAP 1U/ L e 0,5 L de tampão 10x da enzima SAP, com posterior incubação a 37°C por 60 minutos e 85°C por 15 minutos para desnaturar a enzima.
ao padrão de tamanho GS120-LIZ (GeneScanTM – 120 LIZ®, Applied Biosystems, Foster City, CA, USA). O protocolo envolveu a adição de 1µl de produto de minissequenciamento, 0,17µl de padrão de tamanho GS120-LIZ em 8,83µl de Hi-Di. Os parâmetros de corrida foram: polímero POP6 com tensão de 15 volts por 20 segundos de injeção e 15 volts de corrida por 800 segundos. A análise dos eletroferogramas foi realizada com o programa GeneMapperTM 4.0.
4.6 Determinação da ancestralidade genética
A ancestralidade genética foi calculada com o programa ADMIXMAP versão 3.8 (http://homepages.ed.ac.uk/pmckeigu/admixmap/) utilizando 2.500 interações no período de
burn-in e 10.000 interações para medir os parâmetros dos dados. Também foi obtido por este
programa um parâmetro chamado soma de intensidades, o qual equivale ao número médio de gerações desde o processo de miscigenação. Amostras das populações de Botsuana, Camarões, Gana e Senegal (n=120), euroamericanos de Baltimore e de Chicago (n=78) e zapotecos do México (n=29) foram utilizados como representativos das populações africana, europeia e nativo americana, respectivamente.
As estimativas de ancestralidade também foram calculadas pelo programa STRUCTURE 2.3.3 (PRITCHARD et al., 2000), com índices altamente correlacionados à estimativa obtida com o ADMIXMAP: AMR Rho= 0,970, p<0,001; AFR Rho=0,987, p<0,001; EUR Rho=0,980, p<0,001. Estes dois programas são os mais utilizados para estimar a ancestralidade genômica e ambos trabalham com sistema bayesiano coletando primeiramente informação dos dados genéticos das populações parentais e posteriormente estimando as distribuições de ancestralidade utilizando cadeias Markov de Monte Carlo (MCMC – do inglês, Monte Carlo Markov Chain), sendo que diferem ligeiramente na forma como tratam a informação nessa primeira etapa, sendo que o programa STRUCTURE requer uma razão mínima entre o número de ancestrais e de miscigenados inseridos na análise para gerar resultados acurados (TSAI et al., 2005).
4.7 Análise Estatística
africana e ameríndia. Estas variáveis foram ainda testadas quanto à distribuição normal utilizando o teste de Kolmogorov-Smirnov, e, quando necessário, dados pontuais discrepantes (escores com mais de 2 desvios padrão acima ou abaixo da média) foram substituídos por valores iguais à média mais ou menos 2 desvios padrão, respectivamente. Já as variáveis discretas (cor de pele e classificação socioeconômica) foram apresentadas em frequências absoluta e relativa.
A análise gráfica de Bland-Altman (BLAND e ALTMAN, 1986) foi utilizada para avaliar a concordância da ancestralidade genômica e do índice de melanina entre as irmãs. Para análise de concordância da variável categórica cor de pele autodenominada foi utilizado o teste de Kappa (COHEN, 1960).
A associação entre cor de pele e índice de melanina, entre cor de pele e ancestralidade genômica, entre nível socioeconômico e índice de melanina e entre nível socioeconômico e ancestralidade genômica foi testada utilizando o teste ANOVA, já que não foram encontrados grandes desvios dos pressupostos para realização deste teste. Foi também investigado o teste não paramétrico de Kruskal-Wallis para análise das associações supracitadas, no qual foram obtidos resultados praticamente idênticos ao do teste paramétrico.
Para testar a relação entre o índice de melanina e a ancestralidade genômica foi utilizada a correlação não paramétrica de Spearman e para testar a relação entre cor de pele e nível socioeconômico foi utilizado o teste de qui-quadrado.
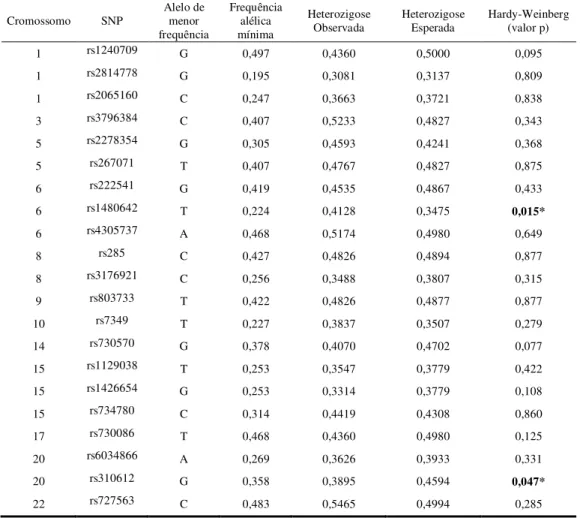
O equilíbrio de Hardy-Weinberg e o desequilíbrio de ligação entre os marcadores genotipados no grupo amostral foi realizado utilizando o programa PLINK versão 1.07 (PURCELL et al., 2007), disponível em http://pngu.mgh.harvard.edu/purcell/plink/. Foi utilizado ainda o programa estatístico ADMIXMAP para avaliar a associação dos marcadores genéticos (SNPs) com o índice de melanina. Os valores de significância obtidos neste teste foram ajustados utilizando a correção de Bonferroni para múltiplas comparações (p = 0,05 ÷ 21 AIMs = 0,002).
Como o estudo teve como amostra pares de irmãs, as análises foram repetidas com amostras não relacionadas (ao dividir a amostra separando aleatoriamente as irmãs em dois novos grupos), e os resultados seguiram mesmo padrão daqueles obtidos com o grupo total, o que viabiliza as análises originais do estudo em termos da possibilidade de correlação de escores entre as irmãs.