Universidade
Católica de
Brasília
PRÓ-REITORIA DE PÓS-GRADUAÇÃO E PESQUISA
STRICTO SENSU EM CIÊNCIAS GENÔMICAS E
BIOTECNOLOGIA
Mestrado
BRASÍLIA
2007
DESENVOLVIMENTO E APLICAÇÃO DE
POLIMORFISMOS DE INSERÇÃO/DELEÇÃO DO
CROMOSSOMO X EM GENÉTICA FORENSE
Autor: Erika Horta Grandi Monteiro
ERIKA HORTA GRANDI MONTEIRO
DESENVOLVIMENTO E APLICAÇÃO DE POLIMORFISMOS DE INSERÇÃO/DELEÇÃO DO CROMOSSOMO X EM GENÉTICA FORENSE
Dissertação apresentada ao Programa de Pós-Graduação “Stricto Sensu” em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito para obtenção de Título de Mestre em Ciências Genômicas e Biotecnologia.
Orientador: Prof. Dr. Rinaldo Wellerson Pereira
ERIKA HORTA GRANDI MONTEIRO
POLIMORFISMOS DE INSERÇÃO/DELEÇÃO NO CROMOSSOMO X
APLICADOS EM GENÉTICA FORENSE.
Dissertação defendida e aprovada como requisito parcial para obtenção do Título de Mestre em Ciências Genômicas e Biotecnologia, defendida e aprovada, em 11 de junho de 2007 pela banca examinadora constituída por:
Prof. Dr. Rinaldo Wellerson Pereira Universidade Católica de Brasília Orientador
Prof. Dr. Dário Grattapaglia Universidade Católica de Brasília Avaliador Interno
Prof. Dr. Márcio Elias Ferreira Universidade Católica de Brasília Avaliador Interno
Prof. Dr. Sidney Emanuel Batista dos Santos Universidade Federal do Pará
Avaliador Externo
À memória da querida Vó Amandina, mulher sempre à frente do seu tempo e eterna fonte de inspiração.
AGRADECIMENTOS
Ao meu orientador, Prof. Dr. Rinaldo Wellerson Pereira, pela paciência, dedicação, orientação e por todos os ensinamentos durante esses anos.
Aos Profs. Drs. Dário Grattappaglia, Marcio Elias e Sidney Santos, membros da banca de avaliação, pelas contribuições, críticas, elogios e por terem aceitado fazer parte dessa banca.
Ao Programa de Pós-Graduação em Ciências Genômicas e Biotecnologia e à Universidade Católica de Brasília, juntamente com os professores e funcionários, pela contribuição na minha formação profissional e ajuda em todos os momentos solicitados e pela atenção despendida.
Ao Dr. Ruy Caldas, coordenador do curso de Pós-graduação em Ciências Genômicas e Biotecnologia,Danielle de Mourae ao secretário Fábio Costa, por todo apoio e incentivo.
A Universidade Católica de Brasília e ao CNPq pelo apoio financeiro que tornou possível a realização desse trabalho.
Aos Dr. Bruno Tommasi e Dr. Henrique Tommasi por terem me dado a grande oportunidade de estar envolvida com a área de Biologia Molecular. Agradeço por terem me proporcionado a oportunidade de realizar o mestrado em Brasília, pela confiança e pela amizade.
A todos os funcionários do Instituto Tommasi, em especial a Dra.Gabriela Nogueira, por todos os conselhos, ajuda e pela imensa amizade.
A todos os funcionários do laboratório Heréditas, pelos amostras de DNA concedidas, tempo e ajuda na realização da minha dissertação.
Ao pessoal do Cenargen, Danielle Faria, Eva Mamani, Juliano Pádua e Marília Pappas pela imensa boa vontade em me atender e ajudar todas as vezes que precisei utilizar o seqüenciador.
Aos técnicos André Ramos, Idacuy Mundin, Márcia e Willian Baião por toda ajuda e pelas boas risadas.
A Breno Abreu e Túlio Lins, colegas de mestrado, de bancada, profissionais competentes, animados e acima de tudo amigos. Obrigada por tudo.
Aos amigos brasilienses, Alessandra Reis, Aline Braga, André Amaral, Joanna de Abreu, Pollyanna Diener e Natália Bueno pela amizade, pela troca de conhecimentos e pelas boas risadas. Obrigada por me receberem tão bem.
de conhecimento, pelos momentos que passamos juntos, pelas críticas, sugestões, ajuda, incentivo e colaboração.
A meus pais, Vânia e Fabiano, pelos momentos de ensinamento e alegria, incentivo, paciência, críticas, carinho e amor.
Aos meus irmãos Juliana e Rubens, sempre com uma palavra de carinho a me incentivar. A toda minha família pelo apoio incondicional e pela amizade. Ao meu primo Alex, por ter me recebido tão bem em Brasília e por fazer tão bem o papel de família.
Aos queridos amigos pela torcida, que apesar de longe, sempre muito perto do coração.
Sou grata pelo apoio, oportunidade, incentivo e ajuda oferecido por todos aqueles que fizeram parte da minha vida nestes dois anos de intenso trabalho. Obrigada!
Quando você partir em direção a Ítaca, Que sua jornada seja longa, Repleta de aventuras, plena de conhecimento. Não temas Laestrigones e Ciclopes nem o furioso Poseidon;
Você não irá encontrá-los durante o caminho, se O pensamento estiver elevado, se a emoção
jamais abandonar seu corpo e seu espiríto. Laestrigones e Ciclopes, e o furioso Poseidon
não estarão em seu caminho se você não carregá-los em sua alma, se sua alma não os colocar diante de seus passos.
Espero que sua estrada seja longa. Que sejam muitas as manhãs de verão, que o prazer de ver os primeiros portos
traga uma alegria nunca vista. Procure visitar os empórios da Fenícia
recolha o que há de melhor. Vá às cidades do Egito,
aprenda com um povo que tem tanto a ensinar. Não perca Ítaca de vista,
pois chegar lá é o seu destino. Mas não apresse os seus passos; é melhor que a jornada demore muitos anos
e seu barco só ancore na ilha quando você já estiver enriquecido
com o que conheceu no caminho. Não espere que Ítaca lhe dê mais riquezas.
Ítaca já lhe deu uma bela viagem; sem Ítaca,você jamais teria partido. Ela já lhe deu tudo, e nada mais pode lhe dar.
Se,no final,você achar que Ítaca é pobre, não pense que ela o enganou.
Por que você tornou-se um sábio, viveu uma vida intensa, e este é o significado de Ítaca.
RESUMO
Atualmente, a identificação genética humana está centrada na utilização de um conjunto de marcadores autossômicos do tipo microssatélites, os quais suprem a maioria das demandas apresentadas aos laboratórios de genética forense. No entanto, para algumas situações, os microssatélites autossômicos comumente utilizados não oferecem resultados satisfatórios. Estas situações passam por casos onde o material genético do suposto pai não está disponível e casos onde a amostra de DNA obtida apresenta-se degradada ou em quantidades abaixo daquelas que são limites para utilização dos sistemas comerciais para amplificação dos microssatélites autossômicos. Com o fito de auxiliar a resolução dessas situações problemáticas, este trabalho objetivou a utilização de marcadores no cromossomo X, permitindo complementar estudos de parentesco, especificamente em situações onde as amostras encontram-se degradadas e/ou em baixas concentrações de DNA. Os marcadores escolhidos foram polimorfismos do tipo inserção/deleção (indel) no cromossomo X, de 2 a 11 pares de base. Foram desenhados iniciadores para 17 indels do cromossomo X que ao amplificar geram produtos de tamanho de 70 a 170 pares de base. Dessas 17 indels apenas 8 permitiram sua utilização em reação em multiplex. O sistema multiplex desenvolvido apresentou alta sensibilidade em suas amplificações, obtendo resultados com concentrações de até 0,1 ng de DNA em um volume de reação de 12,5 l, através de estudos de sensibilidade. Para testar a eficiência do multiplex, foram utilizadas amostras artificialmente degradadas com a utilização de DNAse I. A partir dessas amostras artificialmente degradadas utilizou-se o protocolo de Low Copy Number DNA onde aumentou-se o número de ciclos do protocolo de amplificação de 30 para 34. Essa técnica mostrou-se realmente eficaz. Além disso, também se testou a eficiência da amplificação do sistema multiplex em amostras degradadas submetidas à amplificação de genoma total que se mostrou ineficaz. Um estudo da população brasileira foi conduzido em 200 amostras das cinco regiões geopolíticas brasileiras a fim de estabelecer a freqüência das indels do cromossomo X. Os dados de estrutura populacional indicaram diferença entre regiões (FST= 0,021 P<0,05). Finalmente analisou-se o
funcionamento e a precisão do multiplex em casos de paternidade com trios e suposto pai falecido e compararam-se os resultados previamente obtidos pelo estudo com microssatélites. O sistema de multiplex de indels do cromossomo X provou ser útil e sua aplicação como complemento em casos de parentesco foi aprovado, assim como, seu sucesso na amplificação de amostras degradadas e em amostras com baixa quantidade de DNA, quando analisado em conjunto com a técnica de LCN.
ABSTRACT
Nowadays the human genetic identification it’s based on the utilization of a set of autosomal markers, the microsatellites, which fulfils the majority of the forensic genetic labs requirements. However, there are some situations when that the usual autosomal microsatellites don’t always offer good results. Those situations include cases where the genetic sample of the alleged father is not available and cases where the DNA sample presents itself degraded or in low quantities, rendering the utilization of commercial systems to the amplification of autosomal markers useless. With the aim of assisting the resolution of these problematic situations, this study aimed the use of X-chromosome markers, allowing to improve kinship studies, more specifically, scenarios using degraded or low quantity DNA samples. The markers chosen were insertion/deletion (indel) polymorphisms from X-Chromosome, ranging from 2-11 base pairs. Primers were designed for 17 X-chromosome indels, in order to generate Amplicons ranging from 70 to 170 base pairs. Only eight of these 17 indels allowed their use in multiplex reaction. The X-chromosome based multiplex system has shown to be highly sensitive in their amplifications, being able to amplify as low as 0.1 ng of DNA in a reaction volume of 12.5 l, through sensitivity studies. To test the amplification efficiency of the multiplex, artificially and controlled degraded samples using DNAse I enzyme were used. From these artificially degraded samples, a new amplification protocol based on the increase from 30 cycles to 34 cycles was used. This technique proved to be really effective. Also, degraded samples were amplified using whole genome amplification prior to the PCR reaction for comparison sake that proved ineffective. A Brazilian population study was held on 200 samples from the five geopolitical Brazilian regions in order to establish the frequency of X-Chromosome indels. Population comparison indicated genetic difference between regions (FST= 0.021 P<0.05). Along with that, some kinship case studies,
which were already been analyzed through the use of autosomal marker, were conducted using this novel 8 X-chromosome indel marker multiplex set. This novel multiplex system based on X-chromosome indels markers has proven to be useful and its usage on kinship cases as a complementary tool has been approved, and, in addition, it has proven to be successful in the amplification of degraded samples as well as in low quantity DNA samples along with other technique, Low Copy Number.
LISTA DE FIGURAS
Figura 1 – Primeiras utilizações em casos de imigração e criminal...27
Figura 2 - Marcador STR.. ...29
Figura 3 - Reação de Multiplex...30
Figura 4 – Locos do sistema Codis com suas localizações cromossômicas. ...31
Figura 5 - Cromossomo Y humano.. ...37
Figura 6 - Mapa do genoma mitocondrial humano. ...40
Figura 7 - Cromossomo X humano e seus principais marcadores STRs. ...43
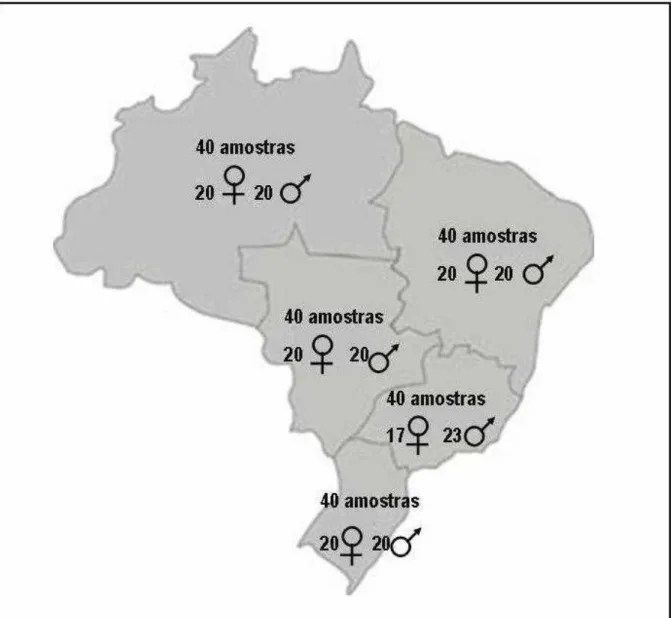
Figura 8 – Distribuição das amostras populacionais brasileiras utilizadas nesse estudo....54
Figura 9 – Localização das 17 indels selecionadas ao longo do cromossomo X...65
Figura 10 – Distribuição dos produtos gerados pelos 17 pares de iniciadores das indels selecionadas ao longo do cromossomo X...66
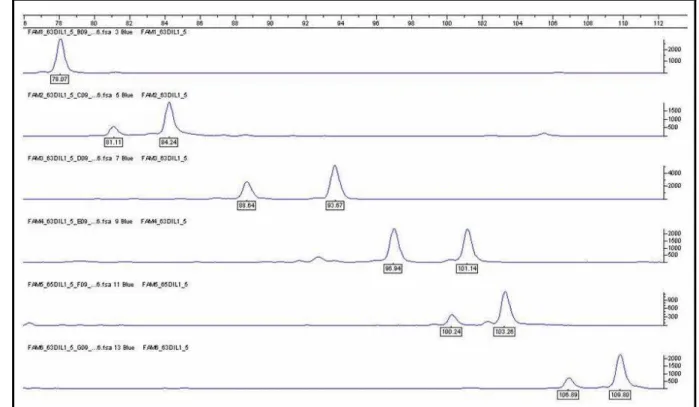
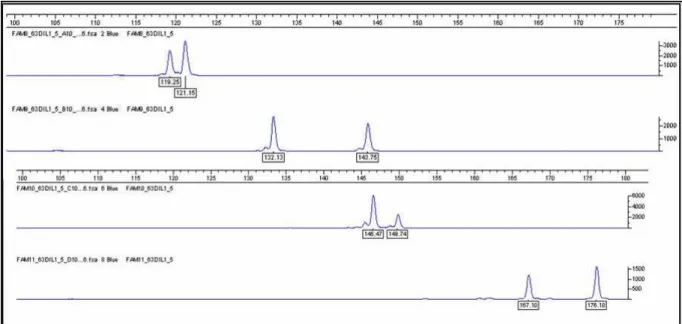
Figura 11 - Eletroferograma das amplificações em singleplex 6-FAM IDX001 a IDX006. ..68
Figura 12 - Eletroferograma das amplificações em singleplex 6-FAM IDX008 a IDX011. ..69
Figura 13 - Eletroferograma das amplificações em singleplex VIC IDX013 a IDX017. ...69
Figura 14 - Distribuição dos produtos gerados pelos 8 pares de iniciadores das indels selecionadas ao longo do cromossomo X, que constituem a reação de multiplex. ...71
Figura 15 - Eletroferograma demonstrando a multiplexação das 8 indels do cromossomo X em 3 indivíduos diferentes...72
Figura 16 - Distribuição das 8 indels constituintes do multiplex no cromossomo X...73
Figura 17 – Resultados AMOVA na população brasileira...77
Figura 18 – Resultados da análise de variância loco a loco das amostras regionais da população brasileira...79
Figura 19 – Heredograma demonstrando configuração do caso Post mortem 1 (PM1).. ...90
Figura 20 - Heredograma demonstrando configuração do caso Post mortem 2 (PM2).. ...93
Figura 21 - Heredograma demonstrando configuração do caso Post mortem 3 (PM3). ...96
Figura 22 - Heredograma demonstrando configuração do caso Post mortem 4 (PM4). ...100
Figura 23 - Heredograma demonstrando configuração do caso Post mortem 5 (PM5).. ...103
Figura 24 - Heredograma demonstrando configuração do caso Post mortem 6 (PM6).. ...107
Figura 26 - Heredograma demonstrando configuração do caso Post mortem 8 (PM8)....113 Figura 27 - Heredograma demonstrando configuração do caso Post mortem 9 (PM9). ...117 Figura 28 - Heredograma demonstrando configuração do caso Post mortem 10 (PM10). ...118 Figura 29 - Heredograma demonstrando configuração do caso Post mortem 11 (PM11).. ..118 Figura 30 - Heredograma demonstrando configuração do caso Post mortem 12 (PM12).. ..119 Figura 31 - Heredograma demonstrando configuração do caso Post mortem 13 (PM13). ...119 Figura 32 - Heredograma demonstrando configuração do caso Post mortem 14 (PM14). ...120 Figura 33 - Heredograma demonstrando configuração do caso Post mortem 15 (PM15). ..120 Figura 34 - Heredograma demonstrando configuração do caso Post mortem 16 (PM16).. ..121 Figura 35 - Heredograma demonstrando configuração do caso Post mortem 17 (PM17). ...121 Figura 36 - Eletroferograma mostrando resultados dos testes de sensibilidade com os
iniciadores marcados com 6-FAM....124 Figura 37 - Eletroferograma mostrando resultados dos testes de sensibilidade com os
LISTA DE TABELAS
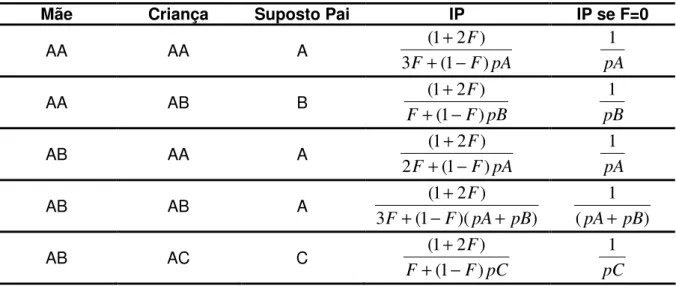
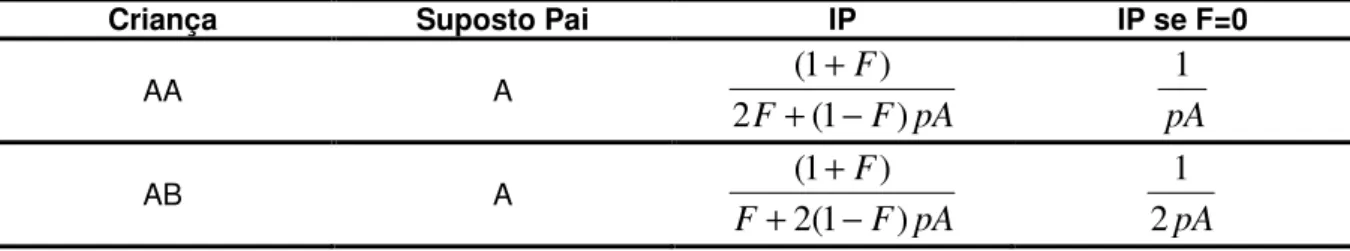
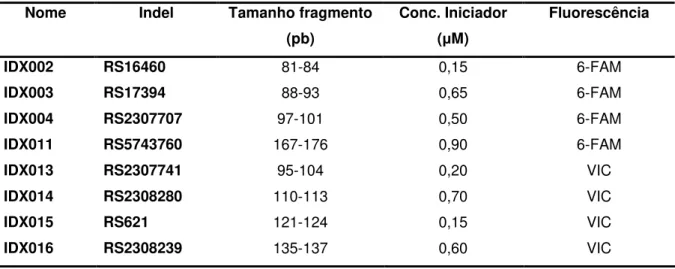
Tabela 1 - Fórmulas para cálculo de IP individual para locos independentes no cromossomo X, em trios. ...58 Tabela 2 - Fórmulas para cálculo de IP individual para locos independentes no cromossomo X, em duos...59 Tabela 3 - Indels selecionadas a partir do NCBI®. ...63 Tabela 4 - Características das 17 indels do cromossomo X selecionadas a partir do NCBI® 64 Tabela 5 - Seqüência dos iniciadores para cada indel selecionada....67 Tabela 6 - Indels do cromossomo X constituintes do multiplex. ...71 Tabela 7 - Caracterização dos 8 locos de indels do cromossomo X baseado em uma amostra de 200 indivíduos....74 Tabela 8 - Desequilíbrio de ligação entre as mini-indels do cromossomo X constituintes do multiplex, calculados apenas entre os homens. ...75 Tabela 9 - Características das 8 indels do cromossomo X utilizadas em multiplex...75 Tabela 10 – Matriz diagonal inferior com valores de Fst par a par e matriz diagonal superior com valores de significância (Pvalor) para população de homens e mulheres....78 Tabela 11 – Matriz diagonal inferior com valores de Fst par a par e matriz diagonal superior com valores de significância (Pvalor) para população de homens...78 Tabela 12 – Matriz diagonal inferior com valores de Fst par a par e matriz diagonal superior com valores de significância (Pvalor) para população de mulheres...78 Tabela 13 - Perfis genotípicos de seis trios de exclusão resultantes da análise com o octaplex (mais amelogenina) de STRs...82 Tabela 14 - Perfis genotípicos de seis trios de exclusão resultantes da análise dodecaplex de STRs.. ...83 Tabela 15 - Perfis genotípicos de seis trios de exclusão resultantes da análise com as mini-indels do cromossomo X.....84 Tabela 16 - Perfis genéticos de seis trios com resultado de inclusão, analisados com o
octaplex (mais amelogenina) de STRs. ...85 Tabela 17 - Perfis genéticos de seis trios com resultado de inclusão, analisados com o
Tabela 18 - Perfis genéticos de seis trios com resultado de inclusão, analisados com com as mini-indels do cromossomo X...87 Tabela 19 - Índice de paternidade combinado (IPC) dos trios com inclusões nos diversos sistemas de multiplex utilizados e suas combinações. ...88 Tabela 20 – Perfis genéticos do caso Post mortem 1 obtidos pela análise de 25 STRs
autossômicos (mais amelogenina). ...91 Tabela 21 - Perfis genéticos do caso Post mortem 1 obtidos pela análise de 8 mini-indels do cromossomo X...92 Tabela 22 - Perfis genéticos do caso Post Mortem 2 obtidos pela análise de 32 STRs
autossômicos (mais amelogenina)....94 Tabela 23 - Perfis genéticos do caso Post mortem 2 obtidos pela análise de 8 mini-indels do cromossomo X...95 Tabela 24 - Perfis genéticos do caso Post Mortem 3 obtidos pela análise de 30 STRs
autossômicos (mais amelogenina)....97 Tabela 25 - Perfis genéticos do caso Post Mortem 3 obtidos pela análise de 7 X-STRs....98 Tabela 26 - Perfis genéticos do caso Post Mortem 3 obtidos pela análise de 8 mini-indels do cromossomo X...99 Tabela 27 - Perfis genéticos do caso Post Mortem 4 obtidos pela análise de 31 STRs
autossômicos...101 Tabela 28 - Perfis genéticos do caso Post Mortem 4 obtidos pela análise de 8 mini-indels do cromossomo X...102 Tabela 29 – Perfis genéticos do caso Post Mortem 5 obtidos pela análise de 32 STRs
autossômicos....104 Tabela 30 - Perfis genéticos do caso Post Mortem 5 obtidos pela análise de 7 X-STRs....105 Tabela 31 - Perfis genéticos do caso Post Mortem 5 obtidos pela análise de 8 mini-indels do cromossomo X...106 Tabela 32 - Perfis genéticos do caso Post Mortem 6 obtidos pela análise de 32 STRs
Tabela 35 - Perfis genéticos do caso Post Mortem 7 obtidos pela análise de 33 STRs
autossômicos...111 Tabela 36 - Perfis genéticos do caso Post Mortem 7 obtidos pela análise de 8 mini-indels do cromossomo X...112 Tabela 37- Perfis genéticos do caso Post Mortem 8 obtidos pela análise de 34 STRs
LISTA DE ABREVIATURAS
A Adenina
BSA Albumina de soro bovino (do inglês, Bovine Serum Albumin)
C Citosina
CODIS Sistema Indexado de DNA Combinando (do inglês, Combined DNA Index System)
D-loop Alça de desdobramento (do inglês, Displacement Loop)
DNA Ácido Desoxirribonucléico (do inglês, Deoxyribonucleic Acid)
dNTP Desoxirribonucleotídeo Trifosfato (do inglês, Deoxyribonucleotide triphosphate)
DOP-PCR PCR com oligonucleotídeos degenerados (do inglês, Degenerate Oligonucleotide Primed PCR)
EDTA Ácido Etileno Diamono Tetrecético (do inglês, Ethylene Diamine TetrAcetic Acid)
FBI Escritório Federal de Investigação (do ingles, Federal Bureau of Investigation)
G Guanina
H Heterozigosidade Indels Inserção/Deleção IP Índice de Paternidade
IPC Índice de Paternidade Combinado
I-PEP PEP melhorado (do inglês, Improved PEP)
ISFG Sociedade Internacional de Genética Forense (do inglês, International Society of Forensic Genetics)
LCN Baixo Número de Cópias (do inglês, Low Copy Number)
LL-DOP-PCR Produtos longos de baixas quantidades DOP-PCR (do inglês, Long Products from low quantities DOP-PCR).
MDA Amplificação de deslocamentos múltiplos (do inglês, Multiple Displacement Amplification)
MSY Região específica de homens do cromossomo Y (do inglês, Male Specific Y Chromosome Region)
NIST Instituto Nacional de Padrões e Tecnologia (do inglês, National Institute of Standards and Technology)
NRY Região não recombinante do cromossomo Y (do inglês, Non Recombining Y-chromosome Region)
PAR Região pseudo-autossômical (do inglês, Pseudo-autosomal region) PCR Reação em Cadeia de Polimerase (do inglês, Polimerase Chain Reaction) PD Poder de Discriminação
PEP Pré-amplificação por extensão do iniciador (do inglês, Primer Extension Preamplification)
PIC Conteúdo de informação de polimorfismo (do inglês, Polymorphism Information Content)
PP Probabilidade de Paternidade
PPE Probabilidade de Exclusão de Paternidade
RFLP Polimorfismos de Fragmentos de Restrição (do inglês, Restriction Fragment Length Polymorphism)
SDA Amplificação com deslocamento da fita (do inglês, Strand displacement Amplification)
SDS Sódio Dodecil Sulfato
SLP Sonda de Loco Único (do inglês, Single Locus Probe)
SNP Polimorfismo de Base Única (do inglês, Single Nucleotide Polymorphism) SSR Repetições de Seqüência Simples (do inglês, Simple Sequence Repeats) STR Repetições Consecutivas Curtas (do inglês, Short Tandem Repeat)
T Timina
Taq Thermus aquaticus
TE Tris-EDTA
Tris Tris-(hidroximetil)-aminometano
VNTRs Repetições Consecutivas de Número Variado (do inglês, Variable Number Tandem Repeat)
LISTA DE SÍMBOLOS
% porcentagem cm centímetro g grama
HCl ácido clorídrico KCl cloreto de potássio kg kilograma
M Molar mg miligrama
MgCl2 cloreto de magnésio
min minuto mL mililitro mM milimolar NaCl cloreto de sódio NaOH hidróxido de sódio ng nanograma
º C grau Celsius pb par de base pg picograma
pH potencial hidrogeniônico rpm rotação por minuto s segundo
U unidade
SUMÁRIO
1 INTRODUÇÃO ...19
2 HIPÓTESE DE TRABALHO...21
3 OBJETIVOS...22
3.1 OBJETIVO GERAL...22
3.2 OBJETIVOS ESPECÍFICOS...22
4 REVISÃO DA LITERATURA ...24
4.1 ASPECTOS HISTÓRICOS DA GENÉTICA FORENSE...24
4.2 MARCADORES POLIMÓRFICOS DE USO CORRENTE EM GENÉTICA FORENSE...28
4.3 MARCADORES DE LINHAGEM EM GENÉTICA FORENSE...33
4.3.1 Cromossomo Y ...33
4.3.2 DNA mitocondrial ...37
4.3.3 Marcadores polimórficos no cromossomo X ...41
4.4 AMOSTRAS COM QUANTIDADES MÍNIMAS DE DNA E/OU DEGRADADAS EM GENÉTICA FORENSE...46
5 MATERIAIS E MÉTODOS...50
5.1 SELEÇÃO DAS INDELS...50
5.2 DESENHO DOS INICIADORES...51
5.3 AMPLIFICAÇÃO DAS INDELS DO CROMOSSOMO X...52
5.4 DETECÇÃO E ANÁLISE DOS PRODUTOS DA PCR...53
5.5 AMOSTRAS DE DNA ...53
5.7 UTILIZAÇÃO EM CASOS DE PATERNIDADE...57
5.8 SENSIBILIDADE...60
5.9 ESTUDOS DE DEGRADAÇÃO...60
5.10 AMPLIFICAÇÃO PELA METODOLOGIA DE LOW COPY NUMBER DNA ...61
5.11 AMPLIFICAÇÃO DE GENOMA TOTAL...61
6 RESULTADOS...62
6.1 IDENTIFICAÇÃO DE INDELS POLIMÓRFICAS AO LONGO DO CROMOSSOMO X ...62
6.2 DESENHO DOS INICIADORES...66
6.3 AMPLIFICAÇÃO DAS INDELS DO CROMOSSOMO X...68
6.4 ESTUDO POPULACIONAL...74
6.5 ESTUDO DE CASOS: EXAMES DE PATERNIDADE EM TRIOS...80
6.6 ESTUDO DE CASOS: EXAMES DE PATERNIDADE EM CASOS DE SUPOSTO PAI FALECIDO89 6.7 TESTE DE SENSIBILIDADE...123
6.8 ESTUDOS DE DEGRADAÇÃO...126
6.9 AMPLIFICAÇÃO DO MULTIPLEX DE INDELS DO CROMOSSOMO X UTILIZANDO PROTOCOLOS DE LOW COPY NUMBER DNA E DE WHOLE GENOME AMPLIFICATION...128
7 DISCUSSÃO...136
8 CONCLUSÃO ...141
9 APÊNDICE...143
10 REFERÊNCIAS ...145
1 INTRODUÇÃO
Há mais de um século, desde a descoberta dos grupos sanguíneos, que polimorfismos em humanos vêm auxiliando a resolução de questões criminais. Passando pelos polimorfismos de grupos sanguíneos, em seguida pelos polimorfismos protéicos/enzimáticos e pelos primeiros polimorfismos em seqüência de DNA (RFLPs), o grande salto deu-se com a identificação dos polimorfismos de seqüência repetitiva do tipo minissatélites. Os minissatélites e sua alta variabilidade permitiram ao Sir Alec Jefreys e seus colaboradores, no início da década de 80 do século XX, as primeiras aplicações de genética forense em um contexto moderno como conhecemos hoje. No entanto, foi a caracterização dos microssatélites e a possibilidade de genotipá-los via PCR, no final dos anos 80 e início dos anos 90, os eventos que permitiram ao campo da genética forense sua consolidação. Atualmente, a base de qualquer laboratório de genética forense é a utilização de 13 a 15 microssatélites autossômicos co-amplificados pela PCR (utilizando sistemas comerciais) e genotipados utilizando a eletroforese em seqüenciadores automáticos de DNA. Em situações especiais a utilização de microssatélites do cromossomo Y e do cromossomo X, oferece auxílio de suma importância para a conclusão de uma investigação. Haplótipos de cromossomo Y são utilizados na identificação de indivíduos relacionados pela mesma linhagem paterna e são importantes ferramentas na identificação individual de acusados de crimes sexuais. Microssatélites do cromossomo X, utilizados como haplótipos ou não, auxiliam no estabelecimento de vínculos de parentesco onde a configuração não é aquela tradicional de mãe, filho e suposto pai. Principalmente naqueles casos onde o suposto pai é ausente, o filho em questão é do sexo feminino e se tem parentes com os quais se podem reconstruir o cromossomo X do suposto pai.
de alterar as condições da PCR e assim obter perfis de DNA para um maior número de locos co-amplificados. Para amostras degradadas, os esforços tem-se concentrado na redução dos fragmentos de microssatélites amplificados pela PCR e na utilização de marcadores polimórficos do tipo SNP (do inglês, Single Nucleotide Polymorphism), que por sua natureza permitem o desenho de sistemas de genotipagem baseados na amplificação pela PCR de fragmentos pequenos.
Os SNPs requerem métodos de genotipagem que envolvem mais que a simples amplificação e eletroforese. Alternativamente, polimorfismos de pequenas inserções/deleções (2-20 pb) podem ser facilmente genotipados utilizando a PCR e eletroforese. Com esta última informação em mente o trabalho apresentado neste documento propôs-se estabelecer um sistema para a co-amplficação de indels do cromossomo X e testá-las como ferramentas de auxílio em identificação humana. Os sistemas de amplificação foram desenhados de forma que os fragmentos tivessem seus tamanhos entre 70 e 170 pares de base. Em analogia ao termo mini-strs que é aplicado para microssatélites amplificados de forma a gerarem fragmentos curtos, as indels genotipadas neste trabalho serão denominadas mini-indels. Além de testar as mini-indels do cromossomo X como ferramentas auxiliares para identificação humana, testou-se também sua aplicação em conjunto a técnicas que buscam solucionar questões relacionadas a baixa concentração de DNA. As técnicas escolhidas foram: a utilização da amplificação pela metodologia de LCN-DNA (do inglês, low copy number DNA) e a amplificação por WGA (do inglês, Whole Genome
Amplification) anterior a amplificação pelas mini-indels.
2 HIPÓTESE DE TRABALHO
Mini-indels do cromossomo X fornecem um poder de exclusão equivalente a microssatélites do cromossomo X em casos de paternidade forense (duos, trios e
3 OBJETIVOS
3.1 Objetivo Geral
Desenhar, otimizar e caracterizar geneticamente um sistema para co-amplificação de polimorfismos do tipo indels ao longo do cromossomo X com a finalidade de aplicação em genética forense.
3.2 Objetivos Específicos
• Identificar indels polimórficas ao longo do cromossomo X utilizando o banco de dados dbSNP;
• Desenhar iniciadores para amplificação de polimorfismos do tipo indel no cromossomo X de tamanho de 70-170 pb;
• Padronizar protocolo para amplificação de indels do cromossomo X em sistemas multiplex;
• Estabelecer a freqüência de indels do cromossomo X em uma amostra da população brasileira;
• Analisar grau de desequilíbrio de ligação entre as indels estudadas;
• Analisar funcionamento e precisão do multiplex em casos de paternidade (trios) e post mortem e comparar os resultados obtidos da análise com as mini-indels do X, com STRs autossômicos e STRs do X;
• Testar a sensibilidade do multiplex em relação à quantidade de DNA;
• Amplificar as amostras provenientes de estudos de degradação;
4 REVISÃO DA LITERATURA
4.1 Aspectos Históricos da Genética Forense
A aplicação da genética em um contexto forense tem suas origens há mais de cem anos, com a descoberta dos polimorfismos do grupo ABO por Karl Landsteiner (LANDSTEINER, 1900; , 1901). A identificação dos padrões de herança do sistema ABO permitiu sua aplicação para a resolução de crimes (JOBLING et al., 2004). Embora seja uma ferramenta rápida, os polimorfismos do grupo ABO apresentam baixa variabilidade, o que os tornam pouco informativos. Existem quatro grupos possíveis: A, B, AB e O, sendo que 40% da população apresentam o tipo “O”. Logo, essa metodologia permite apenas a exclusão de um indivíduo como sendo o doador de uma determinada evidência biológica e não a sua inclusão. Além do sistema ABO, sistemas baseados em polimorfismos sorológicos de outros grupos sanguíneos e em polimorfismos eletroforéticos de proteínas oferecem potencial aplicação em genética forense, embora a baixa informatividade e a rápida degradação das proteínas limitou a ampla utilização dos mesmos (revisado por JOBLING et al., 2004).
Em 1985, este cenário experimentou uma grande revolução em função dos trabalhos do geneticista inglês, Sir Alec Jeffreys, na Universidade de Leicester, UK. Sir Jeffreys descobriu regiões no DNA que se repetiam muitas vezes lado a lado. Além disso, percebeu que o número de repetições presentes numa amostra variava de indivíduo para indivíduo. A estas regiões hipervariáveis foi dado o nome de minissatélites ou VNTRs (do inglês, variable number of tandem repeat). Após a descoberta, Sir Jeffreys demonstrou a aplicabilidade dos minissatélites para a identificação humana, sendo então cunhado o termo “DNA fingerprinting” (GILL et al., 1985; JEFFREYS et al., 1985a; JEFFREYS et al., 1985b; 1985c).
adjacências de várias regiões genômicas que apresentam blocos de DNA repetidos em números variáveis na população. Assim, utilizando uma sonda de DNA em um procedimento de Southern Blotting, um padrão de múltiplas bandas, específico de cada indivíduo podia ser determinado. Esta sonda era definida como sendo multilocal (MLPs, do inglês, multilocal probes). A vantagem apresentada era a alta variabilidade entre indivíduos. A probabilidade de coincidência do padrão multi-bandas entre indivíduos não aparentadas era estimada a 3 X 10-11 e com duas sondas multilocais este valor aumentaria para 5 X 10-19 (revisado por TAMAKI et al., 2005). Entretanto, esta metodologia se mostrava trabalhosa e demorada, além das análises em misturas terem se mostrado um desafio (BUTLER, 2005).
Essa metodologia foi utilizada pela primeira vez em 1985 em um caso de imigração na Inglaterra, onde o envolvido era um jovem de Gana. Preso sob alegações de possuir um passaporte falso, o jovem e sua família foram submetidos aos testes de “DNA fingerprinting” (Figura 1 - A) permitindo assim a verificação da real identidade do jovem. Esse foi o primeiro caso a ser resolvido com a utilização do DNA (JEFFREYS et al., 1985a; JEFFREYS, 2005).
A utilização das sondas multilocais persistiu por algum período, principalmente no estabelecimento de vínculo genético em casos de paternidade. Com a clonagem e caracterização de vários minissatélites, estes começaram a ser investigados individualmente. Neste caso, uma sonda unilocal (SLP, do inglês single
em Cadeia da Polimerase (PCR, do inglês polymerase chain reaction) (JOBLING et al., 2004).
O desenvolvimento da metodologia de PCR em 1985 (SAIKI et al., 1985; MULLIS et al., 1987) revolucionou a tecnologia do DNA e hoje constitui a base para qualquer análise por DNA em genética forense. A PCR consiste em um processo enzimático onde uma seqüência específica de DNA é replicada milhares de vezes (SAIKI et al., 1985; SAIKI, 1988). A sensibilidade e a rapidez permitiram a rápida incorporação da PCR como técnica básica para estudo de amostras forense, onde não é incomum evidências com baixa concentração de DNA. (BUTLER, 2005). O impacto da PCR foi tão grande que em 1993 o inventor Kary B. Mullis recebeu o Prêmio Nobel em Química - menos de 10 anos após a sua descoberta (http://nobelprize.org/nobel_prizes/chemistry/laureates/1993/press.html).
Os primeiros sistemas baseados em PCR tinham como alvo um pequeno número de SNPs (do inglês single nucleotide polymorphisms) no gene HLA-DQA1 (WESTWOOD et al., 1990). Embora esse sistema fosse útil quando a tecnologia de SLPs falhava, o poder de discriminação era muito baixo e com difícil interpretação de misturas. Vários sistemas para estudo de minissatélites baseados na PCR também foram desenvolvidos (revisado por JOBLING et al., 2004; TAMAKI et al., 2005).
Figura 1 – Primeiras utilizações em casos de imigração e criminal. A) A primeira aplicação de DNA fingerprinting – caso de imigração. Duas sondas MLPs para detectar diferentes tipos de minissatélites foram usadas na mãe (M), seus três filhos não envolvidos no caso (U) e seu filho, suposto portador de um passaporte falso (B). O pai não estava presente e o (X) corresponde ao Sir Jeffreys. Todas as bandas presentes em B podem ser vistas em M ou no caracter paternal visualizado em um ou mais Us. B) A primeira utilização de perfis genéticos em um caso criminal - caso Colin Pitchfork. Uma sonda do tipo SLP foi usada para analisar os seguintes DNAs: A, raízes de cabelo da primeira vítima coletadas post-mortem; B, mistura de sêmen e líquido vaginal da primeira vítima; C, sangue da segunda vítima, coletado post-mortem; D, swab vaginal da segunda vítima; E, mancha de sêmen presente na roupa da segunda vítima; S, sangue do primeiro suspeito. Os alelos do sêmen (indicados por setas), não atribuídos às vítimas, aparecem nos dois perfis provenientes dos estupros e não coincide com o perfil do suspeito.
4.2 Marcadores polimórficos de uso corrente em genética forense
No final dos anos 80 os microssatélites, STRs (do inglês short tandem
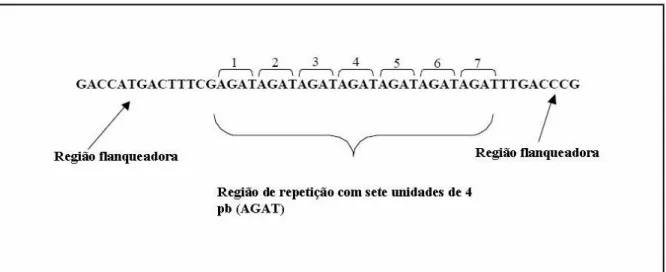
repeat) ou SSR (do inglês, simple sequence repeats) foram descobertos (LITT et al., 1989; WEBER et al., 1989). Assim como os minissatélites, os microssatélites consistem em blocos de seqüência de DNA repetidas consecutivamente. Diferentemente dos minissatélites que apresentam blocos com 70-200 bp, os microssatélites são caracterizados por de 2-6 pares de base (pb), repetidos tipicamente de 5-30 vezes (LITT et al., 1989; WEBER et al., 1989) (Figura 2). Os STRs são encontrados em genomas eucariotos e estão amplamente distribuídos no genoma humano, constituindo cerca de 3% do mesmo, ou seja, aproximadamente um STR a cada 2 kb (LANDER et al., 2001). As seqüências de repetição dos STRs são nomeadas pelo comprimento da unidade de repetição, logo existem, mono-, di-, tri- tetra-, penta- e hexanucleotídeos (TAUTZ, 1993). Devido às características inerentes aos microssatélites com bloco de repetição menor que quatro nucleotídeos, a Sociedade Internacional de Genética Forense (ISFG, do inglês International Society of Forensic Genetics) recomenda a utilização de tetranuleotídeos devido a estes possuírem um grau de polimorfimo razoável, baixa taxa de mutação e a formação reduzida de stutter (comparada aos dinucleotídeos) (BÄR, 1994; BÄR et al., 1997).
alélica e medida de variabilidade entre laboratórios, embora dois kits comerciais incluam o loco complexo hipervariável SE33, conhecido também como ACTBP2 (BUTLER, 2005).
Figura 2 - Marcador STR. Ilustração mostrando a unidade de repetição AGAT do marcador D5S818. O número de repetições AGAT varia entre indivíduos.
Fonte - a autora.
Existem situações onde um alelo de um loco STR contém um número incompleto de repetições. Microvariantes é o termo dado aos alelos que contém unidades de repetição incompleta. Um exemplo comum de uma microvariante é o alelo 9.3 no loco TH01, que contém nove repetições tetranucleotídicas e uma repetição incompleta de três nucleotídeos, pois na sétima repetição falta uma única adenina fora da normal na unidade de repetição AATG (PUERS et al., 1993).
sistemas comerciais para co-amplificação de até 15 microssatélites altamente polimórficos.
Figura 3 - Reação de Multiplex. Amplificações simultâneas de múltiplas regiões de DNA, envolvendo mais de um par de iniciador. Cada iniciador é desenhado para amplificar diferentes locos.
Fonte - a autora.
Figura 4 –Locosdo sistema Codis com suas localizações cromossômicas.
Fonte - http://www.cstl.nist.gov/div831/strbase/fbicore.htm (acessado em 01/2007).
O uso de marcadores adicionais geralmente é utilizado para obter maiores informações sobre determinadas amostras. A grande maioria dos polimorfismos de DNA pode ser dividida em dois grupos: os baseados em substituições nucleotídicas (SNPs, do inglês single nucleotide polymorphism) e os baseados em inserções-deleções de um ou mais nucleotídeos (conhecidos como indels e STRs)(BUTLER, 2005).
10-15 milhões de SNPs com alelo menos freqüente acima de 5% e aproximadamente 3 milhões com alelo menos freqüente entre 30-40% (KRUGLYAK et al., 2001).
O interesse pelos SNPs na área de genética forense é crescente. Esse interesse é devido a um número de características que os tornam muito apropriados para os estudos forenses: primeiramente, a baixa taxa de mutação (interessante para testes de paternidade); segundo, eles são adequados para análises utilizando alta tecnologia e automação; principalmente porque é possível conseguir um amplicon com tamanho reduzido e pela abundância dos mesmos (KWOK et al., 1999; SOBRINO et al., 2005; KIDD et al., 2006). A grande vantagem da utilização dos SNPs é que o tamanho do produto de amplificação pela PCR pode ser a princípio, tão grande quanto os pares de iniciadores específicos (aproximadamente 50 pb). Comparado aos 300 pb necessários para estudo de um STR, os SNPs tornam-se ferramentas de alto interesse para a análise de materiais extremamente degradados (JOBLING et al., 2004).
Entretanto, os SNPs apresentam algumas limitações. Primeiro o número de SNPs necessários é quatro vezes maior do que o número de STRs (CHAKRABORTY et al., 1999; GILL, 2001b). Assim, aproximadamente 60 SNPs serão necessários para se obter um poder de discriminação equivalente aquele obtido com os sistemas multiplex de STRs em uso na área forense (GILL et al., 2004). Segundo, as vantagens de custo ainda não estão claras. A despeito das vantagens e desvantagens da aplicação de SNPs em genética forense, os testes de paternidade ainda podem ser realizados de maneira mais simples e a custo mais baixo utilizando os STRs, sem falar na experiência em STRs ao longo dos últimos 10 anos(AMORIM et al., 2005).
Dificilmente os SNPs substituirão os STRs, pelo menos pelos próximos anos, devido ao grande esforço feito mundialmente para o estabelecimento de banco de dados de perfis de criminosos, onde provavelmente não serão substituídos por outros marcadores sem considerável custo econômico. Entretanto os SNPS constituem uma excelente ferramenta que pode contribuir nas resoluções de casos mais complicados. (GILL, 2001b; BIESECKER et al., 2005).
incorporadas ao arsenal das ferramentas moleculares utilizadas em genética forense (WEBER et al., 2002; BUTLER, 2005). As indels bialélicas variam muito na diferença de comprimento dos alelos, entretanto o maior grupo de indels bialélicas são aquelas que a diferença entre o comprimento do alelo é de apenas alguns nucleotídeos (WEBER et al., 2002) Os dois alelos das indels bialélicas podem ser classificados como curto e longo (BUTLER, 2005). As indels bialélicas tornam-se interessantes para a genética forense quando levado em consideração o tamanho de seus produtos, que podem ser menores do que 100 pb, o que as torna potencialmente co-amplificáveis a um nível maior do que os STRs (WEBER et al., 2002). Em 2002, Weber et al. publicaram um estudo onde relatavam as propriedades básicas das indels bialélicas em humanos através da análise de 2000 polimorfismos (WEBER et al., 2002). Um conjunto de 40 indels selecionadas a partir das 2000 investigadas por Weber et al. foram utilizadas em um estudo populacional (BASTOS-RODRIGUES et al., 2006) envolvendo amostras do painel mundial de diversidade genética humana (HGDP, do inglês, human genetic diversity panel) (CANN et al., 2002)
Em suma, a abundância das indels bialélicas e a facilidade para sua análise tornam-nas de grande interesse na área forense onde ainda não foi publicado nenhum trabalho com a utilização das mesmas.
4.3 Marcadores de linhagem em genética forense
4.3.1 Cromossomo Y
maneiras diferentes. Sendo assim, o cromossomo Y passa a ser responsável pela determinação do sexo (LAHN et al., 2000).
O Y é terceiro menor cromossomo em humanos, com quase 60 Mb(HARRIS et al., 1986), apenas um pouco maior que o cromossomo 21 (47 Mb) e que o cromossomo 22 (49 Mb)(BUTLER, 2005). Aproximadamente 95% de sua seqüência é denominada de NRY (do inglês, non-recombining region), ou região sem recombinação devido ao fato de não sofrer recombinação e estar presente apenas no sexo masculino. A região NRY é repleta de elementos de repetição e pode ser dividida em três: região eucromática, centromérica e heterocromática (FOOTE et al., 1992; TILFORD et al., 2001). A região eucromática é constituída por 24 Mb e a região heterochromática (Yqh) por 30 Mb (FOOTE et al., 1992). As duas pontas do cromossomo Y, conhecidas como regiões pseudo-autossomais (PAR, do inglês pseudo-autosomal regions), recombinam com as regiões homólogas do cromossomo X. A região PAR1 encontra-se na extremidade do braço curto (Yp) do cromossomo Y e possui 2,5 MB, enquanto a região PAR2 que se localiza na extremidade do braço longo (Yq) possui menos de 1 Mb (BUTLER, 2005). Em 2003, Skaletsky et al. renomearam a região NRY de MSY (do inglês, male specific region) devido a evidência da conversão gênica freqüente ou recombinação intracromossomal (SKALETSKY et al., 2003).
linhagem patrilinear. Essas características tornam o cromossomo Y uma ferramenta útil para rastrear a evolução humana através das linhagens masculinas, assim como para aplicação em diversas situações forenses (SANTOS et al., 1993; JOBLING et al., 1995), incluindo aquelas envolvendo evidências de abuso sexual que contenham mistura de DNA masculino e feminino (PRINZ, MECHTHILD et al., 1997; PRINZ et al., 2001).
Em 1992, Roewer et al. descreveram o primeiro marcador polimórfico do tipo microssatélite no cromossomo Y, o Y-27H39, hoje conhecido como o loco DYS19 (ROEWER et al., 1992). Nos 10 anos seguintes, a descoberta de marcadores polimórficos no cromossomo Y não foi tão veloz quanto à caracterização de polimorfismos autossômicos. No início de 2002 existiam apenas 30 marcadores do cromossomo Y disponíveis aos cientistas. Atualmente existem cerca de 200 microssatélites caracterizados no cromossomo Y, assim como em torno de 250 polimorfismos binários (PRINZ, MECHTHILD et al., 1997; BUTLER, 2003; SCHOSKE et al., 2003; KAYSER et al., 2004; BUTLER, 2005).
No ano de 1997, a comunidade forense européia selecionou um grupo de 9 marcadores do cromossomo Y para formar um painel denominado de haplótipo mínimo (KNIJFF et al., 1997; CAGLIÀ et al., 1998; SCHNEIDER et al., 1999). Atualmente em genética forense os microssatélites mais usados são os constituintes do haplótipo mínimo, que incluem: DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393 e DYS385a/b. O haplótipo estendido inclui todos esses marcadores do haplótipo mínimo mais a repetição dinucleotídica altamente polimórfica, YCAII a/b (KNIJFF et al., 1997; ROEWER et al., 2001) (Figura 5).
estendido, além de Y-STRs adicionais, oferecendo um aumento do poder de discriminação (BUTLER et al., 2002; SCHOSKE, 2003). Além desse 20plex, o grupo de Hanson, em 2004, desenvolveu um “megaplex” do cromossomo Y, capaz de amplificar simultaneamente 21 locos (HANSON, 2004).
O maior e mais pesquisado banco de dados de Y-STR, é o criado por Roewer et al. na Humbolt University em Berlim (WILLUWEIT, ; ROEWER et al., 2001). Este banco criado em 2000 é provido de informações provenientes de 89 instituições colaborativas dispersas em 36 países diferentes (BUTLER, 2005). As informações desse banco de dados baseiam-se no haplótipo mínimo e podem ser acessadas através dos sites: http://www.ystr.org ou http://www.yhrd.org. Outros bancos de dados de Y-STR também estão disponíveis na rede mundial de computadores como: http://www.promega.com (12 locos do PowerPlex® Y System) e
http://www.appliedbiosystems.com (17 locos do AmpAmpF STR® Y-filer™ PCR Amplification Kit) entre outros (BUTLER, 2005).
Figura 5 -Cromossomo Y humano. A região NRY que constitui 95% do cromossomo Y é dividida em três: eucromatina, centrômero e heterocromatina. Os outros 5% do cromossomo Y compreendem as regiões que sofrem recombinação, ou seja, a PAR1 e a PAR2 (em laranja). Em azul, estão indicados os locos constituintes do haplótipo mínimo e em preto, outros locos caracterizados no cromossomo Y.
Fonte - www.cstl.nist.gov(figura modificada com base em acesso em janeiro de 2007).
4.3.2 DNA mitocondrial
próprio genoma, muitos genes codificadores de proteínas mitocondriais são encontrados no núcleo celular (TAANMAN, 1999; SCHEFFLER, 2000b).
O genoma mitocondrial humano é organizado em uma fita dupla de DNA circular, com 16569 pb, sendo altamente conservado (ANDERSON et al., 1981). Existe uma média de 4-5 cópias de moléculas de mtDNA por mitocôndria, podendo variar de 1-15 (SATOH et al., 1991) e o número de mitocôndrias é variável de acordo com o tipo celular, podendo chegar a mais de 1000 organelas por célula (ROBIN et al., 1988).
a região hipervariável I (HVI) e a região hipervariável II (HVII), localizadas entre os nucleotídeos 16240-16383 e 73-340, respectivamente.
Figura 6 - Mapa do genoma mitocondrial humano. Os genes codificados pelo DNA mitocondrial (mtDNA) estão indicados por grupos funcionais. As mutações pontuais associadas com doenças mitocondriais são indicadas no centro do genoma. A região não codificante do D-loop está localizada no topo da figura e a posição do nucleotídeo 1 indicada na posição de meio-dia.
4.3.3 Marcadores polimórficos no cromossomo X
Conforme dito anteriormente, os cromossomos sexuais originaram-se de cromossomos autossômicos entre 300 e 350 milhões de anos atrás, logo após a separação de aves e mamíferos (CHOW et al., 2005; BACHTROG, 2006). Os cromossomos X e Y após adquirirem o papel de determinação sexual, pararam de recombinar um com o outro, o que levou o cromossomo Y a se degenerar enquanto o cromossomo X manteve a maioria de seus genes ancestrais (BACHTROG, 2006).
O cromossomo X humano tem aproximadamente 155 Mb e contém 1098 genes (ROSS et al., 2005). Logo, o cromossomo X é um dos maiores cromossomos humanos, mas possui uma densidade gênica um pouco menor do que os cromossomos autossômicos. A baixa densidade gênica é acompanhada por um excesso de elementos repetitivos quando comparado ao resto do genoma (CHOW et al., 2005). Além disso, o cromossomo X possui duas características que o distingue dos cromossomos autossômicos: os machos só carregam um cromossomo X, sendo hemizigotos; cromossomo X passa 2/3 do tempo nas fêmeas e apenas 1/3 do tempo nos machos.
A inativação do X, primeiramente descrito por Lyon em 1961 (LYON, 1961), resulta no silenciamento de um dos cromossomos X da fêmea, a fim de atingir uma equivalência de dosagem com os machos que possuem apenas um cromossomo X e um cromossomo Y, que determina o sexo. Bem no início do desenvolvimento das fêmeas de mamíferos, um dos cromossomos X de cada célula torna-se inativado. O cromossomo X inativado torna-se altamente condensado e é visível como um ponto de coloração escura, chamado corpúsculo de Barr. Esta inativação cromossômica persiste por todas as multiplicações mitóticas subseqüentes que produzem o corpo adulto do animal. O processo de inativação é aleatório, afetando um ou outro cromossomo X. Como resultado desta inativação, o corpo da fêmea adulta é uma mistura, ou mosaico, de células com um ou outro genótipo dos dois cromossomos X diferentes (CHOW et al., 2005).
cromossomo X está na sua fase inicial (EDELMANN et al., 2001; SZIBOR et al., 2003). A idéia do uso de marcadores do cromossomo X para auxiliar a identificação humana é relativamente nova, originando-se em meados da década de 90 e início desta década. Portanto a quantidade de informação disponível sobre o tema ainda é escassa quando comparada com a disponibilidade de informação sobre marcadores autossômicos e do cromossomo Y. Contudo, o cromossomo X tem recebido a atenção de alguns grupos de pesquisadores com o intuito de reverter esse quadro, como mostra a página de internet http://www.chrx-str.org, disponibilizada recentemente (SZIBOR et al., 2006).
Os marcadores do cromossomo X (Figura 7) servem como eficiente complemento nas análises realizadas com marcadores autossômicos e do cromossomo Y, especialmente em casos complexos de parentesco. Esses casos complexos de parentesco foram a grande motivação do aumento de estudos de cromossomo X para sua aplicação em genética forense.
Em contraste com o cromossomo Y, que não sofre recombinação durante a meiose, os marcadores autossômicos e do cromossomo X necessitam de ter sua localização genética determinada precisamente, quando utilizados em testes de parentesco. A determinação da localização dos marcadores do cromossomo X é essencial, pois é a partir desta que se poderá saber a possibilidade de análise de seus marcadores, de forma independente ou de forma dependente (POWLEY, 2002; SZIBOR et al., 2006). Quando os locos apresentam-se independentes uns dos outros, o índice de paternidade pode ser calculado para cada loco e depois combinado por multiplicação dos mesmos (POWLEY, 2002; SZIBOR et al., 2006).
genético gerado, impossibilitando a configuração exata do haplótipo ocorrido realmente (POWLEY, 2002; SZIBOR et al., 2006).
Figura 7 - Cromossomo X humano e seus principais marcadores STRs. Os marcadores DXS6801, DXS6809, e DXS6789, quando analisados conjuntamente constituem um haplótipo. O mesmo acontece quando analisamos os marcadores DXS7424 e o DXS101 em conjunto.
Em testes de paternidade onde não se tem acesso ao material genético do suposto pai, a utilização de microssatélites autossômicos passa pela reconstrução do genótipo do suposto pai utilizando informação de parentes (reconstrução de paternidade) ou pelo teste de relação deste parentesco entre um parente do suposto pai com o filho em questão (teste de Kinship). Em ambos os casos, há uma perda considerável no poder estatístico de se afirmar que o cenário obtido favorece a hipótese de paternidade ou a hipótese de não paternidade. Em situações onde a criança é do sexo masculino, a utilização de marcadores do cromossomo Y é uma ferramenta importante na resolução de casos com suposto pai ausente (SANTOS et al., 1993; GILL et al., 2001; JOBLING et al., 2003; GUSMAO et al., 2006). Quando a criança é do sexo feminino, torna-se interessante e eficiente a utilização de marcadores do cromossomo X, já que os homens são hemizigotos para o cromossomo X, ou seja, carregam apenas um cromossomo X, portanto: a genotipagem do cromossomo X em homens automaticamente revela seu haplótipo; o homem transmite seu cromossomo X inteiro para sua filha, logo o haplótipo paterno do cromossomo X pode ser detectado em suas filhas; todas as irmãs compartilham o haplótipo do cromossomo X paterno; os alelos do cromossomo X não compartilhado por irmãs são obrigatoriamente de origem materna; a genotipagem de duas ou mais irmãs revela o haplótipo do cromossomo X paterno e grande parte do genótipo materno do cromossomo X; a genotipagem de dois ou mais irmãos provê grande parte do genótipo materno do cromossomo X; além disso, é muito provável que haplótipos de grupos de ligação permaneçam estáveis através de muitas gerações, tornando-se um eficaz meio para demonstrar parentesco (SZIBOR et al., 2003).
Além dos casos de paternidade com o suposto pai ausente, a utilização dos marcadores do cromossomo X torna-se mais informativa do que os marcadores autossômicos ou do que microssatélites do cromossomo Y em casos de parentesco onde se tenha somente parentes distantes para serem testados e em casos de maternidade (EDELMANN et al., 1999; BINI et al., 2005).
os marcadores autossômicos com valores de PIC (do inglês, Polymorphism
Information Content) similares. Kishida et al (KISHIDA et al., 1997) modificaram a fórmula original proposta por Krüger et al para a aplicação em marcadores do cormossomo X. Essa modificação levou a um aumento do valor do MEC, uma vez que os homens são diplóides para todos os cromossomos autossômicos e hemizigotos para o cromossomo X (EDELMANN et al., 2001; POWLEY, 2002; SZIBOR et al., 2003) .
Espera-se que a demanda para testes de parentesco, onde apenas parentes distantes estejam disponíveis para teste, cresça, particularmente a partir do desejo de reunir famílias nos contextos de guerra e de migração. Além disso, a identificação de corpos e ossadas de vítimas de guerra ou de desastres de massa necessita de uma combinação de métodos sofisticados. Nesse contexto, deve-se chamar a atenção de que algumas vezes os marcadores do cromossomo X são mais eficientes do que os cromossomos autossômicos, especialmente em casos que envolvam a paternidade de uma mulher ou a maternidade de um homem. Isso é refletido pelas fórmulas usadas para calcular a chance média de exclusão (MEC) para marcadores autossômicos e marcadores do cromossomo X. Se os marcadores possuem um conteúdo de informação de polimorfismo (PIC) comparável, o MEC para o cromossomo X é maior do que para os marcadores autossômicos (SZIBOR, 2007). A opção de utilização de produtos amplificados de tamanho menor, gerados a partir de marcadores do cromossomo X devem ser considerados, especialmente se restos de ossadas humanas estiverem sendo analisadas, ou outras amostras de difícil análise (ASAMURA et al., 2006a).
Em vários países, os geneticistas forenses seguem o princípio de que um teste de DNA forense não deve revelar doenças ou predisposições genéticas. A análise de marcadores do cromossomo Y e do mtDNA podem revelar informações sobre a origem étnica do indivíduo. Entretanto essa informação não é considerada uma intromissão na privacidade do indivíduo. A princípio, o mesmo se aplica na determinação de sexo com os cromossomos X e Y. Contudo, aberrações cromossômicas como a Sídrome de Klinefelter podem ser descobertas quando marcadores do cromossomo X são usados. Essa doença está relacionada ao cariótipo 47XXY e pode ser reconhecida pela detecção de dois cromossomos X em um indivíduo do sexo masculino. Já a Síndrome de Ulrich-Turner, onde o cariótipo é de 45, X, o indivíduo do sexo feminino apresenta apenas um cromossomo X, plenamente funcional, também pode ser diagnosticada pela análise de marcadores do cromossomo X.
Desde 1998, o marcador HumARA descoberto por Desmarais et al. (DESMARAIS et al., 1998)vem sendo utilizado como marcador do cromossomo X. Todavia, desde o início das análises com esse marcador, sabia-se que a repetição CAG do HumARA está localizada em uma região codificante do gene receptor de andrógeno, éxon 1, codificando para um trato poliglutamínico. Em1991, La Spada et a (LA SPADA et al., 1991) provou que uma mutação nesse loco acarretaria no desenvolvimento de Atrofia Muscular Bulbo Espinhal (AMBE), que ocorre quando a repetição trinucleotídica ultrapassa de 43. Além da AMBE, a análise do marcador HumARA pode detectar outros riscos a saúde. Por esse motivos, o marcador HumARA não tem sido usado por diversos geneticistas para análises forenses, embasados na consideração ética citada acima (SZIBOR, 2007) .
4.4 Amostras com quantidades mínimas de DNA e/ou degradadas em Genética Forense
evidências biológicas coletadas na cena de crime, o cientista forense pode deparar-se com cenários onde obter um perfil de DNA torna-deparar-se um enorme desafio, pois amostras com quantidades mínimas e/ou degradadas não são incomuns. Especificamente no que diz respeito à quantidade de DNA, estima-se que sejam necessárias pelo menos 60 cópias de DNA genômico (200 pg) para sua análise (SUN et al., 2005). Com o intuito de maximizar a chance de se obter um perfil de amostras em que a quantidade de DNA esteja abaixo de 100 picogramas, alterações nos protocolos de amplificação são sugeridas. Estas alterações são tratadas dentro do conceito de LCN (do inglês, low copy number) (GILL, 2001b)onde existe um aumento no número de ciclos na reação de PCR de 28-30 para 32-34 ciclos. Geralmente os fabricantes de kits multiplex recomendam 250 pg como o limite mais baixo de sensibilidade. A quantidade ótima de DNA recomendada nos sistemas comerciais disponíveis para co-amplficação de microssatélites é 1 ng de DNA com o número de ciclos de PCR variando entre 28-30 ciclos(GILL, 2001a). Logo, os cientistas forenses, buscaram uma metodologia simples para acrescentar sensibilidade às metodologias padrões, aumentando o número de ciclos na PCR (GILL et al., 2000; WHITAKER et al., 2001). Com uma quantidade de DNA menor do que 100 pg, não há vantagem em acrescer o número de ciclos para mais de 34, pois um aumento maior do que 34 ciclos induz a ampliação da presença de artefatos e o perfil torna-se altamente desbalanceado. Para uma maior confiabilidade nas reações de LCN-DNA, sugere-se que as amplificações sejam repetidas duas ou mais vezes e comparadas a fim de gerar um perfil alélico mais confiável (GILL et al., 2000; GILL, 2001a).
identificação de um homozigoto como heterozigoto (GILL et al., 2000; GILL, 2001a). Deve ficar claro na apresentação dos resultados, que as amplificações de LCN-DNA não podem ser vistas como as mesmas amplificações de uma amostra de DNA dentro das condições padrões.
A necessidade de amplificar seqüências de DNA a partir de células únicas, principalmente no âmbito do diagnóstico em células cancerosas e em células embrionárias, levou ao desenvolvimento de uma série de métodos que amplificam o número de cópias de um determinado genoma. Estas técnicas são conhecidas por WGA (do inglês, Whole Genome Amplification) e consistem na amplificação do genoma total, anterior a uma amplificação pela PCR loco-específica (CHEUNG et al., 1996; WELLS et al., 1999; HOSONO et al., 2003; LASKEN et al., 2003; SCHNEIDER et al., 2004; BERGEN et al., 2005). Em 1992 os dois primeiros métodos de WGA foram publicados, o DOP-PCR (do inglês Degenerate Oligonucleotide Primed PCR) (TELENIUS et al., 1992a; TELENIUS et al., 1992b)e o PEP (do inglês Iniciador Extension Preamplification)(ZHANG et al., 1992; HUGHES et al., 2005). Para aumentar a eficiência do PCR e o rendimento do DNA, foram feitas algumas modificações nessas metodologias levando ao aparecimento do LL-DOP-PCR (do inglês Long products from low quantities DOP-PCR) (KITTLER et al., 2002)e do Improved PEP (I-PEP) (DIETMAIER et al., 1999). Entretanto, nenhuma das metodologias pretende amplificar o DNA em sua totalidade ou dão uma cobertura completa de determinado loco (GILL, 2001a; HUGHES et al., 2005). Ao contrário, estes protocolos produzem uma fonte amplificada para genotipagem ou identificação de marcador de algum tipo, além de serem tendenciosos quanto à modificação da informação contida no DNA(SCHNEIDER et al., 2004). Por essas razões, um novo método para WGA foi introduzido nos últimos anos e tem despertado grande expectativa. Este método é baseado na SDA (do inglês Strand
continua com o processo de amplificação. Novos hexâmeros irão se ligar à fita que foi deslocada, e iniciarão novos sítios de replicação levando praticamente a uma reação exponencial. Além disso, a polimerase 29 tem um aumento na fidelidade da replicação de 10 vezes em relação à Taq polimerase (HUGHES et al., 2005). A taxa de erro da 29 DNA polimerase é de um em 106 em contraste aos três em 104 ou 1.6 em 1 milhão da Taq polimerase (DEAN et al., 2002; HUGHES et al., 2005).O produto de MDA é favorecido quando comparado ao produto de ouras técnicas de WGA, levando em consideração a amplificação não tendenciosa, a reprodutibilidade, a preservação seqüência e a boa cobertura do genoma (HELLANI et al., 2004; HUGHES et al., 2005).
5 MATERIAIS E MÉTODOS
5.1 Seleção das indels
As indels foram selecionadas a partir do NCBI® (do inglês, National Center
for Biotechnology Information) (www.ncbi.nlm.nih.gov) no banco dbSNP® (www.ncbi.nlm.nih.gov/SNP ) (SHERRY et al., 1999; SHERRY et al., 2001) utilizando os seguintes critérios durante a busca: X[chr] "in del"[snpclass] "human"[orgn] 1[weight] "by frequency"[validation] “Minimum frequency allele ranging from” 0,5 to 0,3. Para as indels recuperadas do banco de dados, um novo passo de seleção foi aplicado, descartando aquelas em regiões de baixa complexidade, aquelas com heterozigozidade abaixo de 0,3 e ou constituídas de apenas um nucleotídeo. Também foram descartadas aquelas indels presentes em seqüências de elementos repetitivos. Além disso, verificou-se a presença de SNPs nas vizinhanças das indels. Utilizando freqüências alélicas disponíveis no banco de dados dbSNP foram calculados parâmetros básicos de informatividade das indels selecionadas.
O grau de polimorfismo foi quantificado usando o calculador de conteúdo de informação polimórfica, o PIC (do inglês, Polymorphic Information Content) baseado na seguinte fórmula(BOTSTEIN, 1980):
− −
=1 2 2 2 2
Pj Pi Pi
PIC
Onde: Pi e Pj = Frequência dos alelos i e j de um dado locus
A chance média de exclusão, MECT (do inglês, Mean of Exclusion Chance) para marcadores no cromossomo X em trios familiares (suposto pai, mãe e criança) com a criança do sexo feminino, foi computado segundo (KISHIDA T., 1997), utilizando a fórmula abaixo:
(
fi)
fi(
fi)
fifj(
fi fj)(
fi fj)
fi MEC j i i i
T = − + − + + − −
< 1
1
1 2
3
A chance média de exclusão em duos familiares (suposto pai e criança), MECD para marcadores no cromossomo X, com a criança do sexo feminino, foi
computado segundo (DESMARAIS et al., 1998), utilizando a fórmula abaixo:
3 2
2
1− +
= i i D fi fi MEC
Onde: fi = Frequência populacional do alelo i
A média do poder de discriminação em mulheres (PDM) foi calculada segundo (DESMARAIS et al., 1998), utilizando a seguinte fórmula:
(
)
+ −= i i
F
fi fi
PD 1 2 2 2 4
Onde fi = Frequência populacional do alelo i
E a média do poder de discriminação para marcadores no cromossomo X em homens (PDH), também foi calculada segundo (DESMARAIS et al., 1998), utilizando a fórmula:
− =
i M
fi
PD 1 2
Onde fi = Frequência populacional do alelo i
5.2 Desenho dos iniciadores