Caracterização de uma colecção antiga de trigos portugueses
através de marcadores morfológicos e moleculares
Dissertação de Mestrado em Genética Molecular Comparativa e Tecnológica
Filomena da Conceição Correia Morais
Universidade de Trás-os-Montes e Alto Douro Vila Real, 2010
Este trabalho foi elaborado no âmbito do projecto PTDC/AGR-GPL/65876/2006 "Estrutura genética e filogenias de trigos antigos portugueses: abordagem genómica e de citogenética molecular", coordenado cientificamente pelo Prof. Doutor José Eduardo Lima-Brito e financiado pela Fundação para a Ciência e a Tecnologia.
Agradecimentos
Gostaria de agradecer:
À UTAD, nomeadamente na pessoa do Magnífico Reitor desta Universidade, Professor Doutor Armando Mascarenhas Ferreira, pelo apoio logístico na realização deste trabalho.
Ao Professor Doutor José Eduardo Lima-Brito, por ter orientado este trabalho com empenho e dedicação.
À Doutora Ana Isabel, por todos os ensinamentos, todos os conselhos preciosos e pela franca amizade e ao João, companheiro de longas horas nesta aventura, pelo bom humor e por ser sempre o meu herói de todas as vezes que me esqueço das chaves dentro de casa!
À Professora Doutora Raquel Chaves, pelas aulas da Pós-Graduação que conseguem inspirar até os alunos mais desinteressados e pela confiança que depositou em mim. Ao Sr. Ilídio, por saber tão bem cuidar das minhas plantas!
Ao Sr. Alcídio, à D. Cecília, à D. Alexandrina, ao Sr. Francisco, à D. Marcília, à D. Isabel, ao Cláudio, ao Sr. António e à D. Teresa, por toda a ajuda prestada, pela disponibilidade e pela simpatia.
À “citomalta”, que me recebeu tão carinhosamente e me fez sentir em casa desde o primeiro dia. Ana paço, és uma inspiração! Ana Lu, continua sempre em frente. Su, mantém esse sorriso, é quase uma obrigação! Os pombinhos que sejam sempre muito felizes! Sara, és única. Ana Neta, mantém esse coração sempre derretido. E Karine, sê feliz. Mena és uma verdadeira mulher de sucesso, contas com a minha admiração. A todas as pessoas que trabalham no laboratório dos marcadores moleculares. Tenho impressão que não sabem como foram importantes para o êxito deste trabalho!
À Susana e ao Júlio, verdadeiros responsáveis pelo sucesso da caracterização morfológica dos trigos da colecção! Com humor, simpatia, empenho e eficácia nunca me deixaram desmoralizar face à enorme tarefa que tinha pela frente. Muito obrigada pela ajuda, mesmo!
Aos meus mais queridos e fieis amigos, sempre tolerantes, sempre pacientes, sempre lá quando são precisos. Obrigada!
Ao grupo da Capoeira, por toda a amizade e porque é sempre na capoeira que encontro o meu equilíbrio …
E, finalmente, à minha família, meus alicerces poderosos, meus pilares, meu abrigo em cada segundo da minha vida! AMO-VOS MUITO.
Resumo
O trigo é um dos cereais mais importantes para a alimentação e agricultura a nível mundial, por isso muitos países têm desenvolvido esforços para a caracterização e avaliação da diversidade genética das suas populações ou “landraces” deste cereal.
No IBB-CGB/UTAD tem-se vindo a caracterizar uma colecção antiga de trigos portugueses, composta por 48 cultivares de trigo mole (Triticum aestivum 2n = 6x = 42; AABBDD) e 51 cultivares de trigo rijo (T. turgidum e T. durum; 2n = 4x = 28; AABB), bem como 30 linhas da “landrace” de trigo mole ‘Barbela’.
Os objectivos deste trabalho eram caracterizar esta colecção de trigos com base em marcadores moleculares RAPD e ISSR e em componentes morfológicos e de produção.
Com os marcadores RAPD obteve-se 71,3% de polimorfismo nos trigos moles, 72,7% nos trigos rijos e 54,1% nas linhas de trigo ‘Barbela’. Também se obteve 78,6% de polimorfismo ISSR nos trigos moles, 81,3% nos trigos rijos e 55,7% nas linhas de trigo ‘Barbela’. Os ISSRs evidenciaram maior taxa de polimorfismo do que os RAPDs.
Neste trabalho foram construídos dendrogramas de similaridade genética, utilizando o método de agrupamento UPGMA e o coeficiente de Jaccard para trigos moles, rijos e linhas de ‘Barbela’ com a totalidade dos dados RAPD e com a totalidade dos dados ISSR. No geral, os dendrogramas agruparam os trigos moles por variedades botânicas, os trigos rijos por espécies e as linhas de trigo ‘Barbela’ por região de colheita, quer para RAPDs quer para ISSRs. A combinação dos dois sistemas de marcadores originou resultados similares.
No que concerne aos componentes morfológicos e de produção, estes evidenciaram uma elevada variabilidade genética entre cultivares e entre variedades botânicas. Os dendrogramas UPGMA de similaridade genética, utilizando o coeficiente da distância euclidiana confirmaram a elevada variabilidade genética anteriormente observada, uma vez que não foi possível agrupar os trigos por variedades botânicas, espécies ou regiões de colheita.
Na análise da diversidade genética os ISSRs revelaram ser mais informativos que os RAPDs, sendo os marcadores morfológicos e de produção os menos informativos.
Abstract
The wheat is one of the most important cereals for food and agriculture worldwide, so many countries have developed efforts to the characterization and evaluation of the genetic diversity of their populations or landraces of this cereal.
In IBB-CGB/UTAD we have been characterizing an old portuguese wheat collection, composed by 48 bread wheat cultivars (Triticum aestivum 2n = 6x = 42; AABBDD) and 51 durum wheat cultivars (T. turgidum e T. durum; 2n = 4x = 28; AABB), as well as 30 lines of the ‘Barbela’ bread wheat landrace.
The goals of this work were to characterize this wheat collection based on molecular markers RAPD and ISSR and based on morphological and yield traits.
With RAPD markers we got 71,3% of polymorphism in bread wheats, 72,7% in durum wheats and 54,1% in ‘Barbela’ lines. We also got 78,6% of ISSR polymorphism in bread wheats, 81,3% in durum wheats and 55,7% in ‘Barbela’ lines. The ISSR molecular markers showed higher polymorphism level then RAPD markers.
In this study, UPGMA dendrograms were built using the Jaccard coefficient to the bread wheat, durum wheat and ‘Barbela’ lines, with the RAPD data and with the ISSR data. In general, bread wheats were clustered by botanical varieties, durum wheats by species and ‘Barbela’ lines by sampling area, as for RAPDs as for ISSRs. The combination of the two marker sistems gave rise to similar results.
Morphological and yield traits showed a high genetic variability within cultivars and within botanical varieties. UPGMA dendrograms of genetic similarity, using the euclidian distance coefficient confirmed the high genetic variabitity, once wheats did not cluster by botanical varieties, species or sampling areas.
In the genetic diversity analysis, ISSRs were more informatives than RAPDs, and the morphological and yield markers were the less informatives.
Keywords: Portuguese wheat germplasm; ‘Barbela’; RAPDs; ISSRs; morphological
Índice
Agradecimentos --- i
Resumo --- ii
Abstract --- iii
Índice de Figuras --- iv
Índice de Quadros e Tabelas --- viii
Lista de Abreviaturas --- ix
1. Introdução --- 1
1.1. A Tribo Triticeae, Família Poaceae --- 2
1.2. História evolutiva do trigo --- 3
1.3. Conservação e manutenção do germoplasma --- 6
1.3.1. Conservação do germoplasma --- 6
1.3.2. Bancos de germoplasma --- 7
1.3.3. Manutenção do germoplasma --- 9
1.4. Diversidade genética e o melhoramento do germoplasma--- 11
1.5. Aplicação da biotecnologia no melhoramento do trigo --- 12
1.6. Germoplasma de trigo português --- 15
1.7. Marcadores genéticos --- 16
1.7.1. Marcadores morfológicos --- 17
1.7.2. Marcadores moleculares --- 18
1.7.2.1. Marcadores bioquímicos --- 18
1.7.2.2. Marcadores de DNA --- 19
1.7.2.2.1. Baseados em hibridação de DNA --- 20
1.7.2.2.1.1. RFLPs (“Restriction Fragment Lenght Polymorphism”) --- 20
1.7.2.2.2. Baseados em PCR --- 21
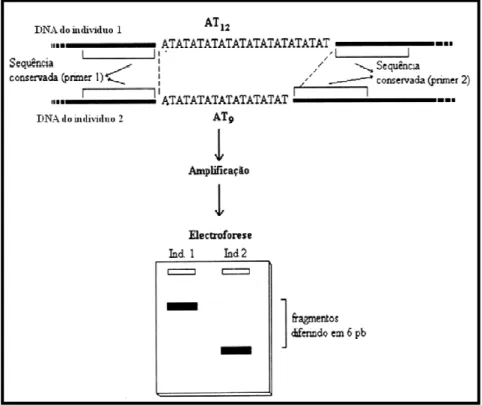
1.7.2.2.2.1. RAPDs (“Random Amplified Polymorphic DNA”) --- 21
1.7.2.2.2.2. AFLPs (“Amplified Fragment Lenght Polymorphism”)--- 25
1.7.2.2.2.3. SSRs (“Simple Sequence Repeat”) ou microssatélites --- 27
1.7.2.2.2.4. ISSRs (“Inter Simple Sequence Repeats”) ou inter-microssatélites -- 28
1.7.2.2.2.5 – Retrotransposões (RTNs) como marcadores moleculares --- 31
2. Objectivos --- 33
3. Material e Métodos --- 34
3.1. Material Vegetal --- 34
3.2. Métodos --- 38
3.2.1. Germinação, crescimento e desenvolvimento do material vegetal --- 38
3.2.2. Colheita de folhas jovens para extracção de DNA genómico --- 39
3.2.3. Extracção de DNA genómico --- 39
3.2.3.1. Extracção de DNA genómico pelo método CTAB --- 39
3.2.3.2. Extracção de DNA genómico com o Kit “DNeasy Plant Mini Kit” (Qiagen) --- 40
3.2.3. Determinação da concentração, pureza e integridade do DNA genómico -- 40
3.2.4. Amplificação de RAPDs --- 41
3.2.4.1. Reacção de amplificação RAPD-PCR --- 41
3.2.5.2. Visualização dos produtos de amplificação ISSR --- 43
3.2.6. Análise estatística dos dados RAPD e ISSR --- 44
3.2.7. Caracterização morfológica e de produção das plantas adultas --- 44
4. Resultados e Discussão --- 46
4.1. RAPDs --- 46
4.2. ISSRs --- 52
4.3. Combinação de RAPDs e ISSRs --- 59
4.4. Caracterização morfológica e de produção --- 63
4.5. Comparação entre os marcadores moleculares e os componentes morfológicos e de produção --- 70
5. Conclusão --- 71
Bibliografia --- 72
Índice de Figuras
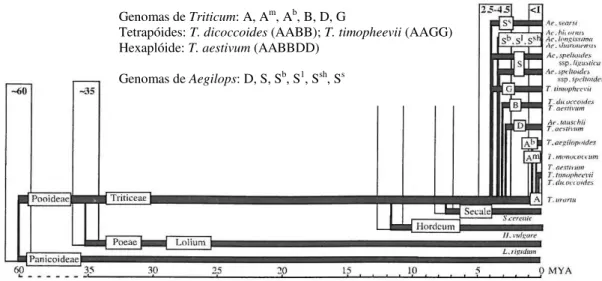
1. IntroduçãoFigura 1. 1 - Estimativa do tempo de evolução das linhas Triticum e Aegilops (adaptado de Huang et al.,
2006).---3
Figura 1. 2 - Etapas da técnica de RFLPs. 1) O dsDNA é fragmentado por enzimas de restrição e
separado por tamanho através de electroforese em gel. 2) Coloca-se o gel num suporte dentro de um recipiente contendo uma solução alcalina. Sobre o gel, coloca-se uma membrana de nitrocelulose ou nylon, papel 3MM e toalhetes por cima. À medida que o tampão sobe por capilaridade, ocorre a transferência dos fragmentos do DNA para a membrana. 3) A membrana é, posteriormente, hibridada com a sonda de DNA marcada por radioactividade. 4) Após lavagens para remoção do DNA não hibridado, procede-se à autorradiografia para visualização das bandas (adaptado de Semagn et al., 2006).---20
Figura 1. 3 - Representação esquemática da amplificação de RAPDs. O “primer” hibrida em locais
complementares aos da sua sequência e que estão situados em cadeias opostas do dsDNA e em direcção convergente. Quando a distância entre esses locais é razoável (não superior a 3000 bp), a região entre eles é amplificável por PCR. As setas representam cópias múltiplas do mesmo “primer” e indicam a orientação da sequência do DNA genómico. Os números representam locais de ligação dos “‘primers” ao DNA molde (adaptado de Semagn et al., 2006).---23
Figura 1. 4 - Representação esquemática da técnica de AFLP. O DNA genómico foi cortado
simultaneamente por duas enzimas de restrição, uma de corte raro e outra com corte frequente. Os fragmentos de restrição ligam-se a adaptadores com extremidades específicas e usam-se “primers” selectivos com extremidades “random” (2-3 nucleótidos). O conjunto de fragmentos obtido pode ser observado por electroforese em gel de agarose ou de poliacrilamida (adaptado de Agarwal et al., 2008).26
Figura 1. 5 - Representação esquemática da técnica de SSR. Os microssatélites compreendem repetições
em tandem de 2-6 nucleótidos. O número de repetições é altamente variável entre indivíduos. São desenhados “primers” complementares às regiões flanqueantes de cada SSR, para amplificar esse locus específico. Devido à hipervariabilidade das repetições, os amplicões vão diferir em comprimento e podem ser analisados por electroforese em gel de poliacrilamida (adaptado de [1]).---27
Figura 1. 6 - Representação esquemática da técnica ISSR-PCR, com um “primer” não-ancorado (AG)8
(a) com um “primer” contendo dois nucleótidos degenerados (N) na extremidade 3’ (b), e com um ‘primer’ contendo dois nucleótidos degenerados (N) na extremidade 5’ (c). A repetição alvo no genoma é do tipo (TC)n e está inversamente orientada no dsDNA. Os produtos de amplificação ISSR obtidos em a),
b) e c) diferem no seu peso molecular (adaptado de Reddy et al., 2002).---30
Figura 1. 7 - Marcadores moleculares baseados em retrotransposões: A) Marcadores IRAP originam-se
pela proximidade de dois LTRs usando “primers outward-facing” que hibridam com sequências LTR alvo. B) Na técnica REMAP utilizam-se “primers” que ligam a microssatélites “outward-facing” LTRs próximos (adaptado de Agarwal et al., 2008).---31
3. Material e Métodos
Figura 3. 1 - Localização geográfica dos locais de colheita do germoplasma ‘Barbela’ em estudo
(Carvalho et al. 2010, submetido).---38
4. Resultados e Conclusão
Figura 4. 1 - Produtos de amplificação RAPD visualizados num gel de agarose 1,5% em 30 cultivares de
trigo mole, com o “primer” OPH-12. M – marcador de peso molecular “Gene Ruller DNA Ladder 100 bp Plus” (Fermentas).---46
Figura 4. 2 – Dendrograma UPGMA de similaridade genética entre as 48 cultivares de trigo mole,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado nos dados RAPD. Os códigos de passaporte, variedades botânicas e nomes das cultivares estão na Tabela 3.1.---49
Figura 4. 3 – Dendrograma UPGMA de similaridade genética entre as 51 cultivares de trigo rijo,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado nos dados RAPD. Os códigos de passaporte, variedades botânicas e nomes das cultivares estão na Tabela 3.2.---50
Figura 4. 4 - Dendrograma UPGMA de similaridade genética entre as 30 linhas de trigo ‘Barbela’,
Figura 4. 5 – Produtos de amplificação ISSR visualizados num gel de agarose 1,5% em 30 linhas de
trigos ‘Barbela’, com o “primer” UBC-15. M – Marcador de peso molecular “Gene Ruller DNA Ladder Mix” (Fermentas).---53
Figura 4.6 - Dendrograma UPGMA de similaridade genética entre as 48 cultivares de trigo mole,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado nos dados ISSR. Os códigos de passaporte, variedades botânicas e nomes das cultivares estão na Tabela 3.1.---55
Figura 4. 7 - Dendrograma UPGMA de similaridade genética entre as 51 cultivares de trigo rijo,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado nos dados ISSR. Os códigos de passaporte, variedades botânicas e nomes das cultivares estão na Tabela 3.2.---56
Figura 4. 8 - Dendrograma UPGMA de similaridade genética entre as 30 linhas de trigo ‘Barbela’,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado nos dados ISSR. Os códigos, ano e regiões de colheita estão na Tabela 3.3.---57
Figura 4. 9 - Dendrograma UPGMA de similaridade genética entre as 48 cultivares de trigo mole,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado na combinação de RAPDs e ISSRs. Os códigos de passaporte, variedades botânicas e nomes das cultivares estão na Tabela 3.1.---59
Figura 4. 10 - Dendrograma UPGMA de similaridade genética entre as 51 cultivares de trigo rijo,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado na combinação de RAPDs e ISSRs. Os códigos de passaporte, variedades botânicas e nomes das cultivares estão na Tabela 3.2.---60
Figura 4. 11 - Dendrograma UPGMA de similaridade genética entre as 30 linhas de trigo ‘Barbela’,
utilizando o coeficiente de Jaccard e o algoritmo SAHN, baseado na combinação de RAPDs e ISSRs. Os códigos, ano e regiões de colheita estão na Tabela 3.3.---61
Figura 4. 12 – Variabilidade genética entre cultivares de trigos moles: a) sementes; b) espigas principais;
c) espiguetas das espigas principais e d) comprimento das plantas adultas.---63
Figura 4. 13 - Variabilidade genética entre cultivares de trigos rijos: a) sementes; b) espigas principais; c)
espiguetas das espigas principais e d) comprimento das plantas adultas.---64
Figura 4. 14 – Dendrograma UPGMA de similaridade genética, utilizando o índice da distância
euclidiana entre as 48 cultivares de trigo mole. Os códigos de passaporte, variedade botânica e nome das cultivares estão na Tabela 3.1.---67
Figura 4. 15 - Dendrograma UPGMA de similaridade genética, utilizando o índice da distância
euclidiana entre as 51 cultivares de trigo rijo. Os códigos de passaporte, variedade botânica e nome das cultivares estão na Tabela 3.2.---68
Figura 4. 16 - Dendrograma UPGMA de similaridade genética, utilizando o índice da distância
euclidiana entre as 30 linhas de trigo ‘Barbela’. Os códigos, ano e regiões de colheita estão na Tabela 3.3. ---69
Índice de Quadros e Tabelas
3. Material e Métodos
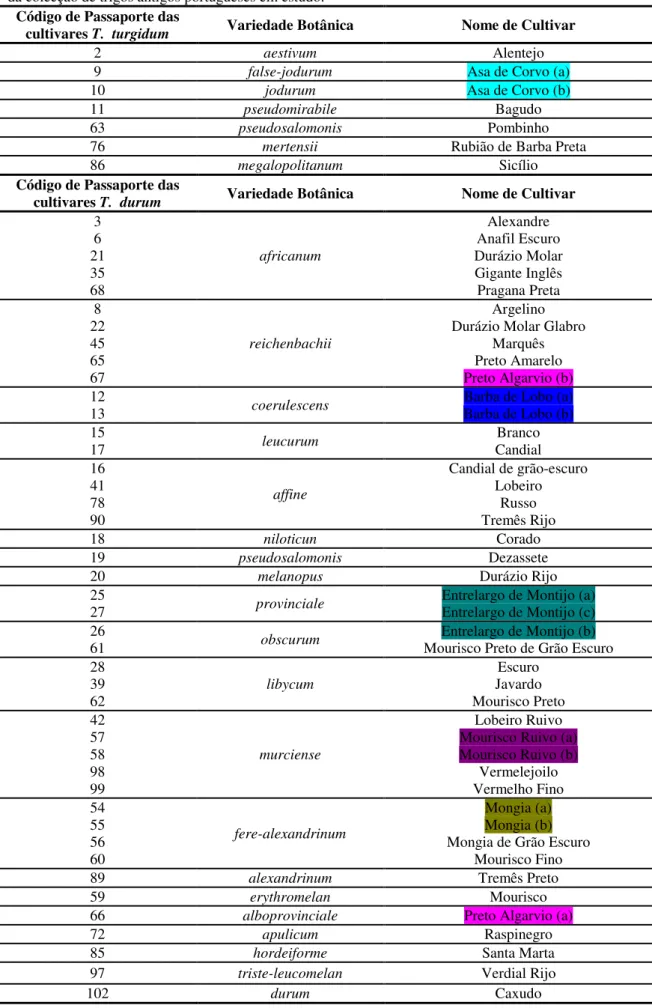
Quadro 3. 1 – “Primers” dos kits OPA, OPH e OPE testados e respectivas sequências. 41 Quadro 3. 2 – “Primers” do set 100/9 (UBC) testados e respectivas sequências. --- 43 Tabela 3. 1 - Código de passaporte, variedade botânica e nome de cultivar das 48 cultivares de trigo mole da colecção de trigos antigos portugueses em estudo. --- 35 Tabela 3. 2 - Código de passaporte, variedade botânica e nome de cultivar das 51 cultivares de trigo rijo da colecção de trigos antigos portugueses em estudo. --- 36 Tabela 3. 3 – Código de campo de 30 linhas de ‘Barbela’ e respectivo local de colheita das espigas individuais que as originaram, no nordeste de Portugal. --- 37
4. Resultados e Discussão
Quadro 4. 1 – Matriz de correlação dos componentes morfológicos e de produção dos trigos moles. --- 64 Quadro 4. 2 – Matriz de correlação dos componentes morfológicos e de produção dos trigos rijos. --- 65 Quadro 4. 3 – Matriz de correlação dos componentes morfológicos e de produção das linhas de trigo ‘Barbela’. --- 66 Tabela 4. 1 – Número de bandas RAPD totais (T), polimórficas (P) e exclusivas (E) e percentagem de polimorfismo RAPD nas cultivares de trigo mole, trigo rijo e nas linhas de ‘Barbela’.---46 Tabela 4. 2 – Número de bandas ISSR totais (T), polimórficas (P) e exclusivas (E) e percentagem de polimorfismo ISSR nas cultivares de trigo mole, trigo rijo e nas linhas de ‘Barbela’. --- 53
Lista de Abreviaturas
A – absorvância
A260 – Leitura de absorvância no comprimento de onda de 260 nm
A280 – Leitura de absorvância no comprimento de onda de 280 nm
AFLPs – “Amplified fragment Lenght Polymorphism” AP-PCR – “Arbitrarily Primed-PCR”
BES – “BAC-End Sequencing”
bp, Mb, Kb – pares de bases, mega pares de bases, kilo pares de bases BSA – “Bulked Segregant Analysis”
CCN – “Cereal Cyst nematodes”, nemátodos do cisto dos cereais cDNA – DNA complementar
CGIAR – “Consultative Group on International Agricultural Research” CIMMYT – “Centro Internacional de Mejoramiento de Maiz e Trigo” cm – centímetros
cp-SSRs – microssatélites em DNA de cloroplastos CTAB – brometo de cetiltrimetilamónio
cv. - cultivar
DAF – “DNA Amplification Fingerprinting” DGB – Departamento de Genética e Biotecnologia
DNA – “DesoxyriboNucleic Acid”, ácido desoxirribonucleico dNTPs – desoxinocleótidos trifosfatados
dsDNA – “double strand DNA”, DNA de cadeia dupla EDTA – ácido etileno diamino tetra acético
ENMP – Estação Nacional de Melhoramento de Plantas ESTs – “Expressed Sequence Tags”
gDNA – DNA genómico
g, mg, µg, ng – gramas, miligramas, microgramas, nanogramas IAA – álcool isoamílico
IBB-CGB – Instituto de Biologia e Bioengenharia – Centro de Genómica e Biotecnologia
IBPGR – “International Board of Plant Genetic Resources”
ICARDA – “International Center for Agricultural Research in the Dry Areas” INIAP – Instituto Nacional de Investigação Agrária e das Pescas
INRA – “Institute National de la Recherche Agronomique” IPK – “Institut für Pflanzengenetik und Kulturpflanzenforschung” IRAPs – “Inter-Retrotransposon amplified polymorphisms” IRGC – “Internacional Rice Germplasm Center”
IRRI – “International Rice Research Institute” ISSRs – “Inter-Simple Sequence Repeat”
ITMI – “International Triticeae Mapping Iniciative”
IWGSC – “International Wheat Genome Sequencing Consortium” LTRs – “Long Terminal Repeats”, repetições terminais longas M, mM – molar, milimolar
MAS – “Marker Assisted Selection”, selecção assistida por marcadores moleculares MgCl2 - cloreto de magnésio
MYA – “Million Years Ago”, milhares de anos min, seg, h – minutos, segundos, horas
ml, µl – mililitros, microlitros
mRNA – RNA mensageiro
mt-SSRs – microssatélites em DNA mitocondrial n – número cromossómico haplóide
NaCl – cloreto de sódio nm – nanómetros
NTSys-pc – “Numerical Taxonomy and Multivariate Analysis System for Personal Computer”
OGM’s – Organismos Geneticamente Modificados ORF – “Open Reading Frames”
PCR – “Polymerase Chain Reaction”, reacção em cadeia da polimerase p/v – peso por volume
PVP – polivinilpirrodona
QTL – “Quantitative Trait Loci”
RAPDs –“Random Amplified Polymorphic DNA”
REMAPs – “REtrotransposon-Microsatellite Amplified Polymorphism” RFLPs – “Restriction Fragment Length Polymorphism”
RTNs – retrotransposões
SAHN – “Sequential Agglomerative Hierarchical Nested Cluster Analysis” SAMPL – “Sequence Amplification of Microsatellite Polimorphic Loci” SRAPs – “Sequence-Related Amplified Polymorphisms”
ssDNA – “single strand DNA”, DNA de cadeia simples SSRs – “Simple Sequence Repeat”, microssatélites
Taq – DNA polimerase I extraída da bactéria termófila Thermus aquaticus
TBE – Tris. Borate EDTA TE – tampão de EDTA
TILLING – “Targeting Induced Local Lesion IN Genome” TRAP – “Target Region Amplification Polymorphism” Tris.HCl –
UBC – “University of British Columbia”
UPGMA – “Unweighted Pair Group Method with Arithmetical Averages” UTAD – Universidade de Trás-os-Montes e Alto Douro
UV – raios ultra-violeta v – volts
v/v – volume por volume
x – número cromossómico base de uma série poliplóide 2n – número cromossómico de um organismo diplóide ºC – graus Célsius
1. Introdução
O trigo é um dos cereais mais importantes para a alimentação e agricultura a nível mundial, pelo que é bastante estudado e utilizado em programas de melhoramento genético por todo o mundo.
Os programas de melhoramento visam a selecção de determinadas características fenotípicas e de produção desejadas levando a um aumento da frequência dos alelos pretendidos ou da sua combinação. Porém, esses programas conduzem simultaneamente a uma diminuição da frequência e mesmo à perda de outros alelos, reduzindo a biodiversidade da população geneticamente melhorada. Para evitar a perda da diversidade genética que pode ocorrer durante os programas de melhoramento, é de vital importância que o melhorador conserve as ‘landraces’ e os seus parentes silvestres, bem como previna o desaparecimento dos recursos nativos (Chen et al., 1994).
A conservação dos recursos genéticos é actualmente uma das questões mais importantes e controversas da humanidade. Em plena época de mudança global, onde os ecossistemas enfrentam grandes mudanças climáticas e fortes pressões antropogénicas, a erosão e perda de diversidade genética é uma realidade cada vez mais actual (Draper
et al., 2004).
Fu et al. (2008) consideram que conhecer as relações genéticas entre as diferentes cultivares que suportam a diversidade genética pode ser de grande utilidade no desenvolvimento de estratégias eficientes de utilização e manutenção do germoplasma.
A diminuição da base genética das cultivares de trigo modernas deve-se essencialmente a pressões selectivas intensivas e à deriva genética. A avaliação e o estabelecimento de estratégias de conservação in situ e ex situ da diversidade genética existente nas populações naturais ou em cultivares locais (Sica et al., 2005) devem ter prioridade máxima a nível mundial visando o desenvolvimento sustentável.
Embora os recursos abundantes de germoplasma de trigo possam ser favoráveis ao melhoramento e à investigação genética, os bancos de sementes enfrentam desafios no que se refere à conservação, avaliação, identificação e utilização das inúmeras ‘accessions’ que detêm (Tanksley e McCouch, 1997). Assim, muitos países têm desenvolvido esforços para a caracterização e avaliação da diversidade genética das suas populações naturais ou “landraces” de trigo.
1.1. A Tribo Triticeae, Família Poaceae
A família Poaceae inclui a tribo Triticeae, que é a divisão dos cereais economicamente mais importantes. Esta tribo inclui 14 géneros, agrupados em duas subtribos – Triticinae e Hordeinae, e compreende caracteres morfológicos que a distinguem das outras tribos da subclasse Graminaea (Sakamoto, 1973).
O trigo (Triticum spp.), o centeio (Secale spp.) e a cevada (Hordeum spp.) pertencem à tribo Triticeae, que inclui também os géneros Agropyron, Aegilops,
Haynaldia, Elymus, Psathyrostachys, Brachypodium e Leymus (Miller, 1987), entre
outros.
Na tribo Triticeae a produção de anfiplóides e de híbridos interespecíficos sugere compatibilidade genética ou citoplasmática entre os diferentes géneros dessa tribo (Sakamoto, 1973).
A compatibilidade genética das triticíneas deriva das relações de homeologia entre os cromossomas dos diferentes genomas que evoluíram, por divergência, de um genoma ancestral (P) (Kihara, 1982; Kimber e Feldman, 1987).
O género Triticum inclui espécies diplóides (2n = 2x = 14), tetraplóides (2n = 4x = 28) e hexaplóides (2n = 6x = 42), que contêm, por gâmeta, um número igual ou múltiplo de sete cromossomas do(s) ancestral(ais) haplóide(s).
A domesticação dos trigos conduziu à reestruturação de ráquis e glumas, convertendo as espigas quebradiças das espécies silvestres em espigas não quebradiças e de grão nu, nas espécies cultivadas (Goncharov, 2005). Em 1753, Linnaeu usou estas características para classificar o género Triticum. O mesmo autor conseguiu classificar os trigos hexaplóides baseando-se apenas em dois critérios: habitat de crescimento (Primavera vs. Inverno) e presença/ausência de aristas. Mais tarde verificou-se que estas características são controladas por um pequeno número de genes (Swaminathan e Rao, 1961).
Nos trigos tetraplóides há duas espécies: Triticum turgidum (AABB) e Triticum
timopheevii (AAGG). O T. turgidum inclui a subespécie silvestre ssp. dicoccoides e
várias subespécies cultivadas, tais como T. turgidum ssp. durum (trigo rijo), que cresce nas regiões semi-áridas da Bacia do Mediterrâneo, Índia e nas grandes planícies do nordeste dos Estados Unidos e Canadá. O trigo tetraplóide T. timopheevii inclui a forma silvestre T. timopheevii ssp. armeniacum e a forma cultivada T. timopheevii ssp.
hexaplóides: T. aestivum (AABBDD) (trigo mole), que inclui várias subespécies, e T.
zhukovskyi (AmAmAAGG).
As espécies poliplóides deste género são alopoliplóides, e podem apresentar heterose ou vigor híbrido, que se manifesta no aumento do tamanho e maior produção de sementes relativamente aos seus progenitores, e cujo efeito resulta da interacção de determinados loci homólogos dos diferentes genomas (Feldman e Sears, 1981).
A alopoliploidia é um fenómeno que ocorre com frequência na natureza (50 a 70% nas angiospérmicas). A tolerância à acumulação de variação genética, quer por mutações quer por hibridação, torna-se facilitada em organismos poliplóides (Feldman e Sears, 1981; Pikaard e Chen, 2000). Segundo os mesmos autores, os poliplóides têm vantagem de viabilidade em relação aos diplóides, ocupando uma maior amplitude de habitats.
1.2. História evolutiva do trigo
A radiação das principais subfamílias da família Poaceae ocorreu há 50-80 milhões de anos (MYA) (Figura 1.1; Huang et al., 2002), enquanto as tribos Triticeae e Poeae (Lolium rigidum) divergiram há cerca de 35 MYA (Figura 1.1) e os géneros Hordeum e
Secale divergiram da linha Triticum/Aegilops há 11 MYA e sete MYA, respectivamente
(Figura 1.1; Huang et al., 2002).
Genomas de Triticum: A, Am, Ab, B, D, G
Tetrapóides: T. dicoccoides (AABB); T. timopheevii (AAGG) Hexaplóide: T. aestivum (AABBDD)
Genomas de Aegilops: D, S, Sb, S1, Ssh, Ss
Figura 1. 1 - Estimativa do tempo de evolução das linhas Triticum e Aegilops (adaptado de Huang et
Análises de emparelhamento meiótico revelaram que a evolução geral da tribo
Triticeae tem sido definida por divergência a nível diplóide a partir de um ancestral
comum, envolvendo a divergência dos genomas diplóides (West et al., 1988).
Várias espécies do género Aegilops contribuíram significativamente para a origem dos trigos poliplóides, que surgiram por hibridação interespecífica, seguida de duplicação cromossómica espontânea (Cox, 1998).
Os géneros Triticum e Aegilops incluem 13 espécies diplóides e 18 poliplóides. Para as espécies diplóides foram designados oito genomas distintos: A (A e Ab/Am), D, S (S, Ss, Sb, S1, Ssh), M, C, U, N e T. Nos trigos poliplóides consideram-se ainda os genomas B e G, uma vez que os seus progenitores diplóides (ancestrais) ainda não são conhecidos (Huang et al., 2006).
As espécies diplóides Triticum urartu e Triticum monococcum foram referidas como sendo dadoras do genoma A. Estes dois trigos diplóides produzem híbridos estéreis quando cruzados, pelo que são considerados de espécies biológicas diferentes (Johnson e Dhaliwal, 1976).
Os estudos genéticos revelaram que os trigos poliplóides constituem duas linhas evolutivas: 1) uma delas resultou nas espécies T. turgidum (AABB) e T. aestivum (AABBDD); 2) enquanto a outra esteve na origem de T. timopheevii (AAGG) e T.
zhukovskyi (AmAmAAGG) (Huang et al., 2006).
Os primeiros estudos citogenéticos propunham a espécie diplóide T. monococcum como dadora do genoma A, nas espécies tetraplóides de ambas as linhas evolutivas (Sax, 1922; Lilienfield e Kihara, 1934). Porém, estudos mais recentes apontam a espécie diplóide T. urartu como dadora do genoma A (Dvorák et al., 1988; Dvorák et
al., 1993; Huang et al., 2006).
A espécie T. zhukovskyi foi originada por hibridação de T. timopheevii com T.
monococcum (Upadhya e Swaminathan, 1963) sendo que um conjunto de genomas A
foi recebido de T. urartu e o outro de T. monococcum (Dvorák et al., 1993).
O T. aestivum surgiu há cerca de 8000 anos, por hibridação espontânea entre T.
turgidum ssp. dicoccon e Aegilops tauschii ssp. strangulata (Kihara, 1944;McFadden e
Sears, 1946; Jaaska, 1981; Lubbers et al., 1991; Dvorák et al., 1998; Huang et al., 2006). Ao compararem alterações de sequência em regiões Glu-1 dos genomas A, B e D de T. aestivum, Gu et al. (2006) sugeriram mais do que um ancestral tetraplóide para T.
A divergiram nos últimos 0,81 MYA. Estes resultados estão de acordo com os estudos realizados por Gu et al. (2004) e Isidore et al. (2005).
A origem dos genomas B e G continua controversa. Há muitas evidências que sugerem a espécie Aegilops speltoides (genoma S) como dadora do genoma B dos trigos moles e duros (Riley et al., 1958; Johnson, 1972; Kerby e Kuspira, 1988; Dvorák e Zhang, 1990; Talbert et al., 1995; Sasanuma et al., 1996; Daud e Gustafson, 1996; Pestsova et al., 1998). A Ae. speltoides pode ser uma forma significativamente divergente do dador ancestral do genoma B (Blake et al., 1999). Alternativamente, o genoma B pode ter resultado da introgressão de várias espécies parentais (Zohary e Feldman, 1962). Feldman (1966) e Dvorák e Appels (1982)sugeriram que o genoma B de T. turgidum e o genoma G de T. timopheevii estavam relativamente próximos, mas também foi sugerido que o genoma G de T. timopheevii está mais estreitamente relacionado com o genoma S de Ae. speltoides do que o genoma B de T. turgidum (Dvorák et al., 1989). Análises de DNA dos organelos em Ae. speltoides indicam que esta espécie pode ser a dadora materna de todos os trigos poliplóides (Wang et al., 1997). No entanto, análises filogenéticas moleculares mais recentes revelaram que alguns loci do genoma S de Ae. speltoides e do genoma G de T. timopheevii estão estritamente relacionados, sugerindo a mesma origem de algumas partes desses genomas, o que não se verifica para o genoma B, cuja espécie dadora permanece desconhecida (Huang et al., 2006). Os mesmos autores sugerem aindaa ocorrência de mosaicismo a nível do genoma (mixogenómio) e de diferenciação interespecífica significativa.
A evolução do trigo continuará a ser alvo de muitos estudos e de desafios permanentes. Um enorme desafio advém do tamanho do seu genoma, que é extremamente grande (cerca de 16000 Mb) quando comparado com as espécies vegetais modelo Arabidopsis thaliana (130 Mb) e Oriza sativa L. (arroz, 430 Mb). Mas a sua especiação faz do trigo uma excelente fonte para estudos de evolução que ocorrem nos genomas pouco após a poliploidia, como remoção de DNA, alterações na expressão génica, reactivação de elementos transponíveis e diversificação funcional de genes duplicados (Ozkan et al., 2001; Shake et al., 2001; Comai et al., 2002; He et al., 2003; Kashkush et al., 2003; Blanc e Wolfe, 2004; Gu et al., 2006).
1.3. Conservação e manutenção do germoplasma
A domesticação dos trigos moles teve início há 12 000 anos atrás no Crescente Fértil. Ao longo dos séculos, novos alelos e novas combinações alélicas foram originados por mutações e por recombinação, respectivamente (Dreisigacker et al., 2005). A variação genética disponível foi subsequentemente sendo reduzida por deriva genética e selecção, pela Natureza e pelos primeiros agricultores, embora tenham sido originadas, por outro lado, “landraces” bem adaptadas a condições específicas dos seus habitats (Mohammadi et al., 2008).
Actualmente, as práticas de melhoramento modernas caracterizadas por pressões selectivas intensas conduziram inevitavelmente à perda da diversidade genética dos cereais no mundo inteiro (Tanksley e McCouch, 1997), uma vez que tais práticas favorecem a homogeneização (Saidi et al., 2001).
1.3.1. Conservação do germoplasma
A conservação dos recursos genéticos tornou-se assim uma prioridade estabelecida e reconhecida a nível mundial podendo agrupar-se em duas categorias: a conservação in
situ e a conservação ex situ, consoante a conservação das espécies, populações ou
habitats forem realizadas, respectivamente, no local ou fora do local de origem (Draper
et al., 2004).
Apesar dos jardins botânicos terem tido desde sempre a preocupação de recolha e conservação de sementes para assegurarem as suas colecções de plantas vivas (Draper et
al., 2004) as iniciativas globais de conservação ex situ começaram apenas na segunda
metade do século XX (Plucknett et al., 1983). Desde essa altura que agrónomos e melhoradores de plantas têm recolhido colecções de germoplasma e depositado essas amostras em centros internacionais, por todo o mundo. Estas colecções representam repositórios de biodiversidade disponíveis para cada espécie e são uma fonte valiosa de genes úteis para os melhoradores (Virk et al., 1995).
A conservação não se limita à colheita e ao armazenamento das sementesdevendo também assegurar a existência do material em condições viáveis e geneticamente íntegras, mantendo as suas características originais (Jaramillo e Baena, 2000). Para que tal seja possível é necessário fazer o controlo das condições de acondicionamento e de armazenamento (nomeadamente, manutenção de um baixo conteúdo hídrico) de forma a reduzir a actividade metabólica e, com ela, os processos envolvidos na perda da viabilidade e do vigor das sementes (Pita Villamil e Martínez-Laborde, 2001).
Existem três tipos de conservação em função do tempo: a curto, a médio e a longo prazo. As colecções conservadas a curto e a médio prazo denominam-se colecções de trabalho e as conservadas a longo prazo designam-se por colecções base. Estas últimas são armazenadas a temperaturas entre os -10ºC e os -20ºC, durante várias décadas. As sementes são cuidadosamente desidratadas e seladas em tubos de ensaio, ou em vácuo em envelopes de folhas de alumínio (Sachs, 2009). No armazenamento a médio prazo as sementes são mantidas a temperaturas que podem variar entre 0ºC e 5ºC, durante 20 anos. A curto prazo as colecções são mantidas à temperatura ambiente ou em refrigeração acima dos 5ºC. Nestas condições as sementes podem conservar-se alguns anos e são constantemente utilizadas para avaliação, distribuição e programas de melhoramento (Plucknett et al., 1983). As sementes de cereais podem manter a sua viabilidade nestas condições durante 10 ou mais anos (Sachs, 2009).
A viabilidade de um germoplasma conservado vai diminuindo com o tempo. Além disso, a sua quantidade vai decrescendo devido à utilização do mesmo (Jaramillo & Baena, 2000). Assim, a renovação e a multiplicação das colecções tornam-se absolutamente necessárias. A renovação consiste no aumento de sementes proveniente de novas colheitas de campo e serve para aumentar a variabilidade genética e a viabilidade da amostra, após longos períodos de armazenamento do material. A multiplicação é efectuada quando se pretende aumentar o número de sementes da amostra inicial (Draper et al., 2004).
Os princípios que guiam a recolha, conservação e troca de germoplasma são basicamente três. Primeiro, quando uma “accession” é recolhida mantém-se sempre uma amostra no país de origem, para uso nacional. Segundo, o germoplasma é disponibilizado gratuitamente a qualquer agricultor interessado. Finalmente, as colecções a longo prazo são duplicadas e mantidas noutro local (Plucknett et al., 1983).
1.3.2. Bancos de germoplasma
Os bancos de germoplasma têm como principais objectivos recolher, manter, preservar e distribuir sementes que representem a diversidade genética das espécies cultivadas (Sachs, 2009).
Nikolai Ivanovich Vavilov foi o primeiro a considerar os parentes silvestres como novas fontes de variabilidade no melhoramento de cultivares (Vavilov, 1926, 1940), o
Botânica Aplicada, que levou à criação do banco de germoplasma actualmente conhecido por “Vavilov All-Union Institute of Plant Industry” (Sprague, 1980; Damania, 2008).
Em 1932 foi formalmente fundado o “Maize Genetics Cooperation Stock Center” (Kass et al., 2005) devido à tradição de partilha de recursos que havia entre os investigadores do milho (Zea mays) para maximizar os esforços de investigação fundamental nesta espécie (Freeling e Walbot, 1994). Esta instituição tinha como objectivos divulgar informação de interesse e dados não publicados e manter e distribuir “stocks” teste. O “Maize Genetics Cooperation Stock Center” tornou-se um modelo para a criação de outros centros de “stock” genético de plantas modelo, bem como na criação de outros bancos de germoplasma (Sachs, 2009).
Após a segunda guerra mundial a “Food and Agriculture Organization” (FAO) das Nações Unidas tornou-se a principal organização a promover a conservação dos recursos genéticos das plantas. O seu grupo de consultadoria em investigação agrária internacional (“Consultative Group on International Agricultural Research” – CGIAR) criou a comissão internacional para os recursos genéticos vegetais (“International Board for Plant Genetic Resources” – IBPGR) em 1973 (Sprague, 1980), quando havia apenas seis países com capacidade de armazenamento de sementes por longos períodos de tempo (Plucknett e Horne, 1992). Desde essa altura o IBPGR trabalhou no sentido de criar uma rede global de bancos de germoplasma (Plucknett et al., 1987). Actualmente existem mais de 100 bancos de germoplasma espalhados pelo mundo, a maioria dos quais situados nos países em desenvolvimento (Sachs, 2009).
Nos bancos de genes, o maior número de “accessions” corresponde aos cereais. No entanto, é de referir que o mais importante não é o tamanho das colecções, mas sim a sua utilidade em programas de melhoramento. O “Centro Internacional de Mejoramiento de Maiz Y Trigo” (CIMMYT), situado no México, mantinha já em 1983 uma colecção a médio prazo de 50 000 trigos e triticales enquanto o “International Center for Agricultural Research in the Dry Areas” (ICARDA), na Síria, armazenava cerca de 17 000 amostras. As colecções de germoplasma de arroz, com cerca de 200 000 “accessions” armazenadas, são menos representativas do que as de trigo (Plucknett
et al., 1983). O “International Rice Germplasm Center” (IRGC) e o “International Rice
Research Institute” (IRRI), nas Filipinas, conservam mais de 75 000 “accessions” de arroz (Virk et al., 1995).
O germoplasma geralmente inclui linhas consanguíneas, “landraces”, variedades de polinização livre, parentes silvestres, cultivares e outros “stocks” provenientes do melhoramento. A importância primária destas “accessions” é a variação indefinida, um recurso valioso para os melhoradores e investigadores (Sachs, 2009).
Outros centros de germoplasma concentram-se em germoplasma especial, como “stocks” genéticos, isto é, uma ou um número limitado de variações definidas como por exemplo: um alelo de um gene específico, combinação de mutações que originam um fenótipo único, uma série de alelos mutantes de genes geneticamente ligados, a variante de uma característica citoplasmática, um inserto transgénico comercialmente não aprovado, uma aberração cromossómica, um aneuplóide ou ainda uma ploidia alternativa. Podem incluir também outros “stocks” genéticos como linhas autofecundadas recombinantes (RILs – “Recombinant Inbred lines”) úteis para o mapeamento de genes, linhas contendo elementos transponíveis activos usados para determinar a função de um gene descoberto por sequenciação (Sachs, 2009).
1.3.3. Manutenção do germoplasma
A conservação de um germoplasma exige a manutenção, caracterização e avaliação da diversidade genética, sendo a caracterização um passo crítico, pois determina a identidade genética de cada “accession” na colecção base (Laurentin, 2009). O elevado número de “accessions” gera problemas de manutenção e de exploração do germoplasma (Virk et al., 1995), pelo que a maioria do germoplasma permanece por caracterizar e, consequentemente, não é utilizado pelos melhoradores (Fernie et al., 2006).
Para a manutenção, investigação e utilização convenientes do germoplasma foi proposto o conceito de colecção nuclear (Brown, 1989), que consiste num subconjunto do germoplasma seleccionado para representar ao máximo a diversidade existente na colecção total, com o mínimo de redundância. A colecção nuclear não envolve a redução da colecção inicial, mas aumenta a eficiência da sua manutenção (Brown, 1989; Andersen e Fairbanks, 1990) sendo utilizada para múltiplos fins e por muitos utilizadores (Brown, 1989; 1995).
Para criar uma colecção nuclear tem que se atender a dois parâmetros fundamentais: 1) a proporção da amostra e 2) a representatividade da variação genética da colecção
ultrapassasse 3000 e que a colecção nuclear representasse pelo menos 70% da diversidade genética da colecção base. Diwan et al. (1994) propuseram que a proporção da amostra fosse sempre maior que 10% enquanto Hintum (1999) sugeriu que a proporção da amostra devia depender do objectivo da colecção nuclear e, por isso, podia variar entre 5 a 20% da colecção base.
Hintum (1994), usando uma proporção de 6,5% da amostra total, estabeleceu, na Holanda, uma colecção nuclear de cevada. Para o germoplasma do arroz na província de Yunnan, China, Li et al. (2000) descobriram que 5% da colecção base representava 96% da variação fenotípica.
Zhang et al. (2002) e Dong et al. (2003) construíram colecções nucleares candidatas para o germoplasma do trigo chinês baseando-se em análises de “cluster” dos dados de passaporte. Dessas análises resultaram 5029 colecções nucleares candidatas. Para essas colecções candidatas do germoplasma 21,5% das “accessions” da colecção base representam 96,4% da variação genética para 21 caracteres botânicos e agronómicos. Posteriormente foi estabelecida uma colecção nuclear com base em análises de microssatélites dessas colecções candidatas (Hao et al., 2006), realçando-se o enorme potencial da combinação dos marcadores moleculares com os dados de passaporte na construção de uma colecção nuclear.
Em França, o “Institute National de la Recherche Agronomique”(INRA) criou uma colecção nuclear mundial de trigo contendo 384 genótipos, baseada na análise molecular de 3942 “accessions” (Balfourier et al., 2007). Outros países e bancos de germoplasma estão também a constituir as suas colecções nucleares, nomeadamente a Rússia (Martynov et al., 2003), Sérvia e Montenegro (Kobiljski et al., 2002). No Brasil a Embrapa Trigo está a desenvolver uma colecção nuclear de trigo baseada em mais de 15 000 ‘acessions’ oriundos de 68 países (Bonow, 2007).
A criação de colecções nucleares baseada na variação genética das “accessions” é de grande utilidade (Virk et al., 1995), embora essa informação nem sempre esteja disponível. No entanto a determinação da variação genética do germoplasma é um passo essencial antes da sua utilização em programas de melhoramento, uma vez que esta informação e as relações entre linhas de elite e cultivares são úteis para o desenvolvimento de novas cultivares (Mohammadi et al., 2008).
1.4. Diversidade genética e o melhoramento do germoplasma
A diversidade genética entre as cultivares adaptadas ou linhas melhoradas tem um impacto significativo no melhoramento das plantas (Fufa et al., 2005), podendo ser avaliada com base em análises do “pedigree”, dados fenotípicos ou moleculares. A análise da diversidade genética com base no “pedigree” pode resultar numa estimativa mais elevada do que a existente no “pool” genético devido a suposições feitas atendendo à deriva genética, às pressões selectivas e ao grau de parentesco com ancestrais com “pedigree” desconhecido (Barrett et al., 1998; Soleimani et al., 2002).
As características morfológicas e fisiológicas têm sido estudadas como critérios de selecção para melhoramento de trigos (Casadesus et al., 2007; Ferrio et al., 2007; Marti
et al., 2007). Algumas características qualitativas também têm sido utilizadas para
analisar a diversidade genética em programas de melhoramento de trigos de inverno (Baenziger et al., 2001) e, em alguns casos, foram úteis na estimativa do nível de diversidade genética e na análise da variação genética em programas de melhoramento (Fufa et al., 2005).
As características morfológicas, fisiológicas e de qualidade têm algumas limitações: incluem polimorfismo e heritabilidade reduzidos; expressão tardia; podem ser controladas por genes com efeitos pleiotrópicos e epistáticos, e dependem de factores ambientais (van Beuningen e Busch, 1997).Assim, os marcadores moleculares têm sido usados complementarmente para a análise da diversidade genética em diversas cultivares e espécies silvestres, pois são inúmeros e não são influenciados por factores externos (Prasad et al., 2000).
A diversidade genética tem sido estudada em trigos e nos seus parentes utilizando dados morfológicos (Lee e Kaltsikes, 1973, entre muitos outros), variação proteica (Cox
et al., 1985; Graybosch, 1992, entre muitos outros) e marcadores de DNA (Chen et al.,
1994; Barrett et al., 1998; Paull et al., 1998; Bohn et al., 1999; Prasad et al., 2000; Soleimani et al., 2002; Almanza-Pinzon et al., 2003; Naghavi et al., 2004, 2007, entre muitos outros).
Nos últimos 40 anos foram desenvolvidos esforços à escala mundial no melhoramento do trigo, que resultaram no aumento de produtividade e estabilidade, bem como no desenvolvimento de cultivares resistentes a stresses bióticos e abióticos (William et al., 2005).
O CIMMYT tem vindo a melhorar cultivares de trigo para uso dos agricultores dos países em desenvolvimento, tendo sido bem sucedido em termos de aumento do rendimento, estabilidade da produção e aumento da resistência a doenças. Contudo este sucesso deu-se à custa de uma redução da diversidade genética das cultivares. Recentemente, os melhoradores do CIMMYT têm tentado expandir a base genética do trigo incluindo em programas de melhoramento “landraces”, materiais de outros programas de melhoramento e trigos sintéticos derivados de espécies silvestres em “pedigrees” de novos materiais avançados. O resultado é um aumento altamente significativo na diversidade genética latente das linhas recentemente melhoradas, aumentando também a produtividade, a resistência a stresses bióticos e abióticos e a qualidade final do produto (Warburton et al., 2006).
Os trigos sintéticos hexaplóides derivados de cruzamentos entre trigos tetraplóides e
Ae. tauschii; bem como a utilização de “landraces”, cultivares tradicionais; e
introgressões “alien”, foram bem sucedidos como novas fontes de variabilidade genética para melhoramento da tolerância dos trigos hexaplóides a stresses abióticos como seca, temperaturas elevadas, salinidade e solos pobres em micronutrientes (Trethowan e Mujeeb-Kazi, 2008).
Apesar do melhoramento poder conduzir a um estreitamento da diversidade genética, alguns estudos mostram que nem sempre tal acontece (Christiansen et al., 2002; Khlestkina et al., 2004).
1.5. Aplicação da biotecnologia no melhoramento do trigo
Tradicionalmente, os métodos de melhoramento incluem hibridação, recombinação e selecção de genótipos com características de interesse (Dong et al., 2009).
Neste milénio, a produção de novas variedades, adaptadas a novos objectivos, será conseguida quer por técnicas de melhoramento convencionais, quer por modificação genética (Snape et al., 2005), de forma a poder responder às exigências cada vez maiores da humanidade.
As ferramentas emergentes permitem a descoberta e manipulação de variação genética desejável nas cultivares futuras, desenvolvidas para responder aos desafios específicos da economia e das condições ambientais (Snape et al., 2005).
Actualmente estão disponíveis mapas genéticos para as principais espécies de cereais, incluindo o trigo, que conta com esforços a nível global no seu mapeamento,
sendo de referir a “International Triticeae Mapping Iniciative” (ITMI), formada em 1989, originalmente como um esforço para desenvolver mapas RFLP em Triticeae, especialmente em trigo e cevada.
A principal utilidade dos mapas genéticos é identificar genes de interesse e anotá-los, para que possam ser directamente utilizados na selecção assistida por marcadores moleculares (“Marker Assisted Selection” – MAS) (Snape et al., 2005).
Nalam et al. (2006) identificaram os genes Br-A2 e Br-A3 nos braços curtos dos cromossomas 3A e 3B, respectivamente. Estes genes afectam a estrutura da ráquis nos trigos tetraplóides silvestres e cultivados, permitindo a sua distinção.
No CIMMYT utiliza-se a estratégia de mapeamento de ligação parcial combinada com “bulked segregant analysis” (BSA) para identificar e caracterizar genes que conferem durabilidade na resistência à ferrugem amarela e à ferrugem das folhas bem como para desenvolver marcadores para esses genes (William et al., 2005). A BSA tem sido utilizada com sucesso na identificação de marcadores moleculares associados a genes de resistência a doenças em várias espécies (Eastwood et al., 1994; Williams et
al., 2001).
Dayteg et al. (2008) desenvolveram um marcador molecular ligado a genes de resistência a nemátodos de cisto dos cereais (CCN) em cevada, com aplicação na selecção assistida por marcadores, que vem complementar os marcadores já conhecidos para esta característica (Kretschmer et al., 1997; Seah et al., 1998; Madsen et al., 2003; Barr et al., 2003; Karakousis et al., 2003).
A reprodutibilidade e a fiabilidade do marcador asseguram a robustez dos ensaios. De uma forma ideal, os marcadores deviam ser utilizados quando a selecção de campo tem custos elevados e/ ou quando essa selecção é laboriosa e pouco fiável, devido a influências ambientais (William et al., 2005).
Depois do mapeamento, a genómica veio facilitar a compreensão das relações entre genes e funções, primeiro com a sequenciação de genomas completos e, posteriormente, com a acumulação de bibliotecas de “Expressed Sequence Tags” – ESTs (Snape et al., 2005).
A sequenciação de genomas de plantas modelo, como a A. thaliana e o arroz, veio revolucionar a compreensão da biologia molecular das plantas, embora esse conhecimento não seja ainda aplicado com eficácia no melhoramento dos principais
O Consórcio Internacional para a Sequenciação do Genoma do Trigo (“International Wheat Genome Sequencing Consortium” – IWGSC) foi fundado em 2005 com o objectivo de desenvolver ferramentas e recursos com vista à sequenciação do genoma do trigo hexaplóide. Os objectivos a médio prazo passam pela construção de um mapa físico dos 21 cromossomas do trigo mole ‘Chinese Spring’, desenvolver novos marcadores, aumentar o conhecimento sobre a organização do genoma do trigo e avaliar as tecnologias de sequenciação (Feuillet e Eversole, 2007).
A sequenciação de um genoma poliplóide com 17 Gb que apresenta uma elevada percentagem de sequências repetitivas (> 80%) é uma tarefa difícil, além de dispendiosa, mesmo utilizando as novas plataformas de sequenciação (Wicker et al., 2006).
Enquanto se aguarda pela consolidação das novas tecnologias de sequenciação (Feuillet e Eversole, 2007) as alternativas passam por clonagem de genes de interesse individuais ou isolamento da sequência dos genes a partir da sua expressão (Snape et
al., 2005).
A modificação genética permite a introdução de genes individuais isolados a partir de qualquer fonte e oferece uma multiplicidade de oportunidades no melhoramento dos cereais. Vários protocolos experimentais foram desenvolvidos com vista à produção em massa de trigo mole transgénico fértil, através de biolística, usando embriões imaturos (Pellegrineschi et al., 2002). As cultivares elite do CIMMYT foram geneticamente modificadas através de biolística com um gene repórter e com o gene de interesse, metodologia essa, que apresentou uma eficiência de co-transformação a rondar os 25 a 30% (William et al., 2005).
Um novo método designado por TILLING (“Targeting Induced Local Lesion IN Genome”) gera mutações alélicas em série, incluindo “knockouts”, nos genes de interesse (Henikoff e Comai, 2003). Este método é útil em estudos de genómica funcional (McCallum et al., 2000) e no melhoramento de cereais como o milho (Till et
al., 2004), o arroz (Wu et al., 2005; Till et al., 2007), a cevada (Caldwell et al., 2004),
entre outras espécies (Hohmann et al., 2005; Perry et al., 2003; Cooper et al., 2008). O trigo tolera frequências de mutações muito mais elevadas do que outras plantas, possivelmente devido à sua natureza poliplóide (Slade et al., 2005), o que se revela vantajoso pois requer uma população mutante relativamente pequena para a selecção das características desejadas. Dong et al. (2009) utilizando o método de TILLING
modificado conseguiram identificar 121 mutantes para os genes Wx-A1 e Wx-D1 em 2 348 plantas.
1.6. Germoplasma de trigo português
Em 1933 o Professor João de Carvalho e Vasconcellos publicou uma monografia denominada “Trigos portugueses ou de há muito cultivados no País”, que corresponde ao primeiro estudo morfológico e taxonómico de trigos portugueses. Este estudo constituiu fonte de orientação e citação obrigatória para os investigadores de outros países que realizavam estudos com trigo (Malato-Beliz, 1972).
A colecção de trigos portugueses foi semeada anualmente até 1936 nos campos de ensaio de Belém. Após a extinção da Estação Agrária, em 1936, a colecção de campo foi transferida para a Estação Nacional de Melhoramento de Plantas (ENMP), em Elvas. Após consulta do Index Seminum, já nessa altura era frequente haver intercâmbio de sementes entre instituições de investigação científica agrária. Assim é provável que tenha ocorrido permuta de material vegetal entre a Estação Agronómica Nacional e o “Institut für Pflanzengenetik und Kulturpflanzenforschung” (IPK), Alemanha. Adicionalmente algumas condições adversas existentes na ENMP conduziram à perda da colecção em Portugal.
A esta colecção de trigos antigos portugueses, podemos associar o trigo mole ‘Barbela’ que é uma “landrace” portuguesa muito antiga, que mostra uma adaptação notável a diferentes condições edafo-climáticas. Esta “landrace” é muito utilizada pelos agricultores devido à sua boa produção de grão e palha, e pela sua tolerância a solos ácidos (Guedes-Pinto e Bernard, 1980, 1983; Ribeiro-Carvalho et al., 2001). Nos últimos anos, a “landrace” ‘Barbela’ tem sido cultivada principalmente no norte de Portugal, na região de Trás-os-Montes especialmente para o fabrico de pão tradicional e produção de palha (Ribeiro-Carvalho et al., 2001).
No Centro de Genómica e Biotecnologia da Universidade de Trás-os-Montes e Alto Douro (CGB/UTAD) têm-se vindo a desenvolver estudos no trigo ‘Barbela’ ao nível citogenético (Ribeiro-Carvalho et al., 1997; 2001), de tolerância a solos ácidos (Pinto-Carnide e Guedes-Pinto, 1999; Martins-Lopes et al., 2009), de características genéticas,
Conjuntamente, esta colecção de trigos antigos, até há poucos anos considerada perdida, constitui um excelente, único e insubstituível repositório de germoplasma que interessa preservar.
Com o objectivo principal de caracterizar essa colecção, o CGB-UTAD viu recentemente aprovado o projecto PTDC/AGR-GPL/65876/2006 “"Estrutura genética e filogenias de trigos antigos portugueses: abordagem genómica e de citogenética molecular", financiado pela Fundação para a Ciência e Tecnologia (FCT). Nesse projecto será efectuada a análise de componentes morfológicos e de produção, bem como a caracterização citogenética e molecular utilizando marcadores moleculares como RAPDs (“Random Amplified Polymorphic DNAs”), SSRs (“Simple Sequence Repeats” ou microssatélites), ISSRs (“Inter-Simple Sequence Repeats” ou inter-microssatélites), e outros baseados em retrotransposões com repetições terminais longas (“Long Terminal Repeats” – LTRs), tais como IRAP (“Inter-Retrotransposon Amplified Polymorphisms”) e REMAP (“Retrotransposon Microsatellite Amplified Polymorphism”).
1.7. Marcadores genéticos
O conceito de marcadores genéticos não é novo. No século XIX, Gregor Mendel foi o primeiro a utilizar um sistema de marcadores baseado no fenótipo, nas suas experiências com as ervilheiras.
Um marcador genético é qualquer característica (fenotípica, proteica ou genotípica) usada como marcador de variação genética, que se manifesta por polimorfismo (identificação de diferentes alelos no mesmo locus). Segundo Ferreira e Grattapaglia (1996) o polimorfismo é a principal característica de um marcador genético.
Em plantas, os marcadores genéticos são utilizados de forma rotineira em estudos de evolução, de taxonomia, filogenéticos, ecológicos e genéticos (Agarwal et al., 2008).
As limitações dos marcadores fenotípicos conduziram à necessidade do desenvolvimento tecnológico de novos sistemas de marcadores, tendo assim surgido os marcadores baseados em DNA.
Em 1994, Solanille classificou os marcadores genéticos em quatro grandes grupos: morfológicos, bioquímicos, citogenéticos e moleculares. Porém, Lefebvre e Chèvre (1995) propuseram uma classificação que consiste em apenas dois grupos de marcadores genéticos: os marcadores morfológicos e os marcadores moleculares.
Segue-se uma descrição detalhada dos respectivos marcadores genéticos, atendendo à classificação de Lefebvre e Chèvre (1995) dando maior ênfase aos marcadores usados neste trabalho: marcadores morfológicos e marcadores moleculares RAPDs e ISSRs.
1.7.1. Marcadores morfológicos
Os marcadores morfológicos revelam polimorfismos genéticos ao nível do fenótipo, estando associados a caracteres morfológicos de fácil identificação, como nanismo, deficiência de clorofila, cor e morfologia foliar (Ryck e Yoder, 1988).
Para além de terem sido os primeiros marcadores a serem utilizados, os marcadores morfológicos também contribuíram para a elucidação dos fundamentos teóricos da análise de ligação génica e para a construção das primeiras versões de mapas genéticos (Knapp, 1991). Sax e McPhee (1923) verificaram que, em feijão, o peso dos grãos estava associado à cor das sementes. Esta foi a primeira tentativa de caracterização individual dos loci envolvidos na expressão de um caracter quantitativo (“Quantitative Trait Loci” - QTL), com auxílio de marcadores morfológicos.
Os marcadores morfológicos têm tido um papel fundamental na divulgação das características agronómicas de novos materiais vegetais e podem influenciar decisivamente a escolha de variedades por parte dos agricultores, embora, em culturas de base genética restrita, possam muitas vezes não distinguir adequadamente as cultivares comerciais (Smith e Smith, 1992; Pecchioni et al., 1996).
Mais recentemente os marcadores morfológicos, em combinação com diversos tipos de marcadores moleculares, estão a ser utilizados em estudos de certificação da autenticidade de “landraces” (Muñoz-Falcón et al., 2009) na distinção de cultivares com importância económica (Rodríguez-Garay et al., 2009), na diversidade genética (Fu et
al., 2008) e na detecção de plasticidade fenotípica em espécies em risco de extinção
(Noel et al., 2007). Em trigos os marcadores morfológicos continuam a desempenhar um papel importante em estudos de híbridos interespecíficos (Lima-Brito et al., 2006), entre outros.
Os parâmetros morfológicos mensuráveis são uma técnica simples de quantificação de variação genética, que pode dar simultaneamente informação sobre a performance de um genótipo em condições ambientais relevantes (Fufa et al., 2005).
No entanto, os marcadores morfológicos apresentam várias limitações, nomeadamente, pequeno número de marcadores diferentes (Rao, 2004), forte influência ambiental, principalmente no que concerne às características quantitativas (Karp et al., 1997; Rao, 2004), estão sujeitos a epistasia e ao pleiotropismo (Tanksley, 1983; Ferreira e Grattapaglia, 1996).
1.7.2. Marcadores moleculares
Os marcadores moleculares abrangem os marcadores bioquímicos e os marcadores de DNA e podem ser classificados em dominantes ou co-dominantes, de acordo com a informação fornecida sobre a heterozigocidade. Os marcadores co-dominantes permitem a distinção entre indivíduos homozigóticos e heterozigóticos, o que constitui uma vantagem (Laurentin, 2009).
Actualmente existe um grande número de marcadores moleculares com aplicabilidade no mapeamento genético (Moore et al., 1995; Gale e Devos, 1998), na selecção assistida por marcadores (Alzate-Marin et al., 2005), em “gene-tagging” (Tanksley, 1983; Ammiraju et al., 2001, 2002; Dayteg et al., 2008), em DNA “fingerprinting” de híbridos interespecíficos (Carvalho et al., 2005, Lima-Brito et al., 2006) e de espécies florestais (Carvalho et al., 2009b), na estimativa de relações
filogenéticas, certificação de clones e estudos evolutivos (Brown, 2004), entre outras.
1.7.2.1. Marcadores bioquímicos
As isoenzimas são formas moleculares estruturalmente diferentes da mesma enzima, com a mesma função catalítica (Kumar, 2009). As isoenzimas originam-se por alteração de aminoácidos, o que resulta em alterações de carga ou de conformação espacial, mudando também a mobilidade electroforética. Pode-se assim detectar a variação alélica por electroforese e posterior coloração específica (Hadacová e Ondrej, 1972; Vallejos, 1983; Soltis e Soltis, 1989).
As isoenzimas são marcadores co-dominantes e altamente reprodutíveis, simples, rápidos e fáceis de usar. Porém, são pouco abundantes e pouco polimórficos, pode ocorrer co-migração de proteínas não homólogas e estão sujeitos às condições ambientais. O perfil de bandeamento obtido pode variar dependendo do tipo de tecido utilizado (raiz vs. folha), uma vez que um gene pode estar a ser expresso num tecido e não noutros (Agarwal et al., 2008).