UNIVERSIDADE DE SÃO PAULO
INSTITUTO DE QUÍMICA
Programa de Pós-Graduação em Ciências Biológicas
(Bioquímica)
MARIA CLAUDIA PEREDA FRANCISCHINI
Caracterização dos fatores sigma da RNA polimerase do
fitopatógeno
Xanthomonas axonopodis
pv. citri
São Paulo
Data de Depósito na SPG:
MARIA CLAUDIA PEREDA FRANCISCHINI
Caracterização dos fatores sigma da RNA polimerase do
fitopatógeno
Xanthomonas axonopodis
pv. citri.
Tese apresentada ao Instituto de
Química da Universidade de São Paulo para
obtenção do título de Doutor em Ciências (Bioquímica).
Orientador: Prof. Dr. Shaker Chuck Farah
São Paulo
MARIA CLAUDIA PEREDA FRANCISCHINI
Caracterização dos fatores sig a da RNA poli erase do fitopatóge o
Xanthomonas axonopodis pv. citri.
Tese apresentada ao Instituto de
Química da Universidade de São Paulo para
obtenção do título de Doutor em Ciências
(Bioquímica).
Aprovado em: __________________
Banca Examinadora
Prof.Dr._______________________________________________________________ Instituição:____________________________________________________________ Assinatura:____________________________________________________________
Prof.Dr._______________________________________________________________ Instituição:____________________________________________________________ Assinatura_____________________________________________________________
Prof.Dr._______________________________________________________________ Instituição:____________________________________________________________ Assinatura_____________________________________________________________
Ao meu marido Carlos pelo amor,companheirismo, pelo incessante apoio e compreensão À minha filha Manuela pelo amor incondicional e por proporcionar momentos maravilhosos na minha vida Ao meu pai Alfonso (“in memorium”) e a minha mãe Francis
AGRADECIMENTO
Ao meu marido Carlos, que sempre esteve disponível, me ajudando, criticando quando necessário, sugerindo idéias e orientando. Muito obrigada, você foi muito importante ao longo do desenvolvimento desse trabalho.
Ao meu orientador, Prof. Dr. Shaker Chuck Farah pela confiança depositada em mim para a realização deste projeto de pesquisa.
À Dra. Cássia Docena com quem tive o grande prazer de trabalhar durante a minha iniciação científica e boa parte do meu doutorado, pelo que me ensinou com sua dedicação, paciência e profissionalismo e principalmente pelas constantes demonstrações de amizade. Muito obrigada
Aos meus amigos e colegas de laboratório: da primeira geração Cássia, Marcos, Éric, D.Cleusa e Alessandra, da segunda geração Paola, Max, Miryam, Luci, Ilda, Camila, Fernando, Diorge, Cristiane e finalmente da terceira geração Pricila, Raphael e Maycon pelo apoio, pela amizade pelo companheirismo, e pelas discussões científicas e pelo bate papo na hora do café.
À minha amiga Kátia por ter me ensinado e apresentado o “mundo do RNA”.
Por fim, aos meus pais Alfonso (“in memoriun”) e Francis pela boa educação, pela dedicação
e muitas vezes o sacrifício para oferecer o melhor. Não foi nada fácil, mas valeu a pena e com certeza tudo o que eu sou eu devo a vocês.
“O tempo é muito lento para os que espera Muito rápido para os que tem medo Muito longo para os que lamentam Muito curto para os que festejam Mas, para os que amam, o tempo é eterno”
RESUMO
Francischini, M.C.P. Caracterização dos fatores sigma da RNA polimerase do
fitopatógenos Xanthomonas axonopodis pv. citri. 2010. 177p. Tese (Doutorado) Programa
de Pós-Graduação em Ciências (Bioquímica). Instituto de Química, Universidade de São
Paulo, São Paulo.
A citricultura é de grande importância para as atividades agrícolas brasileiras, uma vez que o
Brasil é o principal produtor e exportador de suco de laranja. O cancro cítrico, causado pela
bactéria Xanthomonas axonopodis pv. citri (Xac) é um grave problema nesse setor, causando um elevado prejuízo na produção de frutos e seus derivados. O fator sigma é a subunidade da
RNA polimerase que tem a função de direcionar o núcleo da RNA polimerase a uma classe
específica de sequências promotoras. Como a maioria das bactérias sintetiza diversos fatores
sigma, essa característica proporciona à bactéria a oportunidade de manutenção basal da sua
expressão gênica, assim como, a regulação em resposta a alterações ambientais e a sinais
durante o desenvolvimento bacteriano. O genoma de Xac codifica para 14 fatores sigma. Nesse presente trabalho, detectamos interações dos fatores σECF (Xac2814. Xac3989,
Xac0922, Xac1319, Xac1380, Xac1682, Xac4129 e Xac2191) e seus fatores anti-σ cognatos
(Xac2815. Xac3988, Xac0921, Xac1320, Xac1379, Xac1681, Xac4130 e Xac2192). Além
disso, observamos interações entre o fator σFliA (Xac1933) e o anti-σFlgM (Xac1989), seu fator
anti-σ cognato. A caracterização das cepas nocautes para alguns fatores σ apontaram o
envolvimento do fator σ54
Xac1969 no mecanismo de formação de flagelo, a contribuição do fator
σECF
Xac1682 na resposta ao choque térmico e a participação do fator σECFXac2191 no crescimento
bacteriano em condições de carência de ferro.
Palavras-chave: Xanthomonas, fatores sigma, ECF (fator sigma função extracitoplasmática), TTSS
ABSTRACT
Francischini, M.C.P. Caracterization of RNA polimerase sigma factor of phythopathogen Xanthomonas axonopodis pv. citri. 2010. 177p. Ph.D. Thesis – Graduate Program in Biochemistry. Instituto de Química, Universidade de São Paulo, São Paulo.
Citriculture is an important sector of the economy of the State of São Paulo. Citrus canker,
caused by Xanthomonas axonopodis pv. citri (Xac), is a devastating disease responsible for large agribusiness losses every year. several sigma factors. The sigma factor is the subunit of
RNA polymerase that serves to direct the RNA polymerase core to a specific class of
promoter sequences. Most bacteria code for more than one sigma factor, which provides the
cell with the means by which to maintain basal gene expression while at the same time
modulate the expression of specific genes in response in environmental changes and signals
during bacterial growth. The Xac genome codes for 14 sigma factors which are the objects of
study in this thesis. We demonstrate that many of the sigma factors of the σECF family
(Xac2814, Xac3989, Xac0922, Xac1319, Xac1380, Xac1682, Xac4129 e Xac2191) interact
with cognate anti-σ factors (Xacβ815, Xacγ988, Xac09β1, Xac1γβ0, Xac1γ79, Xac1681,
Xac4130 e Xac2192). These sigma-anti-sigma pairs are all coded by neighboring genes.
Interactions between the sigma factor σFliA (Xac1933) and anti-σ
FlgM (Xac1989) were also
observed. Xac strains with gene knockouts for several sigma factors were produced. The
characterization some these knockout strains point to the involvement of σ54
Xac1969 in the
biosynthesis of flagella, participation of σECF
Xac1682 in the ability to survive heat shock and
involvement of σECF
Xac2191 in the response to iron deficiency.
Keywords: Xanthomonas, sigma factors, ECF (Extracytoplasmic function), TTSS (Type III secretion
LISTA DE FIGURAS
Figura 1: Microscopia eletrônica de transmissão mostrando o flagelo polar de
Xanthomonas axonopodis pv. citri (Xac)... 20
Figura 2: Doença cancro cítrico causada por Xanthomonas axonopodis pv.citri... 21
Figura 3. Modelo “ziguezague” ilustrando as etapas de desenvolvimento do sistema
imunológico da planta durante infecção... 24
Figura 4:Arquitetura da sequência primária dos fatores σ... 29
Figura 5: Arquitetura da estrutura primária do fator σ54... 30
Figura 6: Representação das regiões consensos -24 e -1β reconhecida pelo fator σ54.... 30
Figura 7: Esquema de formação do complexo aberto pela RNA polimerase contendo
o fator σ54e as proteínas da família ativadoras do fator σ54... 31
Figura 8: Mecanismo de regulação dos fatores σECF... 34
Figura 9: Modelo de transporte de citrato de ferro e mecanismo de regulação em
E.coli... 37
Figura 10: Mecanismo de transdução de sinal para ativação do fator σE em resposta a
estresses que levam ao desenovelamento de porinas de membrana externa... 41
Figura 11: Esquema simplificado de representação do flagelo... 42
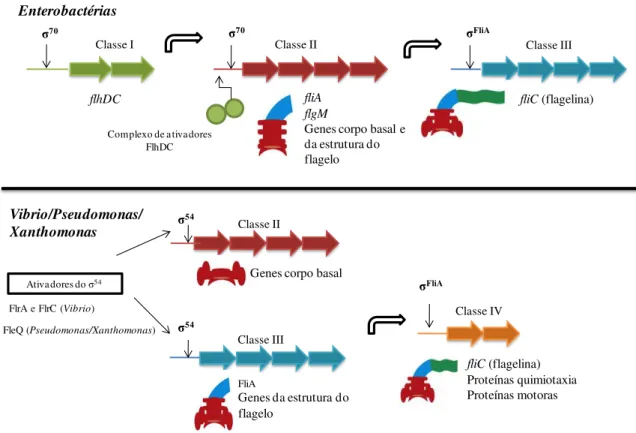
Figura 12: Representação esquemática do modelo de regulação dos genes flagelares... 44
Figura 13: Comparações das estruturas dos flagelos e do Sistema de Secreção do tipo
III... 46
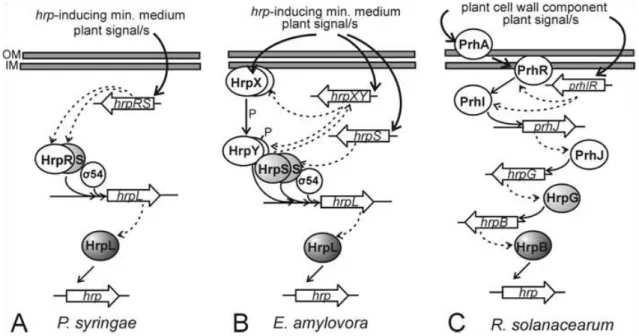
Figura 14: Modelo de regulação da cascata de ativação dos genes hrp.... 50
Figura 15:Mapa gênico do vetor pOBD e pOAD)... 56
Figura 16: Mapa do vetor pET28a(+), mostrando e o sítio múltiplo de clonagem
(Novagen)... 66
Figura 17: Esquema representando as regiões flaqueadoras dos genes selecionados
para a deleção... 71
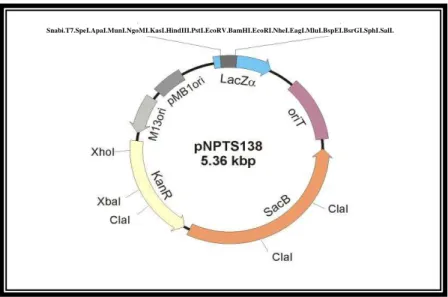
Figura 18: Mapa do vetor suicida pNPTS138 utilizado na produção dos nocautes
apolares... 72
Figura 19: Representação esquemática da seleção das colônias que sofreram a dupla
recombinação homóloga... 74
Figura 20:Mapa do vetor pUFR047... 75
Figura 21:Ensaio de motilidade realizado com a cepa ∆1969 em meio SB semi sólido
0,5 % agar... 89
Figura 22: Quantificação da produção de goma xantana, a porcentagem foi feita em
Figura 23: (A) Ensaio de motilidade do tipo “swimming” das cepas selvagem, ∆Xac1969 e ∆XacFliC realizado em tubos de ensaio de vidro contendo meio XMVβ 0,γ% agar. (B) Gráfico representando a zona de “swimming”, ou seja a distância da
motilidade em milímetros (mm). Os dados apresentados são a média de quatro experimentos independentes e o desvio padrão... 91
Figura 24: Ensaios de patogenicidade da cepa ∆Xac1969 realizados em folhas de
laranja pêra... 92
Figura 25: Representação esquemática dos domínios de transmembrana (TM) dos
fatores anti-σ identificados em Xac.... 94
Figura 26: Alinhamento dos fatores σE de E.coli, de P.aeruginosa e seus homólogos
mais similares em Xac (Xac1319, Xac1682 e Xac1380)... 97
Figura 27: Teste de patogenicidade da cepa mutante ∆Xac1682 realizado em folhas
de laranja pêra... 99
Figura 28: Ensaios de motilidade da cepa ∆Xac168β realizado em meio SB 0,5%
ágar... 99
Figura 29: Investigação da resposta do nocautes do fator σECF
Xac1682 a diferentes tipos
de estresses... 101
Figura 30: Comparação da organização gênica do sistema fecIRfecA de E.coli, prhIR
prhA de R.solancearum e os homólogos em Xac... 103
Figura 31: Alinhamento das regiões 2 dos fatores σ Xac4129 e Xac2191 e seus homólogos PrhI, FecI e PupI... 104
Figura 32: Representação das estruturas primárias dos fatores anti-σ FecR (E.coli),
PrhR (R.solancearum), Xac4130 e Xac2192 (Xac)... 106
Figura 33: Alinhamento dos fatores anti-sigma Xac2192 e Xac4130 com os
homólgos FecR (E.coli). e PrhR (R.solacearum)... 107
Figura 34: Representação das estruturas primárias dos receptores dependentes de TonB, FecA (E.coli), PrhA (R.solancearum), Xac4131 e Xac2193 de (Xac)... 108
Figura 35: Ensaios de duplo-híbrido realizados para confirmar as interações
observadas em ensaios de duplo-híbrido utilizando a biblioteca genômicade Xac... 110
Figura 36: Representação esquemática das interações observadas em ensaios de
triagem utilizando biblioteca genômica de Xac... 110
Figura 37: Gel de SDS-PAGE representando a purificação da proteína Xac2191... 111
Figura 38: Gel de SDS-PAGE 18%. Representação das etapas de purificação da
Figura 39: Gel de SDS-PAGE 15%. Representação da indução em larga escala de
σECF
Xac4129 na cepa BL21(D3)... 113
Figura 40: Gel de SDS-PAGE 18% que apresenta resultado dos testes de mini
indução de Xac41301-117 realizado em diferentes cepas de expressão e em diferentes
temperaturas (30ºC e 37ºC)... 114
Figura 41: Gel de SDS–PAGE 15%. Representação da purificação de Xac4129 em
uma coluna de níquel... 114
Figura 42: “Far-Western blot” ou “overlay” das proteínas Xac21916xHis-Xac2192 1-107... 115
Figura 43: Produtos de PCR gerados à partir do DNA genômico de Xac utilizando par
de oligonucleotídeos que amplifica a região de 300 pb a jusante do códon de iniciação da tradução para cada uma das proteínas homólogas a PrhJ... 118
Figura 44: Ensaios de EMSA utilizando as prováveis regiões promotoras dos
homólogos a PrhJ de R.solancearum... 119
Figura 45: Fotos do gel de agarose das reações de PCR de colônia para confirmar o
nocaute dos genes de interesse... 120
Figura 46: Teste de patogenicidade das cepas mutantes ∆Xacβ19β, ∆Xac41γ0,
∆Xacβ191, ∆Xac41β9 e ∆Xac41β9/∆Xacβ191... 121
Figura 47: Análise por qRT-PCR comparando a expressões dos genes hrpG, hrpX e
hrcU na cepa selvagem de Xac... 123
Figura 48: Análise por qRT-PCR comparando a expressões dos genes hrpG, hrpX e
hrcU nas cepas tipo selvagem e nas mutantes ∆Xacβ19β e ∆Xac4130... 124
Figura 49: Análise por qRT-PCR comparando a expressões dos genes hrpG, hrpX e
hrcU nas cepas selvagem e nas mutantes ∆Xacβ191, ∆Xac41β9 e no duplo mutante
∆Xac41β9/∆Xacβ191.Os RNAs foram extraídos da cepa Wt e mutantes crescida em
meio XMV2... 125
Figura 50: Análise por qRT-PCR comparando as expressões dos genes homólogos a
prhJ (Xac0136, Xac0898, Xac1229, Xac1970, Xac2168, Xac3421,Xac3480, Xac3993
e Xac4180) nas cepas selvagem e mutantes ∆Xacβ191, ∆Xac41β9 e ∆Xac41β9/∆β191.
Os RNAs foram extraídos da cepa Wt e mutantes crescida em meio XMV2... 126
Figura 51: Teste de patogenicidade realizado com a cepa mutante do gene hrpG e com a cepa complementada com o gene hrpG no vetor pUFRO47 (∆hrpG +hrpG) em
folhas de laranja pêra... 127
Figura 52: Gel de agarose. A) Amplificação do fragmento de 1140pb para a clonagem
Figura 53: Análise por qRT-PCR comparando a expressões dos genes hrpG, hrpX e
hrcU nas cepas selvagem e na mutantes ∆hrpG... 128
Figura 54: Curva de crescimento das cepas de Xac Wt, ∆Xacβ191(A), ∆Xac41β9(B)
e ∆Xac41β9/∆Xacβ191(C) na carência de ferro... 131
Figura 55: Análise dos mutantes ∆Xacβ191, ∆Xac41β9 e ∆Xac41β9/∆β191 em
resposta ao choque térmico e ao estresse oxidativo...
132
Figura 56: Curva de crescimento das cepas de Xac Wt, ∆Xacβ191(C), ∆Xac41β9(D)
e ∆Xac41β9/∆Xacβ191(E) na presença de 1,5% etanol...
133
Figura 57: Ensaios de motilidade realizado meio SB 0,5% ágar. Xac Wt, ∆Xacβ191,
∆Xac41β9, ∆Xac41β9/∆Xacβ191, ∆Xacβ19β e ∆Xac4130...
134
Figura 58: Modelo de regulação entre o fator sigma FliA e o anti-sigma FlgM... 138
Figura 59: Modelo de regulação σB em B.subtilis.... 139
Figura 60: Representação esquemática das interações observadas envolvendo o
putativo estressomo (Xac 1269/Xac1270/Xac1271 em Xac e a comparação com as interações envolvendo os dois módulos RsbR/RsbT/RsbT e RsbU/RsbV/RsbW conhecidas em B.subtilis... 141
Figura 61: Representação da organização gênica do lócus de 22kbp no genoma de
Xac, contendo os genes que codificam o fator σECF
Xac1682, o anti-σXac1681 e a serina
protease (Xac1680)... 144
Figura 62: Representação do putativo mecanismo de transdução de sinal que ativa o
fator σECF
Xac1682... 145
Figura 63: Representação da esquemática da via PhoQP de ativação transcricional de
hrpG e da ativação pós-traducional do regulador de virulência HrpG em
LISTA DE TABELAS
Tabela 1: Lista de oligonucleotídeos utilizados nas clonagens no vetor pOBD... 55
Tabela 2: Ensaio de auto-ativação para HIS3 e ADE2... 60
Tabela 3: Lista das proteínas utilizadas como iscas nas triagens de duplo-híbrido, o
número de tranformantes obtidos em cada ensaio e o número de clones positivos... 62
Tabela 4: Lista de oligonucleotídeos utilizados nas clonagens nos vetores pOAD e pOBD para os ensaios de duplo-híbrido e no vetor de expressão pET28a para a expressão das proteínasrecombinantes... 65
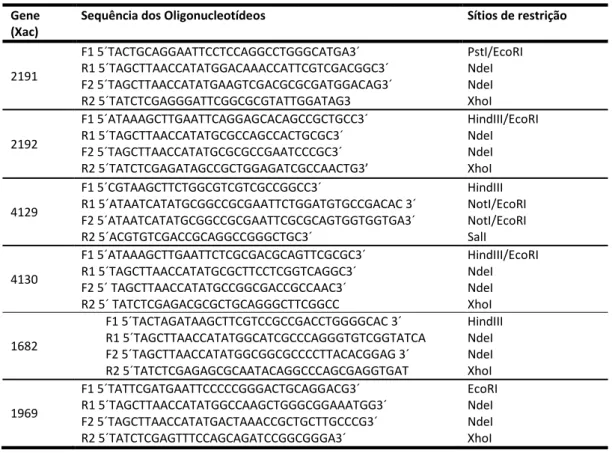
Tabela 5: Sequência dos oligonucleotídeos utilizados para a produção dos nocautes.... 71
Tabela 6: Lista dos oligonucleotídeos utilizados para amplificar os fragmentos de 300pb utilizados nos ensaios de EMSA... 76
Tabela 7: Oligonucleotídeos utilizados nos experimentos de qRT-PCR... 80
Tabela 8: Lista dos genes selecionados para os ensaios de duplo-híbrido... 82
Tabela 9: Resumo das interações obtidas na triagem no sistema duplo-híbrido de
levedura utilizando-se as diferentes iscas de interesse... 84/85
Tabela 10: Lista dos fatores σ ECFs de Xac e seus prováveis fatores anti-σ cognatos..... 94
Tabela 11: Proteínas de Xac homólogas a PrhJ de R. Solanacearum... 117
Tabela 12:Relação de similaridade entre os três fatores σECF (Xac1319, Xac1682 e
Xac1380) de Xac e o fator σE de E.coli, P. aeruginosa, X. fastidiosa e B. pseudomallei.... 143
LISTA DE ABREVIATURAS
Xac-Xanthomonas axonopodis pv. citri 306 Xcc-Xanthomonas campestris pv. campestris Xoo-Xanthomonas oryzae pv. oryzae
Wt- tipo selvagem
ECF-fator sigma (função extracitoplasmática)
TTSS-Sistema de secreção do tipo III
hrp–genes resposta hipersensibilidade e patogenicidade aa-aminoácidos
PCR- reação em cadeia da polimerase
DNA–ácido desoxirribonucléico
dNTPs–dideoxinucleotídeos trifosfato
DO–densidade óptica
kDa–103 Dalton
SUMÁRIO
1. Introdução... 19
1.1 O cancro cítrico e o fitopatógeno Xanthomonas axonopodis pv. Citri... 19
1.1.1 Xanthomonas axonopodis pv. citri (Xac)... 20
1.2 Sistema imune da planta... 22
1.3 Processo de transcrição em procariotos e o papel especial dos fatores sigma... 24
1.4 Classificação dos fatores sigma... 26
1.4.1 Família do fator σ70... 27
1.4.β Família do fator σ54... 29
1.5 Fatores sigma de funções extracitoplasmáticas (σECF)... 33
1.5.1 O fator σFecI e a captação de ferro... 35
1.5.2 O papel dos fatores σECF na resposta ao estresse... 39
1.6 Biossíntese de flagelo... 42
1.7 Sistema de Secreção tipo III (TTSS)... 44
1.8 Fatores sigma de Xanthomonas axonopodis pv. citri... 50
2. Objetivos... 53
3. Materiais e Métodos... 54
3.1 Clonagem dos genes Xac3788, Xac1969, Xac2972, Xac3824, Xac2814, Xac0922 Xac1682, Xac1319, Xac1380, Xac3989, Xac4128, Xac2191, Xac2192, Xac4129, Xac4130, Xac1269, Xac1270, Xac1271 e Xac0571 no vetor pOBD... 54
3.1.1Amplificações dos genes como isca no sistema duplo-híbrido de levedura... 54
3.2 Transformações de células competentes E.coli DH10B... 56
3.3 Extração de DNA plasmidial em pequena escala... 57
3.4 Reações de seqüenciamento... 57
3.5 Identificação de interações proteína-proteína por ensaios de duplo-híbrido em leveduras... 58
3.6. Teste de auto-ativação com os genes de interesse... 59
3.7 Transformação em larga-escala da linhagem PJ69-4a com a biblioteca de Xac... 60
3.8 Extração do DNA plasmidial de levedura em placa... 63
3.9 Cotransformações dos vetores pOAD e pOBD na levedura PJ69-4a... 64
3.10 Expressão de proteínas recombinantes... 65
3.12 Ensaios de “Far-Western blot” ou “Overlay”……… 69
3.13 Produção dos nocautes não polares... 70
3.13.1 Produção do fragmento de 2000pb para a clonagem no vetor pNPTS138... 70
3.14 Transformação em células competentes de Xanthomonas... 72
3.14.1 Preparo de células competentes de Xanthomonas... 72
3.14.2 Transformações das células competentes por eletroporação... 72
3.15 Seleção dos clones positivos... 73
3.16 Construção do plasmídeo de expressão do gene hrpG em Xac... 74
3.17 Ensaios de mobilidade eletroforética (EMSA)... 75
3.18 Caracterizações das cepas de Xac mutantes... 76
3.18.1 Teste de patogenicidade em plantas... 76
3.18.2 Ensaios de motilidade... 76
3.18.3 Teste de sobrevivência a diferentes estresses (choque térmico, estresse oxidativo e presença de etanol)... 77
3.18.4 Curva de crescimento em carência de ferro... 78
3.19 Ensaios de PCR quantitativo em tempo real... 78
4. Resultados... 81
4.1 Seleção das proteínas de Xac para serem analisadas em ensaios de duplo-híbrido.... 81
4.2 Análises das interações proteína-protéina através dos ensaios de duplo-híbrido de levedura... 83
4.3 Interação do fator anti-σXac1989 e o fator σFliAXac1933... 86
4.4 Interações envolvendo o putativo estressomo RsbRST... 86
4.5 Caracterizações dos fatores σ54 de Xac... 87
4.5.1 Interações envolvendo fatores σ54observadas no sistema duplo-híbrido... 87
4.5.β Estudo da relação do fator σ54 Xac1969 na formação de flagelos... 88
4.5.γ Estudo da relação do fator σ54 Xac1969 na virulência... 92
4.6 Interações envolvendo fatores sigma da subfamília ECFs... 92
4.6.1 Interação do fator σECF Xac2814 com o fator anti-σXac2815... 94
4.6.β Interação do σECF Xac3989 com o anti-σXac3988, homólogos de PrtI e PrtR... 95
4.6.γ Interações do σECF Xac0922 com o fator anti-σXac0921... 96
4.6.4 Interações dos fatores σE com os fatores anti-σ... 96
4.6.4.1 Investigação da relação do fator σECF Xac1682 com a virulência e motilidade... 98
4.6.4.β Investigação da participação do fator σECF Xac1682 na resposta aos estresses... 99
4.6.5. Interações dos fatores σECF Xac2191e σECFXac4129 com os fatores anti-σ Xac2192 e anti-σ Xac4130 respectivamente... 102
σECF
Xac2191e σECFXac4129... 103
4.6.5.2 Identificações dos domínios de transmembrana presentes nos fatores
anti-σXac2192 e anti-σXac4130... 104
4.6.5.3 Análise das proteínas sensoras Xac2193 e Xac4131 em comparação com as proteínas FecA de E.coli e PrhA de R.solancearum... 108
4.6.6 Investigações dos fatores σECF
Xac2191 e σECFXac4129 e dos fatores anti-σ Xac2192 e
anti-σ Xac4130 na cascata de ativação do sistema TTSS em Xac... 116
4.6.6.1 Ensaios de EMSA realizado com o fator σECF
Xac2191 e as regiões promotoras
dos possíveis homólogos a prhJ... 116
4.6.6.β Produção dos nocautes não polares dos fatores σECF
Xac2191, σECFXac4129 e dos
fatores anti-σXac2192 e anti-σXac4130... 120
4.6.6.3 Ensaios de patogenicidade realizados em folhas de laranja pêra... 121 4.6.6.4 Análise das expressões dos genes hrpG, hrpX, hrcU e dos homólogos a prhJ
(Xac0136, Xac0898, Xac1229, Xac1970, Xac2168, Xac3421, Xac3480, Xac3993 e Xac4180) por qRT-PCR... 122 4.6.6.5 Efeito da mutação dos genes Xac2192 e Xac4130 nas expressões dos genes
hrpG, hrpX e hrcU... 123 4.6.6.6 Efeito das mutações dos genes Xac2191 e Xac4129 nas expressões dos genes
hrpG, hrpX,hrcU e nos genes homólogos a prhJ... 124 4.7 Efeito da mutação do gene hrpG na patogenicidade... 126 4.8 Caracterizações das cepas mutantes ∆Xacβ191, ∆Xac41β9 e ∆Xac41β9/∆Xacβ191
nas respostas a diferentes estresses... 129 4.8.1 Crescimento na carência de ferro... 129
4.8.β Investigação dos fatores σECF
Xac2191 e σECFXac4129 na resposta ao choque térmico,
ao estresse oxidativo e na presença de etanol... 131 4.8.3 Investigação dos fatores σ ECF
Xac2191e σ ECFXac4129 e dos fatores anti-σXac2192 e
anti-σECF
Xac4130 no processo de motilidade bacteriana... 134
5. Discussão... 135
5.1 Interações dos fatores σECF com os fatores anti-σECF cognatos... 135
5.2 Interação envolvendo o fator sigma FliA responsável pela biossíntese de flagelo.... 137 5.3 O papel do putativo estressomo RsbRST em Xac... 138
5.4 O fator σECF
Xac1682 está envolvido na resposta ao choque térmico em Xac... 141
5.5 O fator σ54
Xac1969 está envolvido no mecanismo de formação de flagelo... 146
fatores anti-sigma Xac2192 e Xac4130 na cascata de ativação do sistema TTSS em
Xac... 150 5.7 A participação do fator σECF
Xac2191 na resposta a carência de ferro... 157
6-Conclusões... 161
7-Referências bibliográficas... 163
1. Introdução
A citricultura é de grande importância para as atividades agrícolas brasileiras, uma vez
que o Brasil é o principal produtor e exportador de suco de laranja, produzindo cerca de 30%
da produção mundial de laranjas-doces (Citrus ssp) e 59% da produção global de suco
concentrado congelado de laranja (Estanislau et.al., 2001). O sistema agroindustrial citrícola nacional movimenta cerca de R$9 bilhões anuais, gerando em torno de 400 mil empregos de
forma direta e indireta (Neves et al., 2004).
Com o avanço da tecnologia no campo no final do século XIX, as plantas cítricas
passaram a ser multiplicadas por enxertia no Brasil, o que trouxe grandes vantagens em
termos de precocidade e uniformidades dos pomares. Por outro lado, essa metodologia
diminuiu a variabilidade, tornando a cultura de citros um alvo constante às inúmeras pragas e
doenças, como por exemplo, as doenças do cancro cítrico, clorose variegada dos Citrus
(CVC), o “greening”, a tristeza e a morte súbita dos citrus.
1.1 O cancro cítrico e o fitopatógeno Xanthomonas axonopodis pv. citri
No ano de 1957, o cancro cítrico foi constatado pela primeira vez no Brasil, mais
especificamente no município de Presidente Prudente (SP) (Rossetti, 1977). A doença foi
posteriormente verificada nos Estados do Paraná (1958), Mato Grosso (1959), Rio Grande do
Sul (1980), Santa Catarina (1985), Minas Gerais (1998) e mais recentemente no estado de
Roraima (2002) (Nascimento et. al., 2003).
Em São Paulo, conseguiu-se evitar até 1979 que a moléstia se alastrasse para a zona
citrícola de exportação, utilizando-se de diversas medidas de controle. Em 2005, o cancro
cítrico foi identificado em 170.165 plantas do Estado, sendo 7,5% em pomares comerciais. Os
sítios e chácaras foram responsáveis por 72% das ocorrências, enquanto que 20,5% foram
1.1.1 Xanthomonas axonopodis pv. citri(Xac)
O gênero Xanthomonas é formado por bactérias Gram-negativas, pertencentes ao grupo Gamma das Proteobactérias. Possui morfologia de bastonete reto, medindo entre 0,2 e
0,6 μm de diâmetro e 0,8 a β,9 μm de comprimento (Swings & Civerolo, 1993; Moreira et al., 2005), respiração aeróbica e motilidade através de um flagelo polar (monotriquia) (Figura 1).
A colônia possui um aspecto mucóide de cor amarela, devido à produção do pigmento
xanthomonadina e à produção de exopolissacarídeos (Goto & Hyodo, 1985).
Figura 1: Microscopia eletrônica de transmissão mostrando o flagelo polar e a morfologia de bastonete reto de
Xanthomonas axonopodis pv. citri (Xac). Cortesia do Prof. Dr.E.W.Kitajima, NAP/MEPA-ESALQ/USP.
A bactéria Xanthomonas axonopodis pv. citri (Xac) é o agente causador do cancro cítrico. O fitopatógeno penetra no hospedeiro através de aberturas naturais como estômatos,
hidatódios e lenticelas ou por ferimentos causados por espinhos, materiais de colheita,
máquinas e também pela larva minadora dos citros (LMC) (Gottwald & Graham, 2000). Ao
atingir o espaço mesofílico do hospedeiro, ocorre a multiplicação da bactéria nos espaços
intercelulares e a produção de exopolissacarídios (EPS), principalmente a goma xantana.
Conseqüentemente, há uma diminuição dos espaços intercelulares e um acúmulo de goma
xantana, resultando em um aspecto de encharcamento (“water-soaking”) no tecido infectado.
O primeiro sintoma visível é a hiperplasia, formada por pequenas manchas amarelas salientes
progridem. A progressão da doença leva a uma queda da produção de clorofila, caroteno e
xantofila na região de encharcamento, formando um halo clorótico (Figura 2A)
(Padmanabham et al., 1973). Quando a doença atinge um estágio avançado, as lesões nas folhas ficam corticosas (Figura 2B).
Nos frutos, a doença se manifesta com o surgimento de pequenas manchas amarelas
com um ponto marrom no centro, podendo ocupar grande parte da casca. As manchas são
salientes, com aspecto verrugoso e de cor marrom, sendo que em estágios mais avançados
podem romper a casca (Figura 2C). Nos ramos, as lesões são salientes e de cor parda (Figura
2D). A desfolhação e a absição prematura dos frutos ocorrem em plantas altamente infectadas
(GOTO et al., 1979), provocando danos irreversíveis que comprometem a produtividade.
Figura 2: Doença do cancro cítrico causada por Xanthomonas axonopodis pv. citri. (A) Folhas com lesões circundadas por um halo clorótico. (B) Escurecimento da lesão com aspecto corticoso. (C) Lesões agluninadas no fruto que podem romper a casca. (D) Lesões salentes e de cor parda presente nos ramos. Fonte: http:// www.
fundecitrus.com.br/doenças/cancro.html.
A forma de disseminação da bactéria é rápida e não depende de um vetor específico.
Um dos maiores agentes disseminadores é o próprio homem, através da circulação
indiscriminada de pessoas pelos pomares, materiais de colheitas, veículos, mudas e outros
materiais contaminados. Outra forma de disseminação pode ser o vento e a chuva,
caracterizando uma disseminação de curta distância, além da larva minadora de citros,
Phyllocnistis citrella, que ao formar galerias nas folhas, facilita a penetração e o desenvolvimento da bactéria na planta.
Os riscos para o agronegócio decorrente da presença do cancro cítrico são
para eliminar o cancro cítrico é a erradicação das plantas contaminadas. O cancro cítrico é
uma doença que possui características fundamentais que torna a forma de erradicação viável e
altamente desejável: 1) incapacidade de sobreviver fora da lesão por períodos prolongados; 2)
não possuir um vetor de disseminação específico; 3) os sintomas são facilmente identificáveis,
o que permite o diagnóstico relativamente rápido; 4) o hospedeiro se restringe a um intervalo
perene de árvores frutíferas (GRAHAM et al., 2004).
1.2 Sistema imune das plantas
As plantas, diferentemente dos mamíferos, não possuem células somáticas
especializadas de um sistema imune adaptativo. No entanto, possuem um sistema de
imunidade inato de cada célula, que permite reconhecer e responder ao patógeno de forma
autônoma. Além disso, uma sinalização sistemática pode ser desencadeada em resposta a
estímulos microbianos que preparam o tecido nativo para o ataque iminente.
Os padrões moleculares associados aos micróbios ou patógenos (M/PAMPs) são
reconhecidos através dos receptores PRRs (“pattern recognition receptors”) localizados na superfície da célula da planta, responsáveis por desencadear a resposta imune primária
(Agrios, 2005; Vivier & Malissen, 2005). A resposta primária induz mudanças na célula
vegetal, como o depósito de calose e o acúmulo de proteínas de defesa, como por exemplo
quitinases, glucanases e proteases, que afetam a colonização do patógeno (Van Loon, 2006).
O segundo mecanismo de defesa desenvolvido pelas plantas está relacionado com os
fatores de virulência. Muitos patógenos desenvolveram mecanismos sofisticados que
reprimem o reconhecimento dos M/PAMPS (Martinon & Tschopp, 2005). Muitos patógenos
bacterianos possuem um sistema de secreção do tipo III (TTSS), importante para injetar
fatores de virulência no interior da célula hospedeira, onde atuam para reprimir a resposta de
patogéno do sistema vascular ou dos espaços intercelulares, elas desenvolveram um
mecanismo de defesa que reconhecem os fatores de virulência (efetores) através das proteínas
NB-LRR, que possuem domínio de ligação a nucleotídeos (NB “nucleotide binding”) e
repetições ricas em leucina (LRR “leucine rich repeat”). O mecanismo de defesa mediado
por proteínas NB-LRR é eficaz contra patógenos que sobrevivem em tecidos vivos
(biotróficos), mas não tem efeito em patógenos que matam o tecido do hospedeiro durante a
colonização (necrotróficos) (Glazebrook, 2005).
O processo de evolução da interação entre os patógenos e o sistema imune da planta
pode ser representado como uma série de adaptações do hospedeiro e do patógeno que tem
características de uma corrida armamentista (Figura 3) (Jones & Dangl, 2006). Na primeira
fase, os M/PAMPs são reconhecidos pelos receptores PRRs (“pattern recognition receptors”),
desencadeando a resposta imune mediada por PAMPs (PTI “PAMP-triggered immunity”), que impede a colonização do patógeno. Na segunda fase, os patógenos conseguem introduzir
efetores no interior da célula, contribuindo para a virulência e interferindo na PTI, resultando
assim em proteínas efetoras suscetíveis (ETS “effector-triggered susceptibility”). Na terceira fase, um efetor é reconhecido especificamente por uma proteína NB-LRR, resultando na
resposta imune desencadeada por efetores ETI (“effector-triggered immunity”). Os efetores ETI amplificam e aceleram a resposta PTI, ocasionando a resistência a doenças e, geralmente,
desencadeando uma morte celular no local da infecção, chamada de resposta de
hipersensibilidade (HR). Na quarta fase, a seleção natural dos patógenos evita a ETI e prioriza
sua sobrevivência no hospedeiro, desencadeando a aquisição de efetores adicionais que
conseguem suprimir a ETI. Uma nova seleção natural desses efetores adicionais permite um
Ef etores patogênicos
Ef etores patogênicos
A
m
p
lit
u
d
e
d
a
d
e
fe
sa
baixa alta
Limear para HR
Limear para resistência efetiva
Figura 3. Modelo “ziguezague” ilustrando as etapas de desenvolvimento do sistema imunológico da planta durante infecção. Neste esquema, a amplitude final da resistência da doença ou suscetibilidade é proporcional à [PTI-ETS+ETI]. Na primeira fase, as plantas detectam a população microbiana (PAMPs, diamantes vermelhos), desencadeando uma resposta de imunidade (PTI). Na segunda fase, patógenos entregam com sucesso efetores que interferem com a PTI, desencadeando a susceptibilidade por efetores (ETS). Na terceira fase, um efetor (indicado na figura em vermelho, Avr) é reconhecido pelas proteínas NB-LRR, ativando a imunidade desencadeada por efetores (ETI), uma versão ampliada de PTI que muitas vezes induz a resposta de hipersensibilidade (HR). Na quarta fase, novos efetores surgem através do fluxo horizontal de genes (em azul), ajudando os patógenos a suprimir a ETI. A seleção por parte das plantas de novos alelos de proteínas NB-LRR, que podem reconhecer um dos efetores recém adquiridos, dispara novamente a ETI. Fonte: Jones e Dangl, 2006.
1.3 Processo de transcrição em procariotos e o papel especial dos fatores sigma
Em procariotos, ao contrário do que ocorre em organismos eucariontes, uma única
RNA polimerase é responsável pela síntese de mRNAs, rRNAs e tRNAs. A RNA polimerase
é uma das maiores proteínas bacterianas, sendo formada por duas subunidades α, uma
subunidade , uma subunidade ' e uma subunidade σ, também chamada fator sigma. As
subunidades alfa, beta e beta' são produtos dos genes rpoA, rpoB, rpoC, respectivamente, e formam o núcleo da RNA polimerase, também chamada de apoenzima (Archambault &
partir de um molde de DNA, mas é incapaz de iniciar a transcrição nos sítios apropriados,
pois não é capaz de distinguir entre promotores e outras sequências de DNA.
A RNA polimerase completa, conhecida como holoenzima, inclui o fator sigma,
responsável pelo reconhecimento do DNA com alta afinidade nas regiões promotoras dos
genes a serem transcritos, formando um complexo binário fechado e estável
(holoenzima/DNA) (Gross et al., 1998). Bactérias utilizam um fator σ70 para a transcrição da maioria dos genes, mas também possuem fatores σ alternativos para expressão de genes
específicos em condições especiais.
Na maioria dos casos estudados, as taxas com que os genes são transcritos variam
diretamente com a taxa com que seus promotores formam complexos de iniciação estáveis
com a holoenzima RNA polimerase. Uma análise das sequências de 300 promotores de E. colireconhecidos pelo fator σ70, identificou duas regiões consenso importantes nas posições
-10 (TATAAT) e -35 (TTGACA) do início da transcrição, separados por 15-19 nucleotídeos,
como sendo as regiões de reconhecimento dos fatores sigma (Hawley & McClure, 1983;
Harley & Reymolds, 1987).
Após a formação do complexo fechado, a holoenzima promove a abertura local da
dupla hélice (bolha), através do rompimento das pontes de hidrogênio e a exposição de cerca
de 17 pares de bases do promotor. Na região consenso -10 predominam nucleotídeos de
adenina e timina, o que facilita a formação do complexo aberto com a RNA polimerase
articulada corretamente à fita que será utilizada como molde. Após a formação da ligação
fosfodiéster entre dois ribonucleotídeos trifosfatos complementares às bases expostas na fita
molde, a enzima move-se e dissocia-se da região promotora, abrindo a dupla hélice mais à
frente e expondo novos nucleotídeos. Este movimento é acompanhado pela liberação do fator
σ através de uma mudança conformacional no núcleo enzimático da RNA polimerase,
para que ocorra processo de elongamento da cadeia, caso contrário, a enzima permaneceria
fortemente ligada ao promotor e não poderia deslocar-se sobre o DNA (Travers et al., 1973; Chamberlim, 1974).
O início da transcrição é o passo mais importante para a regulação da expressão gênica
em bactéria. A transcrição pode ser regulada por fatores transcricionais ativadores ou
repressores, que se ligam na região promotora, pelo próprio promotor, através de variações da
sequência consenso, por pequenos ligantes e também pela estrutura do cromossomo
(Browning & Busby, 2004). Um exemplo de um pequeno ligante que afeta a iniciação da
transcrição é o guanosina tetrafosfato (ppGpp), que é rapidamente sintetizada em resposta a
estresse nutricional. O ppGpp se liga a RNA polimerase nos sítios que incluem o N-terminal
da subunidade ´ e o C-terminal da subunidade (Toulokhonov et al., 2001). O mecanismo de inibição não é totalmente conhecido, porém sabe-se que o ppGpp pode desestabilizar o
complexo aberto (Baker et al., 2001), pode competir com a ligação de NTPs ou pode mudar a afinidade da apoenzima para fatores sigma diferentes (Jores & Wagner, 2003). Outra forma de
regulação gênica ocorre através da inibição do fator σ por um fator anti-σ cognato que impede
que o fator σ reconheça o núcleo da RNA polimerase, resultando na inibição de um conjunto
de promotores.
1.4 Classificação dos fatores sigma
A maioria das espécies bacterianas sintetiza um fator sigma principal (σ70) e outros
fatores sigma alternativos, os quais reconhecem diferentes seqüências consenso do DNA em
famílias de promotores específicos. Esses fatores tornam-se ativos ou tem sua concentração na
célula aumentada em resposta à variações ambientais, competindo com o fator σ principal
pelo núcleo da RNA polimerase (Gruber & Gross, β00γ). Logo, cada fator σ pode direcionar a
bactéria a oportunidade de manutenção basal da sua expressão gênica, assim como sua
regulação em resposta a alterações ambientais e a sinais durante o desenvolvimento. Baseados
na similaridade entre as suas seqüências primárias, os fatores σ podem ser agrupados em duas
famílias: os fatores σ70e os fatores σ54 (Wosten, 1998).
1.4.1 Família do fator σ70
A família do fator σ70 foi subdividida em grupos de acordo com as características
filogenéticas (Lonetto et al., 1992; Wosten, 1998). O grupo 1 é formado pelos fatores sigma primários, presentes em todas as bactérias. Eles são responsáveis pela transcrição da maioria
dos genes que são essenciais para a sobrevivência celular. O fator σ primário em E.coli é codificado pelo gene rpoD, conhecido como fator σ70, enquanto que em bactérias
Gram-positivas, este fator sigma é conhecido como σA (Wosten, 1998). O grupo 2 compreende os
fatores sigma da família σ70
com seqüências similares aos fatores do grupo 1, mas que não
são essenciais para o crescimento da célula, como por exemplo, os fatores σS de E.coli e σB
de B.subtilis, responsáveis pela resposta ao estresse. O grupo 3 compreende fatores sigma alternativos, que diferem significativamente na seqüência de aminoácidos em relação aos
fatores sigma primários. Este grupo inclui também os fatores sigma que controlam a
expressão de genes responsáveis pela biossíntese de flagelo, pela resposta ao choque térmico
e pela esporulação. Os membros do grupo 4, chamado de ECFs (fatores sigma de funções
extracitoplasmáticas) possuem baixa similaridade de sequência em relação aos demais grupos
da família σ70 e estão envolvidos na resposta a sinais extracitoplasmáticos.
Através de uma análise comparativa das sequencias das proteínas da família σ70 em
diferentes bactérias foi possível identificar quatro regiões conservadas (Helmann &
primários, atuando como um domínio inibitório que impede a ligação do fator σ livre ao
promotor reconhecido por ele, além de modular a ligação ao DNA e regular a formação do
complexo aberto, podendo assim induzir ou inibir um processo importante para o início da
transcrição (Wilson & Dombroski, 1997; Vuthoori et al., 2001). A presença dessa região somente nos fatores sigma primários provavelmente é devido à necessidade de uma regulação
mais rígida, uma vez que esses fatores estão em concentrações mais elevadas no citoplasma
em relação aos demais fatores sigma e assim podem competir melhor para a apoenzima. Os
fatores sigma primários reconhecem um maior número de promotores e a subregião 1.1
presente nos mesmos pode proporcionar uma maior flexibilidade no reconhecimento de
promotores distintos (Vuthoori et al, 2001).
A subregião 1.2 auxilia na formação do complexo aberto e no deslizamento da RNA
polimerase sobre o mesmo (Wilson & Dombroski, 1997) e está presente em todos fatores
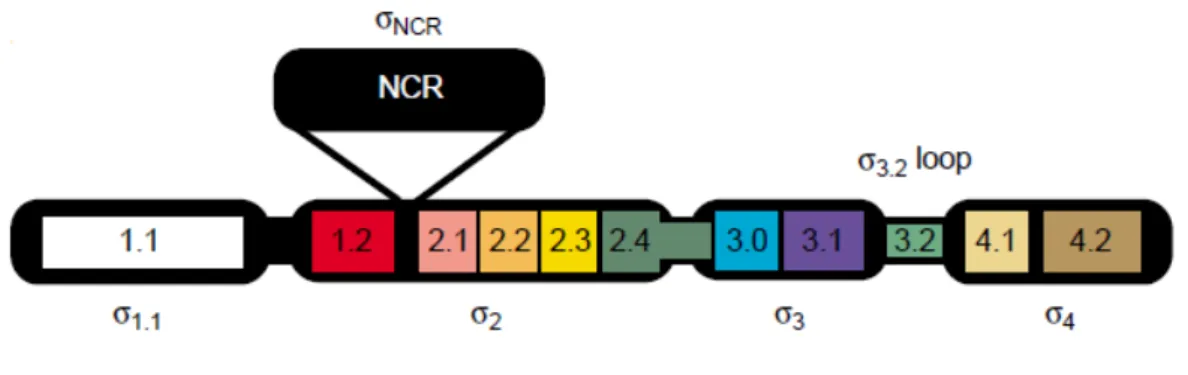
sigma, com exceção da subfamília ECF (Wosten, 1998). A região NCR, localizada entre as
regiões 1 e 2, é bastante divergente e está presente somente nos fatores sigma primários. A
sua função é desconhecida, embora existam evidências de que a NCR tem um papel na
ligação com o núcleo da RNA polimerase (Vassylyev et al., 2002)
A região 2 é a mais conservada e está presente em todos os fatores sigma, sendo
dividida em 4 subregiões (Lonetto et al., 1992). A região 2.1 possui resíduos para a interação com o núcleo da RNA polimerase, a região 2.3 atua na ligação ao DNA e na sua
desnaturação, enquanto que a região 2.4 reconhece a região -10 do promotor (Aiyar et al., 1994; Rong & Helmann, 1994).
A região 3 possui três subregiões e está presente nos fatores sigma ECFs (Lonetto et al., 1992). A região 3.0 reconhece as regiões -14 e -15 do promotor e as regiões 3.1 e 3.2 estão envolvidas na ligação ao DNA e ao núcleo da RNA polimerase, respectivamente (Barne
A região 4 está presente em todos os fatores sigma da família σ70 e possui duas
subregiões. A subregião 4.1 é responsável pela interação com ativadores transcricionais e a
subregião 4.2 reconhece a região -35 do promotor (Sharp et al., 1999; Ross et al., 1993).
Figura 4: Arquitetura da sequência primária dos fatores σ. A região 1.1, apresentada em branco, é a menos conservada. As regiões altamente conservadas σ2e σ3 estão representadas em cores. A região 4 presente em todos os fatores σ está representada em bege para a subregião 4.1 e em marrom para a 4.β. A região NCR presente somente nos fatores σ primários está destacada em preto (Murakami & Darst, 2003)
1.4.2 Família do fator σ54
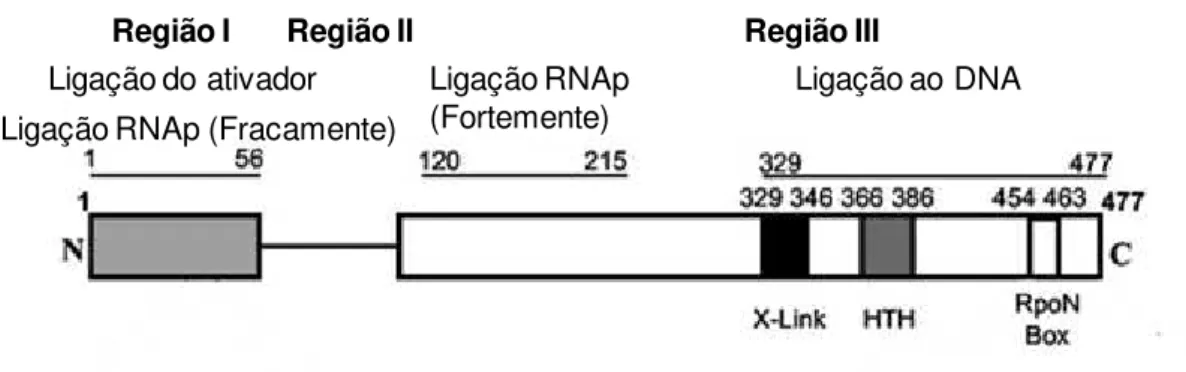
A família do fator σ54 é estrutural e funcionalmente distinta da família do fator σ70.
Baseado na homologia entre as proteínas σ54, três regiões diferentes podem ser destacadas
(Figura 5): A região I, rica em glutamina, que compreende os aminoácidos 1 ao 56; região II,
mais variável e rica em aminoácidos ácidos, que compreende os aminoácidos de 60 a 120 e a
região III, também chamada de C-terminal, que contém 400 aminoácidos e possui a região de
ligação ao DNA que consiste de dois motivos conservados, um domínio HTH
(hélice-volta-hélice) e um “box rpoN”, ambos envolvidos no reconhecimento de regiões promotoras
Ligação RNAp (Fracamente)
Região I Região II Região III
Ligação do ativador Ligação RNAp
(Fortemente)
Ligação ao DNA
Figura 5: Arquitetura da estrutura primária do fator σ54. Os domínios e os motivos envolvidos nas interações com o DNA, núcleo da RNA polimerase e a proteína ativadora estão indicados na figura. Na região III estão em destaque os resíduos importantes para a interação com o DNA: X-link, hélice-volta-hélice (HTH) e boxe RpoN (Burrows et al., 2003). RNAp: RNA polimerase.
A RNA polimerase associada ao σ54 reconhece promotores com regiões conservadas
nas posições -24 (região GG) e -12 (região GC), juntamente ao sítio de início da transcrição
(Figura 6) (Burrows et al., 2003).
Figura 6: Representação das regiões consensos -24 e -1β reconhecida pelo fator σ54 (Burrows et al., 2003).
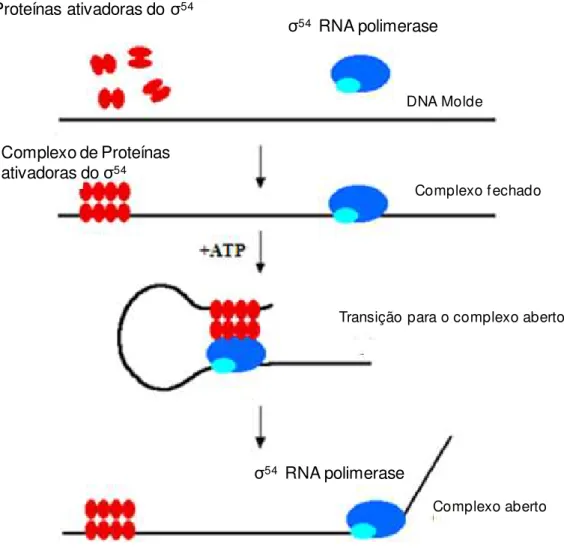
A iniciação da transcrição da holoenzima da RNA polimerase associada ao σ54 difere
da família do σ70. Enquanto a RNA polimerase contendo σ70 pode iniciar a transcrição na
ausência de ativadores transcricionais, a transcrição de todos os promotores dependentes do
σ54 requer a presença de uma proteína ativadora que se liga a uma região de
aproximadamente 100 pb ou mais localizada a montante do início da transcrição (Morris et
al, 1994). Para aproximar a proteína ativadora do complexo RNA polimerase associada ao
fator σ54 é necessária a curvatura do DNA entre os dois, formando um laço. Ao associar-se à
RNA polimerase contendo o fator σ54, as proteínas ativadoras, pertencentes a família de
ATPases AAA (ATPases associadas a várias atividades celulares), hidrolizam ATP,
ocasionando mudanças conformacionais que são associadas à transição entre o complexo
fechado e o complexo aberto (Figura 7), permitindo o início da transcrição (Buck et al., 2000; Zhang et al., 2002).
Proteínas ativadoras do σ54
Complexo de Proteínas ativadoras do σ54
σ54 RNA polimerase
σ54 RNA polimerase
DNA Molde
Complexo f echado
Complexo aberto Transição para o complexo aberto
A família de ativadores de fatores σ54 é composta por proteínas que compartilham
uma organização estrutural semelhante, contendo geralmente três domínios: a região
C-terminal, que contém o motivo HTH (hélice-volta-hélice) de ligação ao DNA; a região
central, responsável pela interação com o fator σ54, sendo a região mais conservada da
proteína e a região N-terminal, composta por um domínio sensor que varia entre as diferentes
proteínas ativadoras e geralmente regula a atividade ATPásica do domínio central
(Studholme & Dixon, 2003).
As diferenças na arquitetura da região N-terminal das proteínas ativadoras do fator σ54
estão relacionadas com a forma e o mecanismo como elas atuam. As proteínas ativadoras que
possuem um domínio sensor regulador de resposta (CheY-like domain) são ativadas através
da fosforilação de um resíduo de aspartato presente na região N-terminal por uma histidina
quinase. Outros ativadores possuem um domínio sensor da família PRD, que pode ser ativado
por fosforilação através do sistema de fosfotransferase dependente de fosfoenol-piruvato
(PTS). Já outros ativadores usam domínios PAS, GAF e V4R como sensores de fatores
ambientais (Studholme & Dixon, 2003).
O fator σ54 foi descoberto em estudos sobre a expressão da glutamina sintetase e a
assimilação de nitrogênio em enterobactérias (Hunt & Magasanik, 1995). Estudos realizados
posteriormente confirmaram o papel do σ54 na assimilação de nitrogênio e também seu
envolvimento em outras funções, incluindo resposta a carência de fonte de carbono,
1.5 Fatores sigma de funções extracitoplasmáticas (σECF)
As bactérias vivem em um ambiente que sofre constantes mudanças, por isso, a
alteração da expressão de proteínas na superfície celular é essencial para a adaptação e a
sobrevivência do organismo. As mudanças ambientais incluem osmolaridade, pressão (Chi &
Bartlett, 1995), fatores ambientais que afeta a composição da membrana (Missiakas et al., 1996), luz (Gorham et al., 1996), temperatura (Erickson & Cross, 1989) e concentração de nutrientes e micronutrientes (Missiakas & Raina, 1998).
Uma variedade de mecanismos é utilizada para sinalizar condições
extracitoplasmáticas para o citoplasma. Uma importante classe de mecanismos envolve a
ativação de fatores sigma específicos (σECF), que por sua vez recrutam a RNA polimerase para
ativar a transcrição de genes com a finalidade de expressar proteínas apropriadas para uma
determinada resposta. Além disso, foram descritos muitos casos em que fatores σECF
controlam a expressão de genes relacionados à virulência. Em Mycobacterium tuberculosis, a
letalidade em camundongos depende do fator ECF σC, enquanto que em Salmonella
typhimirium, a virulência depende do fator σE. Em Pseudomonas aeruginosa , o fator σAlgU
protege a bactéria de antibióticos, estresse oxidativo e da resposta imune do hospedeiro.
Alguns patógenos expressam genes relacionados à virulência em condições de carência de
ferro. Em Pseudomonas aeruginosa, o sideróforo (PvdA), que ajuda na captação de ferro, depende do fator sigma PvdS.
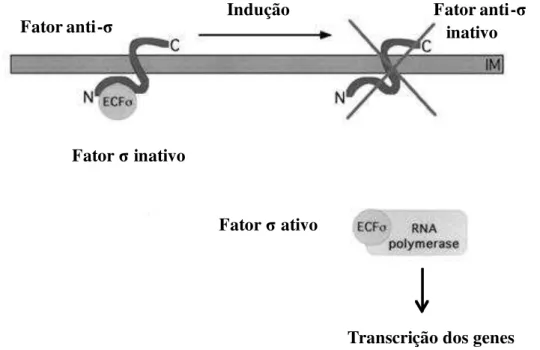
Os fatores σECF respondem a sinais do meio extracitoplasmático por um mecanismo de
transdução de sinal conservado (Figura 8). Vários σECF são co-transcritos com um fator anti-σ
cognato, uma proteína de membrana interna. A maioria dos fatores anti-σ possui um domínio
N-ternimal citoplasmático e o domínio C-terminal periplasmático separados por um segmento
de domínio transmembrana (Hughes & Mathee, 1998). De um modo geral, na ausência do
anti-σ cognato. Esses sinais extracitoplasmáticos específicos são reconhecidos pelo domínio
C-terminal do anti-σ, resultando na sua ativação e conseqüente liberação do fator σECF. O σECF
liberado, em conjunto com o núcleo da RNA polimerase, ativa a transcrição dos genes
relacionados à resposta ao sinal transmitido (Figura 8) (Raivio & Silhavy, 2001).
Indução
Fator σinativo
Fator σativo
Transcrição dos genes
Fator anti-σ Fator anti-inativo σ
Figura 8: Mecanismo de regulação dos fatores σECF. Na ausência do sinal, o fator σ está ligado ao fator anti-σ localizado na membrana interna. Na presença de um sinal extracitoplasmático o fator anti-σ é inativado, liberando o fator σ para interação com o núcleo da RNA polimerase, ativando, portanto, a transcrição dos genes (Raivio & Silhavy, 2001).
Os fatores anti-σ podem ser inativados por três mecanismos diferentes: 1) a eliminação do
fator anti-σ por secreção celular, mecanismo que será descrito na secção 1.6, sobre
biossíntese de flagelo; 2) degradação por protease, processo descrito na secção 1.5.2, sobre
controle do fator σE através de DegS e YaeL; 3) através da interação com um fator anti-anti-σ
que tem como função inibir a atividade do fator anti-σ através da interação proteína-proteína.
fosforilação. Estes tipos de mecanismos ocorrem, por exemplo, no controle da esporulação e
no controle de resposta a fatores estressantes em B. subtillis (Perego, 1998).
1.5.1 O fator σFecI e a captação de ferro
O ferro é um cofator de muitas enzimas com atividade redox e a maioria das bactérias
necessita de quantidades micromolares desse elemento para o crescimento normal (Guerinot,
1994). No entanto, a quantidade de ferro livre disponível é muito baixa no ambiente terrestre e
aquático, devido à rápida oxidação de produtos ferrosos (Fe2+) para íons férricos (Fe3+), na
forma de hidróxidos (Ratledge & Dover, 2000). Em eucariotos superiores existem
quantidades substanciais de ferro, no entanto, o metal se encontra fortemente associado a
proteínas de transporte e de armazenamento de ferro, não sendo disponível na forma livre para
as bactérias parasitas. Conseqüentemente, o nível de ferro livre nos fluídos biológicos é
estimado ser de 10-18M (Griffiths, 1978; Wienk et al., 1999). Dessa forma, as bactérias desenvolveram uma série de estratégias que garante às mesmas adquirir o ferro do ambiente
através da redução de íons férricos para ferrosos, além de obter o metal diretamente das
proteínas que carregam ferro do hospedeiro e captá-lo através de sideróforos.
A captação de ferro mediada por sideróforos é a forma mais comum de aquisição de
ferro entre as bactérias. Os sideróforos são importantes para a sobrevivência e crescimento
desses organismos no solo e em ambientes aquosos (Guerinot, 1994) e são essenciais para a
virulência de muitos patógenos (Ratledge & Dover, 2000). Sistemas para a captação de ferro
mediada por sideróforos foram descritos em bactérias Gram-negativas, embora bactérias
Gram-positivas, como Bacillus, Staphylococcus e Streptomyces, também possuem sistemas de captação de ferro por sideróforos.
A regulação da síntese de sideróforos ocorre através da proteína Fur, que requer íons
uma região especifica do promotor chamada fur-box e inativa a transcrição de genes
relacionados com a captação de ferro. Em situações de carência de ferro, a apo-Fur é incapaz
de se ligar ao DNA, permitindo assim a transcrição dos genes envolvidos no mecanismo de
captação de ferro (Hantke, 2001; Panina et al., 2001). A proteína Fur foi o primeiro regulador da síntese de sideróforos descrito, mas não é o único. Em muitas espécies bacterianas, a
expressão dos genes de captação de ferro depende de ativadores transcricionais (Crossa et al., 1997).
Outra forma de regulação da síntese de sideróforos e dos genes relacionados com a
captação de ferro envolve fatores σda subfamília ECF, mecanismo que permite à bactéria
responder a determinados sideróforos e à disponibilidade de ferro do ambiente. Em E.coli, o
fator σFecI regula a expressão do sistema de transporte de citrato de ferro (Fe3+) (sistema Fec).
A atividade de FecI é regulada por FecA e FecR. FecA é uma proteína de membrana externa
que recebe o sinal extracitoplasmático (presença de Fe3+ na forma de citrato) e transmite esse
sinal para o periplasma. A região N-terminal de FecA é importante para a sinalização de
FecR, um fator anti-σ de membrana citoplasmática que transmite o sinal para o citoplasma e
regula a atividade de FecI (Figura 9) (Braun et al., 2003).
FecA pertence à família de transportadores dependentes de TonB. A ligação da
membrana externa e do citoplasma está acoplada ao conjunto de proteínas TonB, ExbB e
ExbD. A proteína TonB ativa a proteína transportadora através da ligação de um
heptapeptídeo presente na região C-terminal de TonB e um motivo de ligação de TonB (TonB
Box) presente na região N-terminal de FecA (Figura 9) (Braun etal., 2005).
O fator σFecI regula a transcrição dos genes transportadores de citrato de ferro
codificam um complexo ABC de transporte associado à membrana citoplasmática. As
transcrições de fecIR e fecABCDE são reguladas pela proteína Fur (Figura 9).
Figura 9: Modelo de transporte de citrato de ferro e mecanismo de regulação em E.coli. Mudanças estruturais que ocorrem nos “loops” 7 e 8 de FecA após a ligação do citrato férrico capturam o citrato férrico na cavidade de FecA. A região Box TonB no N-terminal de FecA interage com o fragmento periplasmático de TonB. A topologia do sistema de transdução de energia TonB-ExbB-ExbD é mostrada na membrana citoplasmática. A figura representa as interações da região N-terminal de FecA com o domínio chamado motivo de leucina (LLLV) presente na região C-terminal de FecR, a interação do N-terminal de FecR com a região 4 de FecI e também a interação da região 3 de FecI com o núcleo da RNA polimerase. A ativação da transcrição dos genes
fecABCDE ocorre em baixos níveis de Fe2+, que resulta na dissociação da apo-Fur do Fur-boxe (P
fur). O esquema
O fator σFecI direciona o núcleo da RNA polimerase para o promotor dos genes
fecABCDE. A atividade de FecI é controlada por FecR, que protege FecI da degradação e da ação de protease, mantendo-o estável. Na ausência de FecR praticamente não há transcrição
dos genes fecABCDE (Braun et al, 2003). Células mutantes que contêm apenas a região N-terminal de FecR1-87 apresentam transcrição constitutiva dos genes fecABCDE, e as células
que contém uma porção maior do FecR, não contendo a região de interação com FecA,
também apresentam transcrição constitutiva dos genes fecABCDE, porém, mais fraca (Braun, 1997; Enz et al., 2000). Quando o sinal da presença de citrato de ferro (Fe3+) é percebido por FecA e transmitido para FecR, ocorre uma mudança conformacional em FecR e conseqüente
liberação de FecI para associar-se imediatamente ao núcleo da RNA polimerase. Nesse
modelo, o FecR atua tanto como uma chaperona quanto como fator anti-σ, uma vez que FecI
é mantido na forma inativa mas estável quando ligado ao FecR e torna-se ativo ao ser liberado
(Enz et al, 2000).
O mecanismo de regulação da transcrição do sistema Fec foi descrito em outras
espécies. Em Pseudomonas pudita, a expressão do transportador de ferro ocorre através do sideróforo pseudobactina, que induz a síntese de um transportador de membrana externa
Em Pseudomanas aeruginosa, a síntese do sideróforo pioverdina e o transporte da pioverdina férrica são controlados pelo sistema análogo ao FecIR. A pioverdina férrica
funciona como um indutor de pioverdina e também como um ativador dos genes
transportadores de pioverdina férrica, além de induzir a produção da exotoxina A e de
endoproteinase extracelular (PrpL) (Wildermam et al., 2001). O complexo pioverdina férrica é internalizado via proteína de membrana externa FpvA, um receptor dependente de TonB
(Poole et al., 1993). A ligação da pioverdina férrica a FpvA induz um sinal que é transmitido via membrana citoplasmática através do fator anti-σ FpvR para o fator σPvdS (Lamont
et al.,
β00β). O fator σPvdS se liga ao núcleo da RNA polimerase ativando a transcrição dos genes
relacionados.
Sistemas análogos ao FecAIR também foram identificados em Bordetella pertussis (VanderpooL & Armstrong, 2001), B. bronchiseptica (Pradel & Locht, 2001) e B. avium (Kirby et al., 2001). B. pertussis e B. bronchiseptica codificam dois genes reguladores hurI e hurR localizados à montante do conjunto de genes bhuRSTUV. A síntese de um possível transportador de sideróforos férrico de membrana externa (BfrZ) em B.bronchiseptica é regulada por duas proteínas, BupI e BupR, que são homólogas a FecI e FecR,
respectivamente. B. avium possui um sistema de utilização do grupo heme onde a síntese da BhuR, proteína de membrana externa relacionada ao transporte, é induzida na presença do
grupo heme e necessita do fator σ RhuI (Kirbyetal., 2001).
1.5.2 O papel dos fatores σECF na resposta ao estresse
O envelope celular das bactérias está sujeito a várias pertubações ambientais que
necessitam ser controladas para manter a integridade da célula. Em bactérias Gram-negativas,
intercompartimental, uma vez que os fatores de transcrição necessários para a resposta estão
localizados no citoplasma (Raivio, 2005).
Estudos realizados em E.coli monstraram que o fator σE é responsável por respostas à
estresses extracitoplasmáticos como choque térmico e presença de etanol (Mecsas et al., 1993; Missiakas et al., 1996). O fator σE foi caracterizado como um regulador global da
adaptação ao estresse extracitoplasmático e virulência em E.coli, P. aerunginosa e outras
bactérias (Helmann, β00β). O fator σE de P.aerunginosa (também conhecido como AlgU) foi identificado como um regulador do fator σH, relacionado à resposta ao choque térmico e
responsável pela síntese do exopolissacarídeo alginato (Govan & Deretic, 1996; Firoved et al., β00β; Firoved & Deretic, β00γ). O fator σE também tem sido descrito como um regulador
de muitos outros genes, como os relacionados com adesão, genes que codificam proteínas de
membrana, transportadores, proteases e enzimas metabólicas (Firoved et al., 2002; Firoved & Deretic, 2003). Em P. fluorescens, o fator σE foi descrito ser importante para a resposta ao estresse osmótico e por dessecação (Schnider-Keel et al., 2001). Em Salmonella entérica, o
fator σE está envolvido na resposta ao estresse oxidativo, pH ácido, choque térmico e carência
de carbono.
A via de transdução de sinal que ativa o fator σE em resposta a porinas de membrana
externa desenoveladas envolve um fator anti-σ (RseA), uma proteína periplasmática (RseB) e
as proteases DegS e YaeL (RseP) (Figura 10). As proteínas RseA e RseB são codificadas no
operon rpoE-rseABC dependente do fator σE e são autoreguladas pelo σE (Missiakas et al.,
1997). Na ausência do estresse, o fator anti-σ (RseA) mantém o fator σE inativo. Na presença
do choque térmico e na presença de porinas, a quantidade de RseA é reduzida drasticamente
devido à ação das proteases DegS e YaeL. O domínio PDZ de DegS interage com porinas
desenoveladas, ativando a função proteolítoca de DegS e YaeL, que cliva o sítio
associar-se ao núcleo da RNA polimerase e transcrever os genes do seu regulon (Figura 10)
(Ades, 2004; Alba & Cross, 2004; Rowley et al., 2006).