UNIVERSIDADE DE LISBOA
FACULDADE DE CIÊNCIAS
DEPARTAMENTO DE BIOLOGIA VEGETAL
Andreia Sofia Reis Carvalho
Mestrado em Biologia Molecular e Genética
Dissertação orientada por:
Professora Doutora Maria Helena Caria
Professora Doutora Ângela Inácio
2019
Estudo da variabilidade nos genes envolvidos nos mecanismos
de stresse oxidante ligados à perda auditiva em São Tomé e
Príncipe e comparação com a sua distribuição na população
Agradecimentos
Gostaria de agradecer em primeiro lugar à minha orientadora externa, a Professora Doutora Ângela Inácio, por toda ajuda, disponibilidade e orientação dada durante todo este percurso no Instituto Bento da Rocha Cabral, sem o seu apoio e incentivo nunca teria chegado até aqui.
À minha orientadora interna, a Professora Doutora Helena Caria por toda a ajuda, paciência e por me proporcionar todas as condições para a realização deste trabalho em colaboração com o Grupo de Investigação no âmbito do projeto “Saúde para todas as especialidades”.
Não posso deixar de agradecer ao Professor Doutor Manuel Bicho, Diretor do Laboratório de Genética da Faculdade de Medicina de Lisboa (FMUL), a oportunidade e esclarecimentos dados ao longo da realização deste trabalho.
Agradeço igualmente a todas as colaboradoras do laboratório de genética da FMUL, nomeadamente à Doutora Joana Ferreira, pela disponibilidade demonstrada.
Quero ainda agradecer aos meus colegas de laboratório Laura Aguiar, Raquel Carrilho, Sara Hassam, Denise Brito, Francisca Paixão e Alvaro Chao. Sem vocês este caminho não teria sido possível. Obrigada pela paciência, pelos sorrisos e por todos os ensinamentos que me transmitiram.
O meu agradecimento ao Instituto de Investigação Bento da Rocha Cabral e seus funcionários. Não menos importante quero agradecer à Rita, que sempre esteve lá. Obrigada Marta, foste um grande apoio neste processo.
Obrigada aos meus pais pela ajuda e apoio ao longo da vida académica. Obrigada também á minha irmã por tudo.
Resumo
A audição é o sentido responsável pela captação, condução e codificação das ondas sonoras em impulsos nervosos. A perda auditiva (PA) condiciona a integração dos indivíduos na sociedade.
O presente trabalho teve como principal objetivo o estudo da variabilidade em genes envolvidos em mecanismos de stresse oxidante e a sua associação com a PA em São Tomé e Príncipe (STP). Os genes em estudo foram MTHFR, CβS, GST (GSM1 e GSTT1) e NOS3. Para a deteção dos polimorfismos 844ins68 pb do gene CβS, M1*0 e T1*0 dos genes GST e do VNTR no intrão 4 do gene NOS3 foi usada a técnica PCR. Para a deteção do polimorfismo C677T do gene MTHFR foi usada a técnica do RFLP. Os dados estatísticos foram realizados no programa estatístico SPSS, sendo o nível de significância estatístico estabelecido para p <0,05.
Numa primeira fase foi realizado um estudo populacional, entre as populações portuguesa e a de STP, a fim de perceber se diferentes contextos genómicos no que diz respeito ao stresse oxidante poderiam justificar as elevadas taxas de PA em STP. Os resultados da análise populacional mostraram diferenças estatisticamente significativas na distribuição dos genótipos e dos alelos para todos os genes, verificando-se em maioria uma maior frequência dos alelos e genótipos, associados ao stresse oxidante em STP. Contudo, os estudos de associação com a PA na população de STP não mostrou diferenças estatísticas, com exceção do alelo ins do gene CβS que se revelou protetor em relação à doença. Os estudos de epistasia mostraram ainda uma associação entre a combinação de genótipos 4b4b do gene eNOS e CC do gene MTHFR com a PA.
Conclui-se que, apesar da maior incidência das variantes polimórficas em STP, relativamente à população portuguesa, esta parece não justificar a grande prevalência de PA na população africana.
Palavras-chave: Perda Auditiva, Stresse Oxidante, São Tomé e Príncipe, Estudos
Abstract
Hearing is the sense responsible for capturing, conducting and encoding sound waves in nerve impulses. Hearing loss (HL) has consequences in the integration of individuals into society.
The present work had as main objective the study of the genetic variability in genes involved in mechanisms of oxidative stress and its association with HL in São Tomé and Príncipe (STP). The genes under study were MTHFR, CβS, GST (GSM1 and GSTT1) and NOS3. PCR was used for the analysis of the polymorphisms: 844ins68 bp in CβS, M1 * 0 and T1 * 0 of GST and VNTR in intron 4 of the NOS3 gene. For the C677T polymorphism of the MTHFR gene, the RFLP technique was used. Statistical analyses were performed in the SPSS statistical program, and the level of statistical significance was established as p <0.05.
In a first phase of this work, a population study was carried out, between the Portuguese and the STP populations, in order to understand if different genomic contexts with respect to the oxidative stress could justify the high HL levels in STP. The results of the population analysis showed statistically significant differences in the distribution of genotypes and alleles for all genes, with a higher frequency of alleles and genotypes associated with oxidative stress in STP. However, the association studies with HL in the STP population did not reveal statistical differences, except for the ins allele of the CβS gene that was protective against the disease. Epistasis studies also showed an association between the combination of 4b4b genotypes of the eNOS gene and CC of the MTHFR gene with HL.
It is concluded that, despite the higher incidence of polymorphic variants in STP, relative to the Portuguese population, this does not seem to justify the high prevalence of HL in the African population.
key words: Hearing Loss, Oxidant Stress, Sao Tome and Principe, Population Studies,
Lista de Comunicações
Laura Aguiar, Ildegário Semente, Andreia Carvalho, Cristina Caroça, Helena Caria, Paula Faustino, Manuel Bicho, Ângela Inácio. “Genotypic differences between southwestern European and African populations: a comparative study in eNOS, G6PD, GSTM1, GSTT1 and CYB5R3 genes”. Sociedade Portuguesa de Genética Humana (Oral). Porto, 2018.
Laura Aguiar, Ildegário Semente, Andreia Carvalho, Joana Ferreira, Helena Caria, Albertino Damasceno, Paula Faustino, Ângela Inácio, Manuel Bicho.” Diferenças genotípicas entre o sudoeste da Europa e África: Um estudo comparativo em genes relacionados com a hipertensão”. 13º Congresso Português de Hipertensão e Risco Cardiovascular Global.Vilamoura, 2019.
Índice
Agradecimentos ... III Resumo ... III Abstrat ... IV Lista de Comunicações ... V Índice de Tabelas ... VIII Índice de Figuras ... IX Abreviaturas e Acrónimos ... X
1.Introdução...1
1.1. Perda Auditiva ...1
1.2. O Ouvido Humano ...1
1.3. As Principais Causas de Perda Auditiva Neurossensorial ...2
1.4. Stresse Oxidante e Perda Auditiva Neurossensorial ...3
1.4.1. Metabolismo da Homocisteína...4
1.4.2.Metileno Tetrahidrofolato Redutase e Perda Auditiva ...5
1.4.3. Cistationina β-Sintase e Perda Auditiva ...5
1.4.4. Gluatationa S-Transferase e Perda Auditiva ...6
1.4.5. Sintase do Óxido Nítrico e Perda Auditiva ...6
1.4.5. 1 Relação do Metabolismo do Óxido Nítrico com o Metabolismo da Homocisteína ...7
1.4.6. Glucose-6-Fosfato Desidrogenase (G6PD) e Perda Auditiva ...7
1.5. Epistasia ...7
1.6. Genética Populacional ...7
2. Objetivos ...9
3. Metodologia ... 10
3.1. Seleção da Amostra Populacional ... 10
3.2. Análise Genética ... 10
3.2.1. Amplificação dos Fragmentos de DNA a Estudar ... 10
3.2.2. Genotipagem ... 12 3.3. Tratamento Estatístico ... 12 3.3.1 Estudo Populacional ... 12 3.3.2 Estudos de Associação ... 12 3.3.2.1 Análise Isolada ... 13 3.3.2.2 Análise Epistática ... 13 4. Resultados ... 14
4.1. Caraterização da População em Estudo ... 14
4.1.1. Caraterização da População quanto à Infeção por Malária ... 14
4.1.3. Caraterização da População quanto à Perda Auditiva ... 14
4.1.3.1. Caraterização da População quanto à gravidade na Perda Auditiva ... 14
4.1.3.2 Caraterização da População quanto à lateralidade na Perda Auditiva ... 15
4.2. Estudo Populacional das Distribuições Genotípicas e Alélicas nos Genes MTHFR, CβS, GST (M1 e T1) NOS3 e G6PD em STP e Portugal ... 16
4.2.1. Caraterização das Populações em Estudo ... 16
4.2.1.1. Estudo do Gene MTHFR nas Populações de STP e Portuguesa ... 16
4.2.1.2. Estudo do Gene CβS nas Populações de STP e Portuguesa ... 17
4.2.1.3. Estudo do Gene GSTM1 nas Populações de STP e Portuguesa ... 17
4.2.1.4. Estudo do Gene GSTT1 nas Populações de STP e Portuguesa. ... 18
4.2.1.5. Estudo do Gene NOS3 nas Populações de STP e Portuguesa ... 18
4.2.1.6 Estudo do Gene G6PD nas Populações de STP e Portuguesa... 19
4.3 Análise isolada dos genes MTHFR, CβS, GST (M1 e T1) e NOS3 ... 19
4.3.1. Análise do Polimorfismo C677T do Gene MTHFR... 19
4.3.2. Análise do Polimorfismo de Inserção de 68 pb no exão 8 do Gene CβS ... 20
4.3.3. Análise dos Polimorfismos nos Genes GST (M1 e T1) ... 21
4.3.4.Análise do Polimorfismo VNTR do Gene NOS3 ... 22
4.4 Estudo de Relações Epistáticas ... 22
5. Discussão e Conclusão ... 24 Referências Bibliográficas ... 27 Anexos ... 33 Anexo 1 ... 33 Anexos 2 ... 34 Anexos 3 ... 35 Anexos 4 ... 36
Índice de Tabelas
Tabela 3.1 Polimorfismo, primers, condições de amplificação e tamanho esperado dos fragmentos………..11 Tabela 3.2 Condições de Restrição e Possíveis Tamanhos dos Fragmentos do Polimorfismo MTHFR……….…...12 Tabela 4.1. Frequência e número de indivíduos do sexo masculino e feminino quanto ao grau de severidade na PA………...……...15 Tabela 4.2. Média e desvio padrão das idades nas diferentes categorias de gravidade da PA e no grupo de controlo……….……….15 Tabela 4.3. Frequência e número de indivíduos do sexo masculino e feminino relativamente à lateralidade na PA………...15 Tabela 4.4 Média e desvio padrão das idades na PA unilateral e bilateral……….16 Tabela 4.5 Distribuição das frequências genotípicas do polimorfismo C677T nas populações de STP e Portuguesa………...16 Tabela 4.6 Distribuição das frequências alélicas do polimorfismo C677T do gene MTHFR nas populações de STP e Portuguesa……….………16 Tabela 4.7 Distribuição das frequências genotípicas do polimorfismo de inserção de 68 pb do gene CβS na população de STP e Portuguesa………..………17 Tabela 4.8 Distribuição das frequências alélicas do polimorfismo de inserção de 68 pb no intrão 8 do gene CβS na população de STP e Portuguesa………...17 Tabela 4.9 Distribuição das frequências genotípicas dos alelos nulos (M1*0) e não nulo (M1*1) do gene GSTM1 nas populações de STP e Portuguesa………..………...………17 Tabela 4.10 Distribuição das frequências genotípicas dos alelos nulos (T1*0) e não nulo (T1*1) do gene GSTT1 nas populações de STP e Portuguesa………..18 Tabela 4.11 Distribuição das frequências genotípicas do polimorfismo VNTR do gene NOS3 nas populações de STP e Portuguesa………...………...18 Tabela 4.12 Distribuição das frequências alélicas do polimorfismo VNTR do gene NOS3 nas populações de STP e Portuguesa………...19 Tabela 4.13 Distribuição das frequências genotípicas da variante A+ do gene G6PD nas populações de STP e Portuguesa………19 Tabela 4.14 Distribuição das frequências alélicas da variante A+ do Gene G6PD nas populações de STP e Portuguesas………...………...19 Tabela 4.15 Distribuição das frequências genotípicas do polimorfismo C677T do gene MTHFR………...………...20 Tabela 4.16 Distribuição das frequências alélicas do polimorfismo C677T na população ouvinte e na população surda………..20
Tabela 4.17 Distribuição das frequências genotípicas de ins68 pb no intrão 8 do gene CβS na população ouvinte e na população surda………...20 Tabela 4.18 Distribuição das frequências alélicas do polimorfismo de ins68 pb no intrão 8 do gene CβS na população ouvinte e na população surda……….21 Tabela 4.19 Distribuição das frequências genotípicas dos alelos nulos (M1*0) e não nulo (M1*1) do gene GSTM1 na população ouvinte e na população surda………..21 Tabela 4.20 Distribuição das frequências genotípicas dos alelos nulos (T1*0) e não nulos (T1*1) do gene GSTT1 na população ouvinte e na população surda………22 Tabela 4.21 Distribuição das frequências genotípicas do polimorfismo VNTR do gene NOS3 na população ouvinte e na população com PA……….22 Tabela 4.22 Distribuição das frequências alélicas no polimosfismo VNTR do gene NOS3 na população ouvinte e na população não ouvinte………22 Tabela 4.23 Associação significativa entre os genótipos dos genes MTHFR e NOS3 com a PA……..23
Índice de Figuras
Figura 1.1 Esquema representativo do ouvido humano……….2 Figura 1. 2 Esquema simplificado do metabolismo da hcy………5
Abreviaturas e Acrónimos 5-MTHF: 5- Metilenotetrahidrofolato 5,10-MTHF: 5,10- Metilenotetrahidrofolato CAT: Catalase CβS: Cistationina β-Sintase CSE: γ-Cistationase Cys: Cisteína Cx26: Conexina 26 Cx 30: Conexina 30 dB: Decibéis DMSO : Dimetilsufóxido DNA: Desoxyribonucleic acid
eNOS: Sintase do Óxido Nítrico endotelial GJB2: Gap Junction β2
GJB6: Gap Junction β6 GPx: Glutationa Peroxidase
GSH: Forma Reduzida de Glutationa GSSG: Forma Oxidada da Glutationa GST: Glutationa S-Transferase
GSTM1: Transferase da Glutationa, Família mu GSTT1: Transferase da Glutationa, família theta G6PD: Glucose-6-fosfato Desidrogenase Hcy: Homocisteína Hz: Hertz Met: Metionina MS: Metionina Transferase MTHFR: Metilenotetrahidrofolato Redutase OMS: Organização Mundial de Saúde OR: Odds Ratio
PA: Perda Auditiva
PCR: Polymerase Chain Reaction
RFLP: Restriction Fragment Length Polymorphism ERO: Espécies Reativas de Oxigénio
SAM: S-Adenosil Metionina SAH: S-Adenosil Homocisteína SN: Sistema Nervoso
SOD: Superóxido Dismutase STP: São Tomé e Príncipe THF: Tetrahidrofolato
VNTR: Variable Number Tandem Repeat XO: Xantina Oxidase
1.Introdução 1.1. Perda Auditiva
A audição é um sentido importante para a integração dos indivíduos na sociedade, sendo que a perda da mesma contribui para o isolamento social e profissional, depressão, baixo rendimento escolar, comprometimento no desenvolvimento da fala, bem como na qualidade de vida em geral1.
A surdez é a perda parcial ou total da audição, dificultando a compreensão e a comunicação da pessoa afetada, podendo ter causas genéticas ou ambientais. O tipo de perda auditiva (PA) pode ser classificada como:1) surdez por condução, que ocorre quando lesão no ouvido externo e/ou médio, que tem como função conduzir o som até ao ouvido interno; 2) surdez neurossensorial, que ocorre quando há lesão no ouvido interno, impedindo a propagação do estímulo sonoro ao sistema nervoso (SN); 3) surdez mista, a combinação das duas últimas em simultâneo, neste caso, existem lesões tanto no ouvido externo e/ou médio como no ouvido interno2.
Quanto à gravidade, a surdez pode ser categorizada através dos limiares de decibéis (dB) audíveis obtidos no ouvido menos surdo. De acordo com a Organização Mundial de Saúde (OMS), considera-se uma audição normal entre os limiares de 0 a 25 dB, surdez ligeira entre os limiares de 26 a 40 dB, surdez moderada entre os limiares de 41 a 60 dB, surdez severa entre os limiares de 61 a 80 dB e surdez profunda acima do limiar dos 81 dB3. Assim, quando se pretende caracterizar uma perda
auditiva, o correto é classificá-la de acordo com o seu tipo (condução, neurossensorial ou mista) e gravidade (leve, moderada, severa ou profunda)2.
Dados da OMS de 2018 revelam a existência de 466 milhões de indivíduos com deficiência auditiva, correspondendo a 5% da população mundial, dos quais 34 milhões são crianças, prevendo-se que em 2050 existam mais de 900 milhões de indivíduos com perda auditiva incapacitante. A maioria destes indivíduos são oriundos de países subdesenvolvidos localizados no Sul da Ásia e na África Subsariana4,5.Em 2010, nasce em Portugal, a partir do departamento de Otorrinolaringologia da CUF Infante Santo, uma série de ações humanitárias em São Tomé e Príncipe (STP), no âmbito do projeto “Saúde para todas as especialidades”, tendo-se verificado uma elevada prevalência de surdez neurossensorial, com as crianças a formarem o grupo mais afetado, sendo que parte delas não tinha aquisição de linguagem oral e apresentavam surdez bilateral profunda6.
1.2. O Ouvido Humano
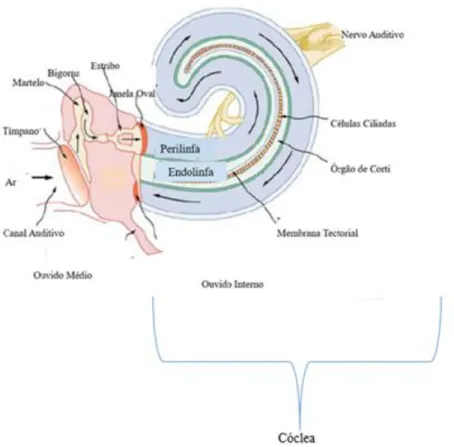
O ouvido humano (Figura 1.1) é o órgão responsável pela audição. O ouvido externo é constituído pelo pavilhão auricular e pelo canal auditivo, sendo que é nesta zona que são captadas as ondas e enviadas através do canal auditivo para o tímpano. O ouvido médio, cavidade preenchida por ar, é constituído pelo tímpano e por três ossículos (martelo, bigorna e estribo) que vibram com a onda sonora. O martelo liga-se fixamente à bigorna que por sua vez se liga de forma flexível ao estribo. Quando a onda sonora chega aos ossículos, estes vibram transmitindo-a ao ouvido interno. O ouvido interno é constituído pela cóclea, com funções na audição e pelo sistema vestibular, responsável pelo equilíbrio. Algumas células do interior da cóclea estão modificadas de maneira a formar o órgão de Corti (Figura 1.1). Este é composto por três fiadas de células, uma mais externa, outra com células de sustentação e uma fiada de células ciliadas internas especializadas na transdução do sinal mecânico em potencial de ação o qual é transmitido ao córtex cerebral e percebido como som7.
Figura 1.1 Esquema representativo do ouvido humano. O canal auditivo (juntamente com o pavilhão auricular) formam o
ouvido externo. O ouvido médio é constituído pelo tímpano e por três ossículos: martelo, bigorna e estribo, que juntos formam o mecanismo de condução do som. O ouvido interno é constituído pela cóclea, cujas células do interior estão modificadas de maneira a formar o orgão de Corti que transforma a vibração em impulso nervoso (Adaptado de Caroça, 2017)
1.3. As Principais Causas de Perda Auditiva Neurossensorial
Existem inúmeros fatores de risco que originam perda auditiva, podendo estes ser inerentes ao sexo, à idade, à etnia ou ao ambiente circundante7. Da sua etiologia, sabe-se que 50% dos casos são de
causa genética, 25% de causas ambientais e os restantes 25% de causas idiopáticas. Relativamente às causas ambientais, alguns estudos indicam que doenças infeciosas, como a rubéola ou a malária possam estar na origem da surdez neurossensorial. A rubéola é uma infeção causada por um vírus. Uma das consequências mais frequentes da infeção por rubéola (60% dos casos) é a PA congénita8, que ocorre quando a mulher grávida é infetada no primeiro trimestre de gravidez. A malária é uma doença causada pelo mosquito anófeles que transmite o parasita Plasmodium spp. A toxicidade do medicamento antimalárico é uma causa amplamente divulgada na PA, no entanto o efeito do próprio parasita em si na PA não é muito claro9,10.
A PA pode ocorrer como um sintoma isolado – perda auditiva não sindrómica – ou associada a uma variedade de sintomas, compondo quadros clínicos de síndromes genéticas – perda auditiva sindrómica. A perda auditiva genética sindrómica é classificada em autossómica dominante (ex: Síndrome de Penred) ou em autossómica recessiva (ex: Síndrome de Usher), sendo esta forma a mais prevalente (70% dos casos)11,12. Mais de metade dos casos de surdez não sindrómica autossómicos recessivos são devidos a mutações no locus DFNB1 - o primeiro locus descoberto como sendo responsável pela PA autossómica recessiva não sindrómica. Este locus localizado no cromossoma 13 codifica para 2 genes – GJB2 e GJB6 – que pertencem ao mesmo cluster e codificam 2 proteínas
altamente conservadas (conexinas) que formam canais intercelulares (gap junction). O gene GJB2 codifica a conexina 26 (Cx 26) e o gene GJB6 codifica a conexina 30 (Cx 30). Estas conexinas são amplamente expressas em vários tipos de células e tecidos13. No ouvido interno, são encontradas nas
células sensoriais epiteliais do órgão de Corti13. No gene GJB2 existe a predominância da mutação
35delG, presente em 60% a 75% dos casos de surdez; enquanto no gene GJB6 existe a predominância de duas grandes deleções de centenas de pb - del (GJB6-D13S1830) e del (GJB6-D13S1854) sendo encontradas frequentemente em associação com a mutação 35delG do gene GJB214,15.
Estudos16–18 revelam que mutações no gene GJB2 não desempenham um papel significativo na
etiologia da PA nas populações da África Subsariana ou nas suas populações descendentes. Outros estudos17,19,20 realizados em STP, mostram que o papel das mutações no gene GJB2 na surdez
neurossensorial sindrómica parece ser pouco significativo21. A acrescentar, o papel das grandes deleções no gene GJB6 na surdez não sindrómica em STP não é significativo21. Neste sentido, os dados remetem para a possível existência de outras variantes genéticas patológicas em STP7.
1.4. Stresse Oxidante e Perda Auditiva Neurossensorial
As células que compõem os tecidos e órgãos do corpo humano são expostos, de forma constante a ataques de natureza oxidante que podem provir de fontes endógenas, como processos metabólicos e/ou de sinalização celular, ou exógenas, por meio de exposição a xenobiontes, como agentes químicos oxidantes, radiações ionizantes e ultra-violeta22,23.
O stresse oxidante pode ser definido como uma condição biológica na qual ocorre desequilíbrio entre a produção de espécies reativas de oxigénio (ERO) e a sua remoção através de sistemas antioxidantes. As ERO, quando em baixas concentrações, têm um papel importante em funções biológicas como a sinalização, migração e divisão celular, no entanto se houver um desvio nas concentrações destas, o seu aumento pode originar efeitos nefastos para o organismo, como lesões no DNA, oxidação lipídica ou desnaturação de proteínas24. Entre as ERO responsáveis pelas inúmeras lesões, podemos destacar o radical superóxido (O
-2), o peroxinitrito (ONOO-) e o radical hidroxilo (OH
-), o mais reativo25. Do metabolismo destes destacam-se o peróxido de hidrogénio (H
2O2) e o ácido
hipocloroso (HOCl)26,27. De forma a equilibrar a quantidade excessiva de ERO e dos seus metabolitos,
o organismo possui sistemas antioxidantes, que estão organizados em três níveis: 1) sistemas que atuam no sentido de prevenir a formação de ERO, por ação de inibidores de enzimas que catalisam essas reações; 2) compostos antioxidantes não enzimáticos como a vitamina A ou a vitamina C que “aprisionam” as ERO diminuindo a sua toxicidade (este nível só entra em ação, no caso do primeiro sistema falhar); 3) sistemas de reparação que reconhecem e decompõem moléculas de DNA danificadas, proteínas que sofreram desnaturação, entre outros (este sistema atua no caso de os dois sistemas anteriores falharem)28–30.
A cóclea é um órgão que produz níveis significativos de ERO, os quais aumentam para níveis prejudicais quando há redução nas atividades enzimáticas dos sistemas antioxidantes. O metabolismo oxidante tem sido associado a diversas patologias, uma vez que pode afetar vários locais do organismo. Lesões e apoptose nas células ciliadas do ouvido interno podem ocorrer como resultado do aumento de ERO31. Clerici e Yang32 estudaram os efeitos da produção de ERO na função coclear, tendo demonstrado que a produção destes no espaço perilinfático (Figura 1.2) pode prejudicar gravemente a produção e transmissão do impulso nervoso. Mais recentemente Gul33 e colaboradores mostraram que a presença de ERO na perilinfa do ouvido interno induz a PA neurossensorial ao causar lesões nas células ciliadas32–
1.4.1. Metabolismo da Homocisteína
A homocisteína (hcy) é um aminoácido não essencial, sulfídrico, derivado da metionina (met), proveniente da dieta ou do metabolismo da via biossintética que converte a met a cisteína (cys) (Figura
1.2). Nesta via, a hcy forma-se num processo onde ocorre a conjugação da met com ATP, resultando na
S-adenil metionina (SAM), seguida de uma reação de desmetilação, formando a S-adenosil homocisteína (SAH) com posterior hidrólise para libertar a adenosina e hcy35–37. A hcy pode ser
convertida irreversivelmente a cys, através da via da transulfuração. Nesta via aproximadamente 50% da hcy é convertida irreversivelmente a ciy, reação catalisada pela cistationina-β sintase (CβS) que tem como cofator a vitamina B6. Nesta reação a hcy liga-se à serina formando a cistationina, seguidamente, a enzima γ-cistationase (CSE) dependente do cofator vitamina B6, vai hidrolisar a cistationina, originando cys e α-cetobutitato35–37. A cys é posteriormente convertida em glutationa (GSH), que possui
um grupo tiol, agente com grande poder redutor. A GSH pode existir sob duas formas: oxidada (GSSG) ou reduzida (GSH). A ação da enzima glutationa S-transferase (GST) facilita a formação e estabilização do anião tiol (GS-), promovendo a ligação da GSH a um substrato eletrofílico. A presença da GSH, em quantidade adequada, é essencial na função protetora contra radicais livres e xenobióticos38.
A hcy pode no entanto, em alternativa à transulfuração, ser remetilada a met através da enzima 5-metiltetrahidrofolato-homocisteína, também designada por metionina sintase (MS), tendo como cofator a vitamina B12. Simultaneamente, a MS converte a 5-metiltetrahidrofolato (forma circulante do folato reduzido-5-MTHF) a tetrahidrofolato (THF). A formação de 5-MTHF requer a redução de 5,10-metil tetrahidrofolato (5,10-MTHF) a 5-MTHF que é catalisado pela enzima 5,10-metileno tetrahidrofolato redutase (MTHFR). Assim, a concentração de hcy depende das atividades enzimáticas de CβS e MTHFR. Deficiências nestas enzimas originam acumulação de hcy intracelular, que ao não ser metabolizada pode comprometer o funcionamento das células endoteliais e da matriz coclear, na medida em que o aumento dos níveis de hcy originam a formação de H2O2, que por sua vez diminui a atividade
Figura 1. 2 Esquema simplificado do metabolismo da hcy. A hcy surge por desmetilação durante o metabolismo da met via
SAM (S-adenosil metionina) e SAH (S-adenosil homocisteína). Depois de formada pode ser metabolizada por remetilação a met através de uma reação catalisada pela enzima MS (metionina sintase) e pelo cofator vitamina B12. A MS requer, como substrato a 5-MTHFR, que é catalizada pela enzima metilenotetrahidrofolato redutase (MTHFR). A hcy pode ainda ser metabolizada a cys pela via de transulfuração, através de uma reação catalisada pela CβS e pelo cofator vitamina B6. A ação da enzima GST promove a ligação da GSH, um agente com grande poder redutor, a um substrato eletrofílico essencial na proteção contra compostos xenobíoticos (Adaptado de: http://lpi.oregonstate.edu/es/mic/vitaminas/folato).
1.4.2.Metileno Tetrahidrofolato Redutase e Perda Auditiva
O gene MTHFR é expresso em vários órgãos e tecidos, nomeadamente no fígado, eritrócitos, rins e cóclea. O gene que codifica a enzima localiza-se no cromossoma 1, na região 1p36.3. A sequência do DNA é de 2,2 kb e contém 11 exões. Os alelos referentes aos polimorfismos C677T e A1298C são os mais frequentes e estão associados a hiperhomocisteinémia40. No que se refere à alteração C677T (rs1801133), esta é caracterizada por uma substituição C→T (ala→val) na posição 677 do gene, tornando a enzima termolábil. Esta alteração leva a uma redução de 30% na atividade enzimática nos indivíduos heterozigóticos e 60% nos indivíduos homozigóticos41.
Martinez-Vega31 e seus colaboradores indicam que a presença do alelo T está associado ao aumento do risco de PA não sendo claro se esta alteração está associada à PA isoladamente31.
1.4.3. Cistationina β-Sintase e Perda Auditiva
O gene que codifica a enzima CβS está localizado no cromossoma 21, na região 21q22.3. A sequência de DNA é de 23,7 kb e contém 17 exões. Este gene é expresso em todos os tecidos, mas
maioritariamente no fígado42. Uma das mutações mais frequentes envolve a inserção de 68 pb (844ins68) que duplica a região intrão7/ exão 8.
Embora o mecanismo exato pelo qual o alelo 844ins68 afeta a função da enzima CβS não esteja perfeitamente esclarecido, foi mencionado que a presença deste alelo aumenta a atividade enzimática.
1.4.4. Gluatationa S-Transferase e Perda Auditiva
As enzimas GST estão codificadas numa família de genes de enzimas responsáveis por catalisar a conjugação da forma reduzida de glutationa (GSH) a substratos xenobióticos, indutores de stresse oxidante. As GST humanas, expressas em todos os tecidos, podem ser divididas em cinco classes principais: alfa, mu, pi, theta e zeta43–45.
Nos seres humanos o gene GSTM1 (classe mu) está localizado no cromossoma 1, na região 1p13.3. A sequencia de DNA é de 20 Kb, é constituído por 7 exões e possui 3 formas alélicas, GSTM1*A, GSTM1*B e GSTM1*0. Os alelos GSTM1*A e GSTM1*B diferem num nucleótido, de C→T, na posição 534, resultando na troca de uma lisina por uma aspargina, não tendo implicações funcionais. O alelo GSTM1*0 é um alelo nulo resultante de uma deleção que compromete a transcrição de RNAm e consequentemente a produção da enzima. Assim, o GSTM1*A e GSTM1*B possuem um fenótipo ativo e o GSTM1*0 é responsável pelo fenótipo nulo que leva à ausência da atividade enzimática46.
Nos seres humanos o gene GSTT1 (classe theta) está localizado no cromossoma 22, na região 22q11.2. A sequencia de DNA é de 20 kb constituído por 5 exões e possui 2 formas alélicas, GSTT1*1 e GSTT1*0. O alelo GSTT1*1 corresponde à forma alélica funcional enquanto o alelo GSTT1*0, corresponde a uma forma não funcional ou nula. O alelo nulo é responsável pelo fenótipo nulo que leva à ausência da atividade enzimática45,47.
A existência de genótipos nulos comprometem a conjunção do GSH com metabolitos ou toxinas oxidantes. Sabe-se também que níveis elevados de ERO modulam a expressão e atividade de enzimas antioxidantes, incluindo as GST, o que promove a diminuição dos níveis de GSH, originando lesões nas células da cóclea e consequentemente PA48,49.
1.4.5. Sintase do Óxido Nítrico e Perda Auditiva
A sintase endotelial do óxido nítrico (eNOS) é a principal enzima responsável pela síntese de óxido nítrico (NO) no endotélio vascular, o qual tem um papel vasoprotetor, antioxidante, intervindo no metabolismo de substâncias endógenas e exógenas50. O NO é sintetizado a partir do aminoácido
L-arginina, sendo para isso necessária a presença de O2, NADPH e do cofator tetrahidrobiopterina (BH4).
A enzima eNOS é codificada pelo gene NOS3, localizado no cromossoma 7, na região 7q35-36. A sequência de DNA é de 4,4 kb e contém 26 exões50. Foram já caracterizados vários polimorfismos no gene NOS3, incluindo polimorfismos de base única (SNPs), inserções/deleções e repetições de número variável em tandem (VNTR). Existe um VNTR descrito com repetições de 27 pb, localizada no intrão 4, sendo os alelos mais frequentes o alelo 4b (5 repetições) e o alelo 4a (4 repetições), embora outros alelos mais raros tenham já sido relatados51.O alelo 4a está associado a baixa produção de nitrato plamático, em que os indivíduos homozigóticos para o referido alelo apresentam uma redução de 20% na concentração dos compostos do NO em comparação com os indivíduos com genótipo 4b4b, bem como a diminuição da expressão da proteína eNOS52.
O stresse oxidante é considerado um dos principais mecanismos da redução de disponibilidade de NO50-55 uma vez que num processo denominado desacoplamento da eNOS leva à produção de O-2 em
vez de NO. Um dos principais determinantes do desacoplamento da eNOS é a perda de biodisponibilidade do cofator BH4, promovida pelas ERO50,53,54.
Trabalhos de Xiaorui55 e seus colaboradores demonstraram a existência de eNOS no endotélio dos vasos das células ciliadas internas e externas do órgão de Corti e trabalhos de Popa56 detetaram a presença de eNOS nas células ciliadas e de suporte.
Estudos de Lubos e colaboradores57 sugerem que o alelo 4a contribui para a PA, devido à baixa
biodisponibilidade de NO.
1.4.5.1 Relação do Metabolismo do Óxido Nítrico com o Metabolismo da Homocisteína
Sabe-se que as propriedades vasodilatadoras das células endoteliais são alteradas em condições de hiperhomocisteinemia, especificamente no que se refere à produção de NO, uma vez que que a oxidação da hcy origina ERO que inativam a enzima e NOS335,58.
1.4.6. Glucose-6-Fosfato Desidrogenase (G6PD) e Perda Auditiva
A G6PD é uma enzima citoplasmática expressa em todos os tecidos, mas a sua deficiência manifesta-se essencialmente nos eritrócitos59. Catalisa o primeiro passo da via das pentoses fosfato,
transformando a glicose-6-fosfato em 6-fosfogluconato com redução do NADP a NADPH, o qual, está estreitamente relacionado com o metabolismo da glutationa, mantendo esta no seu estado reduzido (GSH). De facto, o NADPH formado irá atuar no ciclo da glutationa, uma vez que a glutationa redutase irá necessitar de NADPH para reduzir a GSSG a GSH59. Também, para que ocorra síntese de NO é necessária a presença do cofator NADPH. Alterações na atividade da G6PD afetam a produção de NADPH, o que origina a diminuição da produção de NO e consequentemente a redução da sua biodisponibilidade e aumento do stresse oxidante72,87.
O gene que codifica a enzima G6PD localiza-se no cromossoma X, na região Xq28. A sequência de DNA é de 20 kb e contém 13 exões. As diversas mutações no gene G6PD resultam em diferentes proteínas com níveis de atividade enzimática variáveis, associadas a um largo espetro de fenótipos bioquímicos e clínicos. As variáveis mais frequentes denominam-se do tipo A, enquanto o tipo B corresponde ao alelo normal. As variantes do tipo A são predominantemente encontradas na população africana, onde a malária é endémica. Por exemplo, uma substituição de A→G na posição 376 (rs1050829, ou variante A+) causa uma redução de 15% da atividade enzimática. A substituição de G→A na posição 202 (rs1050828, ou variante A-) causa uma redução de 85% da atividade enzimática A variante A- é tipicamente definida pela presença tanto do alelo rs1050829 como do alelo rs105082859,60. A icterícia neonatal e a anemia hemolítica aguda são as manifestações clínicas mais frequentes da variante A-, havendo estudos que associam esta enzimopatia à PA neurossensorial 21. De
facto, uma das consequências da icterícia neonatal é o elevado nível plasmático de bilirrubina, o qual induz toxicidade originando danos no sistema auditivo e consequentemente PA61.
1.5. Epistasia
A epistasia descreve a interação entre dois ou mais genes que não fazem parte do mesmo locus, a interação tem uma consequência fenotípica, e um dos genes pode modificar a expressão do (s) outro (s) (epistasia antagonista), ou por outro lado pode reforçar a expressão do outro gene (epistasia sinérgica)62,63. Os estudos epistáticos permitem obter mais informações acerca de como os genes afetam a suscetibilidade a doenças e assim avançar com descobertas nomeadamente na área da medicina63. No presente trabalho pretendeu-se testar a associação entre a PA com a interação entre 2 genes candidatos.
1.6. Genética Populacional
A diversidade genética das populações africanas atuais é o resultado de uma história antiga e complexa. A chegada de exploradores e comerciantes portugueses à costa subsaariana durante o século
XV representou um capítulo novo na história do continente africano. Um exemplo é a história do povoamento de STP. Este arquipélago é constituído por um grupo de ilhas de origem vulcânica, localizadas na zona equatorial africana, com aproximadamente 187 000 habitantes e uma área total de 1001 km2. O isolamento de STP em relação aos outros países do continente africano gerou uma
diversidade biológica singular, contribuindo para isso, o facto de não ter fronteiras terrestres, mas situar-se relativamente próximo das costas do Gabão, Guiné Equatorial, Camarões e Nigéria64–66.
As ilhas de STP permaneceram desabitadas até 1470, ano em que os navegadores portugueses João de Santarém e Pêro Escobar, numa tentativa de alcançar a rota marítima para a Índia, descobriram as ilhas. A colonização teve início no final do século XV, liderada por Álvaro Caminha, tendo sido feita em conjunto com cristãos novos que tinham sido expulsos de Portugal pela Inquisição. A localização estratégica das ilhas permitiu o estabelecimento de importantes portos para os portugueses nos séculos seguintes64–66.
Durante o século XVII e XVIII, STP viveu-se um período de enorme despovoamento e a atividade local destinava-se quase exclusivamente ao negócio de escravos e mantimentos para os nativos. Apesar deste período de estagnação que afetou STP, o país acabou por recuperar a economia e a população com a introdução do café, em 1787, e do cacau, em 1821, levando a que muitas famílias portuguesas fossem viver para STP. Em 1876 a escravatura foi abolida, contudo o trabalho escravo continuou por várias décadas, sendo a mão-de-obra originária de diferentes regiões da costa ocidental do continente africano até à independência em 1975. Assim, as raízes dos são-tomenses remontam aos escravos trazidos para as ilhas durante o período inicial de ocupação, que entraram em STP em massa desde o início do século XIX, para suprir as necessidades das emergentes economias de café e cacau.
As populações são as unidades sobre as quais se medem as variações genéticas e evoluem quando ocorre uma alteração no seu fundo genético, ou no conjunto de alelos que a caracteriza. STP foi ao longo dos séculos, colonizado, recebeu escravos e sofreu despovoamentos, tendo sido por isso alvo de fatores relevantes na alteração do seu fundo genético, como as migrações e a deriva genética.
2. Objetivos
O presente estudo tem como objetivo principal verificar se polimorfismos genéticos em genes que codificam para proteínas envolvidas no processo de stresse oxidante, influenciam a suscetibilidade para a surdez neurosensorial em STP.
Mais especificamente pretende-se numa fase inicial testar a existência de diferenças nas distribuições genótipicas e alélicas em genes relacionados com o stresse oxidante, entre a população Portuguesa e a população de STP. Numa segunda fase pretende-se perceber se as diferenças encontradas poderão estar a contribuir para as elevadas frequências de Perda Auditiva em STP. Com esse fim serão realizados estudos de associação na população de STP, entre as variantes genéticas e a perda auditiva.
Os polimorfismos em estudo são:
a) Substituição C677T no gene MTHFR; b) Inserção de 68 pb no intrão 8 no gene CβS; c) Deleção das isoformas GSTM1 e GSTT1; d) VNTR no intrão 4 no gene NOS3;
3. Metodologia
3.1. Seleção da Amostra Populacional
Este estudo incide sobre uma amostra de 316 indivíduos de STP constituída por 2 grupos, um de 136 indivíduos com PA (ligeira, moderada, severa ou profunda) em pelo menos um dos ouvidos e outro grupo de 180 indivíduos, que corresponde ao grupo controlo, com audição normal em ambos os ouvidos.
Todos os participantes responderam a um questionário de forma a caraterizar na história clínica os fatores de risco. Dos fatores de risco constam a história familiar de PA, consanguinidade, historial pré- e peri natal, conhecimento de infeção por malária, infeção por rubéola e outros agentes. O estado de audição foi avaliado através de audiogramas (entre 200 Hz e 8000 Hz).
Do estudo foram excluídos indivíduos com menos de 2 anos e mais de 35 anos. Foram também excluídos indivíduos com PA por condução ou mista de causas identificadas, indivíduos que apresentavam atrasos no desenvolvimento psicomotor e indivíduos que tiveram complicações neonatais.
3.2. Análise Genética
3.2.1. Amplificação dos Fragmentos de DNA a Estudar
A amplificação dos fragmentos de DNA foi efetuada pela reação de PCR (Polymerase Chain Reaction) que consiste numa série de ciclos, cada um dos quais constituído por um conjunto de reações efetuadas a diferentes temperaturas. Cada ciclo de PCR envolve a desnaturação do DNA, o emparelhamento (annealing) de primers e a síntese de DNA. Para tal, utilizou-se um termociclador (GenoSmart2, VWR) e as condições de amplificação descritas (Tabela 3.1). Amplificaram-se fragmentos dos genes MTHFR, CβS, GST (GSTM1 e GSTT1) e NOS3.
Tabela 3.1 Polimorfismo, primers, condições de amplificação e tamanho esperado dos fragmentos
Gene (polimorfismo) Primers Condições de
PCR Mistura Reacional Tamanho dos fragmentos MTHFR (C677T) 5’TGAAGGAGAAGGTGTCTGCGGGA 3’ 5’AGGACGGTGCGGTGAGAGTG 3’ 35 Ciclos Desnaturação 30 seg a 94ºC Annealing 30 seg. a 61ºC Extensão 45 seg. a 72ºC Extensão final 7 min. a 72ºC Primer F: 1 µl (10pmol/µl) Primer R: 1µl (10pmol/µl) Master mix*: 12,5 µl H2O: 3 µl DMSO: 2,5 µl DNA: 5 µl V. final: 25µl 198 pb CβS (ins68pb) 5’GCAGTTGTTAACGGCGGTATTG 3’ 5’ GCCGGGCTCTGGACTCGACCTA 3’ 40 Ciclos Desnaturação 30 seg a 94ºC Annealing 30 seg a 6ºC Extensão 40 seg a 72ºC Extensão final 10 min a 72ºC Primer F: 1µl (10pmol/µl) Primer R: 1µl (10pmol/µl) Master mix*: 12,5 µl H2O: 5,5 µl DNA: 5 µl V. final: 25 µl Sem inserção 252 pb Com inserção 320 pb GST (T1 e M1) (nulo/não nulo) Primer 1 (GSTM1): 5’GCCATCTTGTGCTACATTGCCCG3’ Primer 2 (GSTM1): 5’ATCTTCTCCTCTTCTGTCTCCCC 3’ Primer 3 (GSTM4): 5’TTCTGGATTGTAGCAGATCATGCCC 3’ Primer 4 (GSTT1): 5’TTCCTTACTGGTCCTCACATCTC 3’ Primer 5 (GSTT1): 5’TCACCGGATCATGGCCAGCA3’ 40 Ciclos Desnaturação 45 seg a 94ºC Annealing 45 seg a 58ºC Extensão 45 seg a 72ºC Extensão final 5 min a 72ºC Primer F: 1µl (10pmol/µl) Primer R: 1µl (10pmol/µl) Master mix*: 12,5 µl H2O: 5,5 µl DNA: 5µL V.final: 25 µl GSTM1- 230pb GSTM4 - 157 pb GSTT1- 480 pb NOS3 (VNTR do intrão 4) 5’AGGCCCTATGGTAGTGCCTT 3’ 5’TCTCTTAGTGCTGTGGTCAC 3’ 35 Ciclos Desnaturação: 30 seg. a 94ºC Annealing: 30 seg. a 55ºC Extensão: 45 seg. a 72ºC Extensão final: 5 min. a 72ºC Primer F: 1 µl (10pmol/µl) Primer R: 1µl (10pmol/µl) Master mix*: 12,5 µl H2O: 5,5µl DNA: 5µl V. final: 25 µl Variante a 393 pb Variante b 420 pb
*Dream Taq Green PCR Master Mix 2x - Thermo Scientific
A confirmação da amplificação foi efetuada por eletroforese em gel de agarose. Os géis foram feitos a uma concentração de agarose a 3% para o gene MTHFR e de 2% para os genes CβS, GST (M1 e T1) e NOS3 e feitos num volume de 120 ml de TAE 1x concentrado (TAE 10x: 20 mM Tris-Acetato, 1 mM EDTA, pH 8) e uma concentração de brometo de etídeo de 10 mg/ml. Para os polimorfismos NOS3 e MTHFR, a eletroforese foi realizada a 120 V durante 90 minutos, para os polimorfismos GST (M1 e T1) a eletroforese foi realizada a 120 V durante 60 minutos e para o polimorfismo CβS a
eletroforese foi realizada a 130 V durante 50 minutos. Um transiluminador (GenoSmart, VWR) foi utilizado para visualizar o perfil de migração.
3.2.2. Genotipagem
Dos polimorfismos estudados, quatro – nos genes NOS3, CβS, GST (M1 e T1) - são detetados por variação do tamanho do produto amplificado, sendo apenas necessário recorrer à técnica do PCR e eletroforese. Os tamanhos esperados encontram-se descritos na Tabela 2.1. O polimorfismo MTHFR é um polimorfismo de um único nucleótido, pelo que a genotipagem é efetuada recorrendo à técnica de RFLP (Restriction Frangment Length Polymorphism), a qual consiste numa restrição enzimática que permite discriminar os alelos através do padrão de migração dos fragmentos de restrição obtidos em eletroforese de gel de agarose. O gel de 2 a 3%, foi feito com 120 ml de TAE 1x concentrado (TAE 10x: 20 mM Tris-Acetato, 1 mM EDTA, pH 8) e 10 mg/ml de brometo de etídio. A restrição foi efetuada num termociclador (GenoSmart2, VWR) nas condições descritas na (Tabela 3.2) e a eletroforese realizada a 130 V durante 60 minutos. O perfil eletroforético foi visualizado num transiluminador (GenoSmart, VWR).
Tabela 3.2 Condições de Restrições e Possíveis Tamanhos dos Fragmentos do Polimorfismo MTHFR
Gene Polimorfismo Componentes Condições da Restrição
Tamanho dos fragmentos
MTHFR C677T
Enzima: 1µl Hinf I (5U/µl)
Tampão R+: 2 µl 10X buffer H2O: 12 µl Produto de PCR: 10 µl V. final: 25 µl 18 h, 37ºC CC -198 pb CT -198, 175 e 23 pb TT -175 e 23 pb 3.3. Tratamento Estatístico 3.3.1 Estudo Populacional
Para a realização do estudo populacional foi analisada uma população africana (STP) e uma população do Sul da Europa (Portugal). Para os genes MTHFR, CβS, GST (M1 e T1), NOS3 e G6PD foram comparadas as frequências genotípicas observadas com as frequências esperadas em equilíbrio Hardy-Weinberg, utilizando o teste do Qui-quadrado (ꭓ 2). Para avaliar a existência de diferença significativa na distribuição dos genótipos e dos alelos entre as populações africana e portuguesa foi realizado o teste do ꭓ 2. O teste estatístico aplicado aos genes CβS e G6PD foi o teste exato Monte-Carlo
(uma vez que existiam em ambos os casos mais de 20% de células com frequência esperada inferior a 5). Todos os resultados foram obtidos usando o programa estatístico SPSS 25, sendo o nível de significância estatística estabelecido para p <0,05.
3.3.2 Estudos de Associação
Neste trabalho foram realizados estudos de associação entre as variantes genéticas e a PA, usando o teste de ꭓ2 e de Fisher, sendo o nível de significância estatística estabelecido para p <0,05.
A análise foi inicialmente realizada com foco em cada variante isoladamente – análise isolada - e posteriormente com foco numa combinação de genótipos de 2 genes – análise epistática.
3.3.2.1 Análise Isolada
Para cada polimorfismo, utilizou-se o teste do ꭓ2 para testar se a população estava em equilíbrio Hardy-Weinberg.
Compararam-se as distribuições das frequências genotípicas e alélicas entre o grupo com PA e o grupo de controlo, utilizando o teste do ꭓ2 para todos os genes exceto para o gene GSTT1, no qual foi
usado o teste de Fisher uma vez que uma célula esperava uma contagem menor que 5 sendo o nível de significância estatística estabelecido para p <0,05. O nível de proteção ou risco foi avaliado através de Odds Ratio (OR).
A avaliação da normalidade da variável idade foi efetuada através do teste Kolmogorov-Smirnov. Para testar se havia diferenças na idade relativamente à gravidade na PA utilizou-se o teste ANOVA.
3.3.2.2 Análise Epistática
Foi realizado o teste do ꭓ2 de modo a estudar a associação entre uma combinação de genótipos
para 2 genes e o fenótipo em estudo. Foram analisadas todas as combinações possíveis de genótipos entre 2 genes, num universo de 6 genes. O nível de proteção ou risco foi avaliado através de OR, sendo o nível de significância estatística estabelecido para p <0,05.
4. Resultados
4.1. Caraterização da População em Estudo
A população estudada é constituída por 316 indivíduos com idades compreendidas entre os 2 os 35 anos, com uma média de 17,4 anos e desvio padrão de 9,7 anos, dos quais 172 (54,4%) são do sexo masculino e 144 (45,6%) são do sexo feminino.
4.1.1. Caraterização da População quanto à Infeção por Malária
Dentro da população de 316 indivíduos, 194 (61,4%) já tinham contraído malária, os quais apresentaram em média 20,6 anos de idade com desvio padrão de 9,0 anos. Destes, 83 (42,8%) são do sexo masculino e 111 (57,2%) são do sexo feminino. Cento e treze (35,8%) indivíduos nunca contraíram malária, e apresentaram uma média de 12,3 anos de idade com desvio padrão de 8,4 anos. Destes, 58 (51,3%) são do sexo masculino e 55 (48,7%) são do sexo feminino; os restantes 9 (2,8%) indivíduos não souberam responder à questão, os quais apresentaram média de 10,4 anos de idade, com desvio padrão de 6,9 anos. Destes, 6 (66,7%) são do sexo feminino e 3 (33,3%) são do sexo masculino. A infeção por malária não apresenta diferenças estatisticamente significativas relativamente ao sexo (F=1,750; p=0,193). Existem diferenças estatisticamente significativas no que diz respeito à idade entre os indivíduos que já tinham contraído malária e os que nunca contraíram (t=7,910; p <0,001).
4.1.2. Caraterização da População quanto à Presença da Mutação Drepanocítica
Dentro da população de 316 indivíduos, 5 (3,3%) indivíduos têm mutação para a drepanocitose, apresentando uma média de 17,4 anos de idade com desvio padrão de 9,6 anos. Dentro destes, 3 (60,0%) são do sexo masculino e 2 (40,0%) são do sexo feminino. Os restantes 311 (96,7%) indivíduos não têm mutação para a drepanocitose, e apresentam uma média de 16,8 anos de idade com desvio padrão de 13,8 anos. Destes, 170 (54,7%) são do sexo masculino e 141 (45,3%) são do sexo feminino. A presença da mutação drepanocítica não está associada ao sexo (F=0,427; p=0,663) nem à idade (t=0,140; p=0,889).
4.1.3. Caraterização da População quanto à Perda Auditiva
Na população de 316 indivíduos, 180 (56,9%) são ouvintes e 136 (43,1%) têm PA (unilateral ou bilateral). Dentro do grupo de ouvintes, 87 (48,3%) são do sexo masculino e 93 (51,7%) são do sexo feminino. Dentro do grupo de indivíduos com PA, 57 (41,9%) são do sexo masculino e 79 (58,1%) são do sexo feminino. A presença de PA não apresenta diferenças estatisticamente significativas relativamente ao sexo (F=1,238; p= 0,266). Não existem diferenças estatisticamente significativas na idade entre os individuo ouvintes e os indivíduos com PA (t= 53,315; p=0,427).
4.1.3.1. Caraterização da População quanto à gravidade na Perda Auditiva
Na população constituída por 316 indivíduos, 180 (70,9%) têm audição normal bilateral e 136 (43,0%) indivíduos têm PA. Destes, 37 (27,2%) indivíduos têm PA ligeira, 23 (17,0%) indivíduos têm PA moderada, 21 (15,4%) indivíduos têm PA severa e 55 (40,4%) indivíduos têm PA profunda.
Tabela 4.1. Frequência e número de indivíduos do sexo masculino e feminino quanto ao grau de severidade na PA Controlo (n=180) (%) PA Ligeira (n=37) (%) PA Moderada (n=23) (%) PA Severa (n=21) (%) PA Profunda (n=55) (%) Valor de p* Feminino Masculino 93 (51,7%) 87 (48,3%) 27 (73,0%) 10 (27,0%) 9 (39,1%) 14 (60,9%) 15 (71,4%) 6 ( 28,6%) 28 (50,9%) 27 (49,1%) 0,032 *F
A severidade na PA apresenta diferenças estatisticamente significativas relativamente ao sexo (ꭓ 2
= 10,575; p=0,032).
Tabela 4.2. Média e desvio padrão das idades nas diferentes categorias de severidade da PA e no grupo de controlo
Controlos (n=180) (μ;σ) PA Ligeira (n=37) (μ;σ) PA Moderada (n=23) (μ;σ) PA Severa (n=21) (μ;σ) PA Profunda (n=55) (μ;σ) Valor de p* Idade 17,78; 9,77 18,00; 9,45 16,57; 10,29 18,00; 10,18 15,87; 9,66 0,735 *ANOVA μ: média,σ:Desvio Padrão
Quanto à idade, não existem diferenças significativas entre os grupos e a gravidade de PA.
4.1.3.2 Caraterização da População quanto à lateralidade na Perda Auditiva
Da amostra de 136 indivíduos com PA 44 (32,4%) têm PA unilateral e 92 (67,6%) têm PA bilateral.
A lateralidade na PA não apresenta diferenças estatisticamente significativas relativamente ao sexo (ꭓ2 =1,650; p=0,438) (tabela 4.3).
4.3. Frequência e número de indivíduos do sexo masculino e feminino relativamente à lateralidade na PA
PA Unilateral (n=44) (%) PA Bilateral (n=92) (%) Valor de p* Feminino Masculino 27 (61,4%) 17 (38,6%) 52 (56,5%) 40 (43,5%) 0,202 *t
Não existem diferenças estatisticamente significativas na idade quanto à lateralidade na PA (t=100,207; p=0,202) (tabela 4.4).
Tabela 4.4 Média e desvio padrão das idades na PA unilateral e bilateral PA unilateral (n=44) (μ, σ) PA bilateral (n=92) (μ, σ) Idade 20,09; 9,28 15,37;9,61
4.2. Estudo Populacional das Distribuições Genotípicas e Alélicas nos Genes MTHFR, CβS, GST (M1 e T1) NOS3 e G6PD em STP e Portugal
4.2.1. Caraterização das Populações em Estudo
Para este estudo foi realizado uma comparação das frequências genotípicas e alélicas em genes envolvidos no stresse oxidante, entre uma população do Sul da Europa – Portugal - e uma população africana – STP. A população de STP encontra-se caracterizada no capítulo 3.1. A população portuguesa é constituída por 178 indivíduos, destes 92 (51,7%) são do sexo feminino e 86 (48,3%) são do sexo masculino com uma média de 54,3 anos e um desvio padrão de 12,1 anos.
Os genes analisados foram: MTHFR, CβS,GSTM1, GSTT1, NOS3 e G6PD.
4.2.1.1. Estudo do Gene MTHFR nas Populações de STP e Portuguesa
Um exemplo representativo da obtenção de dados relativos à genotipagem do gene MTHFR encontra-se no Anexo 1. De referir que neste caso, a enzima de restrição Hinf I corta o alelo T.
Para o gene MTHFR a população de STP não está em equilíbrio Hardy-Weinberg (ꭓ 2 = 38,315; p <0,001), já a população portuguesa está em equilíbrio Hardy-Weinberg ( ꭓ 2 = 0,542; p=0,461). Foram calculadas as frequências genotípicas e alélicas para as duas populações, tendo-se observado a existência de diferenças estatisticamente significativas na distribuição dos genótipos e dos alelos do gene MTHFR nas populações de STP e Portuguesa, como mostram as Tabelas 4.5 e 4.6, respetivamente.
Tabela 4.5 Distribuição das frequências genotípicas do polimorfismo C677T nas populações de STP e Portuguesa
MTHFR STP (n=129) (%) Portugal (n=120) (%) Valor de p* CC CT TT 109 (84,5%) 15 (11,6%) 5 (3,9%) 61 (50,8%) 52 (43,3%) 7 (5,8%) <0,001 *ꭓ2
Tabela 4.6 Distribuição das frequências alélicas do polimorfismo C677T do gene MTHFR nas populações de STP e Portuguesa
Alelos STP (n=258) (%) Portugal (n=240) (%) Valor de p*
C T 233 (90,3%) 25 (9,7%) 174 (72,5%) 66 (27,5%) <0,001 * ꭓ2
4.2.1.2. Estudo do Gene CβS nas Populações de STP e Portuguesa
Um exemplo representativo da obtenção de dados relativos à genotipagem do gene CβS encontra-se no Anexo 2.
Para o gene CβS as populações de STP e Portugal não se encontram em equilíbrio Hardy-Weinberg (ꭓ2=2,011; p <0,001; (ꭓ2=162,070; p <0,001).
Foram calculadas as frequências genotípicas e alélicas para as mesmas populações, tendo-se verificado a existência de diferenças estatisticamente significativas na distribuição dos genótipos e dos alelos do gene CβS nas populações, como mostram as Tabelas 4.7 e 4.8, respetivamente.
Tabela 4.7 Distribuição das frequências genotípicas do polimorfismo de inserção de 68 pb do gene CβS na população de STP e Portuguesa CβS STP (n=85) (%) Portugal (n=114) (%) Valor de p* del/del ins/del ins/ins 33 (38,8%) 43 (50,6%) 9 (10,6%) 112 (98,2%) 0 (0,0%) 2 (1,8%) <0,001
*Teste exato Monte-Carlo
Tabela 4.8 Distribuição das frequências alélicas do polimorfismo de inserção de 68 pb no intrão 8 do gene CβS na população de STP e Portuguesa Alelos STP (n=170) (%) Portugal (n=228) (%) Valor de p* del ins 109 (64,1 %) 61 (35, 9%) 224 (98,2%) 4 (1,8%) <0,001 *ꭓ2
4.2.1.3. Estudo do Gene GSTM1 nas Populações de STP e Portuguesa
Um exemplo representativo da obtenção de dados relativos à genotipagem do gene GSTM1 encontra-se no Anexo 3.
Para o gene GSTM1 não pode ser calculado o equilíbrio Hardy-Weinberg nem as frequências alélicas uma vez que não se consegue distinguir um homozigótico não nulo de um heterozigótico.
Foram calculadas as frequências genotípicas para as mesmas populações, tendo-se verificado a existência de diferenças estatisticamente significativas na distribuição dos genótipos do gene GSTM1 nas populações de STP e Portuguesa, como mostra a Tabela 4.9, sendo mais frequente a presença de genótipo nulo (M1*0) na população Africana do que na população Portuguesa.
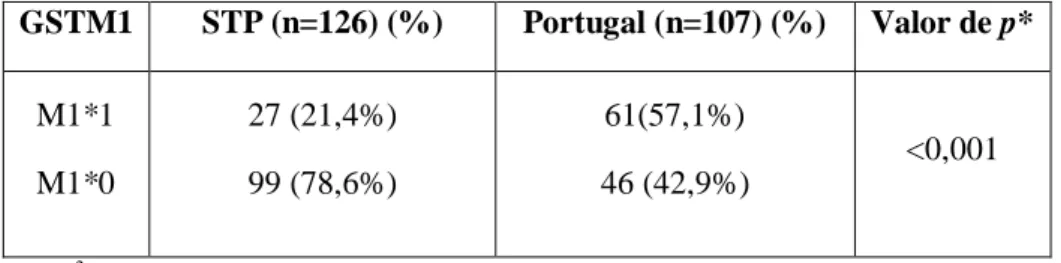
Tabela 4.9 Distribuição das frequências genotípicas dos alelos nulos (M1*0) e não nulo (M1*1) do gene GSTM1 nas populações de STP e Portuguesa GSTM1 STP (n=126) (%) Portugal (n=107) (%) Valor de p* M1*1 M1*0 27 (21,4%) 99 (78,6%) 61(57,1%) 46 (42,9%) <0,001 *ꭓ2
4.2.1.4. Estudo do Gene GSTT1 nas Populações de STP e Portuguesa.
Um exemplo representativo da obtenção de dados relativos à genotipagem do gene GSTT1 encontra-se no Anexo 3.
Para o gene GSTT1 também não foi possível calcular o equilíbrio Hardy-Weinberg nem as frequências alélicas uma vez que não se consegue distinguir um homozigótico não nulo de heterozigótico.
Foram calculadas as frequências genotípicas para as mesmas populações, tendo-se verificado a existência de diferenças estatisticamente significativas na distribuição dos genótipos do gene GSTT1 nas populações de STP e Portuguesa, como mostra a Tabela 4.10, sendo mais frequente a presença de genótipo nulo (T1*0) na população Africana do que na população Portuguesa.
Tabela 4.10 Distribuição das frequências genotípicas dos alelos nulos (T1*0) e não nulo (T1*1) do gene GSTT1 nas populações de STP e Portuguesa GSTT1 STP (n=126) (%) Portugal (n=107) (%) Valor de p* T1*1 T1*0 3 (2,4%) 123 (97,6%) 99 (92,5%) 8 (7,5%) <0,001 *ꭓ2
4.2.1.5. Estudo do Gene NOS3 nas Populações de STP e Portuguesa
Um exemplo representativo da obtenção de dados relativos à genotipagem do gene NOS3 encontra-se no Anexo 4.
Para o gene NOS3, a população de STP não se encontra em equilíbrio Hardy-Weinberg (ꭓ2
=15,079; p <0,001); já a população portuguesa encontram-se em equilíbrio Hardy-Weinberg e ꭓ2
=0,692; p=0,405).
Foram calculadas as frequências genotípicas e alélicas para as duas populações, tendo-se observado a existência de diferenças estatisticamente significativas na distribuição dos genótipos e dos alelos nas populações de STP e Portuguesa, como mostram as Tabelas 4.11 e 4.12, respetivamente.
Tabela 4.11 Distribuição das frequências genotípicas do polimorfismo VNTR do gene NOS3 nas populações de STP e Portuguesa
NOS3 STP (n=53) (%) Portugal (n=157) (%) Valor de p*
4b4b 4b4a 4a4a 23 (43,4%) 24 (45,3%) 6 (11,3%) 106 (67,5%) 46 (29,3%) 5 (3,2%) 0,003 *ꭓ2
Tabela 4.12 Distribuição das frequências alélicas do polimorfismo VNTR do gene NOS3 nas populações de STP e Portuguesa
4.2.1.6 Estudo do Gene G6PD nas Populações de STP e Portuguesa
Para o gene G6PD as populações de STP e portuguesa não estão em equilíbrio Hardy-Weinberg (ꭓ2
= 24,557; p <0,001; ꭓ2=81,112; p <0,001, respetivamente).
Foram calculadas as frequências genotípicas e alélicas para as mesmas populações, tendo-se observado a existência de diferenças estatisticamente significativas na distribuição dos genótipos e dos alelos do gene G6PD, como mostram as Tabelas 4.13 e 4.14, respetivamente.
Tabela 4.13 Distribuição das frequências genotípicas da variante A+ do gene G6PD nas populações de STP e Portuguesa
G6PD STP (n=178) (%) Portugal (n=140) (%) Valor de p* AA/A AG GG/G 94 (52,8%) 42 (23,6%) 42 (23,6%) 137 (97,9%) 2 (1,4%) 1 (0,7%) <0,001
*Teste exato Monte-Carlo
Tabela 4.14 Distribuição das frequências alélicas da variante A+ do Gene G6PD nas populações de STP e Portuguesas
Alelos STP (n=270) (%) Portugal (n=260) (%) Valor de p*
A G 173 (64,1%) 97 (35,9%) 256 (98,5%) 4 (1,5%) <0,001 *ꭓ2
4.3 Estudos de associação para os genes MTHFR, CβS, GST (M1 e T1) e NOS3 4.3.1. Análise do Polimorfismo C677T do Gene MTHFR
Para o gene MTHFR, foi possível obter resultados para 230 indivíduos da amostra tendo sido calculadas as frequências genotípicas e alélicas e comparadas as suas distribuições entre a população ouvinte e a população com PA, não se observando diferenças estatisticamente significativas, como mostram as Tabelas 4.15 e 4.16 respetivamente.
As populações ouvinte e surda não estão em equilíbrio Hardy-Weinberg (ꭓ2 =38,315 p <0,001)
(ꭓ2 = 50,767; p <0,001) para o polimorfismo em análise.
NOS3 STP (n=106) (%) Portugal (n=314) (%) Valor de p*
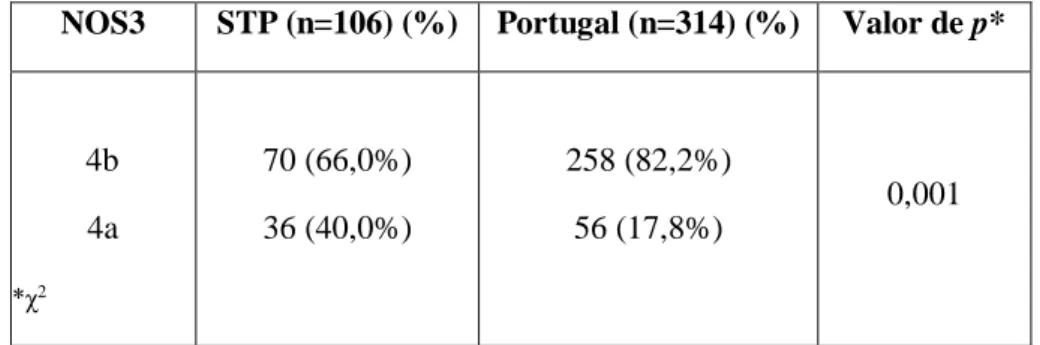
4b 4a 70 (66,0%) 36 (40,0%) 258 (82,2%) 56 (17,8%) 0,001 *ꭓ2
Tabela 4.15 Distribuição das frequências genotípicas do polimorfismo C677T do gene do gene MTHFR
Genótipo População Ouvinte
(n=129) (%) População Surda (n=101) (%) Valor de p* CC CT TT 109 (84,5%) 15 (11,6%) 5 (3,9%) 88 (87,1%) 9 (8,9%) 4 (4,0%) 0,799 *ꭓ2
Tabela 4.16 Distribuição das frequências alélicas do polimorfismo C677T na população ouvinte e na população surda
Alelos População Ouvinte
(n=258) (%) População Surda (n=202) (%) Valor de p* C T 233 (90,3%) 25 (9,7%) 185 (91,6%) 17 (8,4%) 0,637 *ꭓ2
4.3.2. Análise do Polimorfismo de Inserção de 68 pb no intrão 8 do Gene CβS
Para o gene CβS, foi possível obter resultados para 154 indivíduos da amostra tendo sido calculadas as frequências genotípicas e alélicas e comparadas as suas distribuições entre a população ouvinte e a população com PA, observando-se diferenças estatisticamente significativa na distribuições dos genótipos e alelos do gene CβS, como mostram as Tabelas 4.17 e 4.18. As populações ouvinte e surda não estão em equilíbrio Hardy-Weinberg (ꭓ2 = 2,011; p <0,001 e ꭓ2 = 17,969; p < 0,001, respetivamente) para o polimorfismo em análise.
Tabela 4.17 Distribuição das frequências genotípicas de ins68 pb no intrão 8 do gene CβS na população ouvinte e na população surda
Genótipo População Ouvinte
(n=85) (%) População Surda (n=69) (%) Valor de p* del/del del/ins ins/ins 33 (38,8%) 43 (50,6%) 9 (10,6%) 42 (60,9%) 22 (31,9%) 5 (7,2%) 0,024 *ꭓ2
Através do teste Odds Ratio (OR) verificou-se que a presença do alelo ins representa um fator de proteção para o desenvolvimento da PA (OR=0,41; IC= [0,2127-0,7824]) (Tabela 4.18).
Tabela 4.18 Distribuição das frequências alélicas do polimorfismo de ins68 pb no intrão 8 do gene CβS na população ouvinte e na população surda
Alelos População Ouvinte
(n=170) (%) População Surda (n=138) (%) Valor de p* del ins 109 (64,1%) 61 (35,9%) 106 (76,8%) 32 (23,2%) 0,007 *ꭓ2
4.3.3. Análise dos Polimorfismos nos Genes GST (M1 e T1)
Para o gene GSTM1 foi possível obter resultados para 244 indivíduos da amostra tendo sido calculadas as frequências genotípicas e comparadas as suas distribuições entre a população ouvinte e a população com PA, não se observando diferenças estatisticamente significativas na distribuição dos genótipos, como mostra a Tabela 4.19.
Não foi possível calcular as frequências alélicas nem o equilíbrio Hardy-Weinberg uma vez que não se consegue distinguir um homozigótico não nulo de um heterozigótico.
Tabela 4.19 Distribuição das frequências genotípicas dos alelos nulos (M1*0) e não nulo (M1*1) do gene GSTM1 na população ouvinte e na população surda
Genótipo (GSTM1) População Ouvinte (n=126) (%) População Surda (n=118) (%) Valor de p* M1*1 M1*0 27 (21,4%) 99 (78,6%) 33 (27,9%) 85 (72,1%) 0,236 *ꭓ2
Para o gene GSTT1, foi possível obter resultados para 244 indivíduos da amostra tendo sido calculadas as frequências genotípicas e comparadas as distribuições entre a população ouvinte e a população com PA, não se observando diferenças estatisticamente significativas, como mostra a Tabela
4.20.
Não foi possível calcular as frequências alélicas nem o equilíbrio Hardy-Weinberg uma vez que não se consegue distinguir um homozigótico não nulo de um heterozigótico.
Tabela 4.20 Distribuição das frequências genotípicas dos alelos nulos (T1*0) e não nulos (T1*1) do gene GSTT1 na população ouvinte e na população surda
Genótipo (GSTT1) População Ouvinte (n= 126) (%) População Surda (n= 118) (%) Valor de p* T1*1 T1*0 3 (2,4%) 123 (97,6%) 7 (5,9%) 111 (94,1%) 0,204 *F
4.3.4.Análise do Polimorfismo VNTR do Gene NOS3
Para o gene NOS3, foi possível obter resultados para 75 indivíduos da amostra tendo sido calculadas as frequências genotípicas e alélicas e comparadas as suas distribuições entre a população ouvinte e a população com PA. Tanto a população com PA como a população ouvinte não se encontram em equilíbrio de Hardy-Weinberg, (ꭓ2= 15,079; p <0,001 e ꭓ 2= 31460; p < 0,001 respetivamente) para o polimorfismo em análise.
Não se observaram diferenças estatisticamente significativas na distribuição dos genótipos e dos alelos do gene NOS3, como mostram as Tabela 4.21 e 4.22, respetivamente.
Tabela 4.21 Distribuição das frequências genotípicas do polimorfismo VNTR do gene NOS3 na população ouvinte e na população com PA
Genótipo População Ouvinte
(n=53) (%) População Surda (n=22) (%) Valor de p* 4b4b 4b4a 4a4a 23 (43,4%) 24 (45,3%) 6 (11,3%) 8 (36,4%) 12 (54,5%) 2 (9,1%) 0,765 *ꭓ2
Tabela 4.22 Distribuição das frequências alélicas no polimosfismo VNTR do gene NOS3 na população ouvinte e na população não ouvinte
Alelo População Ouvinte
(n=106) (%) População Surda (n=44) (%) Valor de p* 4b 4a 70 (66,0%) 36 (34,0%) 28 (63,6%) 16 (36,4%) 0,778 *ꭓ2
4.4 Estudo de Relações Epistáticas
Foram realizados estudos de associação para todas as combinações possíveis de genótipos entre 2 genes e o fenótipo PA.
Apenas obtivemos um resultado com associação significância à PA (Tabela4.23) referente à
Tabela 4.23 Associação significativa entre os genótipos dos genes MTHFR e NOS3 com a PA
Combinações Ouvintes (n=32)
(%) Surdos (n=10) (%) Valor de p* OR [IC,95%]
0 1 25 (78,1%) 7 (21,9%) 3 (30,0%) 7 (70,0%) 0,008 8,333 [1,697-40,911] * F
0 - Combinações MTHFR-NOS3 (CC-4b4a | CC-4 a4a| CT-4b4b | CT-4b4a |CT-4 a4a | TT-4b4b| TT-4b4a | TT-4a4a)