SILENCIAMENTO GÊNICO DE QUITINA SINTASES
DE Anthonomus grandis: POTENCIAL
BIOTECNOLÓGICO NO CONTROLE DE
INSETOS-PRAGA
Autor: Leonardo Lima Pepino de Macedo
Orientadora: Dra. Maria Fátima Grossi de Sá
LEONARDO LIMA PEPINO DE MACEDO
SILENCIAMENTO GÊNICO DE QUITINA SINTASES DE ANTHONOMUS GRANDIS:
POTENCIAL BIOTECNOLÓGICO NO CONTROLE DE INSETOS-PRAGA
Tese apresentada ao Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para obtenção do Título de Doutor em Ciências Genômicas e Biotecnologia.
Orientadora: Maria Fátima Grossi de Sá
7,5cm
Ficha elaborada pela Biblioteca Pós-Graduação da UCB
M141s Macedo, Leonardo Lima Pepino de.
Silenciamento gênico de quitina sintases de Anthonomus grandis: potencial biotecnológico no controle de insetos-praga. / Leonardo Lima Pepino de Macedo – 2012.
153 f.; il : 30 cm
Tese (doutorado) – Universidade Católica de Brasília, 2012. Orientação: Profa. Dra. Maria Fátima Grossi de Sá
1. Biotecnologia agrícola. 2. Controle biológico. 3. Pragas – controle. 4. Ácido ribonucleico. 5. Controle genético. I. Sá, Maria Fátima Grossi de, orient. II. Título.
Tese de autoria de Leonardo Lima Pepino de Macedo, intitulada “SILENCIAMENTO GÊNICO DE QUITINA SINTASES DE Anthonomus grandis: POTENCIAL BIOTECNOLÓGICO NO CONTROLE DE INSETOS-PRAGA”, apresentada como requisito parcial para obtenção do grau de Doutor em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 04/12/2012, defendida e aprovada pela banca examinadora abaixo assinada:
À Deus, pelo seu amor e pela grande oportunidade de convivência com os amigos.
À Carol, um grande presente de Deus, pela paciência, companheirismo e dedicação ao nosso amor, e agora ao nosso amado filho João Vitor.
Aos meus pais Francisco Pepino de Macedo e Adelaide Maria Ferreira de Lima, que não mediram esforços para ajudar em minha formação, pelo amor, paciência e apoio incondicional. Dedico também a meus irmãos, Linardo e Cristina, principalmente pela alegria de vivermos em família acima de tudo nos amando.
À EMBRAPA Recursos Genéticos e Biotecnologia, por ser um centro de excelência, que me ofereceu as condições necessárias para o desenvolvimento da pesquisa.
Aos meus inestimáveis amigos Fernando Fonseca, Alexandre Firmino e Roberta Coelho pelos longos e prazerosos momentos de convivência, discussões, tentativas, muitos erros e finalmente acertos.
Ao Dr. Osmundo Brilhante pelo exemplo de seriedade e amor ao trabalho. Obrigado pela amizade, pelos ensinamentos e pelos inesquecíveis momentos de convivência.
À Dra. Cristina Mattar, pelas palavras amigas, orientações, sugestões e dicas importantíssimas para a execução deste trabalho. Sou grato também pelas importantes sugestões durante a qualificação.
Ao Dr. José Dijair A. de Souza Jr, vulgo, Juuunioo, pela amizade e por importantes sugestões durante a qualificação.
Aos grandes amigos de jornada José Edilson e o Ludovico Migliolo muito obrigado pela paciência e amizade.
A equipe do Laboratório de Interação Molecular Planta-Praga I, Ariane Lacerda, Vanessa Olinto, a Dra. Regina, Thales Rocha, Isabela Tristán, Jéssica, Tati, Jorge Arboleda, Aulus Barbosa, Érico Vasconcelos, Gustavo Oliveira, Magda Beneventi, André Júlio, Itamara Mezzalira, Felipe Redorat, Erich Nakasu, Tiago Siqueira, Angelina Basso, José Cesamildo, Thiago Gonçalves, Diogo Sá, Vivian Miranda, Eduardo e Flávia Mulinari, Stephan Dohms, Uriele, Maíra Grossi, Bruna Araújo, Hudson Ramos, Rafael Del Sarto, Cleiton Cruz, Rodrigo Fragoso, Luciane Guimarães, PH Costa, por serem grandes colegas.
Aos saudosos amigos e profissionais que passaram pelo laboratório durante esse período do doutorado Hermógenes, Davi Farias, Ticiana Amorim e o Grauber Pacelli.
Às amigas Maria Eugênia, Joseilde, Raquel Sampaio pelos momentos prazerosos de estudo e cumplicidade.
Meus sinceros agradecimentos a todos que trabalham na plataforma de criação de insetos do Cenargen, Francisco Schmidt, Helio Santos, Isabella Grisi, Raphael, sem vocês estávamos perdidos!!!
À Lecir Nascimento, pela sua paciência e pela amizade. Não esquecendo também, da Angélica Bússolo, que a substituiu, demonstrando grande capacidade. À inesquecível Sineide Rangel; À pessoa que mais... Sheiska Silva. À todas um grande abraço e um muito obrigado especial.
“Fracassos? Não sei do que falas, em cada experiência descubro um dos motivos pelo qual a lâmpada não funciona. Agora sei
mais de mil maneiras de como não fazer a lâmpada.”
Macedo, Leonardo Lima Pepino. Silenciamento gênico de quitina sintases de Anthonomus grandis: potencial biotecnológico no controle de insetos-praga. Tese (Doutorado em ciências genômicas e biotecnologia) – Universidade Católica de Brasília, 2012.
O bicudo-do-algodoeiro (Anthonomus grandis) é a praga mais destrutiva da cultura do algodão (Gossypium hirsutum L.) no Brasil. As larvas deste inseto têm hábitos endofíticos o que dificulta o seu controle químico. Assim, se faz necessário o desenvolvimento de novas estratégias para controle desta praga. Com a disponibilidade do transcritoma do bicudo-do-algodoeiro, foi possível a busca por genes essenciais deste inseto-praga que pudessem ser silenciados durante o seu desenvolvimento por meio da técnica de RNA interferente, visando seu controle. Dentre as vias metabólicas detectadas, a via biossintética da quitina foi focada neste estudo. Este homopolímero estrutural de N-acetil-D-glucosamina é um dos principais componentes do exoesqueleto e da membrana peritrófica dos insetos. Portanto, a síntese de quitina apresenta-se como alvo de grande potencial para o desenvolvimento de estratégias de controle de inseto-pragas. Os objetivos do presente estudo foram 1) identificar e obter sequências dos genes de quitina sintases de A. grandis; 2) avaliar a expressão dos genes durante o desenvolvimento do bicudo-do-algodoeiro e 3) avaliar o efeito do silenciamento por RNAi sobre o desenvolvimento do inseto. Neste trabalho é descrito a obtenção e a caracterização das sequências completas de dois cDNAs de quitina sintase de A. grandis, denominados AntgCHS1 e AntgCHS2. Suas sequências de aminoácidos apresentam grande similaridade com outras quitina sintases de insetos. Ambos os genes possuem somente uma cópia no genoma de A. grandis. A análise transcricional indica que o gene AntgCHS1 foi altamente expresso na cutícula, predominantemente durante as fases de ovo, terceiro instar larval e pupa. Já o gene AntgCHS2 teve sua maior expressão restrita ao intestino médio do inseto durante as fases de alimentação. O silenciamento de AntgCHS1 causou má formações na cápsula cefálica e a inviabilidade da prole. Já o silenciamento de AntgCHS2 causou mortalidade e redução na oviposição. A interrupção e/ou a diminuição na síntese dos constituintes das estruturas quitinosas por meio da tecnologia do RNAi, visando como alvo os genes AntgCHS1 e AntgCHS2, apresentam-se como uma estratégia promissora para o controle genético e especifico do bicudo-do-algodoeiro.
The cotton boll weevil (Anthonomus grandis) is the most destructive insect-pest that attacks cotton crop in Brazil. The endophytic habit of these insect larvae make difficult their chemical control. Therefore, it is necessary to develop new strategies to control this pest. The boll weevil transcriptome availability makes possible the search for insect essential genes that could be silenced in its development using RNA interference, aiming its control. Among the metabolic pathways identified in the A. grandis transcriptome, chitin biosynthetic pathway was the target of this study. Chitin, a structural N-acetyl-D-glucosamine homopolymer, is a major component of exoskeleton and peritrophic membrane found in insects. Consequently, the chitin synthesis represents a target of great potential for development of strategies to control insect pests. In this context, the objectives of this study were 1) indentify and obtain cDNA sequences of A. grandis chitin synthase, 2) evaluate their expression during insect development and 3) check the silencing effect on insect development by RNAi. Our results describe the cloning and characterization of two chitin synthase cDNAs sequences from A. grandis, denominated AntgCHS1 and AntgCHS2, which were predicted to encode two proteins with 1566 and 1404 amino acid residues, respectively. These protein sequences exhibited high similarity with other insect chitin synthases. Both genes showed only one copy in the A. grandis genome. The transcriptional analysis indicated that AntgCHS1 gene was highly expressed in insect cuticle, predominantly during the egg, larval and pupa stages. On the other hand, the AntgCHS2 gene demonstrated an expression restricted to the insect midgut during the third instar and adult stage, the feeding phases. The AntgCHS1 silencing caused head capsule malformations and also egg inviability. Concerning to the AntgCHS2 knocking-down it was observed adult mortality as well as reduced oviposition. The interruption of chitinous structures synthesis using RNAi technology through AntgCHS1 and AntgCHS2 genes silencing, represent a promising strategy to be used in the development of GM cotton resistant to the cotton boll weevil.
Figura I-1. Esquema do mecanismo molecular da via do RNAi.. ... 27
Figura I-2. Via biossintética da quitina em insetos.. ... 34
Figura I-3. Representação esquemática da organização dos domínios da quitina sintase de insetos. ... 36
Figura II-1. Esquema da organização espacial do sistema digestório de insetos ... 41
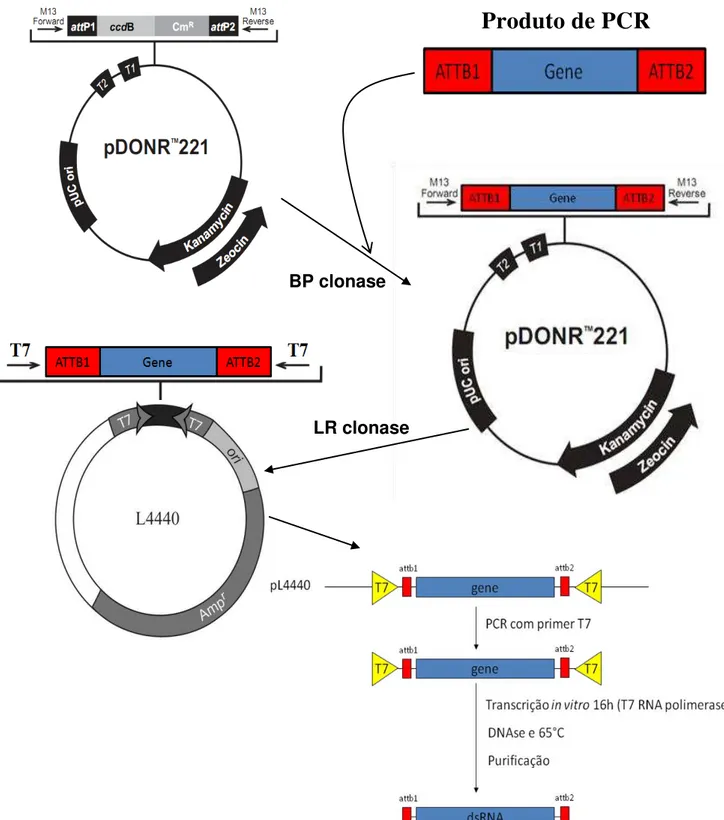
Figura II-2. Estratégia de clonagem do cDNA de AntgCHS2. ... 47
Figura II-3. Esquema metodológico para a sintese in vitro de dsRNA ... 52
Figura II-4. Procedimento de microinjeção em bicudo-do-algodoeiro. ... 53
Figura II-5. Organograma metodológico da clonagem, caracterização e análise funcional de AntgCHS2 ... 58
Figura II-6. Sequências de nucleotídeos e de aminoácidos deduzida do cDNA de AntgCHS2. ... 61
Figura II-7. Representação esquemática da organização dos domínios de AntgCHS2 ... 62
Figura II-8. Análise filogenética baseada na sequência de aminoácidos das quitina sintases de insetos. ... 64
Figura II-9. Análise de southern blot do DNA genômico de A. grandis para detecção do número de cópias do gene AntgCHS2. ... 65
Figura II-10. Padrão de expressão do gene de quitina sintase 2 durante o desenvolvimento de A. grandis determinado por PCR em tempo real. ... 66
Figura II-11. Padrão de expressão de AntgCHS2 em diferentes tecidos de A. grandis. ... 67
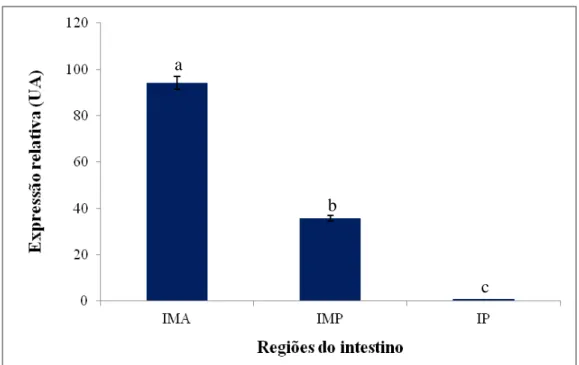
Figura II-12. Padrão de expressão de AntgCHS2 em diferentes regiões do intestino de larvas de 3º instar de A. grandis ... 68
Figura II-13. Análise do silenciamento gênico por qPCR de AntgCHS2 em adultos de A. grandis após microinjeção de dsRNA sob diferentes formas de administração. ... 69
Figura II-14. Análise do silenciamento gênico de AntgCHS2 em adultos de A. grandis após microinjeção de dsRNA de diferentes regiões do cDNA por qPCR. ... 70
Figura II-15. Efeito de diferentes concentrações de dsRNA sobre o silenciamento gênico de AntgCHS2 em adultos de A. grandis por qPCR. ... 71
Figura II-18. Efeito do silenciamento de AntgCHS2 sobre oviposição de fêmeas de A. grandis no decorrer do tempo. ... 74 Figura II-19. Efeito do silenciamento de AntgCHS2 sobre oviposição total de ovos de
fêmeas de A. grandis. ... 75 Figura II-20. Efeito do silenciamento de AntgCHS2 sobre a sobrevivência de fêmeas de A.
grandis ... 76 Figura II-21. Efeito do silenciamento de AntgCHS2 sobre a atividade especifica de
amilases em intestinos de fêmeas de A. grandis. ... 77 Figura II-22. Efeito do silenciamento de AntgCHS2 sobre a atividade especifica de
enzimas tipo tripsina presentes em intestinos de fêmeas de A. grandis ... 78 Figura II-23. Efeito do silenciamento de AntgCHS2 sobre quantidade de proteína presente
na hemolinfa de fêmeas de A. grandis ... 79 Figura II-24. Análise morfológica de intestinos de insetos adultos tratados com dsRNA
AntgCHS2 ... 80 Figura II-25. Análise histológica de intestinos de insetos adultos tratados com dsRNA
AntgCHS2 ... 81 Figura III-1. Modelo esquemático da estrutura da cutícula de insetos. ... 93 Figura III-2. Estratégia de clonagem do cDNA de AntgCHS1 ... 98 Figura III-3. Organograma metodológico da clonagem, caracterização e análise funcional
de AntgCHS1 ... 104 Figura III-4. Sequências de nucleotídeos obtida e de aminoácidos deduzida do cDNA de
AntgCHS1. ... 106
Figura III-5. Representação esquemática da organização dos domínios de AntgCHS1. ... 107
Figura III-6. Análise filogenética baseada na sequência de aminoácidos das quitina sintases de insetos ... 109 Figura III-7. Análise de southern blot do DNA genômico de A. grandis para detecção do
número de cópias do gene AntgCHS1 ... 110 Figura III-8. Padrão de expressão do gene de quitina sintase 1 durante o desenvolvimento
Figura III-11. Efeito do RNAi parental sobre o silenciamento gênico de AntgCHS1 em ovos de A. grandis por qPCR ... 114 Figura III-12. Efeito do dsRNA de AntgCHS1 sobre larvas neonatas provenientes de insetos
adultos microinjetados de A. grandis. ... 115 Figura IV-1. Padrão de expressão de AntgCHS1 e AntgCHS2em diferentes tecidos de A.
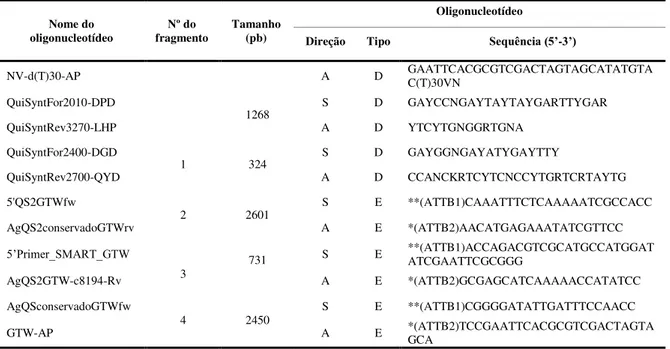
Tabela I-I Eventos de algodão GM aprovados no Brasil ... 24 Tabela II-I Lista de oligonucleotídeos utilizados na clonagem de AntgCHS2 ... 48 Tabela II-II Oligonucleotídeos utilizados nas reações de PCR em tempo real para análise
de AntgCHS2 ... 50 Tabela II-III Enzimas da via biossintética da quitina encontradas no transcritoma de A.
grandis ... 59 Tabela III-I Lista de oligonucleotídeos utilizados na clonagem de AntgCHS1 ... 98 Tabela III-II Oligonucleotídeos utilizados nas reações de PCR em tempo real para análise
µg micrograma µL microlitro
Algodão GM Algodão Geneticamente Modificado AntgCHS1 Quitina sintase 1 de Antonomus grandis AntgCHS2 Qquitina sintase 2 de Antonomus grandis Bt Bacillus thuringiensis
cDNA DNA complementar
Contigs Sequências contíguas montadas por alinhamento de reads DNA Ácido desoxirribonucléico
dNTP deoxinucleotídeo dsRNA RNA dupla fita
EC Códigos de classificação enzimática esiRNA siRNA preparado enzimaticamente in vitro EST expressed sequence tag, etiquetas de transcritos GAPDH Gliceraldeído-3-Fosfato Desidrogenase
GM geneticamente modificado kb Quilobase - 1000 pares de bases
miRNAs dsRNA com 21 a 26 nucleotídeos com bases despareadas mRNA RNA mensageiro
NCBI National Center for Biotechnology Information ºC Graus Celsius
pb Pares de bases
PCR Reação de polimerização em cadeia ("polymerase chain reaction") PIB Produto Interno Bruto
Primers Oligonucleotídeos iniciadores
qRT-PCR RT-PCR quantitativa (em tempo real) RDRP RNA Polimerase dependente de RNA RISC RNA-Induced Silencing Complex RNA Ácido ribonucléico
RNAi RNA interferente
RT-PCR Transcrição reversa seguida de PCR
INTRODUÇÃO GERAL ... 18
CAPÍTULO I – REVISÃO BIBLIOGRÁFICA ... 20
1 Revisão bibliográfica ... 20
1.1 Aspectos gerais da cultura de algodão no Brasil ... 20
1.2 Insetos-praga da cultura do algodão ... 21
1.3 O bicudo-do-algodoeiro (Anthonomus grandis) ... 22
1.4 Cultivares de algodão geneticamente modificadoS ... 23
1.5 RNA interferente (RNAi) no controle de pragas ... 25
1.6 Transcritoma do bicudo-do-algodoeiro: Descoberta e seleção de genes com potencial para RNAi ... 30
1.7 Quitina: ocorrência e estrutura ... 31
1.8 Síntese de quitina em insetos ... 33
2 Justificativa ... 38
3 Hipótese geral da tese ... 39
4 Objetivos ... 39
4.1 Objetivo geral ... 39
4.2 Objetivo específico... 40
CAPÍTULO II – CLONAGEM, CARACTERIZAÇÃO E ANÁLISE FUNCIONAL DA QUITINA SINTASE DO TIPO II ENVOLVIDA NA SÍNTESE DA MEMBRANA PERITRÓFICA EM Anthonomus grandis. ... 41
1 Introdução ... 41
1.1 O Sistema digestório dos insetos ... 41
1.2 Membrana peritrófica como alvo para controle de insetos-praga ... 42
2 Objetivos ... 45
2.1 Objetivo geral ... 45
2.2 Objetivos especificos ... 45
3 Material e Métodos ... 46
3.1 Insetos ... 46
3.2 Clonagem e sequenciamento de quitina sintase II de A. grandis ... 46
3.2.1 Clonagem de região conservada de quitina sintases ... 46
3.2.2 5’ e 3’ RACE da quitina sintase 2 de A. grandis ... 47
3.5 Determinação do padrão de expressão de AntgCHS2 ... 49
3.6 Localização tecidual da expressão do gene AntgCHS2 em A. grandis ... 50
3.7 Desenho e produção de RNA dupla fita (dsRNA) in vitro ... 51
3.8 Microinjeção de dsRNA em A. grandis ... 51
3.9 Avaliação do silenciamento de AntgCHS2 por PCR em tempo real ... 53
3.9.1 Avaliação do método de administração do dsRNA por microinjeção ... 53
3.9.2 Localização da sequência alvo dentro da sequência de cDNA da AntgCHS2 e do tamanho do segmento de dsRNA para o silenciamento ... 54
3.9.3 Efeito da dose de dsRNA microinjetado ... 54
3.9.4 Persistência do silenciamento durante as fases de desenvolvimento do inseto ... 55
3.10 Avaliação do silenciamento de AntgCHS2 sobre parâmetros fenotípicos de fêmeas de A. grandis ... 55
3.11 Avaliação do silenciamento de AntgCHS2 sobre a atividade enzimática em intestinos de fêmeas de A. grandis ... 56
3.11.1 Avaliação da atividade tripsínica ... 56
3.11.2 Avaliação da atividade -amilásica ... 56
3.12 Avaliação da quantidade de proteína na hemolinfa de A. grandis ... 57
3.13 AnáliseS morfológica e histológica de intestinos de A. grandis provenientes dos tratamentos com dsRNA ... 57
4 Resultados ... 59
4.1 Identificação da via biossintética da quitina no transcritoma do bicudo-do-algodoeiro . 59 4.2 Clonagem e sequenciamento da quitina sintase 2 de A. grandis (AntgCHS2) ... 60
4.3 Análise filogenética de AntgCHS2 ... 62
4.4 Análise por southern blot do DNA genômico de A. grandis para detecção de genes de quitina sintase II ... 65
4.5 Análise da expressão de AntgCHS2 durante o ciclo de vida de A. grandis ... 66
4.6 Localização tecidual da expressão de AntgCHS2 em A. grandis ... 67
4.7 Avaliação do silenciamento de AntgCHS2 em A. grandis ... 68
4.7.1 Otimização do procedimento de microinjeção de RNA interferente em adultos de A. grandis ... 68
4.7.2 Efeito de diferentes regiões do cDNA de AntgCHS2 no silenciamento gênico ... 70
4.7.5 Avaliação da persistência do RNAi larval sobre AntgCHS2 ... 73
4.8 Avaliação do silenciamento de AntgCHS2 sobre o fenótipo de fêmeas de A. grandis .. 74
4.8.1 Efeito do silenciamento de AntgCHS2 sobre a oviposição ... 74
4.8.2 Efeito do silenciamento de AntgCHS2 sobre a mortalidade de fêmeas... 76
4.9 Avaliação do silenciamento de AntgCHS2 sobre a atividade enzimática em intestinos de fêmeas de A. grandis ... 77
4.9.1 Efeito do silenciamento de AntgCHS2 sobre as atividade amilolítica e tripsínica em intestinos de fêmeas de A. grandis ... 77
4.10 Avaliação do silenciamento de AntgCHS2 sobre a quantidade de proteína na hemolinfa de fêmeas de A. grandis ... 79
4.11 Avaliação do silenciamento de AntgCHS2 sobre a morfologia do intestino de A. grandis80 5 Discussão ... 82
6 Conclusões ... 91
CAPÍTULO III – CLONAGEM, CARACTERIZAÇÃO E ANÁLISE FUNCIONAL DA QUITINA SINTASE I DE Anthonomus grandis ... 92
1 Introdução ... 92
1.1 Cutícula de insetos ... 92
1.2 Síntese de quitina na cutícula de insetos como alvo para controle de insetos-praga ... 93
2 Objetivos ... 96
2.1 Objetivo geral ... 96
2.2 Objetivos especificos ... 96
3 Material e Métodos ... 97
3.1 Clonagem e sequenciamento de quitina sintase I de A. grandis ... 97
3.1.1 Clonagem de região conservada de quitina sintases ... 97
3.1.2 5’ e 3’ RACE da quitina sintase 2 de A. grandis ... 97
3.2 Análise da sequência de DNA e proteína utilizando ferramentas de bioinformática ... 99
3.3 Análise por southern blot do DNA genômico de A. grandis para detecção de genes de quitina sintase I ... 99
3.4 Determinação do padrão de expressão de AntgCHS1 ... 100
3.5 Localização tecidual da expressão do gene AntgCHS1 em A. grandis ... 101
3.6 Desenho e produção de RNA dupla fita (dsRNA) in vitro ... 101
3.7 Microinjeção de dsRNA em A. grandis ... 102
3.8.3 Efeito do silenciamento de AntgCHS1 sobre larvas neonatas ... 103
4 Resultados ... 105
4.1 Clonagem e sequenciamento da quitina sintase 1 de A. grandis (AntgCHS1) ... 105
4.2 Análise filogenética de AntgCHS1 ... 107
4.3 Análise por southern blot do DNA genômico de A. grandis para detecção de genes de quitina sintase I ... 110
4.4 Análise da expressão de AntgCHS1 durante o ciclo de vida de A. grandis ... 111
4.5 Avaliação do silenciamento de AntgCHS1 em A. grandis ... 112
4.5.1 Avaliação fenotípica da microinjeção de dsRNA AntgCHS1 em larvas de A. grandis 112 4.5.2 Efeito do silenciamento de AntgCHS1 sobre a oviposição ... 113
4.5.3 Análise do silenciamento de AntgCHS1 sobre ovos obtidos de fêmeas microinjetadas (RNAi parental) ... 114
4.6 Efeito do silenciamento de AntgCHS1 sobre o desenvolvimento larval ... 115
5 Discussão ... 116
6 Conclusão ... 118
CAPÍTULO IV – DISCUSSÃO GERAL E PERSPECTIVAS ... 119
1 Discussão geral e perspectivas ... 119
2 Perspectivas ... 125
3 Referências ... 126
Apêndice A - Determinação dos instars larvais do bicudo-do-algodoeiro ... 142
Apêndice B - Caracterização morfológica do trato digestório de Anthonomus grandis 144 Apêndice C - Sequências de AntgCHS2 e AntgCHS1 e das sequências utilizadas para a síntese do dsRNA ... 145
Apêndice D - Síntese do dsRNA de AntgCHS2 e AntgCHS1 ... 150
ANEXOS ... 151
Publicações do Aluno ... 151
Artigos relacionados à tese ... 151
Outros Artigos ... 151
INTRODUÇÃO GERAL
No Brasil a cultura do algodão (Gossypium hirsutum L.) vem assumindo um importante
papel na economia agrícola brasileira, destacando-se como uma das principais commoditíes cultivada e tendo um importante papel na geração de divisas, desenvolvimento social e abertura de novos postos de trabalho de forma direta e indireta.
Com a crescente expansão na última década da cultura algodoeira para áreas do cerrado, grandes áreas em sistema de monocultura foram criadas favorecendo o aparecimento de pragas e doenças. Dentre as pragas, o bicudo-do-algodoeiro (Anthonomus grandis Boheman) é a praga de maior incidência na cultura do algodão e com maior potencial de dano. Os métodos de controle do bicudo são baseados na utilização de grandes quantidades de agrotóxicos, cuja aplicação exige elevados investimentos, onerando e às vezes inviabilizando a produção.
A importância do controle de pragas do algodoeiro e o aumento da consciência da população para os efeitos diretos e indiretos dos pesticidas na saúde pública e no meio ambiente, em geral, têm demandado a criação de novas formas de controle de pragas, que sejam mais econômicas e menos danosas ao ecossistema. Nesse cenário, o desenvolvimento de novos produtos e a perspectiva da utilização de variedades transgênicas com resistência a insetos são alternativas a serem consideradas, que poderão ser um fator de vantagem competitiva para o cultivo de algodão, além de evitar danos ao meio ambiente.
Deve-se ressaltar que as variedades de algodão GM disponíveis no mundo não são resistentes ao bicudo-do-algodoeiro. E, portanto, há a necessidade do desenvolvimento de eventos adequados às demandas nacionais. Este fato reforça a necessidade da busca por novas moléculas para uso na geração de novos eventos de algodão GM. Assim, muitos esforços têm sido despendidos pela equipe de pesquisadores da Embrapa com o intuito de obter plantas transgênicas resistentes ao bicudo-do-algodoeiro. Em particular, a equipe do Laboratório de Interação Planta Praga (Embrapa Recursos Genético e Biotecnologia) tem dado ênfase na busca de fatores de defesa com potencial uso no controle do bicudo-do-algodoeiro. Atualmente, duas estratégias moleculares se destacam: i) a superexpressão de proteínas entomotóxicas e ii) o silenciamento gênico via RNA interferente (RNAi).
moléculas alvos, essenciais ao bicudo-do-algodoeiro, visando sua aplicação em plantas de algodão GM resistentes ao bicudo-do-algodoeiro.
CAPÍTULO I – REVISÃO BIBLIOGRÁFICA
1 REVISÃO BIBLIOGRÁFICA
1.1 ASPECTOS GERAIS DA CULTURA DE ALGODÃO NO BRASIL
No Brasil, a agricultura possui grande importância sócio-econômica, é responsável pela geração direta e indireta de empregos, seja na cadeia de produção ou em produtos derivados (AVELAR; VILELA, 2006), além da destacada contribuição ao agronegócio brasileiro. Em 2011, a agricultura contribuiu com 15,42% do PIB (CENTRO DE ESTUDOS AVANÇADOS EM ECONOMIA APLICADA DA UNIVERSIDADE DE SÃO PAULO - CEPEA/USP, 2011). No país, as culturas que mais se destacam são: soja, milho, feijão, café, cana-de-açúcar e o algodão (BRASIL, 2012).
A utilização da fibra de algodão pelo homem remonta a séculos, sendo que os primeiros fragmentos de tecido datam de mais de sete mil anos. Há sólidos indícios de que populações ancestrais do Vale do Nilo, no Egito, e do Peru eram bastante familiarizadas com o cultivo e uso do algodão (HUCKELL, 1993; MOULHERAT et al., 2002). Sendo a principal fibra natural utilizada no Brasil e no mundo, e que abastece cerca de 50% da indústria mundial de fibras têxteis para a produção de tecidos e confecções (SANTOS et al., 1999), o algodão não apresenta grandes exigências com relação ao clima e ao solo, e, portanto, pode ser produzido em praticamente todos os continentes. Atualmente, esta planta, é cultivada em mais de 60 países, ocupando uma área de 33,5 milhões de hectares e produzindo 26,8 milhões de toneladas de pluma (USDA: UNITED STATES DEPARTMENT OF AGRICULTURE, 2012).
No cenário mundial, o Brasil é o quinto maior produtor de algodão (INTERNATIONAL COTTON ADVISORY COMMITTEE - ICAC, 2012b). A área destinada à cultura do algodão no Brasil historicamente era superior aos quatro milhões de hectares até o início dos anos 80 do século XX, quando começou o seu declínio. Estagnando ao redor dos três milhões de hectares, e recua para menos de dois milhões de hectares já no início dos anos 90 e para menos de um milhão (657,6 mil hectares) na safra 1996/97. A partir de então volta a crescer e atualmente o Brasil planta algodão em cerca de 1,4 milhões de hectares (DESER, 2007; USDA, 2012).
Mato Grosso (50,2%), Bahia (29,7%) e Goiás (8,4%) (INSTITUTO BRASILEIRO DE GEOGRAFIA E ESTATÍSTICA - IBGE, 2012). Na última década, a cotonicultura avançou em áreas de cerrado do Brasil central (SUINAGA, 2003). Este avanço, nos últimos anos, culminou com o aparecimento de uma nova estrutura de produção em que são utilizadas grandes áreas planas para o cultivo, alta tecnologia, produtores com perfil empresarial, ação direta das instituições de pesquisa e excelente clima para o desenvolvimento da cultura (BELTRÃO; AZEVEDO, 2008b). No entanto, para que a cultura continue em expansão é necessário atentar para alguns fatores que interferem diretamente na produtividade e no custo de produção, sendo que o ataque de pragas em geral e o uso excessivo de agrotóxicos exigem grandes investimentos, onerando e às vezes inviabilizando a produção.
1.2 INSETOS-PRAGA DA CULTURA DO ALGODÃO
O algodoeiro é considerado uma das plantas cultivadas mais susceptíveis ao desenvolvimento e ataque de insetos-praga, com mais de 20 espécies relatadas e conhecidas (GALLO et al., 2002). Estes insetos que atacam o algodoeiro podem ser divididos em dois grupos: i) os que ocorrem, principalmente, no estabelecimento da cultura, destacando-se a broca-da-raiz (Eutinobothrus brasiliensis), broca-do-ponteiro (Conotrachelus denieri), percevejo castanho (Horcias nobilellus) e o pulgão (Aphis gossypii), e ii) os que ocorrem, principalmente, no florescimento e na frutificação, destacando-se o curuquerê (Alabama argillacea), a mosca branca (Bemisia argentifolii), a lagarta-da-maçã (Heliothis virescens), ácaro branco (Polyphagotarsonemus latus), ácaro rajado (Tretanychus urticae), percevejo rajado (Horcias nobilellus), percevejo manchador (Dysdercus ruficollis), lagarta militar (Spodoptera frugiperda), lagarta rosada (Pectinophora gossypiella) e o bicudo-do-algodoeiro (Anthonomus grandis) (BRASIL, 2007). Esses artrópodes coexistem com o algodoeiro num complexo relacionamento por um longo período evolucionário, o que resultou em adaptações morfológicas e bioquímicas de ambas as partes (HEDIN; MCCARTY, 1995; BASTOS et al., 1999; SCATAGLINI et al., 2000; MAO et al., 2007).
mais assustadora com a demanda da cotonicultura, responsabilizada por 27% do total de inseticidas utilizados no país (BARROSO; HOFFMANN, 2007). Grande parte dos inseticidas utilizados na cotonicultura no Brasil é empregada no controle do bicudo-do-algodoeiro.
1.3 O BICUDO-DO-ALGODOEIRO (Anthonomus grandis)
O bicudo-do-algodoeiro é uma das mais destrutivas pragas do algodoeiro, devido a sua rápida capacidade reprodutiva e de destruição de botões florais e maçãs (HAYNES; SMITH, 1992). O bicudo-do-algodoeiro tornou-se uma praga-chave da cultura do algodão no país devido a sua rápida disseminação, grandes danos causados nas áreas cotonicultoras, capacidade de adaptação a novos ambientes, reforçado por suas características biológicas e comportamentais favoráveis (ALMEIDA; SILVA, 1999).
A subfamília Anthonominae, a qual pertence o bicudo, é composta de aproximadamente 33 gêneros e cerca de 500 espécies descritas. Anthonomus é o maior gênero da subfamília e contém cerca de 350 espécies descritas, com muitas outras a serem descritas. O indivíduo adulto do bicudo é de coloração cinza ou castanha, com aproximadamente 7 mm de comprimento, apresentando um bico com cerca de 2 mm. As fêmeas tem uma vida média de 20 a 30 dias e podem ovipor de 100 a 300 ovos. Do ovo até a emergência do adulto, todas as fases da vida do bicudo processam-se no interior do botão floral ou da maçã, tendo, pois, um desenvolvimento em habitat relativamente bem protegido. Este inseto possui uma extraordinária capacidade reprodutiva podendo produzir de três a sete gerações no ciclo normal da cultura, dependendo das condições de umidade e temperatura. Isto significa que de 50 bicudos que entra em diapausa, espera-se uma população de 500.000 adultos ao fim da próxima safra (BRAGA SOBRINHO; LUKEFAHR, 1983). Portanto, os níveis de infestação crescem rapidamente e os prejuízos podem a tingir até 100% da produção, caso as medidas de controle não sejam adequadas e realizados no momento correto.
algodoeiro. Desde sua introdução no Brasil e , particularmente no Nordeste, estudos têm sido conduzidos visando gerar tecnologias que sejam econômica e ecologicamente viáveis para serem utilizadas no seu controle. Entretanto, a maioria dos esforços realizados até hoje tem se concentrado no aspecto agronômico incluindo: o manejo integrado de pragas, o melhoramento clássico visando o desenvolvimento de cultivares de ciclo curto e maturação rápida, o desenvolvimento de armadilhas e a apresentação de novos inseticidas (ALMEIDA; SILVA, 1999; BASTOS et al., 1999; BUSOLI et al., 2004).
Mesmo com o emprego das atuais formas de controle, o bicudo-do-algodoeiro se mantém como a praga que causa os maiores prejuízos à cultura do algodão no Brasil, devido, principalmente, ao fato de a fase larval do desenvolvimento do inseto ocorrer endofiticamente, no botão floral. Assim, este inseto não é passível de controle por inseticidas ou métodos convencionais. Desta maneira, o uso da biotecnologia se apresenta como uma alternativa promissora, por meio da incorporação de genes inseticidas, para a obtenção de plantas de algodão GM resistente a inseto-praga (OLIVEIRA et al., 2011).
1.4 CULTIVARES DE ALGODÃO GENETICAMENTE MODIFICADOS
De fato, o algodão é uma das culturas que mais tem se beneficiado dos avanços da biotecnologia, sendo o terceiro principal cultivo transgênico (JAMES, 2011a). Atualmente, a resistência a inseto e tolerância a herbicida são as duas características agronômicas aprovadas e amplamente comercializadas que foram introduzidas no algodão através de engenharia genética (MEHBOOB-UR-RAHMAN et al., 2012).
Tabela I-I – Eventos de algodão GM aprovados no Brasil
Adaptada de: (ISAAA, 2012)
Desde a adoção comercial de algodão Bt em 1996 nos Estados Unidos e Austrália, o número de países que o produzem comercialmente e a área cultivada vêm aumentando consistentemente. Em 2011, o algodão Bt foi cultivado em 17,9 milhões de hectares (ha), o que corresponde a mais de 72 % da área total do algodão cultivado em todo o mundo (JAMES, 2011b). A Índia é hoje o país com a maior área plantada de algodão Bt (8,4 milhões de ha), seguido pela China (3,7 milhões ha) e os EUA (2 milhões ha). Cerca de 8,25 milhões de produtores de algodão utilizam plantas geneticamente modificadas, dos quais 90% são pequenos agricultores. A ampla adoção do algodão Bt é explicada dado aos múltiplos benefícios alcançados pelos agricultores que adotam esta tecnologia, incluindo o aumento de produtividade, redução no uso de inseticidas e aumento na margem de lucro, resultando em benefícios econômicos, sociais e ambientais. Segundo estudo realizado na Índia, o algodão Bt
foi responsável pelo aumento do rendimento da produção e da lucratividade em 24% e 50%, respectivamente (QAIM, 2010; KOUSER; QAIM, 2011; KATHAGE; QAIM, 2012).
Porém, apesar dos eventos de algodão GM disponíveis atualmente serem eficientes no controle de vários insetos lepidópteros que atacam o algodoeiro, estes não apresentam resistência ao bicudo-do-algodoeiro e, portanto, há a necessidade do desenvolvimento de
Evento Gene inserido Característica Companhia
Bollgard Cry1ac Resistência a insetos Monsanto
Bollgard II Cry1Ac + Cry2Ab2 Resistência a insetos Monsanto
WideStrike Cry1ac + Cry1f Resistência a insetos/Tolerância a herbicida Dow AgroSciences
GlyTol™ 2mepsps Tolerância a herbicida Bayer CropScience
GlyTol™ Liberty Link™ 2mepsps + Bar Tolerância a herbicidas Bayer CropScience
Glytol™ x Twinlink™ 2mepsps + Bar +
Cry1Ab +Cry2ae Resistência a insetos/Tolerância ao herbicida Bayer CropScience
Fibermax™ Liberty
Link™ Bar Tolerância a herbicidas Bayer CropScience
RoundUp Ready (RR) CP4 epsps Tolerância a herbicida Monsanto
Roundup Ready™ Bollgard II™ Cotton
CP4 epsps+ Cry1Ac
+ Cry2ab2 Resistência a insetos/Tolerância a herbicida Monsanto Roundup Ready™ Flex™
Cotton CP4 epsps Tolerância a herbicida Monsanto
Roundup Ready™ Flex™ Bollgard II™ Cotton
CP4 epsps+ Cry1Ac
+ Cry2ab2 Resistência a insetos/Tolerância a herbicida Monsanto
TwinLink™ Cotton Bar + Cry1Ab
eventos GM de acordo com a demanda do Brasil. Este fato reforça a necessidade da busca por novas moléculas com potencial inseticida para a geração de novos eventos de algodão GM. Assim, esforços têm sido despendidos pela Embrapa, com o intuito de obter plantas de algodão GM resistentes ao bicudo-do-algodoeiro.
1.5 RNA INTERFERENTE (RNAI) NO CONTROLE DE PRAGAS
O potencial acúmulo de resíduos de pesticidas no agrossistema e nos produtos vegetais causa uma crescente preocupação mundial. Além das exigências impostas pela lei à não presença ou aceitação de resíduos tóxicos nos alimentos, a sociedade está, a cada dia, exigindo produtos que estejam seguramente livres de agrotóxicos (SALLES, 2000). Com o objetivo de diminuir os aspectos ambientais negativos e de superar a resistência de populações de insetos resistentes aos inseticidas neurotóxicos convencionais, a tendência nas pesquisas é o desenvolvimento de métodos eficientes com baixa toxicidade para a saúde humana e para o meio ambiente (DHADIALLA et al., 1998; GRAPOV, 1999).
A adoção cada vez maior de plantas transgênicas resistentes a insetos tem mostrado diversos benefícios econômicos, ambientais e para a saúde humana (CHRISTOU et al., 2006). As principais variedades transgênicas com efeitos entomotóxicos bem estabelecidos expressam toxinas do Bacillus thuringiensis (CHRISTOU et al., 2006). Em outras variedades genes que expressam inibidores de proteinases e lectinas também são utilizados (HILDER; BOULTER, 1999; FERRY et al., 2006). Nos últimos anos, especial foco tem sido dado à utilização de RNA de interferência para o controle de pragas (PRICE; GATEHOUSE, 2008).
O fenômeno do RNA interferente (RNAi), no qual um RNA fita dupla (dsRNA – do inglês double-stranded RNA) induz o silenciamento gênico, foi documentado pela primeira vez em 1990, quando ao acaso, a introdução do gene da calcona sintase, visando aumentar a pigmentação de flores de petúnia, resultou na redução da expressão do gene endógeno, resultando em flores parcialmente ou totalmente despigmentadas (NAPOLI et al., 1990). Alguns anos depois, o silenciamento de RNAm por RNA sense e antisense foi descrito em um modelo animal, o nematoide Caenorhabditis elegans (GUO; KEMPHUES, 1995).
(VOINNET; BAULCOMBE, 1997; WATERHOUSE et al., 2001). Entretanto, atualmente sabe-se que esse processo participa de forma integral na regulação da expressão gênica em várias plantas e outros eucariotos (LILLEY et al., 2007).
Desde a identificação do dsRNA como a molécula gatilho do processo de silenciamento gênico, uma década de trabalho resultou na elucidação do mecanismo molecular da via "clássica" do RNAi. O processo básico da via "clássica" do RNAi pode ser dividido em três etapas principais (TOMARI; ZAMORE, 2005). Na primeira etapa, um dsRNA endógeno ou exógeno, que é expresso ou introduzido na célula é processado em pequenas moléculas de dsRNA com 21 a 26 nucleotídeos, denominados siRNA (do inglês -
short interfering RNA), por uma ribonuclease do tipo RNAse III, a Dicer. Dependendo do organismo pode existir uma ou mais Dicers, cada uma responsável pela produção de um diferente tipo de dsRNA (MEISTER; TUSCHL, 2004). Por exemplo, em D. melanogaster, Dicer-1 está envolvida, principalmente, na produção de miRNA (do inglês - micro interfering RNA); enquanto, Dicer-2 é responsável pelo processamento de dsRNAs longos em siRNA (LEE et al., 2004). Durante a segunda etapa do mecanismo de RNAi, esses duplexs são transportados para o complexo efetor RISC (do inglês - RNA-induced silencing complex), onde ocorre a separação das fitas e uma delas, denominada de fita guia, é preferencialmente acoplada no complexo proteico chamado RISC. Na última etapa, a fita guia é utilizada para reconhecer os RNAm alvos pelo pareamento de bases Watson-Crick. O silenciamento do gene é um resultado da degradação do RNAm alvo pela enzima Argonaute (Ago), uma RNaseH que faz parte do complexo RISC (GAYNOR et al., 2010). Se o duplex siRNA/mRNA contém bases despareadas no sítio de clivagem, muitas vezes o caso de miRNAs, o RNAm não é clivado. Em vez disso, o silenciamento do gene é um resultado da inibição da tradução (HAMMOND, 2005; GHILDIYAL; ZAMORE, 2009) (Figura I-1).
silenciamento do RNAm por siRNA; (ii) silenciamento do RNAm ou inibição da tradução por miRNA (GHILDIYAL; ZAMORE, 2009).
Na natureza, o dsRNA pode ser produzido pela polimerização de RNA em molde de RNA (por exemplo, a partir de eventos de transcrição ou replicação vírus de RNA) ou pela hibridização de transcritos sobrepostos (por exemplo, a partir de sequências repetitivas, tais como cópias de transgene ou transposons). Tais dsRNAs originam siRNAs ou rasiRNAs (do inglês - repeat-associated short interfering RNAs), que geralmente guiam a degradação de RNAm e/ou modificação da cromatina .
O estudo do RNAi resultou na elucidação de um sistema altamente conservado e complexo da regulação da expressão gênica que é mediado por meio de uma variedade de produtos à base de RNA (siRNAs, os miRNAs, ou rasiRNAs). Com o progresso dos estudos, a vasta abrangência desta via ainda está por ser revelada. Atualmente, sabe-se que a via de RNAi é um mecanismo essencial de proteção contra infecções virais (WATERHOUSE et al., 2001; ZAMBON et al., 2006), previne a inserção aleatória de elementos transponíveis (OBBARD et al., 2009), também possui implicações importantes no papel biológico da heterocromatina e manutenção do genoma (LIPPMAN; MARTIENSSEN, 2004). Ademais, miRNAs desempenham papéis importantes no desenvolvimento e processos celulares básicos, estima-se que esses RNAs influenciem a expressão de 30% de todos os genes codificadores de proteínas (OUELLET et al., 2006)
Além do papel essencial do RNAi na expressão de genes endógenos, ele também tem sido muito explorado em estudos genéticos como uma ferramenta poderosa para se obter a perda de função de fenótipos, já que a clivagem do RNAm reduz a sua quantidade disponível para tradução e, assim, mimetiza a perda de função por mutação. Áreas como biologia do desenvolvimento, biologia celular, biologia evolutiva e genômica funcional têm sido drasticamente impactadas pela capacidade de se analisar rapidamente a função do gene. Além da vantagem de usar o RNAi como uma ferramenta para a análise funcional dos genes em processos biológicos, atualmente, diferentes aplicações biotecnológicas são discutidas, incluindo a terapia médica para a infecção viral ou doenças genéticas (SHANKAR et al., 2005), manejo de pragas (BAUM et al., 2007; MAO et al., 2007) dentre outras.
Com relação ao manejo de insetos-pragas, os insetos-modelos são considerados ferramentas eficientes para o estudo de genômica funcional (BELLES, 2010; NOH et al., 2012). Muitos destes cumprem os critérios para um organismo modelo, incluindo pequeno tamanho corporal, curto tempo de geração e quantidade da ninhada. Os insetos representam um grupo extremamente diversificado em relação à morfologia e as características dos seus ciclos de vida, o que os torna modelos ideais para estudos comparativos nas áreas de fisiologia, biologia evolutiva, biologia do desenvolvimento e biologia populacional. Além disso, o conhecimento da biologia do inseto é crucial para se resolver os problemas causados, por exemplo, por vetores de doenças e pragas agrícolas.
aplicadas em insetos, resultando em diversos relatos na literatura que a ingestão de dsRNA pode reprimir os genes-alvo nesses animais (ZHOU et al., 2008; WALSHE et al., 2009; CHEN et al., 2010; LI et al., 2011). Por exemplo, Araújo e colaboradores (2006) demonstraram que a administração de dsRNA contra o gene da nitroforina salivar 2 (NP2) na alimentação do barbeiro Rhodnius prolixus resultou numa redução de 42% dos transcritos de NP2. Bautista e colaboradores (2009) visaram como alvo o gene do citocromo P450, CYP6BG1, da traça das crucíferas, Plutella xylostella, via RNAi oral. A redução do número de transcritos de CYP6BG1 foi evidente no intestino médio, bem como na carcaça e nos músculos. As larvas tratadas com dsRNA mostraram um aumento significativo na susceptibilidade a permetrina, a qual é normalmente inativada pelo citocromo P450. Estes resultados sugerem que, pelo menos, em alguns insetos, a ingestão de dsRNAs ativa a via do RNAi que pode se espalhar a partir do intestino para outros tecidos, resultando na resposta sistêmica do RNAi.
Trabalhos recentes têm demonstrado que plantas transgênicas que produzem shRNAs (do inglês - small harpin RNA) podem proteger tais plantas contra o ataque de insetos-pragas (BAUM et al., 2007; MAO et al., 2007; ZHA et al., 2011). Em um desses trabalhos, Baum et al. (2007) identificaram 290 genes de uma biblioteca de cDNA da lagarta da raiz do milho, Diabrotica virgifera, que poderiam ter funções biológicas críticas. Foram sintetizados dsRNAs contra cada gene, destes apenas dsRNAs para 14 dos genes selecionados produziram nanismo e mortalidade significativa quando administrados por via oral em dieta artificial. Os autores selecionaram apenas um destes, V-ATPase A, que mostrou rápido silenciamento do gene em uma baixa concentração de dsRNA, e gerou um milho transgênico expressando V-ATPase shRNA. As plantas transgênicas mostraram uma significativa proteção contra danos causados pela infestação de D. virgifera.
1.6 TRANSCRITOMA DO BICUDO-DO-ALGODOEIRO: DESCOBERTA E SELEÇÃO DE GENES COM POTENCIAL PARA RNAI
A pesquisa sobre insetos, especialmente nas áreas de fisiologia, bioquímica e biologia molecular, passou por transformações notáveis durante as últimas duas décadas. A conclusão do sequenciamento do genoma do primeiro inseto, a mosca da fruta Drosophila melanogaster, em 2000, foi seguida por uma enxurrada de atividades voltadas para o sequenciamento dos genomas de várias outras espécies de insetos. Até o presente, genomas de insetos de diferentes ordens já foram sequenciados dentre estes Bombyx mori, Aedes aegypti, Apis mellifera, Acyrothosyphon pisum, Drosophila melanogaster, Tribolium castaneum (ADAMS et al., 2000; THE HONEY BEE GENOME CONSORTIUM, 2006; NENE et al., 2007; RICHARDS et al., 2008; THE INTERNATIONAL SILKWORM GENOME CONSORTIUM, 2008; THE PEA APHID GENOME CONSORTIUM, 2010).
O sequenciamento cada vez mais rápido destes genomas foi favorecido devido ao desenvolvimento de tecnologias de sequenciamento massal, também denominadas tecnologias de Sequenciamento de Nova Geração (NGS, do inglês - Next Generation Sequencing). A partir de 2005 essas novas tecnologias começaram a ser utilizadas como prestação de serviços e evoluíram rapidamente (MARGULIES et al., 2005; METZKER, 2010).
Quando comparado com o sequenciamento automatizado de Sanger (SANGER; COULSON, 1975), a tecnologia de NGS acelerou dramaticamente a velocidade de obtenção de sequências por meio do aumento do número de reações de sequenciamento e da redução do volume de reação em um único equipamento de sequenciamento. Ao contrário de sequenciamento de Sanger, os eventos de incorporação dos nucleotídeos marcados com fluorescência aos moldes de DNA são continuamente monitorados e registrados. Mais de 100 milhões sequências curtas (variando entre 35pb a mais de 300pb) podem ser obtidas por corrida usando algumas das tecnologias NGS, o que acarreta uma grande economia de tempo e custo por base para o sequenciamento. Várias plataformas NGS, incluindo Roche/454 Life Sciences’ GS FLX, Illumina’s Solexa GAII, e ABI’s SOLiD, estão disponíveis comercialmente. Cada plataforma tem seus próprios métodos de sequenciamento e características próprias (MARDIS, 2008; METZKER, 2010).
do custo do sequenciamento de cromossomas completos, além de possibilitar o estudo simultâneo da expressão e da regulação de vários genes em diferentes tecidos e em diferentes processos biológicos, possibilitando avanços na identificação e caracterização de genes (EMRICH et al., 2007; MARDIS, 2008). A plataforma de sequenciamento Roche/454 tem sido amplamente utilizada para sequenciar o transcritoma de espécies de insetos pouco estudadas (VERA et al., 2008) e uma das principais vantagens do pirosequenciamento 454 é a redução de problemas de montagem associados às leituras curtas, crucial, quando não existe um genoma de referencia (EMRICH et al., 2007).
Diversos aspectos ecológicos e fisiológicos sobre o bicudo-do-algodoeiro já foram bem documentados (BRAGA SOBRINHO; LUKEFAHR, 1983; HEDIN; MCCARTY, 1995), porém dados de estudos genéticos deste inseto praga são escassos na literatura. Dentro da estratégia de silenciamento gênico é imprescindível o conhecimento de sequências expressas num determinado organismo, buscando encontrar em sua fisiologia, genes e proteínas imprescindíveis para o seu desenvolvimento e sobrevivência. Assim, o pirosequenciamento por 454 permitiu a construção de um banco de dados com 20.841 de sequências expressas (ESTs) envolvidas no desenvolvimento do inseto praga, bicudo-do-algodoeiro. Este banco de dados tornou-se, portanto, um recurso poderoso para estudos moleculares de um inseto sem informações de sua bagagem gênica, podendo auxiliar, dentre outras aplicações, na detecção das moléculas-alvo para o silenciamento gênico (FIRMINO et al., in press).
Com base na anotação funcional do transcritoma do bicudo-do-algodoeiro foi possível identificar diversas rotas metabólicas essenciais ao desenvolvimento do inseto, dentre estas, a via envolvida na síntese dos compostos quitinosos será abordada neste trabalho. Diversos estudos mostram que alterações no metabolismo de quitina por RNAi causam danos no desenvolvimento de insetos, o que coloca os genes da biossíntese da quitina como possíveis alvos para o uso em estratégia potencial para o controle especifico de insetos-praga (ALVES et al., 2010; AMPASALA et al., 2011; NOH et al., 2012).
1.7 QUITINA: OCORRÊNCIA E ESTRUTURA
A quitina é um polissacarídeo linear formado por resíduos de N-acetil-D-glucosamina
uma grande variedade de grupos taxonômicos. É encontrado, por exemplo, na parede celular de fungos, na parede cística de Entamoeba histolitica (CAMPOS-GONGORA et al., 2004), no bico de cefalópodes (HANLON; MESSENGER, 1996), na faringe e na casca dos ovos de nematóides (VERONICO et al., 2001), assim como no exoesqueleto de artrópodes (MERZENDORFER, 2006).
A quitina é amplamente difundida entre os insetos, os quais utilizam este versátil biopolímero em várias estruturas anatômicas. As duas principais estruturas extracelulares onde ocorre a deposição de quitina são a cutícula que reveste a epiderme e a membrana peritrófica que recobre o intestino médio. Em adição, significante deposição de quitina ocorre na traquéia, no intestino anterior, no intestino posterior, nas glândulas salivares, e nas estruturas bucais de larvas e adultos (MUTHUKRISHNAN et al., 2012).
Apesar de sua composição conter apenas resíduos de N-acetil-D-glucosamina, os insetos fazem uso das propriedades químicas da quitina para montar diferentes estruturas, que dependendo da função, necessitam de rigidez e flexibilidade características essas relacionadas com a organização molecular das cadeias de quitina.
Sabe-se que as cadeias de quitina tendem a formar microfibrilas de aproximadamente 3nm de diâmetro e que são estabilizadas por pontes de hidrogênio formadas entre o os grupamentos amino e carbonila do N-acetil-D-glucosamina. Análises por difração de raios-X sugerem que a quitina é uma substancia polimórfica que ocorre na natureza em três diferentes formas cristalinas denominadas de e . Na forma , todas as cadeias estão arranjadas de forma antiparalela; na forma as cadeias estão arranjadas paralelamente; e na forma existe uma mistura alternada de cadeias paralelas com cadeias antiparalelas. A principal diferença entre as formas é o grau de hidratação, que confere às estruturas diferentes propriedades físicas (RUDALL; KENCHINGTON, 1973).
estrutura mais relaxada, ambas e quitina formam estruturas quitinosas mais flexíveis como as da membrana peritrófica e do casulo (PETERS, 1992). O modelo de arranjo das fibras de quitina descrito acima é certamente simplista e não explica as propriedades físico-químicas das cutículas e da matriz peritrófica adequadamente porque é reduzido para apenas um dos componentes de uma estrutura complexa. No entanto, as diferenças na disposição dos microfibrilas de quitina, entre cutícula e matriz peritrófica, podem ajudar a compreender a sua função. A cutícula é secretada sob a forma de camadas finas a partir das microvilosidades apicais das células da epiderme, sendo abordada com mais detalhes no Capitulo III. As microfibrilas de quitina na cutícula são embebidas em uma matriz de proteína e estabilizadas de uma maneira que se assemelha a construções de concreto armado, em um padrão altamente organizado, o que fornece rigidez e estabilidade (BOULIGAN.Y, 1972). Em contraste, na matriz peritrófica, as microfibrilas são normalmente montadas como uma rede organizada de forma aleatória, embebida em uma matriz amorfa, o que permite a característica de semi-permeabilidade (TELLAM et al., 1999).
1.8 SÍNTESE DE QUITINA EM INSETOS
Relativamente pouco se conhece sobre a via biosintética da quitina em insetos ou outros invertebrados, sendo atribuída, principalmente, à dificuldade de se obter preparações solúveis de quitina sintases e a instabilidade da glutamina-frutose-6-fosfato aminotransferase, enzima que fornece o precursor glucosamina da via biossintética da quitina (MUTHUKRISHNAN et al., 2012). A síntese de quitina em inseto é iniciada com a produção de glucose, proveniente da clivagem da trealose pela enzima trealase (EC 3.2.1.28). A última reação da via é realizada pela quitina sintase (EC 2.4.1.16), que catalisa a polimerização da quitina a partir de monômeros ativados de UDP-N-acetilglucosamina (Figura I-2) (MERZENDORFER, 2011).
Figura I-2. Via biossintética da quitina em insetos. A via inicia com trealose, o principal açucar da hemolinfa dos insetos, e termina com o polimero de quitina. A trealose é inicialmente convertida em glucose pela trealase (1) (3.2.1.28). A conversão da glucose em frutose-6-P envolve a hexoquinase (2) (EC 2.7.1.1) e a glucose-6-P-isomerase (3) (EC 5.3.1.9). A partir da Frutose-6-P a via biosintética da quitina se ramifica. A primeira enzima que catalisa este ramo é a glutamina-frutose-6-fosfato aminotransferase (4) (GFAT, EC 2.6.1.16). A reação catalisada pela GFAT converte a frutose-6-P em glucosamina-6-fosfato, pela transferência de um o grupamento amina a partir da L-glutamina e a isomerização frutosamina-6-fosfato resultante. Em seguida, um grupo acetil da coenzima A é adicionado pela glucosamina-6-P-acetiltransferase (5) (EC 2.3.1.4), para se obter a N
-acetilglucosamina (GlcNAc)-6-P, cujo fosfato é então transferido a partir do C-6 para o C-1 catalisado pela Fosfo-acetil-glucosamina mutase (6) (EC 5.4.2.3). O resultante GlcNAc-1-P, por fim, é uridinilado pela UDP-GlcNAc pirofosforilase (7) (EC 2.7.7.23), resultando em UDP-GlcNAc, que serve como o substrato para a quitina sintase (8) (CHS, EC 2.4.1.16) (MERZENDORFER; ZIMOCH, 2003). Trealase (1) Trealose -D- glucose Hexoquinase (2) Fosfo-acetil-glucosamina mutase (6) glucose 6-fosfato isomerase
(3) Glutamina-frutose-6-fosfato aminotransferase (4) glucose 6-fosfato Frutose 6-fosfato Glucosamina 6-fosfato
N-acetil Glucosamina
6-fosfato Glucosamina-6-fosfato
N-acetiltransferase (5)
N-acetil Glucosamina
1-fosfato UDP-N-acetilglucosamina pirofosforilase (7) Quitina sintase (8) Quitina
UDP-N-acetil
Diversos projetos de sequenciamento de genomas de fungos revelaram a presença de dois a vinte genes de quitina sintases por espécie, as quais foram categorizadas em sete classes (LENARDON et al., 2010). Em fungos, as diferentes quitina sintases possuem funções distintas, incluindo os processos de divisão celular e esporulação, e sua expressão varia ao longo dos estágios de desenvolvimento (HENAR VALDIVIESO et al., 1999; RONCERO, 2002; MARTIN-UDIROZ et al., 2004; KONG et al., 2012).
Em contraste com os genomas de fungos, em todos os genomas de insetos sequenciados até o presente somente dois genes de quitina sintase foram encontrados por espécie, os quais foram agrupados em duas classes de genes, classe A e classe B. A divisão das quitina sintases de insetos em duas classes possui relevância funcional, assim como é bem relatada em fungos a especialização funcional de suas quitina sintases (MARTIN-UDIROZ et al., 2004; KONG et al., 2012). Em insetos, os genes de quitina sintases da classe A especializaram-se na síntese de quitina da cutícula, enquanto os da classe B são especializadas na produção de quitina para a membrana peritrófica (ARAKANE et al., 2008).
As quitina sintases pertencem à família de glicosiltransferases 2 (GT2), da qual também fazem parte as celulases sintases (COUTINHO et al., 2003). As enzimas desta família geralmente utilizam um mecanismo catalítico em que ocorre a inversão da configuração anomérica do açúcar doador (MERZENDORFER, 2011).
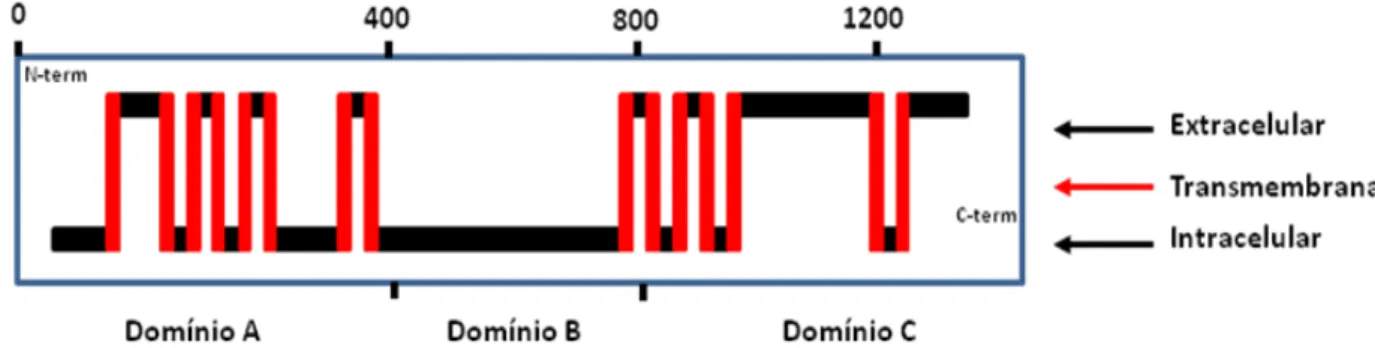
Figura I-3. Representação esquemática da organização dos domínios da quitina sintase de insetos. Estrutura dos domínios e topologia de membrana da quitina sintase 1 de Manduca sexta (Acesso nº. AY062175).
As hélices transmembranas estão representadas como barras verticais, as regiões citoplasmática ou extracelular estão descritas como barras horizontais. Os domínios N-terminal, catalítico e C-terminal estão marcados com A, B e C, respectivamente, seguindo a nomenclatura sugerida anteriormente (Tellam et al., 2000). Blocos altamente conservados estão marcados com as respectivas sequências
conservadas. A caixa cinza sombreado destaca a região que é afetada pelo exon alternativo nos genes da classe A. As caixas de vermelho, azul e amarelo indicam os supostos locais de N-glicosilação catálise, e coiled-coils, respectivamente. Adaptado de Merzendorfer et al., 2003.
Atualmente, há dois modelos alternativos para a síntese de quitina em insetos (MERZENDORFER; ZIMOCH, 2003). No primeiro modelo, a quitina sintase é transportada para a membrana plasmática por vesículas intracelulares. Após a fusão com a membrana, a quitina sintase é ativada e subsequentemente transloca as cadeias de quitina para o espaço extracelular. No segundo modelo, mais especulativo, a quitina é secretada no lúmen de vesículas especializadas que se acumulam logo abaixo à teia terminal das microvilosidades das células epidérmicas e se funde com a membrana plasmática, quando a quitina precisa ser liberada. Este modelo permite o armazenamento do polímero de quitina e sua rápida liberação, o que pode ser importante para a secreção da matriz peritrófica logo após a alimentação em mosquitos sugadores de sangue (IBRAHIM et al., 2000). Apesar de todo o conhecimento acumulado na última década sobre a síntese de quitina em insetos, muitas questões referentes ao mecanismo de síntese e sua translocação através da membrana ainda não foram esclarecidas.
2 JUSTIFICATIVA
3 HIPÓTESE GERAL DA TESE
A síntese de quitina é um processo essencial para a formação de diversas estruturas morfológicas do corpo dos insetos. A redução da expressão das quitina sintases de Anthonomus grandis, por RNAi pode causar danos incompatíveis com o desenvolvimento do inseto, acarretando sua morte. Assim, o silenciamento gênico por RNAi pode se tornar um método eficiente no controle desse inseto-praga.
Em se confirmando essa hipótese, o principal benefício desse novo método para o controle do bicudo-do-algodoeiro é a redução dos prejuízos causados por este inseto na cotonicultura nacional, além de reduzir a utilização de agrotóxicos e seus efeitos nocivos ao homem e ao meio-ambiente e, assim, contribuir para a competitividade do agronegócio brasileiro.
Essa hipótese está baseada em uma série de relatos científicos que mostraram o potencial deletério causado pelo silenciamento de genes de quitina sintases em outros insetos-praga (ARAKANE et al., 2005; ALVES et al., 2010; AMPASALA et al., 2011).
4 OBJETIVOS
4.1 OBJETIVO GERAL
4.2 OBJETIVO ESPECÍFICO
Para se atingir o objetivo geral da tese foram executados trabalhos experimentais que estão agrupados e descritos em dois capítulos:
- Capitulo II – “Clonagem, caracterização e análise funcional da quitina sintase da classe II envolvida na síntese da membrana peritrófica em A. grandis”. Este capítulo teve como objetivo descrever o sequenciamento do cDNA da quitina sintase II de A. grandis, bem como obter a caracterização funcional de sua expressão ao longo do desenvolvimento do inseto e avaliar o seu potencial como alvo para o silenciamento gênico, visando desenvolver uma estratégia de controle desta praga”.
CAPÍTULO II – CLONAGEM, CARACTERIZAÇÃO E ANÁLISE FUNCIONAL DA QUITINA SINTASE DO TIPO II ENVOLVIDA NA SÍNTESE DA MEMBRANA PERITRÓFICA EM Anthonomus grandis.
1 INTRODUÇÃO
1.1 O SISTEMA DIGESTÓRIO DOS INSETOS
A habilidade dos insetos de se alimentar de diferentes tipos de matéria orgânica é o maior fator para o seu sucesso evolutivo, capacitando-os para os mais diversos nichos ecológicos. A grande variedade de alimentos que podem ser ingeridos pelos insetos é refletida na diversidade de estruturas das peças bucais bem como, na diversidade do trato digestório, com um alto grau de especialização, que varia com o tipo particular de dieta (WIGGLESWORTH, 1972). O trato digestório dos insetos é constituído por um tubo de células epiteliais que se estende da boca até o ânus (WIGGLESWORTH, 1972; TERRA; FERREIRA, 1994). Está dividido em três principais regiões, baseado na origem embrionária e na sua função fisiológica em estomodeu (intestino anterior), mesêntero (intestino médio) e proctodeu (intestino posterior), onde o principal local de absorção e digestão é o intestino médio (WIGGLESWORTH, 1972; TERRA; FERREIRA, 1994; TERRA et al., 1996) (Figura II-1).
1.2 MEMBRANA PERITRÓFICA COMO ALVO PARA CONTROLE DE INSETOS-PRAGA
Um dos principais objetivos de investigações básicas associadas ao manejo integrado de pragas é a perspectiva de entender e interferir nos processos vitais do inseto (genéticos, bioquímicos e morfofisiológicos), de modo que tal interferência possua escassa ou nenhuma influência sobre outros animais e, ao mesmo tempo, possa servir como instrumento potencial para o controle de espécies prejudiciais (CRUZ et al., 2000). Dentro desta perspectiva o sistema digestório dos insetos é uma importante região de exposição com o meio ambiente, e dessa forma, estratégias que possam interferir na bioquímica e fisiologia desta região, visando à redução de absorção de nutrientes, seriam ferramentas potencialmente eficientes no manejo de pragas (SHEWRY; LUCAS, 1997).
O trato digestório animal está exposto a uma variedade de agentes nocivos de natureza química, física e biológica, necessitando de mecanismos para a sua proteção. Nos vertebrados, o muco é uma secreção que recobre e protege o epitélio intestinal, enquanto auxilia o processo de digestão. Nos insetos, entretanto, não se observa uma camada mucosa propriamente dita recobrindo o epitélio intestinal, em seu lugar, o intestino médio dos insetos é protegido por uma estrutura acelular e semipermeável denominada membrana peritrófica ou matriz peritrófica ou, ainda, gel peritrófico (PETERS, 1992; LEHANE, 1997; TERRA, 2001). A membrana peritrófica é uma estrutura mucosa (WANG; GRANADOS, 1997), que difere do muco dos vertebrados pela incorporação de quitina, resultando em uma estrutura protéica reforçada por fibrilas de quitina (PETERS, 1992; TELLAM et al., 1999). É constituída principalmente por glicoproteínas e proteoglicanos (20-55%) e por quitina (3-40%) (KRAMER et al., 1995; LEHANE, 1997) em uma organização que fornece semi-permeabilidade e elasticidade a estrutura (LEHANE, 1997). A quitina é um importante componente estrutural da membrana peritrófica, além de fornecer rigidez, serve também como sítio de ancoragem para proteínas, como as peritrofinas (WANG; GRANADOS, 2001).
Essas proteínas interagem com outras peritrofinas mediada por sua ligação a oligossacarídeos contendo N-acetil-D-glucosamina e, desta maneira, uma malha tridimensional de interação de glicoproteínas poderia ser formada e contribuir para as características estruturais como força, elasticidade e porosidade que são observadas nas membranas peritróficas (TELLAM et al., 1999).
membrana peritrófica do tipo I, produzida por muitos insetos adultos hematófagos e coleópteros, é sintetizada por células epiteliais do intestino médio, sendo produzida em resposta a ingestão de alimento, que por descamação da superfície epitelial, dá origem a uma estrutura em forma de bolsa que recobre o alimento (LEHANE, 1997). O Tipo II de membrana peritrófica é produzido a partir de um pequeno órgão altamente especializado chamado de cárdia situado na região anterior do intestino médio. Este tipo de membrana peritrófica é constitutivamente produzido como um contínuo tubo com estrutura altamente organizada (PETERS, 1992; TELLAM; EISEMANN, 2000).
As principais funções atribuídas a esta membrana são a de proteção mecânica contra
injúria às células do intestino médio (WIGGLESWORTH, 1972), uma barreira física contra
microorganismos (PETERS, 1992), uma barreira seletiva para enzimas digestivas e produtos
de digestão (DAY; WATERHOUSE, 1953) e atua no mecanismo de reciclagem de enzimas
digestivas, fenômeno conhecido como circulação endo-ectoperitrófica (TERRA, 1988;
TERRA; FERREIRA, 1994; BOLOGNESI et al., 2001; TERRA, 2001).
Devido suas importantes funções fisiológicas, a membrana peritrófica é considerada um importante alvo estrutural para o estabelecimento de estratégias de controle de insetos (WANG; GRANADOS, 2001). Diversos estudos de caracterização da estrutura e composição das membranas peritróficas de insetos das diferentes ordens culminaram no acúmulo de conhecimento para o desenvolvimento de alternativas, visando causar-lhes alterações estruturais.
Diversos relatos demonstram que a interrupção da formação da membrana peritrófica por agentes capazes de se ligar na quitina presente nessa estrutura, e competir pelos sítios de ligação com as peritrofinas, ou ainda, degradar a quitina, podem alterar suas propriedades funcionais como a proteção química e mecânica do epitélio intestinal. Danos a membrana peritrófica acarreta um aumento da suscetibilidade e mortalidade dos insetos por agentes infecciosos como vírus e bactérias, além de interferir na assimilação de nutrientes (POWELL et al., 1998; WANG; GRANADOS, 2000; BOLOGNESI et al., 2001). Assim, a quitina, sendo um dos componentes majoritários do arcabouço estrutural da membrana peritrófica, é considerada a principal molécula alvo da membrana peritrófica.