L'étude de l'implication des facteurs de traduction lors de la mobilisation des points de contrôle a été réalisée en tirant parti du modèle d'embryon d'oursin. Nos études renforcent l’intérêt et les connaissances sur l’implication des facteurs de traduction lors de la mobilisation des points de contrôle du cycle cellulaire chez l’embryon d’oursin soumis à un stress et/ou à des dommages à l’ADN.
LISTE DES PUBLICATIONS
AVANT PROPOS
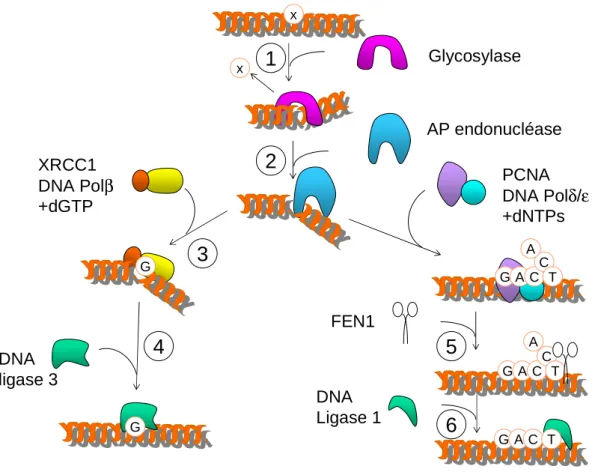
La première partie présente la régulation du cycle cellulaire et discute des différents mécanismes de surveillance qui existent pour répondre aux dommages à l'ADN. Nous avons ensuite décrit les mécanismes de réparation de l’ADN et d’apoptose, qui font partie de ces points de suivi.
INTRODUCTION
1 Point de surveillance du cycle cellulaire
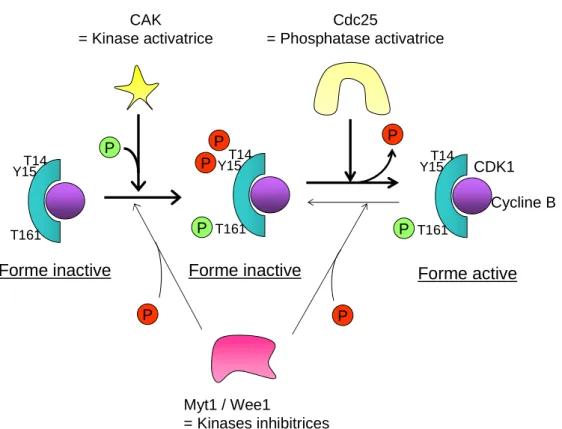
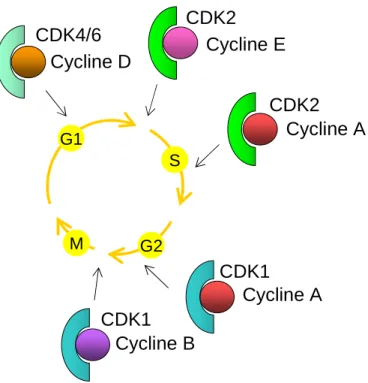
Les cyclines sont essentielles à l'activité des kinases CDK (Cyclin Dependent Kinase) et seul le complexe CDK-Cycline est actif. L’activité des complexes CDK-Cycline est également régulée par des protéines appelées CKI (Cyclin dependant Kinase Inhibitors).
CDK2
Les protéines de la famille de CKI Cip1/Waf1/Kip1-2, notamment p21Cip1/Waf1 et p27Kip1, peuvent se lier et inhiber le complexe CDK2-Cycline E (Figure 3). Les protéines p21Cip1/Waf1 et p27Kip1 pourraient également avoir une fonction activatrice et permettre l'assemblage du complexe CDK4-Cycline D (Coqueret 2003).
CDK4/6 CDK1
L'action de ces protéines induit un arrêt du cycle dans G1 par la formation de complexes ternaires INK4-CDK4/6-Cycline D ou binaires INK4-CDK4/6 (Jeffrey et al. Il a également été démontré que p27Kip1 est capable de s'associer au Complexe CDK2-cycline A kinase (Russo et al. 1996).
CDK1
CDK2 p27
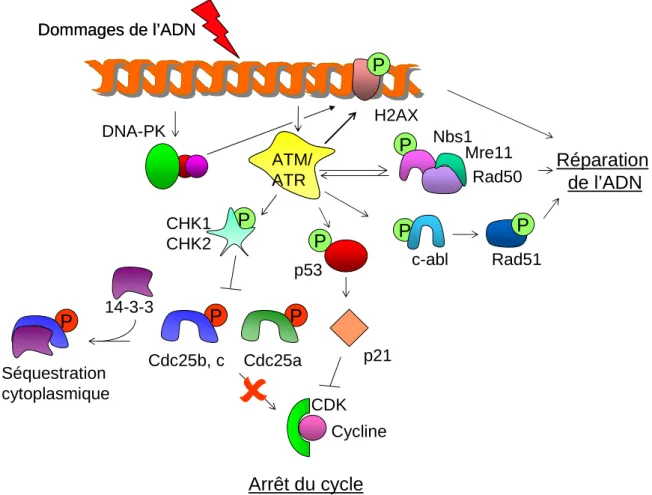
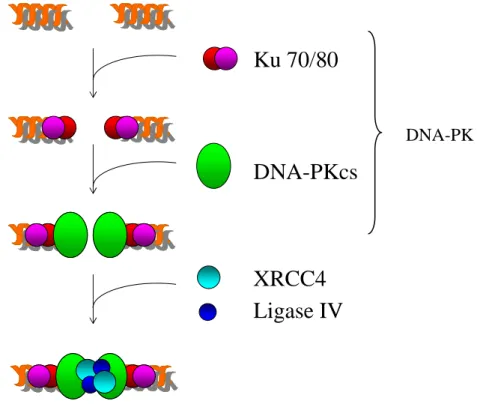
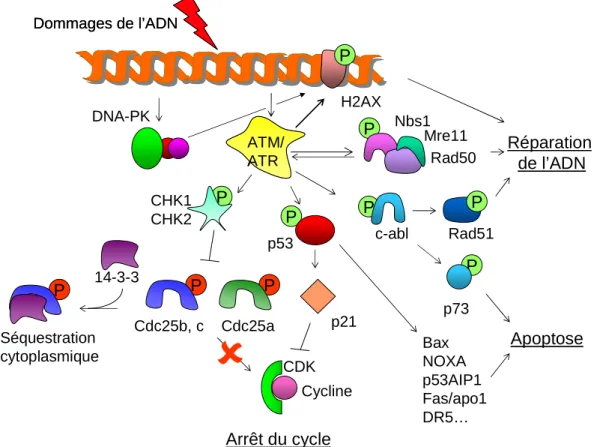
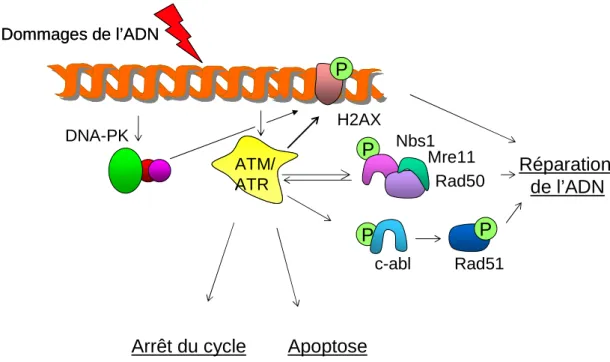
Les kinases ATM/ATR, activées lors des dommages à l'ADN, phosphorylent la protéine p53 (Vousden et Lane 2007), soit directement, soit via les kinases CHK1/CHK2 (Protéines Checkpoint 1 et 2). L’acteur clé de la réparation du NHEJ est la protéine kinase dépendante de l’ADN (ADN-PK).
XRCC4 Ligase IV
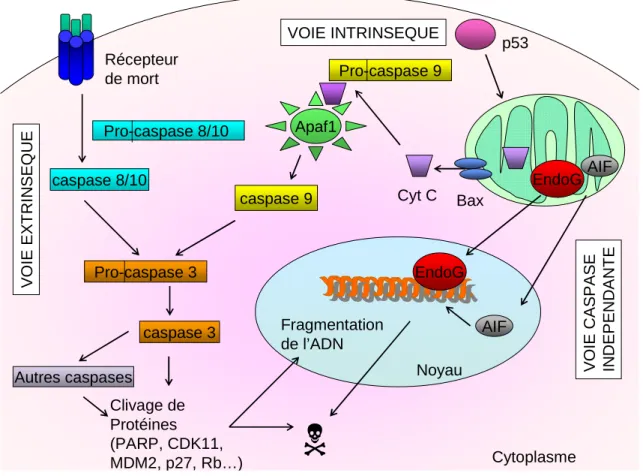
La protéine c-abl peut également être activée par la voie JNK (c-jun N-terminal kinase) et MAPKp38 (Kharbanda et al. 1998). Lorsque l'apoptose est induite, la protéine de 57 kDa est libérée par les mitochondries (Susin et al. 1999).
2 La traduction
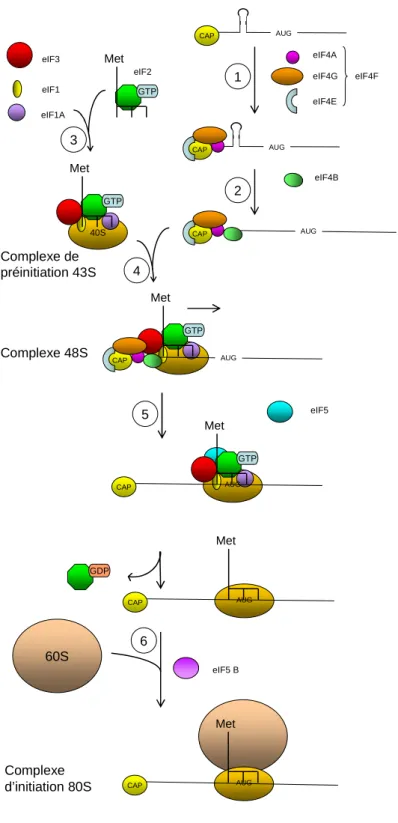
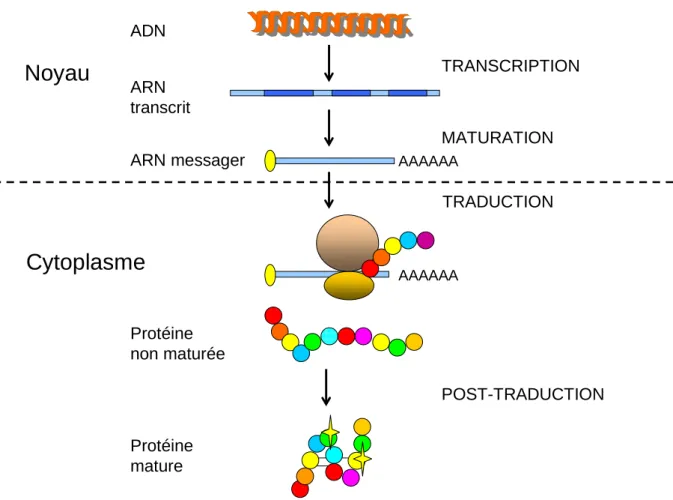
Implication des facteurs de traduction lors de l'initiation de la traduction dépendante du capuchon. La synthèse protéique dépendante de la casquette est régulée à toutes les étapes de l'initiation de la synthèse.
40S eIF4G
La synthèse des protéines est essentielle à l'expression de messagers codant pour des protéines régulatrices du cycle telles que les cyclines (Cao et Richter 2002 ; Salaun et al. 2003). La synthèse des protéines en mitose est bien régulée et permet l'expression de messagers spécifiques (Pyronnet et Sonenberg 2001).
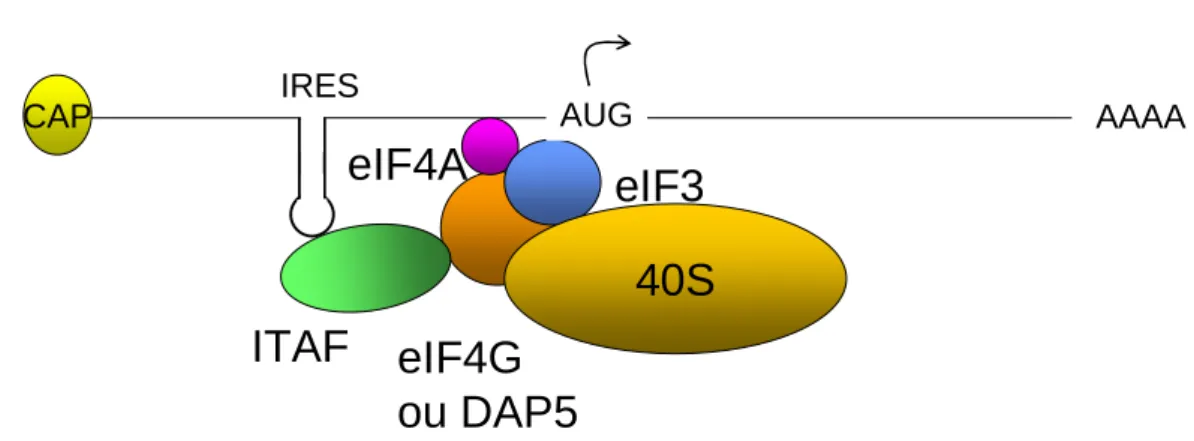
ITAF
La protéine messagère p27Kip1 est spécifiquement traduite à l'aide d'une séquence IRES dans la région 5'UTR (Miskimins et al. 2001). La protéine p110PITSLRE est impliquée dans l'épissage des pré-messages et la régulation transcriptionnelle (Trembley et al. 2002).
GCN4
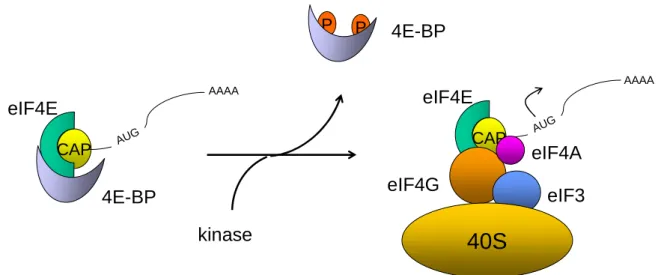
D'autres messagers sont également régulés de cette manière, comme le messager codant pour la protéine ATF4 (Activating Transcription Factor 4) (Harding et al. 2000). La protéine eIF4G est spécifiquement clivée par les caspases au cours du processus apoptotique (Clemens et al. 2000). La surexpression artificielle de la protéine eIF4E entraîne la transformation des cellules NIH-3T3 en cellules tumorales et l'apparition de tumeurs après leur injection à des souris nues (Lazaris-Karatzas et al. 1990).
La surexpression de eIF4E est corrélée à l'expression de TLK1B (Tousled-like kinase 1B) dans le cancer du sein (Byrnes et al. 2007). L'expression artificielle de plusieurs protéines du complexe multiprotéique eIF3 entraîne la transformation des cellules NIH3T3 en cellules malignes (Zhang et al. 2007).
La réparation des dommages à l'ADN fonctionne chez l'oursin Sphaerechinus granularis (Le Bouffant et al. 2007). Des gènes codant pour les protéines de la famille Bcl-2 ou IAP ont été trouvés chez les oursins. Le facteur 4E-BP a été impliqué dans la suppression de l'inhibition de la synthèse protéique en réponse à la fécondation (Cormier et al. 2001).
La première division de l'embryon d'oursin dépend de la protéine 4E-BP car l'inhibition de la dégradation de la protéine 4E-BP en réponse à la fécondation empêche la synthèse de la cycline B (Salaun et al. 2003). Le répresseur traductionnel 4E-BP est exprimé dans l'embryon d'oursin en réponse à un stress hypoxique, radiomimétique (bléomycine) et au chrome (Le Bouffant et al.
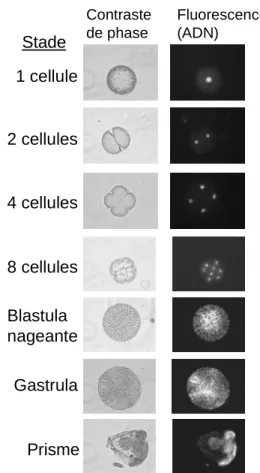
RESULTATS
Chromium impedes cell cycle progression and induces the apoptotic response
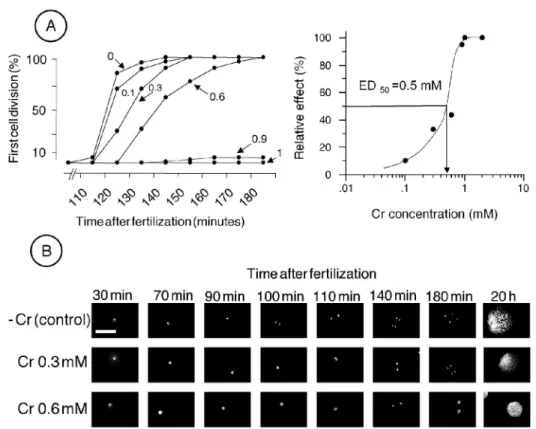
Cell cycle progression was drastically arrested as judged by the kinetics of the first cell cycle of early development (Figure 1A, left). Inhibition of cell cycle progression was dose-dependent with an ID50 of 0.5 mM chromium (Figure 1B, right). When observed after 20 hours, the number of nuclei in treated embryos was approximately half of that in control embryos, indicating a specific effect of chromium (< 0.6 mM), which is a delay in early development cell cycles without acute toxicological damage to the embryos (Fig. 1B, see 20 hours).
When chromium was applied at higher concentration (1 mM), the cell cycle was fully arrested (fig. 1A) and significant cytological changes were observed after 5 hours of development (fig. 2A). Caspase activity of embryo extracts prepared at different times after fertilization increased from 5 h and remained high until 30 h (fig. 2B), thus strengthening the conclusion that chromium 1 mM induced the apoptotic response that occurs after the DNA damage checkpoint cell cycle arrests in the event of DNA repair failure (28).
Chromium provokes an increase in the amount of 4E-BP
The 4E-BP chromium-induced increase is functional
Sea urchin eggs (5% suspension) were fertilized and treated (+Cr) or not (-Cr) after 20 min with 1 mM chromium. Sea urchin eggs (5% suspension) were fertilized and treated (+Cr) or not (-Cr) after 20 min with 1 mM chromium. Timing of protein synthesis required for mitosis in the cell cycle of the sea urchin embryo.
34; Inhibition of translation and modification of translation factors during apoptosis induced by the DNA damaging agent MMS in sea urchin embryos." Submitted. Title: Inhibition of translation and modification of translation factors during apoptosis induced by the DNA damaging agent MMS in sea urchin Inhibition of translation and modification of translation factors during apoptosis induced by the DNA-damaging agent MMS in sea urchin embryos.
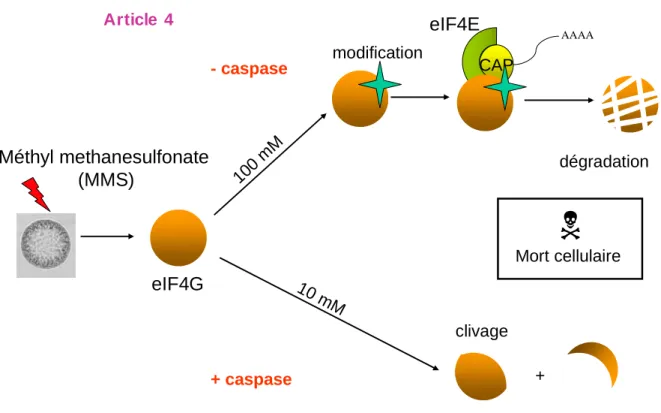
A later response to MMS exposure of sea urchins and embryos is the degradation of eIF4G.
CONCLUSIONS
ET PERSPECTIVES
CONCLUSION ET PERSPECTIVES
1 L’embryon d’oursin et endommagements de l’ADN
Les dommages à l'ADN provoquent un arrêt du cycle cellulaire impliquant le complexe CDK1-Cycline B, une réparation de l'ADN adaptée au type de dommage ou à l'apoptose de l'embryon d'oursin.
2 La protéine eIF2 αααα , un inhibiteur de la traduction dans l’embryon d’oursin
Ainsi, la protéine eIF2α participe à la régulation de l'activité de traduction de l'embryon pendant la fécondation et lors de la mobilisation des points de contrôle de l'ADN endommagé par le MMS. Il serait intéressant d'étudier la voie de signalisation responsable de la phosphorylation de eIF2α, d'une part en réponse à la fécondation et d'autre part, lors du traitement par MMS. Les résultats de l’annotation du génome de l’oursin ont permis de prédire que trois des quatre eIF2-kinases sont présentes chez l’oursin.
Les kinases GCN2, PERK et HRI ont été annotées dans cet organisme alors que la kinase PKR est absente. La kinase GCN2, impliquée dans la réponse cellulaire initiée après une lésion de l'ADN (Deng et al. 2002 ; Jiang et Wek 2005), est la candidate la plus prometteuse dans l'action du MMS.
3 La protéine 4E-BP, entre survie et apoptose
Ovule non fécondé
Le mécanisme responsable de la dégradation de la protéine 4E-BP dans le modèle oursin reste à identifier. Nos travaux montrent que la protéine 4E-BP est surexprimée lorsque les embryons d'oursins sont placés dans des conditions hypoxiques. Nous associons le rôle de la surexpression de la protéine 4E-BP à la survie cellulaire, comme cela a déjà été suggéré dans d'autres modèles (Teleman et al.
Il est intéressant de noter que les variations de l’expression de la protéine 4E-BP sont spécifiques au type de stress. Une voie d’analyse possible serait de forcer la surexpression de la protéine 4E-BP dans l’embryon d’oursin et de la traiter avec du MMS.
4 La protéine eIF4G dans tous ses états
La protéine eIF4G (orange) est modifiée en réponse à de fortes doses de méthanesulfonate de méthyle (MMS). La protéine eIF4G est clivée en réponse à de faibles doses de MMS et d'une manière corrélée à l'activation de la caspase. Nous avons montré qu'un fragment de la protéine eIF4G apparaît (fragment ~ 100 kDa) après traitement des embryons avec 10 mM de MMS.
Des sites potentiels de clivage des caspases sont observés dans la protéine eIF4G de l'oursin. Un test de clivage in vitro pourrait également être réalisé en incubant la protéine eIF4G avec des caspases actives.
5 Endommagement de l’ADN et synthèse protéique spécifique
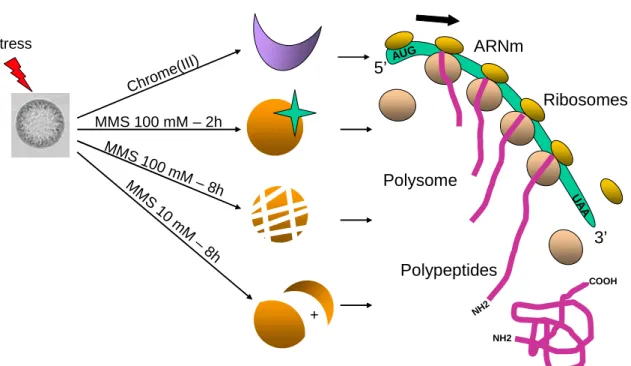
Enfin, nous avons montré que lorsque les embryons sont traités avec de fortes doses de MMS (100 mM), la protéine eIF4G est dégradée précocement alors qu'aucune activité caspase n'est détectée. Le rapporteur codant pour la protéine 4E-BP devrait, par exemple, être trouvé au niveau du polysome, car il est exprimé malgré l'inhibition globale de la traduction. La purification des fractions polysomales peut également être réalisée dans des embryons traités avec du MMS utilisé à des concentrations de 10 ou 100 mM, afin de comparer les profils obtenus lorsque la protéine eIF4G est modifiée, dégradée ou clivée.
Puisque la protéine eIF4G modifiée est capable de s'associer à eIF4E même si la synthèse protéique globale est inhibée (Article 4), nous émettons l'hypothèse que cette modification permet la traduction de messagers spécifiques. Enfin, lorsque la protéine eIF4G est dégradée ou clivée après le traitement des embryons avec du MMS, des messagers spécifiques de la mort cellulaire peuvent avoir été recrutés dans les polysomes.
6 Conclusions générales
ANNEXES
The interaction domains of eEF1Bγ with eEF1Bα and eEF1Bδ were located in the N-terminal region (domain I) of the protein. Schematic structure of the eEF1Bγ protein component of elongation factor 1B illustrating the structural and functional domains (boxed), the phosphorylation sites detailed in single-letter amino acids and their position with arrows in the sequence (red) according to the different species, and the localization of the binding domain (blue arrow) to the associated partners. Furthermore, purification of the trypanothione (a derivative of glutathione) transferase activity from Leishmania led to a parasite eEF1B complex, with the S-transferase activity associated with the eEF1Bγ subunit [73,74].
Schematic structure of the eEF1Bα protein component of elongation factor 1B illustrating the structural and functional domains (box), the phosphorylation site position arrowed in red, and the localization of the binding domain (blue arrow) to the associated partners. Schematic structure of the eEF1Bα(top) and eEF1Bβ(bottom) protein components of rice elongation factor 1B illustrating the structural and functional domains (encased), the phosphorylation sites position in red arrow, and the localization of the binding domain (blue arrow) to the associated partners .