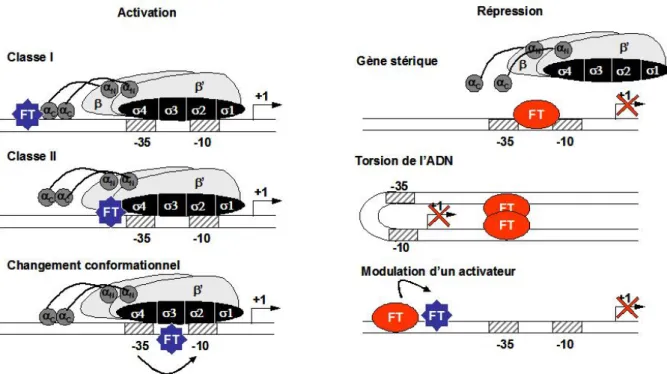
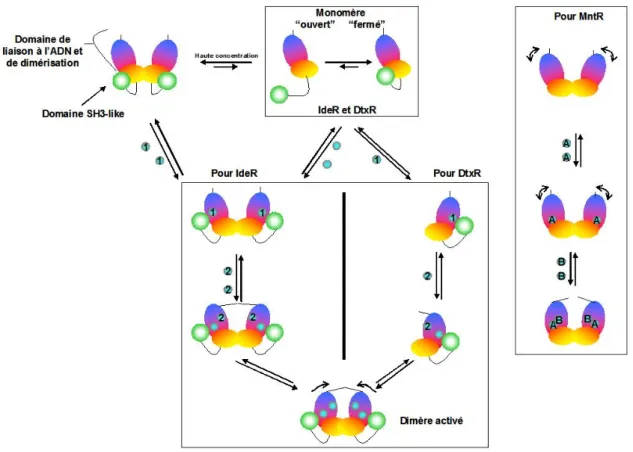
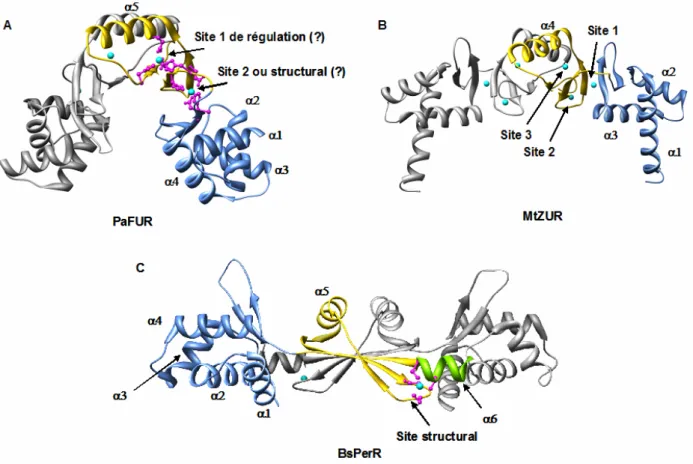
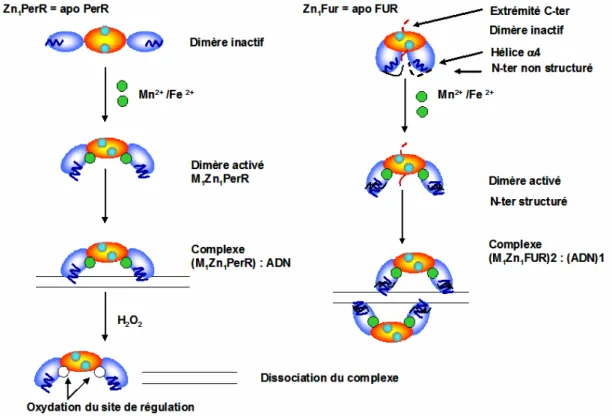
Le domaine C-ter de la sous-unité α (αC) interagit ici avec une région en amont du promoteur (élément UP) plus précisément en amont du motif -35 (séquence riche en A/T). Un mécanisme simplifié de régulation de l'homéostasie du nickel est présenté à la figure I.34. La structure secondaire de la chaîne B de HpNikR a été élucidée sur la base de la structure RX (Dian et al., 2006).
Cela a été confirmé par résolution de la structure par diffraction des rayons X (Dian et al., 2006). Régulation de la synthèse protéique par l'uréase et les protéines accessoires Cette synthèse est régulée à deux niveaux (Pflock et al., 2006b).
Présentation du travail
Par ailleurs, des travaux publiés en 2005 par Delany et al., ont mis en évidence une potentielle régulation croisée entre les deux principaux métallorégulateurs de H.pylori NikR et FUR, en fonction de la disponibilité des ions métalliques (Delany et al., 2005). Afin d'étudier plus spécifiquement cette régulation croisée, des travaux préliminaires de caractérisation de la FUR de H.pylori ont été lancés et seront présentés dans le chapitre V. Enfin, une conclusion générale concernant l’ensemble des résultats sera développée au chapitre VI.
Characterization of the Helicobacter pylori NikR-P(ureA) DNA interaction: metal ion requirements and sequence specificity. NikR mediates nickel-responsive transcriptional repression of the Helicobacter pylori outer membrane proteins FecA3 (HP1400) and FrpB4 (HP1512). Binding of the fur (ferri uptake regulator) repressor of Escherichia coli to arrays of the GATAAT sequence.
Nickel binding and immunological properties of the C-terminal domain of the Helicobacter pylori GroES homologue (HspA). Characterization of metal receptor sites in Escherichia coli Zur, an ultrasensitive zinc(II) metalloregulatory protein. CueR (YbbI) of Escherichia coli is a regulator of the MerR family that controls the expression of the copper exporter CopA.
Characterization of the DNA binding site in the regulatory protein for iron uptake from Escherichia coli by UV cross-linking and mass spectrometry. Crystal structure of the Staphylococcus aureus pI258 CadC Cd(II)/Pb(II)/Zn(II)-responsive repressor.
Les fondements de l’étude d’EcNikR
En l'absence d'un tel composé, la détermination de l'affinité et l'étude de la spécificité ionique ne sont pas possibles. L'affinité du site dit de haute affinité pour le nickel a été décrite comme étant de l'ordre du picomolaire. Dépendance du « coefficient d'extinction apparent » du NikR lié au Ni(II) en fonction de la concentration de NikR Q2E.
Les données (lignes noires), corrigées pour l'absorbance de fond, ont été ajustées globalement (lignes rouges) à un mécanisme de liaison en deux étapes. B) Constantes de vitesse exponentielles pour la phase lente (λ1) en fonction de la concentration de NikR. Figue. 2A, C et B, D) conduira à une surestimation de l'affinité de NikR pour Ni(II) dans un test compétitif. Ce phénomène débute à partir de 0,8 équivalent de nickel ajouté, et est maximal au-dessus de 1,5 équivalent de nickel, comme le montre la figure II.2, qui montre le suivi de l'agrégation des protéines.
Ce comportement suggère que le nickel responsable de l'agrégation est lié aux sites exposés au solvant. L'action de l'EDTA sur le nickel issu du site dit de haute affinité est beaucoup plus lente (t1/2 : 5h), ce qui est conforme à la littérature (Chivers & Sauer, 2002), mais peut être accélérée en présence de cyanure de potassium. .

La deuxième technique consistait à mesurer la quantité de nickel lié à la protéine par FBA. Cette différence de sensibilité peut être corrélée à la présence de sites supplémentaires décrits dans la structure de HpNikR (site externe) et absents dans EcNikR et à la plus faible richesse en histidines (28 par protéine native, dont 20 sont impliquées dans la liaison du Ni(II) . ) du site de haute affinité et du site externe). Nos résultats nous ont amenés à proposer qu'un cluster d'histidine exposé au solvant (H77, H78, H79) présent à l'embouchure du site de haute affinité dans EcNikR, mais absent dans HpNikR, est un site potentiel impliqué dans l'agrégation de la protéine.
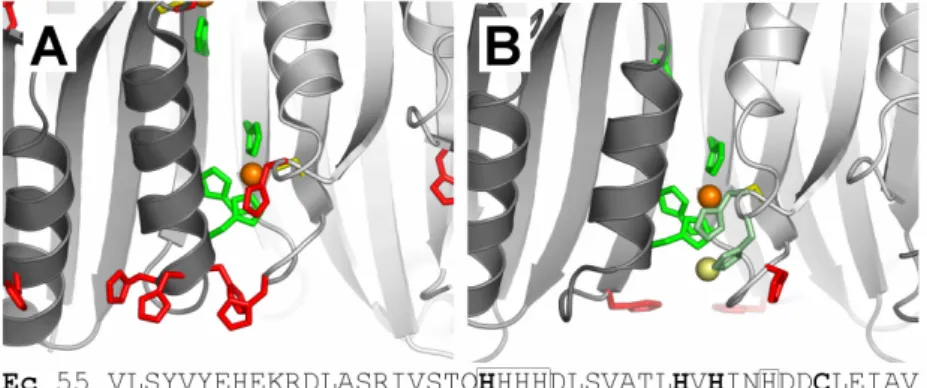
Les histidines et les cystéines impliquées dans la liaison du Ni(II) au site HA sont respectivement colorées en vert et en jaune. Les histidines impliquées dans la liaison au site externe Ni(II) sont vert pâle. Les histidines non impliquées dans la liaison du Ni(II) sont colorées en rouge.
Une comparaison de séquence entre Ec et HpNikR est présentée, les résidus impliqués dans la liaison du Ni (II) sont en gras. Afin de vérifier notre hypothèse, une étude des propriétés de métallation d'un triple mutant de EcNikR ne possédant plus ce groupe a été entreprise et sera présentée en II.5 p-153.
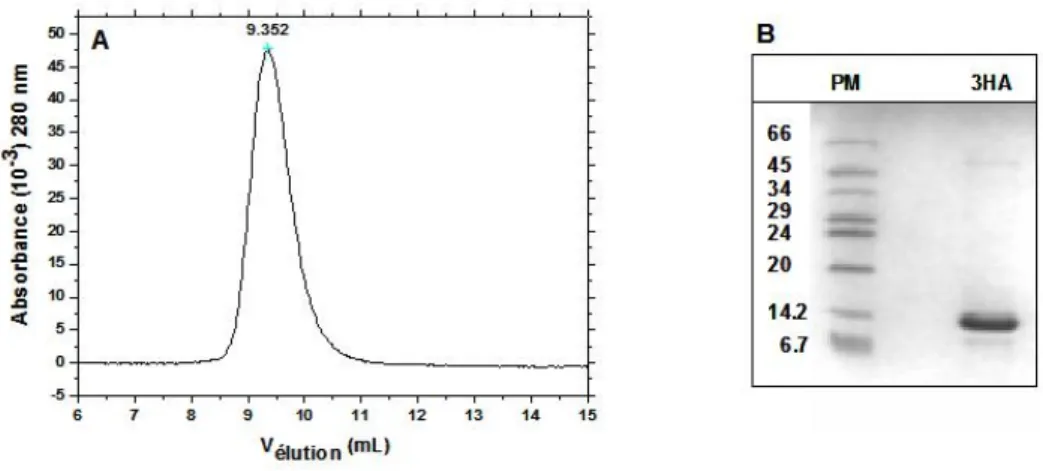
Le mutant EcNikR 3HA
Afin de garantir la masse exacte de la protéine dans des conditions dénaturantes, un échantillon a été analysé par spectrométrie de masse (ESI-TOF). En effet, selon les données RX, la présence de nickel favorise la structuration de l'hélice α 3 et provoque le mouvement de la boucle située entre les brins β3 et β4 (Schreiter et al., 2003). Le spectre CD proche UV de EcNikR 3HA apo présente une large bande centrée à 278 nm qui reflète la structure tertiaire de la protéine.
Ces bandes seraient équivalentes aux bandes observées dans l'UV-visible lors de la métallation du site de haute affinité (Chivers & Sauer, 2000). La suppression du cluster histidine pourrait également modifier la structure de la protéine au « niveau tertiaire ». Le mutant EcNikR 3HA a été construit à l'origine pour tester l'hypothèse selon laquelle le groupe d'histidine H77 à 79 était impliqué dans l'agrégation des protéines.
Après ajout de nickel à la protéine apo EcNikR 3HA, des bandes apparaissent à 302 et 460 nm caractéristiques de la métallation du site dit de haute affinité avec Ni(II) (Figure II.12A et B). Ce résultat suggère que la suppression du cluster histidine ne modifie pas la sensibilité de la protéine à l'agrégation Ni-dépendante.
La liaison à l’ADN de EcNikR
Les conditions utilisées rassemblaient 50 nM d'ADN spécifique (100 pb) avec 35 µM de Ni(II) et une série de protéines allant de 25 nM à 500 nM†. La liaison maximale à l'ADN a été observée pour des rapports protéine:ADN égaux ou supérieurs à 4. Une telle hypothèse expliquerait l'observation de complexes à 190 et 224 kDa après saturation de l'ADN concurrent.
La liaison de l'ADN EcNikR WT et Q2E à la séquence pnikA-Fl (utilisée dans le gel retard ci-dessus) a été mesurée par anisotropie de fluorescence, dont le principe est brièvement expliqué dans Matériels et méthodes. La liaison totale de EcNikR WT et Q2E à pnikA-Fl a été surveillée pendant les titrages au nickel en l'absence d'ADN concurrent (Figure II.15). Les liaisons non spécifiques et spécifiques des protéines à l'ADN sont simultanées et sont observées dans des conditions d'agrégation.
Les conditions expérimentales imposées supposent que le facteur limitant pour la liaison à l’ADN n’est pas la protéine (excès de 62,5 fois par rapport à la concentration en ADN), mais la concentration en nickel. La liaison à l'ADN devrait également être maximale compte tenu du Kd moyen de liaison à l'ADN de 20 nM (Bloom & Zamble, 2004) en présence de 1 ainsi que de 1,5 équivalents de Ni (II).

Discussion et Conclusion
Le fait qu'EcNikR présente une constante de dissociation pour Ni(II) de l'ordre d'une centaine de nanomolaires semble plus cohérent avec les aspects physiologiques. Selon ce modèle, la liaison du Ni(II) au site de haute affinité d'EcNikR a permis à la protéine de se lier à l'ADN avec une affinité modérée. La métallation des sites annexines présentant une affinité d'ordre micromolaire pour le métal a permis une liaison de haute affinité de la protéine à l'ADN.
En considérant seulement que KdNi1 n'est pas de 7 pM, mais de l'ordre de 100 nM, il est très probable qu'une partie de la protéine utilisée dans les expériences de liaison à l'ADN n'était pas inactive dans des conditions stoechiométriques. Nous augmenterions l'affinité d'EcNikR pour l'ADN en augmentant la charge positive de la protéine (Schreiter et al., 2006). Nous pensons que les sites Ni(II) de faible affinité sont directement responsables de l’agrégation des protéines.
Il peut empêcher l’agrégation des protéines in vivo en liant l’excès de métal. Le Kd de EcNikR-Ni en ADN sera réduit en présence d'un large excès de Ni(II) du fait de l'activation totale de la protéine et/ou du fait de l'effet attractif imposé par le métal (caractère stabilisant du complexe) .
L’objectif de l’étude
Purification d’HpNikR WT
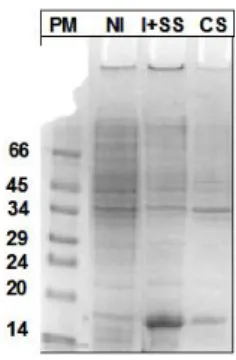
Pour limiter la protéolyse des protéines, un mélange d'inhibiteurs de protéase (fluorure de 4-(2-aminoéthyl)benzènesulfonyle (AEBSF), pepstatine A, E-64, bestatine et EDTA) (Sigma) a été ajouté à la suspension bactérienne avant la rupture cellulaire par sonication. . Le choix de la première étape de purification a été déterminé par le point isoélectrique théorique de HpNikR, pI = 5,2 (Swiss prot). La protéine ainsi purifiée est ensuite concentrée avant de passer à la deuxième étape de purification.
La figure III.3 présente le profil d'élution obtenu lors de l'étape d'exclusion moléculaire d'un extrait concentré de la protéine préalablement purifiée sur Ressource Q. Afin de garantir la masse de la protéine, un échantillon a été analysé par spectrométrie de masse. La teneur en métaux de HpNikR après purification est déterminée par un dosage colorimétrique au PAR.
Ce protocole de purification est efficace pour obtenir une protéine pure sans métal (trace métallique <5%), par rapport à un protocole de purification qui n'inclut aucun traitement "lourd" EDTA de la protéine et utilise dans un premier temps une colonne d'affinité chargée en Ni (II ). 50 mg de protéines pures sont obtenus par litre de culture après toutes les étapes de purification.
Liaison des métaux
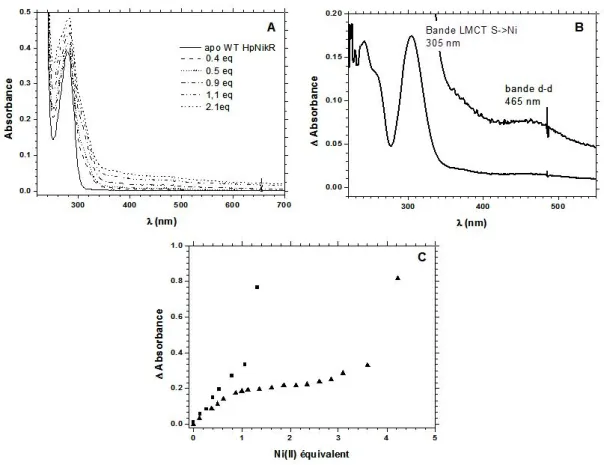
De plus, la détermination de la constante de dissociation du site de haute affinité pour Ni(II) a été réalisée à nouveau avec une technique indirecte : compétition de liaison de Ni(II) en présence d'EGTA. Cette bande est similaire à celle observée lors de la métallation du site de haute affinité d'EcNikR par Cu(II) (Wang et al., 2004). Après cette métallation, l'état de saturation est observé comme lors de la métallation avec Ni(II) (Figure III.5C).
L'intensité de la bande à 309 nm indique que le site de métallation implique une cystéine dans la coordination de l'ion métallique. Ces bandes à 309 et 370 nm sont similaires à celles observées lors de la métallation du site de haute affinité d'EcNikR avec Co(II) (Wang et al., 2004). Ces données confirment le fait que la trempe observée dépend de la métallation du site de haute affinité.
Ces coefficients pourraient démontrer une coopérativité positive impliquée lors de la métallation des sites de haute affinité de la protéine. Un tel comportement a été observé lorsque HpNikR a été mis en contact avec 50 µM d'ANS (Figure III.19A).
Caractéristiques du mutant H99A-H101A-C107S : M1
Une étude préliminaire, réalisée par nos collaborateurs, a montré qu'une bactérie exprimant un tel mutant devenait sensible au stress du métal Ni(II) (Dian et al., 2006).