L'intégration et la dégradation de virus (ou phages) au sein des génomes bactériens (appelés alors prophages) génèrent un flux génétique favorisant la diversification génétique de leurs hôtes. Ces résultats ont mis en évidence la forte conservation des positions d'intégration des prophages, ainsi que les différentes adaptations des phages tempérés à l'organisation chromosomique de leurs hôtes. La deuxième partie du travail portait sur l'origine de la diversité génétique des phages tempérés.
Diversité des bactéries et de leurs génomes
A titre d'exemple, citons le cas de la bactérie parasitaire obligatoire Mycobacterium leprae, dont la présence d'un grand nombre de pseudogènes témoigne d'une réduction récente de son génome (Gomez-Valero, et al. 2007). Il est intéressant de souligner qu’il existe de grandes différences dans la taille du génome au sein d’une même espèce bactérienne (Touchon, et al. 2009). Les genres Escherichia et Salmonella sont relativement proches et auraient divergé il y a environ 100 millions d'années (bien que ces estimations soient imprécises) (Battistuzzi et al. 2004).
Le chromosome bactérien: une structure organisée
- L'organisation associée à la réplication
- La ségrégation
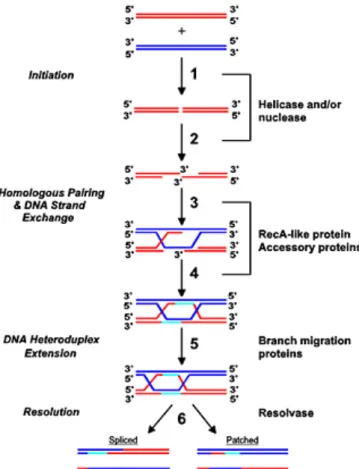
- La recombinaison homologue
- La structure du chromosome
- Implications évolutives
Il est largement co-orienté avec la fourche de réplication (sur le brin précoce) et est présent environ tous les 12 kb (Figure 2) (Touzain et al. 2011). Il a également été démontré que ces différentes régions chromosomiques occupent des positions spécifiques dans la cellule (Meile et al. 2011). Ces différentes contraintes affectent donc directement la plasticité de la structure des chromosomes (Esnault et al. 2007).

Plasticité et évolution du génome bactérien
- La sélection
- La dérive génétique
- La recombinaison
- Les pertes de gènes
- Les gains de gènes
- Les éléments mobiles
- Points chauds de transfert
Enfin, ce processus permet d'incorporer des séquences d'ADN exogènes dans un réplicon (Ochman, et al. 2005). En général, ces éléments sont appelés îlots génomiques et leur taille varie généralement de 10 kb à 200 kb (Juhas et al. 2009). Différents gènes liés à leur motilité peuvent également aider à identifier certains types d'îlots (Langille, et al. 2010).

Diversité des phages
De la découverte de Twort et d'Hérelle à la biologie synthétique
Classification de Baltimore et classification de l'ICTV
Phages virulents et phages tempérés
Au cours de la lysogénie, le prophage n'exprime presque aucun gène et peut se répliquer passivement à mesure que le chromosome hôte se réplique. Le prophète peut ainsi bénéficier de la propagation de son hôte sur plusieurs générations. Après induction ou de manière stochastique, un prophage peut quitter le cycle lysogène et commencer un cycle lytique (Ball et Johnson 1991 ; Gottesman et Yarmolinsky 1968).
Description d'un phage tempéré modèle: Lambda
- Généralités
- Un génome très organisé
- Régulation et cycle
- Intégration et excision
- Stabilité de la lysogénie
"Early Left" permettra au génome Lambda de s'intégrer dans le génome de l'hôte si le site correspondant à l'intégrase est disponible, conduisant à l'établissement de la lysogénie. Dans les deux cas, l'excision ou la circulation de l'ADN Lambda conduit à la formation d'un épisome circulaire. dans la cellule (Guarneros et Echols 1970). L'origine de la réplication est située dans la séquence du gène O elle-même.

Evolution des lambdoïdes et le mosaïcisme phagique
Définition des lambdoïdes et conflits taxonomiques
Enfin, il a été récemment démontré que le facteur de terminaison de l'hôte Rho pourrait également jouer un rôle important dans le maintien de la lysogénie (Menouni, et al. 2013). Ce phénomène peut être attribué aux variations stochastiques du niveau d'expression de CI, ainsi qu'à la stabilité des ARNm et des protéines produites (Arkin et al. 1998). Enfin, le niveau de condensation de l'ADN chromosomique peut affecter le taux d'induction Lambda (Norregaard et al. 2014).
Organisation génomique modulaire conservée et morons
La fonction de ces gènes est souvent inconnue et n'est généralement pas essentielle (voir (Court et Oppenheim 1983)). La taille et le contenu de ces régions varient considérablement d'un lambdoïde à l'autre (Juhala, et al. 2000). J'ai mentionné plus tôt que la majorité des gènes lambdoïdes sont regroupés en trois opérons majeurs et que les différents gènes sont regroupés en modules fonctionnels au sein de ces opérons.
Mécanismes à l'origine du mosaïcisme
De plus, un nouveau mécanisme de recombinaison par l'enzyme Redβ a été récemment proposé et permettrait des échanges d'ADN importants (Maresca, et al. 2010). Si la conservation de telles séquences aux limites des modules a bien été observée dans certains génomes (Clark, et al. 2001), cela ne semble pas expliquer la majorité des cas (Hendrix et Casjens 2006). Pour cette raison, il a été suggéré que la recombinaison illégitime joue un rôle dominant dans le mosaïcisme des phages (Hendrix et al. 1999).
Ecologie et course aux armements
L'organisme le plus abondant et son impact
La co-évolution et la course aux armements
Un autre système peut également dégrader l’ADN des phages : les CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) associés aux gènes cas (protéines associées aux CRISPR) (Deveau, et al. 2010). Ce dernier point est d’autant plus important que les phages possèdent souvent des gènes qui leur permettent de contourner les défenses bactériennes (Samson, et al. 2013). Il a été récemment démontré que les phages peuvent disposer de leur propre système CRISPR-Cas pour échapper au système de défense de l'hôte (Seed et al. 2013).
L'ambigüité des phages tempérés: faux ennemis ou faux amis?
Par exemple, les phages peuvent parfois coder l’antitoxine et ainsi empêcher l’avortement prématuré du cycle lytique (Otsuka et Yonesaki 2012). Il est intéressant de noter que les phages peuvent parfois utiliser les mêmes systèmes que les bactéries pour échapper à leurs défenses. Les phages et leurs hôtes sont donc en constante coévolution, car ils entretiennent une relation hôte-parasite.
Impact des prophages à court terme: un génotype bactérien étendu
- La transduction
- Protection contre la sur-infection
- Gènes augmentant la croissance de l'hôte
- Gènes liés à la pathogenèse
- Utilisation des prophages comme armes
- Effets délétères liés à l'intégration
Ainsi, jusqu'à 2 % des phages P22 peuvent remplir leur capside d'ADN bactérien (Ebel-Tsipis, et al. 1972a). Une étude récente suggère que les phages sont également des vecteurs de gènes de résistance aux antibiotiques (Modi et al. 2013). La réponse SOS est déclenchée lorsque la bactérie a subi des dommages génomiques importants (Bjedov et al. 2003).
Impact des prophages à long terme: source de nouveaux gènes et domestication
Les prophages cryptiques: reliques ou entités fonctionnelles?
Cependant, certains génomes bactériens peuvent contenir plusieurs prophages (18 dans E. coli O157:H7 Sakaï) (Asadulghani, et al. 2009). Si certains éléments correspondent à des prophètes a priori complets et fonctionnels, de nombreux prophètes semblent en voie de dégradation (Asadulghani, et al. 2009) et sont généralement qualifiés de prophètes « cryptiques ». Plusieurs études ont également étudié de manière exhaustive la fonctionnalité des prophages présents dans certains génomes bactériens (Asadulghani et al. 2009 ; Matos et al. 2013).
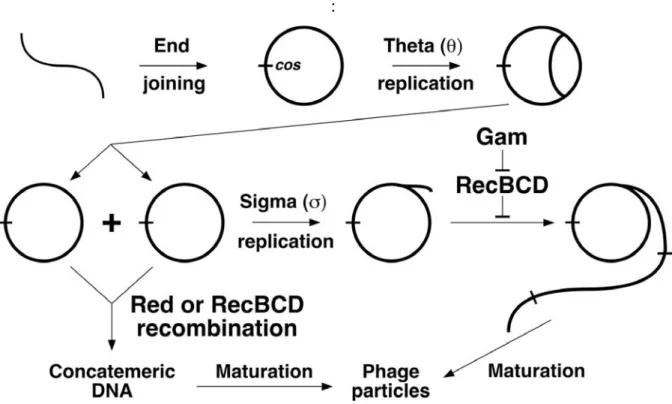
Le renouvellement des prophages: une source d'ADN pour innover
En effet, il a été démontré que les génomes de phages hautement dégradés peuvent utiliser des particules de phages liées pour se mobiliser (Enea et al. 1977). La vue générale de sa structure, du mécanisme de sécrétion ainsi que de la séquence de certains de ses composants suggèrent que cet élément provient de l'atténuation des gènes de queue du phage Myoviridae (Fig 13) (Bonemann, et al. 2010). Depuis, il a été observé chez diverses bactéries, mais sa fonction reste inconnue (Bonemann, et al. 2010).

Ambigüités entre prophages défectifs et systèmes domestiqués
Nous avons vu que les ATG permettent le transfert horizontal de gènes bactériens (Lang, et al. 2012). En fait, il a été observé que les phages caudés contiennent souvent (~ 25 %) des protéines structurelles avec des domaines d'immunoglobuline (de type Ig) (Fraser, et al. 2006). Le mosaïcisme génomique des phages apparaît comme un obstacle important au principe même de ces méthodes (Lawrence, et al. 2002).

These results show how natural selection for seamless integration of prophages into the chromosome shapes the evolution of the bacterium and the phage. Third, the phage endures selection for DNA motifs that relate exclusively to the biology of the prophage in the bacterial chromosome. The integration of phages can affect a number of the organizational features of the bacterial chromosome (Reyes-Lamothe et al. 2008; Rocha 2008).
Thexaxis indicates the position of the foci of phage integration in the genomes of Escherichia coli (top) and Salmonella enterica (bottom). Putative targets” of integration are also indicated in the middle part of the figure (details in the keys). Most genes are silent in the prophage state, with the notable exception of the repressor of the lytic cycle.
The MatP protein interacts with the 13 bpmatSsites to organize the terminus of replication of the chromosome in the Ter macrodomain (Mercier et al. 2008). The motif is therefore concentrated in the Ter macrodomain and absent from the rest of the chromosome. Therefore, integration of a mat-missing prophet probably has no disruptive effect in the formation of the macrodomain.
ThematSmotif defines the Ter macrodomain and is absent from the rest of the chromosome (Mercier et al. 2008).
Most of the identified recombinases (68%) are from the Redb family, the ones encoded by Lambda (Figure S1). Association between the presence of phage recombination functions and the abundance of Chi sites. (A) Number of lambdoid phages encoding RecBCD inhibitors (Inh+/Inh2) and recombinases (Rec+/Rec2). B) Distribution of the number of observed/expected (O/E) ratios of Chi sites among lambdoid phages. The central vertical lines indicate the data range, with a maximum distance of 1.5 interquartile ranges (i.e. the distance between the first and third quartile values).
The overrepresentation of Chi sites in Rec+Inh2phages is not significantly different from that of the nuclear genome of E . We tested whether imprecise delineation of the latter might have affected the number of Chi motifs found in our data set. We constructed the pangenomes (i.e., the set of all different gene families) of Rec+and Rec2lambdoids (see Materials and Methods).
Pangenomes of the lambdoid phages encoding recombination functions (Rec+) are larger than those lacking them (Rec2). The pan-genome size (y-axis) of each type of phage genome was calculated for increasing numbers of genomes (x-axis). The absence of Chi in phage Lambda was instrumental in discovering the function of this motif [46]. Hence, the study of the roles of Chi sites in phages may illuminate further functional associations between recombination, phage replication and packaging.
Among the 131 non-lambdoid genomes, 80 phage genomes from the Caudovirale order (69 virulent and 11 temperate) were downloaded from RefSeq (when classified in a genus defined by the ICTV).

In contrast, small deletions (and the experimental deletion spectrum) lead to a much weaker dependence of deletion probability with position in the prophage. Third, orthologous prophages replicate like any part of the bacterial chromosome and should therefore exhibit similar neutral substitution rates. Histogram of the ratio of nonsynonymous to synonymous substitutions (dN/dS) among orthologous prophages.
It is possible that many of the orthologous prophages correspond to R/F-type bacteriocins or phage killer particles. Steinberg KM & Levin BR (2007) Grazing protozoa and the evolution of the Escherichia coli O157:H7 Shiga toxin-encoding prophage. Replication stalls during a single round of replication of the Escherichia coli chromosome in the absence of DnaC activity.
At the end of the procedure, we identified 100 families of orthologous prophages, including 372 different prophages. This introduces imprecision in the estimation of the ratios of non-synonymous (dN) versus synonymous (dS) substitution rates. For each family of orthologs, the size of the pangenome was compared to the largest prophage of the family.
Histogram of the ratios of non-synonymous to synonymous substitutions (dN/dS) in orthologous prophages defined with stricter parameters. Average gene conservation (top) and dN/dS values (bottom) of the different functional categories of orthologous prophages.