Des moteurs de jeux ` a la physique des chromosomes
Pascal Carrivain
Laboratoire de Physique Th´eorique de la Mati`ere Condens´ee, Universit´e Pierre et Marie Curie, 4, Place Jussieu, 75252 Paris Cedex 05, France
JURY:
Aur´elien Bancaud (LAAS Toulouse) David Bensimon (ENS Paris)
Ralf Everaers (ENS Lyon) Jean-Fran¸cois Joanny (Institut Curie) S´ebastien Neukirch (Institut Jean Le Rond d’Alembert)
Directeur de th`ese: Jean-Marc Victor (LPTMC Paris)
19 mars 2013
1/42
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
3/42
Pr´ esentation du laboratoire et de l’´ equipe M3V
Mod´elisationMulti-´echelle de laMati`ereVivante
1. Directeur du laboratoire de Physique Th´eorique de la Mati`ere Condens´ee : Pascal Viot 2. ´Equipe M3V :
I Chef d’´equipe : Jean-Marc Victor (Directeur de recherche au CNRS)
I Annick Lesne (Directeur de recherche au CNRS)
I Maria Barbi (Maitre de conf´erences `a l’UPMC)
I Julien Mozziconacci (Maitre de conf´erences `a l’UPMC)
I Laurence Signon (Charg´e de recherche au CNRS et stagiaire au LPTMC)
I Axel Cournac (Post-doctorant) 3. Anciens membres :
I Fabien Paillusson
I Hua Wong
I Eli Ben-Ha¨ım
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
5/42
ADN nu sous pinces magn´ etiques : contexte biologique
(a) (b) Wong & al, BIOESSAYS. 2009 Oct 29 ;31(12) :1357-1366
ADN nu sous pinces magn´ etiques : montage exp´ erimental
Figure:Vue sch´ematique d’une exp´erience de pinces magn´etiques.
7/42
ADN nu sous pinces magn´ etiques : premi` ere exp´ erience
Propri´et´e de courbure de l’ADN : persistance en courburep= 50nm Cette exp´erience ne mesure pas la capacit´e de l’ADN `a se tordre : persistance en twistt
M´ ethode du Pivot et du Krankshaft pour la simulation d’ADN
(a) Vologodskii, Macromolecules1994, 27, 5623-5625
●●
●●●
●
●
●
●
●
●
●
●
●
●●
●●
●●
●
●●
●
●●●
●
●
●
i N
●●
●●●
●
●
●
●
●
●
●
●
●
●●
●●
●
●
●
●
●
●●● ●
●●
● Pivot Rotation Ru(θ)(i→N)
●●
●●●
●
●
●
●
●
●
●
●
●
●●
●●
●
●
●
●
●
●●● ●
●●
i j
●●
●●●
●
●
●
●●
●
●●
●
●●
●
●
●
●
●
●
●
●●● ●
●● Krankshaft Rotation Rv(φ)(i→j)
(b) Vue sch´ematique des m´ethodes du Pivot (de rouge
`
a bleu) et du Krankshaft (de vert `a magenta) + algo- rithme de Metropolis
(c) Vue sch´ematique d’un polym`ere greff´e dans un tube
9/42
Mod` ele du “Bead spring” (1)
Mod` ele du “Bead spring” (2)
(a) (b)
11/42
Mod` ele du “Bead spring” (3)
(a) (b)
SHAKE
t
δ = d
− f
δf
δt + ∆ t
− f
δf
δt + ∆ t
Figure:Vue sch´ematique d’un algorithme type SHAKE.
Edberg &al, J. Chem. Phys.84(12), 15 June 1986
Forester &al, Journal of Computational Chemistry, Vol. 19, No. 1, 102-111 (1998) Hess &al, Journal of Computational Chemistry, Vol. 18, No. 12, 1463-1472 (1997) Ryckaert &al, Journal of Computational. Physics23, 321-341 (1977)
13/42
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
Examples
(a) Starcraft 2 (b) Crysis 3
15/42
Joints m´ ecaniques
ODE is Copyright 2001-2004 Russell L. Smith. All rights reserved.
(a) Vue sch´ematique d’un joint “Ball-in- Socket”
(b) Vue sch´ematique d’un joint “Hinge”
(c) Vue sch´ematique d’un joint de Cardan (d) Vue sch´ematique d’un joint de contact
Joints m´ ecaniques : formulation math´ ematique
I ´Equation de la dynamique d’un syst`eme de corps rigides
S = {r1,q1. . .rN,qN}T V = {v1,ω1. . .vN,ωN}T Fe = {f1,Γ1. . .fN,ΓN}T M = {m1,I1. . .mN,IN}
L˙ = Fe avec L=MV
I Contrainte holonomique :δ(S,t) =0(joints m´ecaniques)
I Contrainte cin´ematique : ˙δ=J V= 0
I Torseur dynamique des contraintesFc=JTλ, avecλ`a calculer !
I Le torseur des contraintes ne travaille pasFTcV= 0 !
I
h
J M−1JT+k∆tcfmi
λ=−kerp
∆t2δ−Jh
V
∆t +M−1
Fe−MV˙ i
I kerpetkcfmservent `a contrˆoler la stabilit´e du syst`eme
17/42
Les diff´ erentes ´ etapes de simulation
1. N cylindres de longueurl = 3.34nmet de rayon effectifreff=rc+lD (rc repr´esente le rayon cristallographique de l’ADN etlD la longueur de Debye de la solution en sel) connect´es par des joints “Ball-in-Socket”
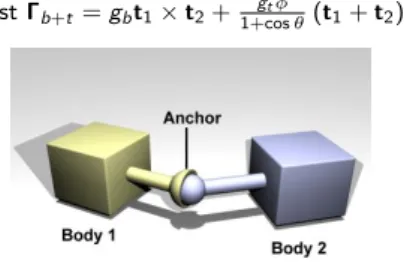
2. Couples de courbure et de twistΓb+t=gbt1×t2+1+cosgtφθ(t1+t2)
Figure:Vue sch´ematique d’un joint “Ball-in-Socket”
3. Dynamique de Langevin-Euler locale de corps rigidesG=−ΣL+ΞW˙ 4. ODE calcule les contraintesλ
5. Le syst`eme ´evolue du tempstau tempst+ ∆tavec une int´egration semi-implicite
Dynamique de Langevin-Euler avec un thermostat global (1)
Berendsen &al, J. Chem. Phys. 81 (8), 15 October 1984 Bussi &al, The Journal of Chemical Physics126, 014101 (2007) Bussi &al, Computer Physics Communications 179 (2008) 2629
I Thermostat global versus thermostat local
I Hamiltonien du syst`eme :H(S,V) =T(v) +R(ω) +U(S)
I Distribution dans l’ensemble canonique :P(S,L)dSdL∝e−βH(S,bsmcV) I Vitesse de thermalisation de l’´energie cin´etique :
dH =
6N
X
i=1
Σ?ii
β − Σ?ii M?iiL?2i
! dt+
s 2Σ?iiL?2i
βMii dWi
= (hEi −2T) Σ?Tdt+ (hEi −2R) Σ?Rdt+ 2 s
Σ?TT+ Σ?RR
β dW
I “Disturbance”
L˙−Fe
T
M−1 L˙−Fe
=GTM−1G
I Dynamique de Langevin-Euler globale :Ge=h Σ?hhEi
E
1−2βhEi1
−1i +q
Σ? βEW˙i
L
I Gen’intervient pas dans le calcul du torseur des contraintesFc
19/42
Dynamique de Langevin-Euler avec un thermostat global (2)
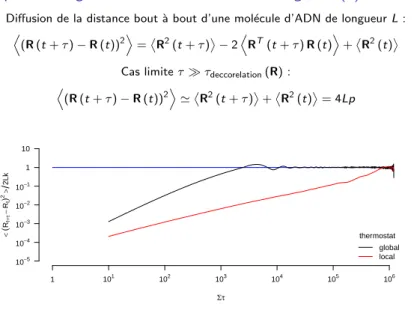
Diffusion de la distance bout `a bout d’une mol´ecule d’ADN de longueurL: D
(R(t+τ)−R(t))2E
=
R2(t+τ)
−2D
RT(t+τ)R(t)E +
R2(t)
(1) Cas limiteττdeccorelation(R) :
D
(R(t+τ)−R(t))2E '
R2(t+τ) +
R2(t)
= 4Lp (2)
Στ
< (Rt+τ−Rt)2 >2Lk
1 101 102 103 104 105 106
10−5 10−4 10−3 10−2 10−1 1 10
thermostat global local
Figure:Diffusion de la distance bout `a bout normalis´ee de la mol´ecule d’ADN (en ordonn´ee) en fonction du temps adimensionn´e de simulation (en abscisse).
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
21/42
Calibration de la longueur de persistance en courbure (1)
Corr´elation tangentielle le long de la mol´ecule d’ADN et validation des moyennes angulaires. Param`etres de la simulation p= 50nm,t= 95nm, etLADN= 1µm.
●
●
●
●
●
●
●
●
●
●
●
●
●
●
●
●
●●
●●●
●●●●
●●●●●●
●●●●●●●●●●●●
●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●●
0 20 40 60 80 100
0.0 0.2 0.4 0.6 0.8 1.0
j−i
correlation
● 300x10bp 200x15bp 150x20bp fit
T
Calibration de la longueur de persistance en courbure (2)
Mol´ecule d’ADN sous force de traction. Param`etres de la simulationp= 50nm,t= 95nm, etLADN= 1µm.
0.0 0.2 0.4 0.6 0.8 1.0
0.1 0.5 1.0 5.0 10.0 50.0 100.0
ε =z L
βfp
●
●
●
●
●
●●●●●
●
●
●
●
●●●●●
●
●
●
●
●●
●
numerical WLC
analytical WLC fit to exp (10 mM salt) LDNA=300×10 bp 10 mM salt analytical FJC
LDNA=10×300 bp
Figure:L’extension relative de la mol´ecule d’ADN (en abscisse) est montr´ee en fonction de la force de traction adi- mensionn´ee (en ordonn´ee). Les r´esultats de simulations sont confront´es aux r´esultats exp´erimentaux de Bustamante 1992, Siggia 1995.
23/42
Vue sch´ ematique de plecton` emes ` a nombre de tours fix´ es obtenus avec ODE
Param`etres de la simulationp= 50nm,t= 95nm,LADN= 1µm, avec une force de tractionf= 0.74pNet un nombre de tour de bille magn´etiquen= 15.
Figure:Vue sch´ematique de plecton`emes `a nombres de tours fix´es(vid´eo).
Calibration de la persistance en twist : simulation ` a nombre de tours fix´ e
Extension relative de la mol´ecule d’ADN `a nombre de tours de bille magn´etique fix´e. Param`etres des simulationsp= 50nm, t= 95nm,reff'2nmetLADN= 1µm.
0.00 0.02 0.04 0.06 0.08 0.10
0.0 0.2 0.4 0.6 0.8 1.0
σ
< z >L
●●
●
●
●
●
●
●
●
●
●
●
●
●
●
●
●
●●●●●●
●
L=1 µm r~=2 nm f=0.35 pN f=0.50 pN f=0.61 pN f=0.74 pN f=0.91 pN f=1.13 pN f=1.42 pN f=1.80 pN Mosconi100mM(2009)
Figure:L’extension relative de la mol´ecule d’ADN (en ordonn´ee) est montr´ee en fonction du nombre de tours de bille magn´etique (en abscisse) pour une diff´erentes forces de traction. Les symboles repr´esentent les r´esultats de simulations et les lignes pleines repr´esentent les r´esultats exp´erimentaux (Mosconi &al, PRL102, 078301 (2009) solution de 100mMde sel).
25/42
Vue sch´ ematique de plecton` emes ` a couple fix´ e obtenus avec ODE
Param`etres de la simulationp= 50nm,t= 95nm,LADN= 1µm, avec une force de tractionf= 0.74pNet un couple sur la bille magn´etique Γn= 15pN·nm.
Figure:Vue sch´ematique de plecton`emes `a couple fix´e(vid´eo).
Calibration de la persitance en twist : simulation ` a couple fix´ e
Nombre de tours de la bille magn´etique en fonction du couple appliqu´e. Param`etres des simulationsp= 50nm,t= 95nm, reff'2nmetLADN= 1µm.
0.00 0.05 0.10 0.15
0 5 10 15 20 25
< σ >
Γ (pN.nm)
●
●
●
●
●
●
●
●
●
●● ● ● ● ●●
●
●
●
●
●
●
●
●
●
●
●
●
●
●
● f=0.74 pN/r=2.00 nm ● f=0.91 pN/r=2.00 nm f=1.13 pN/r=2.00 nm Γ(σ)=tplectonemekBTω0(σ − σ0)
(a) L’overtwist (en abscisse) est montr´e en fonction du couple appliqu´e sur la bille magn´etique. Les lignes en poin- till´ees sont des estimations analytiques des couples de flam- bage (Mosconi &alPRL102, 078301 (2009)). Le couple est d´eduit des exp´eriences `a nombre de tours fix´es (Zhang
&al, PRE77, 031916 (2008)).
0.0 0.2 0.4 0.6 0.8 1.0
F=0.74 pN
t T 0
0.2 0.4 0.6 0.8 1 0 0.2 0.4 0.6 0.8 1 0 0.2 0.4 0.6 0.8
1 z L Γ =25 pNnm σ
Γ =9.7 pNnm
Γ =0 pNnm
0 0.05 0.1 0.15 0.2 0 0.05 0.1 0.15 0.2 0 0.05 0.1 0.15 0.2
(b) L’overtwist (en rouge) et l’extension relative (en bleu) de la mol´ecule d’ADN sont montr´es (en ordonn´ee) en fonc- tion du temps de simulation (en abscisse) et ce pour trois couples Γ = 0,9.7,25pN·nmet une force de traction f = 0.74pN.
27/42
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
Comment compacter l’ADN ?
(a) Configuration d’une mol´ecule d’ADN (b) Configuration d’une mol´ecule d’ADN surenoul´ee
(c) 7 t´etrasomes (d) 4 nucl´eosomes
Figure:Vue sch´ematique des deux diff´erents m´ecanismes de compaction d’une mol´ecule d’ADN de 900 bp.
29/42
Pinces magn´ etiques et fibre de chromatine
:Bancaud &al, Nature Structural & Molecular Biology, Vol. 13 No. 5, (2006), Bancaud &al, Molecular
Mod` ele gros-grain de nucl´ eosome
Analyse des modes normaux (J. Mozziconacci) et structuration m´ecanique (P. Carrivain et H. Wong)
(a) Nucl´eosome crystallogra- phique Luger & al, Current Opinion in Structural Biology 1998, 8 :33-40
(b) T´etrasome (c) Nucl´eosome
Figure:Vue sch´ematique du mod`ele gros-grain de nucl´eosome construit `a partir de la structure crystallographique.
L’ADN nucl´eosomal est enroul´e `a gauche. Les dim`eres H2A-H2B sont repr´esent´es en rouge et jaune tandis que les dim`eres H3-H4 sont repr´esent´es en bleu et vert. Les sph`eres noires repr´esentent les “Four-Helix-Bundle”.
31/42
Nucl´ eosome en traction
(a) Nucl´eosome ferm´e (b) Nucl´eosome un tour de d´egraf´e (c) Nucl´eosome deux tours de d´egraf´es
Figure:Vue sch´emtatique des trois ´etats du nucl´eosome sous force de traction, mod`ele commun`ement admis dans la litt´erature.
I Kruithof &al, Biophysical Journal Volume 96 May 2009 37083715
I Ettig &al, Biophysical Journal Volume 101 October 2011 19992008
I ...
Nucl´ eosome en traction : donn´ ees d’E. Praly
Avec D. Bensimon, V. Croquette, F. Ding, J. Mozziconacci, E. Praly et J-M. Victor
Figure:Figure tir´ee de la th`ese d’´E. Praly (´Etude du m´ecanisme d’action des facteurs de remodelage de la chro- matine, `a l’´echelle de la mol´ecule unique). Nous pouvons distinguer trois ´etats d’extension associ´es `a deux sauts de 12nmpuis 24nm.
33/42
Nucl´ eosome en traction : d´ egraffage sym´ etrique de l’ADN
Avec D. Bensimon, V. Croquette, F. Ding, J. Mozziconacci, E. Praly et J-M. Victor
0.0 0.2 0.4 0.6 0.8 1.0
550 600 650 700
t
z(nm)
550 604 619.8 626.6 630 654.6 700
rien 6.5 6.5 & 5.5 6.5 & 5.5 & 4.5 6.5 & 5.5 & 4.5 & 3.5 & 2.5 & 1.5
Figure:Sch´ema de d´egraffage sym´etrique de l’ADN
Nucl´ eosome en traction : d´ egraffage assym´ etrique de l’octam` ere (1)
Avec D. Bensimon, V. Croquette, F. Ding, J. Mozziconacci, E. Praly et J-M. Victor
(a) (b) (c)
(d) (e) (f)
Figure:Ces images illustrent les diff´erentes conformations accessibles au nucl´eosome pour un d´egraffage assym´etrique de l’octam`ere. La simulation est r´ealis´ee pour une force de traction de 3.2pNet un couple nul Γ = 0.
35/42
Nucl´ eosome en traction : d´ egraffage assym´ etrique de l’octam` ere (2)
Avec D. Bensimon, V. Croquette, F. Ding, J. Mozziconacci, E. Praly et J-M. Victor
0.0 0.2 0.4 0.6 0.8 1.0
550 600 650 700
t
z(nm)
550 604.3 618.4 630.2 653.2 653.9 700
rien 2x6.5
2x6.5 & 1xdocking domain & 1xFHB flexible 2x6.5 & 2xdocking domain & 2xFHB flexibles 2x6.5 & 2xdocking domain & 3FHB flexibles
Figure:Sch´ema de d´egraffage assym´etrique de l’octam`ere. Deux sauts en extension de 12nmet 23nm.
Nucl´ eosome en traction : mod´ elisation dynamique
Avec D. Bensimon, V. Croquette, F. Ding, J. Mozziconacci, E. Praly et J-M. Victor
Mod´elisation des interactions avec des potentiels de Morse
Nucl´eosome sous une force de tractionf = 3.2pNet un couple de Γ = 0pN·nm Calcul de structure pour obtenir les interactions au sein du nucl´eosome
SHLs D (kBT) a nm−1
re (nm)
±6.5 2 3.46 0.20
±5.5 10 0.80 0.87
±4.5 15 0.51 1.35
±3.5 20 0.78 0.89
±2.5 15 0.85 0.81
±1.5 15 0.62 1.12
±0.5 20 0.85 0.81
(a) Param`etres pour les SHLs.
Interaction dim`ere-dim`ere D (kBT) a nm−1
re (nm)
Docking Domain 5 0.69 1.00
Docking Domain 5 0.69 1.00
Four-Helix-Bundle 15 0.39 1.76
Four-Helix-Bundle 20 0.40 1.70
Four-Helix-Bundle 15 0.39 1.76
(b) Param`etres pour les DDs et FHBs.
Figure:Tableau r´ecapitulatif des param`etres des potentiels de Morse utilis´es pour reproduire les SHL, FHB et DD.
37/42
Nucl´ eosome en traction et torsion
Avec D. Bensimon, V. Croquette, F. Ding, J. Mozziconacci, E. Praly et J-M. Victor
R´eversome : nucl´eosome sous une force de tractionf = 3.2pNet un couple de Γ = 25pN·nm
Figure:Vue sch´ematique d’un r´eversome au milieu de plecton`emes : nucl´eosome sous une force de tractionf = 1pN et un couple de Γ = 10pN·nm.
R´ eversome ` a l’int´ erieur d’une fibre de chromatine
Assemblages de nucl´eosomes et t´etrasomes sous une force de traction def= 1pNet un couple Γ = 10pN·nm
(a) R´eversome `a l’int´erieur d’une fibre de chromatine (b) R´eversome `a l’int´erieur d’une “fibre” d’Archaea
39/42
Fibre de chromatine sous force de traction et couple appliqu´ e sur la bille magn´ etique
0.00 0.01 0.02 0.03 0.04 0.05
0 100 200 300 400
σ
zL
● ●●
●
●
●
●
● ●● ●● ●
● f=0.3 pN
N=1N=2 N=5
(a) L’extension de la fibre de chromatine (avec 1,2,5 nucl´eosomes) est montr´ee en fonction du nombre de tours pour une force de tractionf= 0.3pN.
0.00 0.01 0.02 0.03 0.04 0.05
0 2 4 6 8 10 12
σ
Γ (pN.nm)
●
●
●
●
●
●
●
●
●
●
●
●
●
● f=0.3 pN
N=1 N=2N=5
(b) Le couple appliqu´e sur la bille magn´etique est montr´e en fonction du nombre de tours de la bille magn´etique. Le couple de flambage est de l’ordre de Γp= 6pN·nm
Figure:
Sommaire
Pr´esentation du laboratoire et de l’´equipe M3V Contexte
Introduction
M´ethode du Pivot et du Krankshaft pour la simulation d’ADN Mod`ele du “Bead spring”
SHAKE
Open Dynamics Engine Examples
Joints m´ecaniques
Les diff´erentes ´etapes de simulation
Manipulation de mol´ecule uniqueIn silicod’ADN nu Calibration de la longueur de persistance en courbure Calibration de la longueur de persitance en twist SimulationIn silicode fibre de chromatine
Comment compacter l’ADN ?
Pinces magn´etiques et fibre de chromatine Nucleosome
Conclusion et perspectives
41/42
Conclusion et perspectives
1. Nouvel outil pour la simulation de macromol´ecules : moteurs de jeux
2. Images des structures complexes telles que les fibres de chromatine sous contraintes 3. Finir le calcul de structure du nucl´eosome
4. Mesures de la diffusion des plecton`emes
5. Simulation du “crosslink” des exp´eriences de HiC (Avec A. Cournac, J. Mozziconacci et J-M.
Victor)
6. Simulation de fibre de chromatine inhomog`ene : les cinq couleurs de la chromatine chez la drosophile (Avec G. Cavalli et C. Vaillant)
7. Simulation des complexes de prot´eines “polycomb” et du noyau de drosophile (Avec G. Cavalli et C. Vaillant)