Etude du mécanisme d'action des facteurs de remodelage de la chromatine par micromanipulation et visualisation de l'ADN. A dix minutes de la fin, ajoutez le foie de l'animal préalablement cuit dans l'eau pendant 5 minutes.
Introduction
1.1 – A gauche, représentation schématique des différents niveaux de compaction de l'ADN à la base de la structure de la chromatine dans la cellule eucaryote. A droite, quelques images de microscopie électronique illustrant les différents niveaux de compaction. Nous reviendrons maintenant plus en détail sur les différents niveaux de compaction de la chromatine après avoir fait quelques rappels sur l’ADN.
Architecture nucléaire
- Structure de l’ADN
- Structure des histones
- Premier niveau de condensation de l’ADN : les nucléosomes
- L’ADN de jonction et l’histone H1 de jonction
- Deuxième niveau de compaction : la fibre de 30 nm
- Niveaux supérieurs de compaction de la chromatine
- Compartimentation structurale du noyau à grande échelle
La protéine de liaison TATA (TBP) se lie à la boîte TATA dans le petit sillon de l'ADN. Les interactions entre l'ADN et les histones introduisent des distorsions qui modifient la structure de l'ADN.
Les modifications covalentes des histones
- Acétylation des histones
- Méthylation des histones
- Phosphorylation des histones
- ADP-ribosilation, Ubiquitination et Sumolation
- Les variants des histones
- La méthylation de l’ADN
La méthylation des arginines dans les histones H3 et H4 est corrélée à un état transcriptionnel actif [77]. La méthylation de l'arginine 3 de l'histone H4 facilite l'acétylation de H4 et augmente l'activation transcriptionnelle via le récepteur nucléaire de l'hormone (78).
Les facteurs de remodelage de la chromatine
2.6 – Tous les complexes de remodelage de la chromatine ont pour sous-unité catalytique une AT-Pase ADN-dépendante appartenant à la famille des protéines Swi2/Snf2. Tous les facteurs de remodelage de la chromatine sont caractérisés par la présence d'une sous-unité ATPase dépendante de l'ADN, qui appartient à la superfamille SNF2 (Sucrose Non Fermenters)[ 125 , 108 ].
La famille SWI/SNF
Le complexe yRSC
RSC est un complexe de remodelage de la chromatine de la famille Swi/Snf, fortement exprimé dans S. Les effets sur l'intégrité de la paroi cellulaire sont cohérents avec d'autres expériences sur le lien entre RSC et la voie de la protéine Kinase C.
Les sous-unitées ATPase Brg1 et Brm
Des études réalisées sur des souris mutantes BRM-/- montrent une augmentation de la prolifération cellulaire [154]. Brg1 affecte directement le cycle cellulaire [164] grâce à son interaction avec la protéine du rétinoblastome (pRB) et à la répression des gènes de la cycline A et du cdc.
La famille ISWI
La protéine ISWI (Drosophile)
Le gène ISWI de Drosophila est essentiel au développement normal et à la viabilité cellulaire (175). L'activité ISWI peut être fortement stimulée par la présence d'une autre sous-unité du complexe, la protéine ACF1 de Drosophila (172).
La famille CHD
A noter que ce complexe a la particularité d'avoir une activité de remodelage de la chromatine combinée à une activité histone désacétylase. Cependant, la protéine Chd1, homologue de Mi-2 chez la drosophile, est localisée dans la chromatine transcriptionnellement active, suggérant un rôle positif de cette protéine dans la transcription (187).
Les enzymes avec le domaine Split ATPase
L'ancrage de la molécule d'ADN à la surface du capillaire est réalisé par l'interaction entre le couple digoxigénine/antidigoxigénine. On peut donc ainsi mesurer l’extension de la molécule d’ADN en temps réel. 3.22 – Analyse de la longueur d'une molécule d'ADN On suit le contour de la molécule (tracé avec le programme open source ImageJ).
La translocation d'ADN entraîne des changements transitoires dans le nombre d'entrelacements de la molécule d'ADN. L'activité de l'ISWI est profondément affectée par l'ampleur de la force appliquée à la molécule d'ADN. La protéine ISWI est capable de s'attacher à la molécule d'ADN en réduisant l'allongement de la molécule d'ADN.
Cette translocation s'accompagne également d'un changement transitoire du degré de surenroulement de la molécule d'ADN.

Comparaison biochimique de l’activité
Mécanismes d’action proposés pour le remodelage
Force en Biologie
Force entropique
Pour l’ADN par exemple, ces fluctuations entraînent une perte d’orientation de la molécule sur une distance appelée longueur de persistance ξ, qui est d’environ ξ = 50 nm. Si nous étudions l’ADN sur des échelles de taille supérieures à ξ, l’ADN a la forme d’une sphère statistique. Par la suite, nous verrons que la force typique à laquelle on observe l'activité des facteurs de remodelage de la chromatine est de l'ordre du pico-Newton (pN) : c'est une force suffisamment élevée pour obtenir un étirement important de l'ADN et des fluctuations limitées (faible bruit ). ), mais un voltage suffisamment bas pour ne pas (trop) contrarier l'activité de ces protéines.
Force de rupture de liaison covalente F max
Force de rupture de liaisons non-covalentes
Ce dispositif permet également la rotation de la micropipette et donc la torsion de la molécule d'ADN. Avec ces molécules d'ADN, nous observons des résultats différents selon la topologie de la molécule. Ces superturns augmentent le nombre de pléctonomes négatifs présents dans la molécule d'ADN et diminuent donc l'allongement de la molécule.
Micromanipulation de l’ADN
Les pinces optiques
Dans le système microfibre, une molécule d’ADN est tirée avec une micropipette et la force exercée par la déviation de la microfibre est mesurée. Dans une pince magnétique, les molécules d’ADN sont tirées et mises en rotation à l’aide d’une paire d’aimants. La limite inférieure des forces appliquées est déterminée en fonction de la force avec laquelle les fluctuations thermiques déstabilisent la chute.
Les dispositifs à microfibres
3.3 - Quelques exemples de méthodes de micromanipulation utilisées pour étudier l'interaction ADN-protéine Dans une pince optique, un faisceau laser capte et projette une bille qui possède un indice de réfraction élevé par rapport à l'eau. La constante de rigidité de la pince ktrap peut être déterminée avant la mesure, par exemple, en utilisant le flux hydrodynamique (vitesse et viscositéη), agissant avec une force FS sur la particule piégée à rayonner : Fs = 6πηrv. La limite supérieure est déterminée par l’absorption du faisceau par la particule piégée, ce qui provoque un échauffement local pouvant endommager les échantillons biologiques.
Les pinces magnétiques
Différences entre les différentes techniques de micromanipulation de l’ADN 70
3.4 – Aperçu des principales techniques utilisées dans les études d'ADN à molécule unique avec les caractéristiques clés de chaque technique. Au cours de ma thèse, j'ai utilisé trois techniques différentes pour manipuler et visualiser des molécules d'ADN uniques, ce qui m'a permis d'étudier les facteurs de remodelage de la chromatine.
Pinces magnétiques
- Microscope
- Molécule d’ADN utilisée
- Ancrage de la molécule sur les billes et sur la surface du capillaire
- Mesure du déplacement de la bille et de l’extension de la molécule d’ADN 73
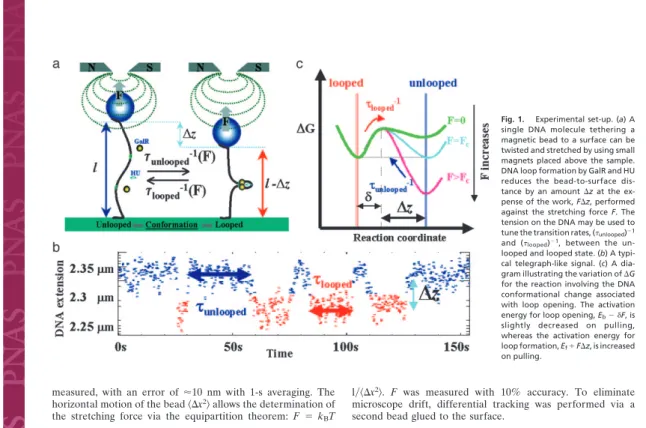
Si l’enzyme transloque la molécule d’ADN, elle formera avec le temps des boucles d’ADN qui réduiront l’allongement de l’ADN d’une quantité égale à la taille de la boucle. Ce signal résulte de l’introduction de boucles d’ADN, qui réduisent la longueur de la molécule et donc le mouvement brownien. L'article suivant présente une étude détaillée du bouclage de l'ADN dans la région promotrice opérongale.
TPM
Montage expérimental
Les échantillons d'ADN de billes ont été observés à l'aide d'un microscope à contraste interférentiel différentiel (DIC). Les images ont été collectées avec une caméra vidéo CCD (CV-M10) ayant une fréquence d'enregistrement de 25 Hz. Toutes les images ont été enregistrées sur vidéocassettes et numérisées avec un ordinateur (Fig.3.11).
Préparation des chambres de mesure et de l’ADN
3.10 – Schéma montrant un exemple d'étude au niveau d'une molécule unique utilisant la technique TPM de translocation de RecBCD par le groupe Gelles[239]. a) Une molécule d'ADN de 1,3 à 1,5 kpb, fonctionnalisée avec une digoxigénine à une extrémité, a été fixée à la surface d'un capillaire recouvert d'antidigoxigénine. Les extrémités d'une molécule d'ADN sont fonctionnalisées par PCR avec une biotine d'un côté et une digoxigénine de l'autre. Grâce aux liaisons spécifiques entre digoxigénine/antidigoxigénine et biotine/streptavidine, l'ADN est spécifiquement attaché à la bille et à la surface de la microchambre.
Analyse des images
3.14 – Schéma montrant la relation entre la distance bout à bout (E2E) et le déplacement δx de la balle. Si la balle est attachée par une seule molécule, les fluctuations browniennes le long des deux axes seront symétriques (Fig. 3.13c et d).
Calibration et mesure de la distance bout-à-bout (E2E)
3.15 – Courbe d'étalonnage obtenue en mesurant l'amplitude des oscillations browniennes en fonction de la taille de la molécule d'ADN. Il est possible de relier l'amplitude de mouvement δx de la bille à la taille de l'ADN qu'elle ancre à la surface en calibrant les oscillations browniennes de plusieurs billes fixées avec des molécules d'ADN de différentes tailles. Comme le montre la figure 3.15, l'amplitude des oscillations browniennes observées correspond à la courbe théorique obtenue en modélisant l'ADN avec le modèle de chaîne en forme de ver (WLC).
AFM
Montage expérimental
Le microscope utilisé pour acquérir les images est un microscope Multimode Nanoscope IV de Digital Instruments. Pour contrôler l'humidité de l'air, le microscope a été placé dans une enceinte en plexiglas, dans laquelle règne une atmosphère saturée d'azote, permettant d'éliminer toute trace d'eau de la surface du mica et ainsi de réduire la force de Laplace entre la pointe et la surface du mica. champion. ce qui améliore les images obtenues [281].
Dépôt d’ADN sur le mica
Analyse des images
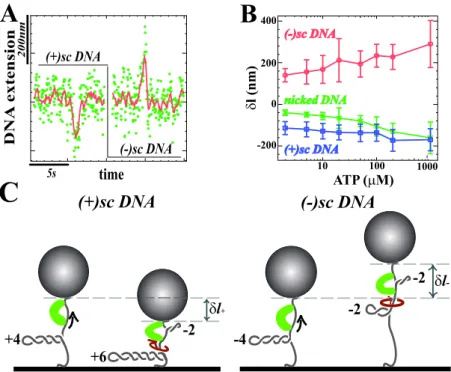
Une molécule d’ADN est dite relâchée si son indice d’intrication Lk est égal à l’indice d’intrication naturel Lk0, qui est déterminé par la relation suivante. La valeur Lk0= 6 signifie que la molécule d'ADN présente 6 tours de double hélice ou 6 croisements d'hélice. Une molécule d'ADN superenroulée peut donc être formée en réduisant la torsion de deux unités (I) ou en augmentant la torsion de l'ADN de la même quantité (II).
Comportement de l’ADN sous torsion
Si on augmente la force (F > 1 pN) ces courbes deviennent asymétriques : l'extension de l'ADN n'est plus réduite lorsque la molécule est sous-enroulée (∆Lk < 0). Au-delà du seuil de torsion, la molécule d'ADN forme à chaque tour un plectone qui réduit l'allongement de la molécule (in. 4.5 – Principe de mesure avec une pince magnétique Avec une pince magnétique on mesure l'allongement de la molécule d'ADN au cours du temps.
Micromanipulation d’une seule molécule d’ADN
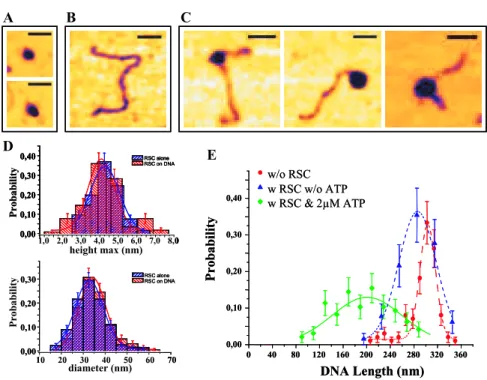
À une concentration très élevée d'ISWI (plus de 25 nM), un compactage de la molécule d'ADN est observé en raison de la formation de nombreux complexes le long de la molécule (Figure 4.10A). Ainsi, nous pouvons mesurer la variation de l'extension des molécules d'ADN en présence d'ISWI mais sans ATP (Figure 4.11D), et constater que l'ISWI la réduit de 46,5 ± 2,2 nm. Cela suggère que le nombre de tours compensatoires introduits dans l’ADN enroulé est indépendant de la concentration en ATP.
Cet effet a également été observé pour d'autres enzymes qui transloquent la molécule d'ADN (la protéine FtsK [288]). La taille de la boucle d'ADN transloquée par les deux familles augmente avec la concentration en ATP selon la cinétique de Michaelis-Menten.
Article RSC
Expériences avec la protéine ISWI
- AFM
- Étude de l’accrochage d’ISWI sur une molécule d’ADN linéaire nu
- Translocation d’ISWI sur l’ADN linéaire en présence d’ATP
- Pinces magnétiques
- Accrochage d’ISWI sur une molécule d’ADN "nickée"
- Accrochage d’ISWI sur une molécule d’ADN superenroulable
- Translocation d’ISWI sur une molécule d’ADN "nickée"
- Translocation d’ISWI sur une molécule d’ADN superenroulable
- Translocation sur l’ADN simple brin
- Effet de la force
- Modèle de translocation proposé
Concernant le complexe RSC, nous avons étudié les propriétés de liaison de l'ISWI sur une molécule d'ADN nickelée. Comme le montre la figure 4.12A, (à F = 0,4 pN), l'attachement d'une seule protéine ISWI à l'ADN sous-enroulé s'accompagne d'une réduction de la longueur de la molécule d'ADN. Cet effet peut s'expliquer par un phénomène de relâchement partiel de la torsion dans la boucle d'ADN transloquée (glissement).
Expériences faites avec la technique TPM
Contrôles effectués
Sur la trace B), on constate l'adsorption de la bille en surface en fin de capture. En vert se trouvent les points expérimentaux, et en rouge les points moyens sur 4 s. D) Répartition du bruit d'une balle collée à une surface. En convertissant le mouvement brownien de ceux-ci en distance effective, j'ai pu déterminer le niveau seuil de mouvement brownien (15 nm), en dessous duquel le mouvement est caractéristique du mouvement d'une balle adsorbée à la surface (Fig. 4.22 D). .
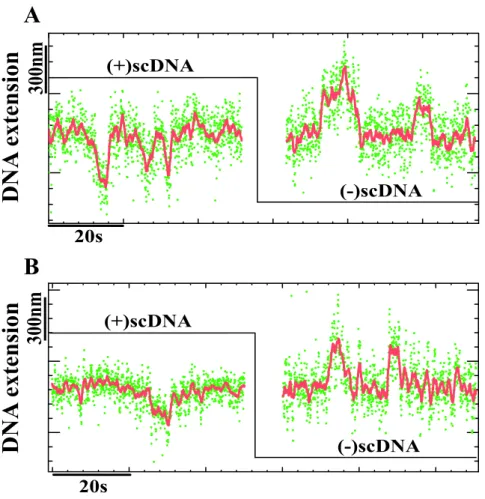
Mesures effectuées en présence d’ATP
Expérience avec la technique TPM, les mécanismes d'exploitation ISWI et RSC. 4.23 – Signal télégraphique en présence de ISWI.A). 4.26 - Pinning d'ISWI sans ATP ou en présence d'ATP-γ-S.A) et B) Signaux obtenus avec ISWI sans ATP. Sur les figures 4.26A et B, nous pouvons voir deux traces obtenues en présence d'ISWI sans ATP.
Pinces magnétiques vs. TPM
Dans le cas de l'ISWI, comme dans le cas du RSC, la formation de boucles est affectée de manière exponentielle par la force et aucune boucle d'ADN n'a été observée au-dessus d'environ 2,3 pN. La translocation d'ADN introduit de manière transitoire des changements dans le nombre d'enveloppement de la molécule d'ADN, comme dans le complexe RSC, mais avec la chiralité opposée (bobines superposées stockées dans la boucle ; bobines inférieures compensatoires dans le reste de la molécule). La cinétique de rétention des protéines sur une molécule d’ADN est en effet différente de celle obtenue par des expériences réalisées avec des pinces magnétiques.
L’ADN pour les pinces magnétiques
Préparation de l’ADN pour les expériences TPM et AFM
Préparation des capillaires pour les pinces magnétiques
Ensuite, il est placé sur une platine chaude pendant quelques minutes, après quoi il est incubé avec du Sigmacote (Sigma-Aldrich), qui forme une couche de polysilane en surface. Enfin, la surface est passivée et stockée à 4◦C jusqu'à utilisation dans une solution contenant du PBS, 1% de BSA, 1% de PluronicF127 (Sigma-Aldrich) et 0,01% de NaN3.
Préparation des microchambres pour les expériences TPM
Préparation des disques de Mica pour l’AFM
Tampon de la protéine
Analyse de données
Subunits of the yeast SWI/SNF complex are members of the actin-related protein (ARP) family. SWI/SNF-related chromatin remodeling complex, E-RC1 is required for tissue-specific transcriptional regulation by EKLF in vitro. Roles of the histone H2A-H2B dimers and (H3/H4)2 tetramer in nucleosome remodeling by the SWI/SNF complex.
![FIG. 2: Dependence of δl n A) on ATP concentration at fixed force F = 0.3 pN and B) on stretching force F at fixed [ATP]=100µM](https://thumb-eu.123doks.com/thumbv2/1bibliocom/465153.70487/105.892.338.662.327.494/dependence-concentration-fixed-force-stretching-force-fixed-100µm.webp)