Une classe d’enzymes appelées topoisomérase est responsable de la régulation de la topologie de l’ADN dans la cellule. Outre leur propre importance physique, ces expériences constituent un prélude nécessaire à l’étude de la décaténation de l’ADN par les topoisomérases à molécule unique.
Introduction
Structure de l’ADN
- Structure primaire
- Structure secondaire
- Structure tertiaire
- Indice d’enlacement
- Vision intuitive
- Intégrale de Gauss
- Super-enroulement
- La Torsade (ou T wist (T w))
- La vrille (ou W rithe (W r))
- Théorème de Calugareanu
- Extension au cas de molécules linéaires
Toutes ces règles impliquent des modifications géométriques locales, mais aussi des changements topologiques globaux de la molécule d'ADN. De plus, l’état tordu de l’ADN est défini par une autre variable appelée twistTw.
Le super-enroulement de l’ADN
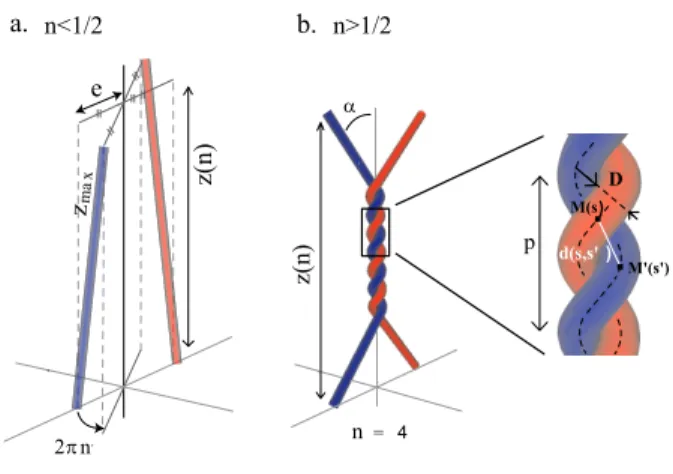
Géométrie des molécules super-enroulées
Cependant, il a été suggéré (voir [90] pour une revue) qu'il pourrait y avoir des régions où la topologie de l'ADN est bloquée. Un solénoïde est constitué d'une seule hélice b). Les données expérimentales (voir la figure 2.6 pour un exemple) montrent que les molécules d'ADN déroulées forment des structures plectonémiques [134].
Le super-enroulement in vivo
- Compaction de l’ADN
- Transcription
- Réplication
L’enroulement négatif de l’ADN favorise la déstabilisation de la double hélice, en particulier dans les régions riches en AT. Au fur et à mesure que le complexe enzymatique progresse, un excès d’enveloppement d’ADN se crée en amont de la fourche de réplication et un déficit en aval.
Noeuds et liens
Les acteurs de la régulation de la topologie de l'ADN sont des enzymes appelées topoisomérases (on les appellera aussi « topo »). Enregistrement typique de l'extension d'une tresse lors de la décaténation par topo 3 (20 nM) à 1 pN.
Méthodes usuelles d’études de l’ADN
Electrophorèse de l’ADN
Sur la ligne 1, on distingue deux bandes correspondant aux molécules d'ADN circulaires détendues (en haut) et aux molécules superenroulées (en bas) (les molécules migrent de haut en bas). Dans les 2ème et 3ème rangées, chaque bande représente des molécules d'ADN dont le nombre d'entrelacement diffère d'une unité (appelés topoisomères).
Microscopie électronique
La figure 2.9 montre une image d'un gel d'agarose obtenu après migration de molécules d'ADN circulaires ayant des topologies différentes. La rangée 1 montre deux populations de molécules d’ADN circulaires : certaines sont détendues et d’autres sont superenroulées.
Microscopie à force atomique
Dans les lignes 2 et 3, en plus des deux populations précédentes, un ensemble de bandes isolées indique la présence de topoisomères dans des états super-enroulés intermédiaires entre les bandes de la ligne 1. Ces topoisomères ont été obtenus après l'action de la topoisomérase sur les molécules initialement super-enroulées de ligne 1.
Intérêt de la micromanipulation de molécule unique
Les caténanes ont été obtenues par l'action simultanée de l'hélicase RecQ et de la topoisomérase III (voir Partie III).
Dispositifs pour étirer l’ADN
Ordres de grandeur de force
- Force de rupture de liaison covalente F max
- Force de rupture de liaisons non-covalente
- Force entropique
- Force de Langevin
Puisque la viscosité de l’eau ne peut être réduite et que l’échelle de temps τ ∼ 1/∆f est déterminée par les processus biologiques étudiés, nous verrons ci-dessous que la seule façon d’améliorer le rapport signal sur bruit est de réduire la taille de l’eau. capteur. . Ensuite nous verrons que la force typique avec laquelle on observe l'activité des topoisomérases est de l'ordre du picoNewton (pN) : c'est une force suffisamment élevée pour permettre un étirement important de l'ADN et obtenir des fluctuations avec une extension limitée, mais qui est faible. suffisant pour ne pas contrarier (trop) l’activité des topoisomérases.
Dispositif pour tirer et tordre l’ADN
Microfibres
Piège optique
Pince magnétique
Construction d’ADN et accrochage des molécules
Construction d’ADN
1.2 – Construction de l’ADN et attachement au ballon et à la surface. a) L'ADN utilisé est le résultat de la ligation de trois fragments : le fragment principal est obtenu par extraction de l'ADN plasmidique de bactéries. Une telle construction permet la fixation spécifique d'une extrémité à la surface d'une bille magnétique recouverte de streptavidine, et de l'autre extrémité à la surface du capillaire, préalablement incubé avec un anticorps anti-digoxygénine.
Accrochage bille-ADN et surface-ADN
Les deux fragments terminaux sont synthétisés par réaction de polymérisation en chaîne (PCR) sur le plasmide pBlueScript en présence d'oligonucléotides dUTP marqués soit à la digoxigénine, soit à la biotine. Fragments terminaux synthétisés par PCR en présence d'oligonucléotides dUTP marqués à la biotine ou à la digoxigénine.
Blocage en rotation
Une fois digérés, ces fragments (d'environ 600 paires de bases) sont ligaturés au fragment central (d'environ 10 000 paires de bases).
Montage expérimental
Le capillaire, qui contient le mélange billes-ADN, est noyé dans une plaque de cuivre vissée sur la plaque. Lorsque les aimants sont placés à proximité du capillaire, les billes fixées par l'ADN s'éloignent de la surface et oscillent librement dans la solution.
Mesure de l’extension de l’ADN
L'autocorrélation du profil de la balle permet de déduire en temps réel le déplacement d~ de la balle. Une référence absolue à la position de la balle est obtenue en laissant tomber la balle sur la surface.
Mesure de force par mouvement brownien
- Analyse temporelle
- Analyse fréquentielle
- Quel modèle pour l’ADN?
- La chaîne gaussienne
- Le modèle de Kratky-Porod
- La chaîne librement jointe (FJC)
- Le modèle du ver (ou worm-like chain (WLC))
- Simulation Monte-Carlo d’une chaîne semi-flexible
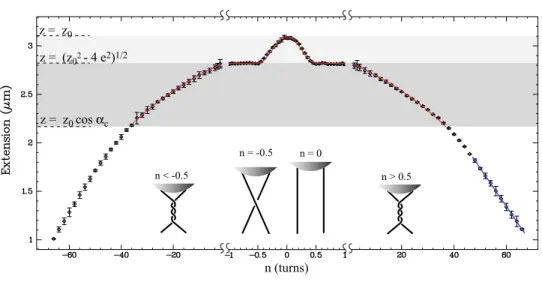
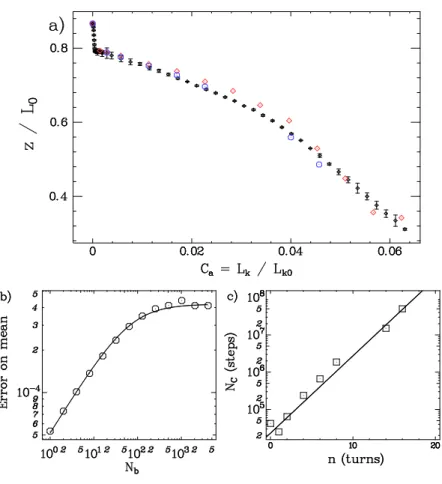
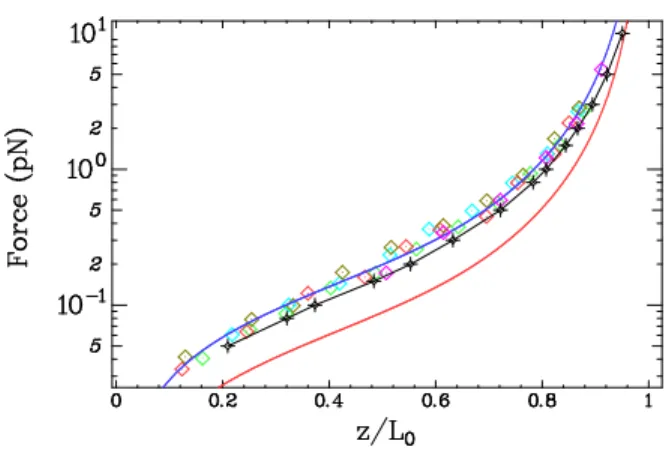
2.1 – Expansion de la force de réponse d'une molécule d'ADN dans son régime entropique Les points expérimentaux sont verts. La courbe force-extension d'une molécule d'ADN peut également être obtenue à partir d'une simulation Monte Carlo d'une chaîne semi-flexible [135].
L’ADN étiré sous torsion
- Modèle simple de l’instabilité de flambage
- Modèles statistiques pour le super-enroulement de l’ADN
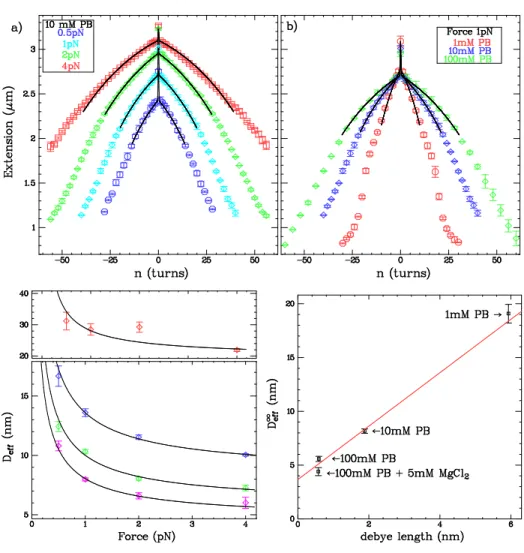
- Influence de la concentration ionique sur les courbes extension-rotation
- Modification de la torsade spontanée T w0
- Modification de la pente des courbes extension-superenroulement
- Modification du nombre de tours critique de flambage
- Simulation Monte-Carlo d’une molécule d’ADN super-enroulé
- Estimation de la constante de torsion
- Courbes extension-rotation et force-extension
- Effet du diamètre effectif de l’ADN
- Transition induite par le couple
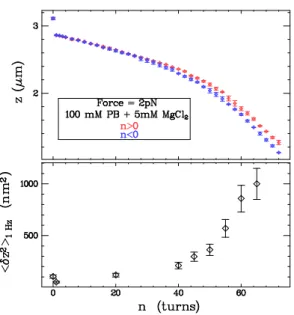
La figure 2.9 montre les courbes d'étirement-rotation obtenues dans du PB 10 mM (rouge) et dans un tampon topo 4 à 0,7 pN (bleu). Premièrement, la présence d’ions magnésium dans le tampon topo 4 affecte les propriétés élastiques de la molécule d’ADN.
Intérêt pour l’étude des topoisomérases
Le comportement de l’ADN sous l’influence d’une force et d’une contrainte de torsion est désormais bien compris. Dans ce chapitre, nous étudions la caténation par micromanipulation de deux molécules d'ADN recouvertes de nickel (qui présentent une cassure simple brin).
Expériences et modèles préliminaires
The scheme of [55], based on the structural particles of the underlying units GyrA (in blue bright) and GyrB (in violet) of the enzyme. Relaxation of the (⫹) supercoils that form before the replication fork (by gyrase and Topo IV) is thought to be the major route for uncoupling in the early stages of replication, while decatenation of the entangled sister chromosomes becomes increasingly important as replication progresses. progresses to the final stage (9, 10).
Article : "Braiding DNA..."
Réplication d’une molécule circulaire
Les topoisomérases
- Clivage de l’ADN
- Classification
- Type I
- Type II
- topoisomérases procaryotes
- topo 1
- topo 3
- Gyrase
- topo 4
- Résumé
- topoisomérases eucaryotes
- topo 3
- topo 1B
- topo 2
- Résumé
Cette coupure est réalisée par deux groupements tyrosine (appartenant à deux sous-unités identiques de l'enzyme) sur chaque squelette de la molécule d'ADN. Une partie importante du topo 2 de levure (792 acides aminés) a été cristallographiée avec une résolution de 2,7 [9].
Questions fondamentales
Quel lien existe entre l'hydrolyse de l'ATP et le passage du segment T à travers le segment G. L'hydrolyse d'une autre molécule d'ATP serait impliquée dans la reformation de l'enzyme, ce qui nécessite la séparation de ses extrémités N-terminales.
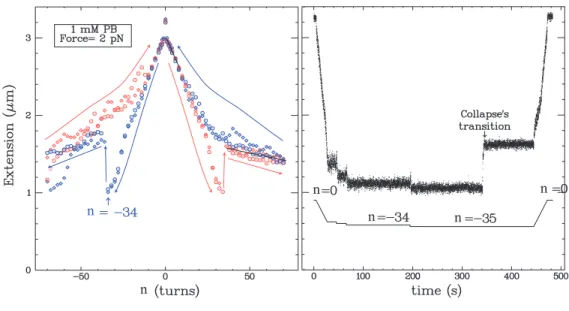
Relaxation du super-enroulement
Cette expérience montre également que l'enzyme agit de manière processive (la processivité ne peut être mesurée dans ces conditions car le nombre de tours générés mécaniquement semble être systématiquement inférieur à la processivité de l'enzyme). De ce fait, la probabilité que l’enzyme reste coincée entre deux cycles enzymatiques semble assez faible.
Cycles enzymatiques uniques
Pour mesurer réellement la processivité, il faut être capable d'enrouler rapidement la molécule au fur et à mesure qu'elle est déroulée par l'enzyme. Ceci est lié au mécanisme cinétique par lequel l'enzyme réalise sa catalyse (comme nous l'expliquerons plus loin).
Article : On the relation between noise spectre and the distribution of time between
Enfin, il semble clairement établi que l'angle entre croisements est un paramètre déterminant de l'activité du topo IV. Tous ces événements entraînent des interruptions dans la topologie de l’ADN que doivent gérer les topoisomérases.
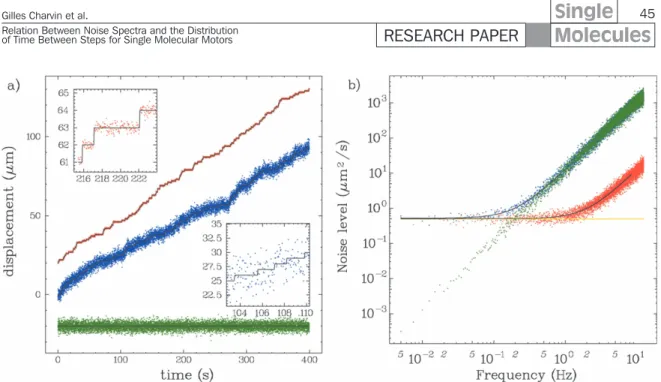
Observation des cycles enzymatiques
Influence de la concentration d’ATP
- Vitesse de réaction l’enzyme en fonction de [ATP]
- Coopérativité de l’accrochage des molécules d’ATP
- Modélisation du cycle enzymatique
- Distribution des temps de cycle et stochasticité
Dans ce régime de concentration en ATP ([AT P]<< KM), l'étape limitante du cycle est associée à la fixation de l'ATP. C'est également cette propriété qui fait varier la vitesse de réaction comme le carré de la concentration en ATP et qui fait que la réaction est coopérative par rapport à l'ATP.
Influence de la force sur l’activité de l’enzyme
2.10 – Taux de relaxation pour le superenroulement et la décaténation des topoisomérases en fonction de la force appliquée Les expériences réalisées avec topo 4 (en haut) et topo 2 (en bas) ont été équipées des fonctions v = v0exp(−F∆/kBT) )etv = v0/( 1 +kexp(F∆/kBT)). A noter que le taux de décaténation à F = 0 est très proche du taux de relaxation du supercoiling à F = 0, confirmant sa capacité égale à travailler sur les deux substrats.
Etude d’enzymes mutantes de la topo 2 de S. cerevisiae
- Activité du double mutant E66Q/E66Q en molécule unique
- Activité du mutant E66Q/WT en molécule unique
- Relaxation du super-enroulement
- Lien avec la fonction de la topo 4 in vivo
- Quel(s) modèle(s) pour expliquer la chiralité de la topo 4?
- Reconnaissance globale de la topologie
- Modèle de Klenin-Langowski-Vologodskii (KLV)
- Sensibilité au couple
- Reconnaissance d’un croisement d’ADN
L’idée la plus simple est de penser que l’enzyme est capable de reconnaître la géométrie générale de la molécule d’ADN. Il émet l’hypothèse que l’enzyme aurait une certaine affinité pour la géométrie des jonctions d’ADN attendues dans un plectonème.
Accrochage de la topoisomérase 4 sur l’ADN
Propriétés d’accrochage de la topo 4
On distingue donc clairement l'angle entre le croisement d'un superturn (+) et d'un superturn (-), mais aussi des tresses d'ADN dont le sens d'enroulement diffère (voir Figure 3.4). Ce modèle est particulièrement adapté au mécanisme d'action de l'enzyme, qui interagit très localement avec deux molécules d'ADN.
Article : Bending effect by topoisomerase IV
Addition of topo IV (0.3 nM) to the reaction chamber (thereby leading to a leak into the chamber) leads to a smooth, gradual (on time scale≈500s) increase in DNA extension (Fig. 3a). Data obtained in the absence of topo IV were fitted (black line) using a powerlaw function[17]:δl0=AF−0.4withA≈30.

Décaténation par la topo 4
- Le "paradoxe" de la topo 4
- Article : "Single molecule study of DNA unlinking by eukaryotic and proka-
- Données supplémentaires publiées avec l’article
- Comparaison avec l’article de Stone et al
- Expériences complémentaires : 2 molécules, dont une au moins super-enroulable 123
- Principe de l’expérience
- Résultats
Interestingly, the force dependence of the Topo IV uncoupling rate differs from that of Topo II. In Salmonella, a defect in the separation of the daughter chromosomes (a partition defect) is associated with a mutation in a membrane protein, ParF (24).
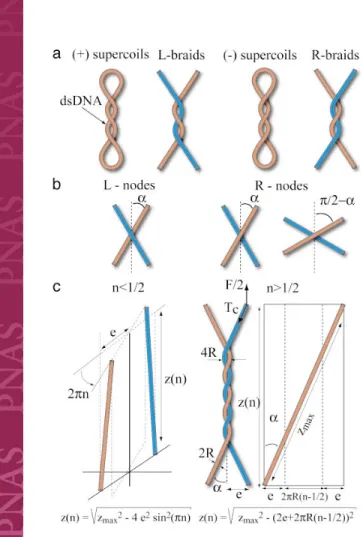
Modèles
Modèle à trois sites d’accrochage
Ainsi, de manière surprenante, il existe des différences significatives (mais corrélées entre les différentes expériences) selon les topoisomérases de type II utilisées. Cependant, le mécanisme liant cette propriété de reconnaissance active des topoisomérases de type II à l’utilisation de l’ATP reste flou.
Modèle de Vologodskii
Topo 3, topoisomérase de type I, est également incapable de maintenir une population plasmidique dans un état déplié en dessous du niveau d'équilibre thermodynamique[ 102 ]. Ce modèle est basé sur le fait que le passage du brin se fait toujours de l'intérieur vers l'extérieur de l'épingle à cheveux.
Modèle d’amplification cinétique
Selon les auteurs, l'ATP, dont l'hydrolyse sert à garantir l'unidirectionnalité, permet ainsi de simplifier la topologie à l'équilibre. Pour concilier ce modèle avec les observations sur le rôle de l'ATP dans le cycle enzymatique (voir chapitre 2 de la partie III), les auteurs proposent que l'étape 2->1* soit rendue irréversible par l'attachement d'une molécule d'ATP à la juxtaposition du G et T.
Principe de l’expérience
Résultats
- Le signal de décaténation
- Fréquence de passage de brin
- Détermination de P (Ca) par simulation numérique
- Distribution des temps τ (Ca)
- Nécessité d’un contrôle expérimental : la topo 3 d’E. coli ?
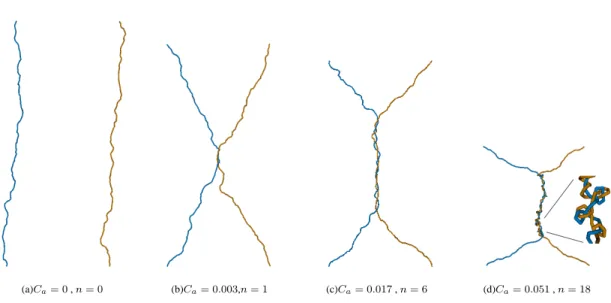
La figure 4.9 montre la comparaison de la fréquence de croisement des cordes obtenue à 2 pN et P(Ca) obtenue à partir d'une simulation numérique. En ajustant une courbe exponentielle, nous avons renormalisé les temps de transit des brins obtenus lors de la cinétique.
A gauche : évolution de la position des moteurs en fonction du temps lors de l'enregistrement des temps de décaténation. Après notre étude en 4 billes, il nous a semblé prudent d’examiner l’activité de la gyrase sur une seule molécule.
Relaxation du super-enroulement (+)
Topo 4 et gyrase sont les deux topoisomérases de type II trouvées chez les procaryotes. Ensuite, la gyrase possède des propriétés uniques : elle fait tourner le solénoïde d’ADN sur elle-même, ce qui lui permet de générer un surenroulement négatif en présence d’ATP.
Génération de super-enroulement négatif
Perspectives
Perspectives pour l'étude des topoisomérases par micromanipulation de molécules uniques Au cours de cette thèse nous avons utilisé la micromanipulation de molécules uniques d'ADN pour étudier des enzymes de la classe des topoisomérases de type II. De plus, nous avons étudié les propriétés de reconnaissance et de discrimination des topoisomérases.
Préparation des capillaires
Parallèlement, une molécule d'ADN d'environ 1000 paires de bases est synthétisée par PCR à partir du plasmide pKS (pBlueScript) autour du site MCS. Finalement, la ligation de ces trois fragments donne une molécule d'environ 11 kb a priori super-enroulée.
Tampon
Cependant, une fraction variable de molécules présente généralement au moins une cassure simple brin. A noter que nous avons utilisé à plusieurs reprises des molécules synthétisées par Marie-Noëlle Dessinges, Giuseppe Lia et Jean-François Allemand.
- topo 2 de Drosophilia melanogaster
- topo 2 de Saccharomyces cerevisiae
- topo 4 de E. coli
- Gyrase de E. coli
- topo 3 de E. coli
L'utilisation du chlorure de magnésium ne pose aucun problème, mais le remplacement du glutamate de potassium par du chlorure de potassium inhibe complètement l'activité de l'enzyme. L'enzyme provient de John Innes Enterprises en Angleterre, dont le site Internet est à l'adresse suivante : http://www.john-innes.co.uk/gyrase/.
Analyse de données
Nous utilisons la procédure Metropolis[80] pour générer une série de configurations caractéristiques de l'équilibre thermodynamique, comme décrit dans[61]. L'ADN est décrit comme une chaîne semi-flexible de N longueurs KuhnlK, constituée de N. segments élémentaires (voir figure C.1), telle que k >>1.
Energie de courbure
Force
Mouvements de la chaîne
Rotation d’un segment de la chaîne
Mouvement de Crankshaft
Critère d’acceptation/rejection
Gestion des erreurs numériques
Echantillonnage de la molécule d’ADN
Mesure de vrille et contraintes topologiques
Calcul de vrille W r
Indice d’enlacement
Noeuds
Contraintes géométriques
Auto-évitement
Murs
Ces parois provoquent une déviation significative de l'extension de la molécule pour les faibles extensions < z > /L < 0,25, due à une répulsion entropique exercée par la molécule (voir figure C.4). L'allongement typique à forces nulles est donné par la formule de pas aléatoire et croît donc comme L0,5.
Distance entre molécules
Juxtaposition et calculs d’angles entre segments
Optimisation
Compléxité des routines
Temps de relaxation
Interface et programmation
Role of DNA topology in mu-transposition: mechanism for recording the relative orientation of two DNA segments. A human topoisomerase II alpha heterodimer with only one ATP binding site can undergo successive catalytic cycles.