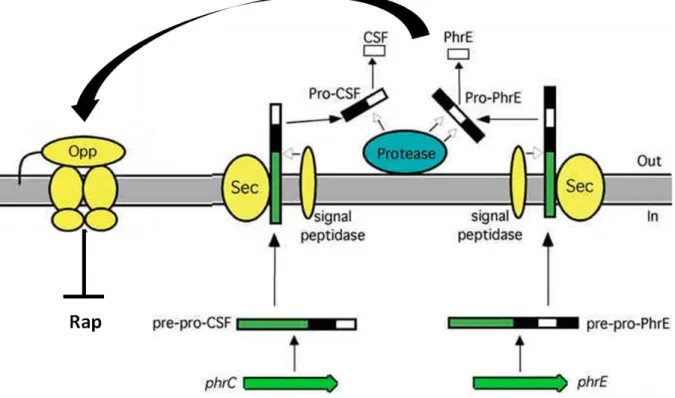
INTRODUCTION
L'activité antagoniste de CheR permet aux bactéries de mesurer les gradients chimiques et la phosphatase CheZ déphosphoryle spécifiquement CheY-P pour mettre fin à la signalisation.
La communication cellulaire chez les bactéries
- Interactions entre les bactéries et l’environnement
- Interactions entre les bactéries : le quorum sensing
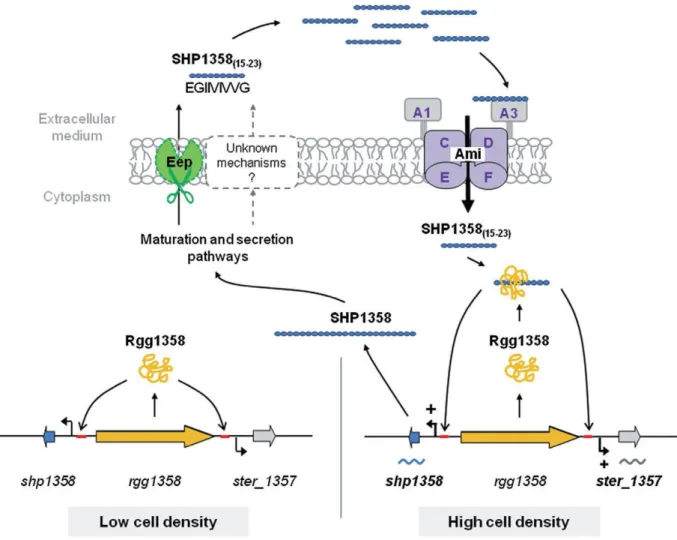
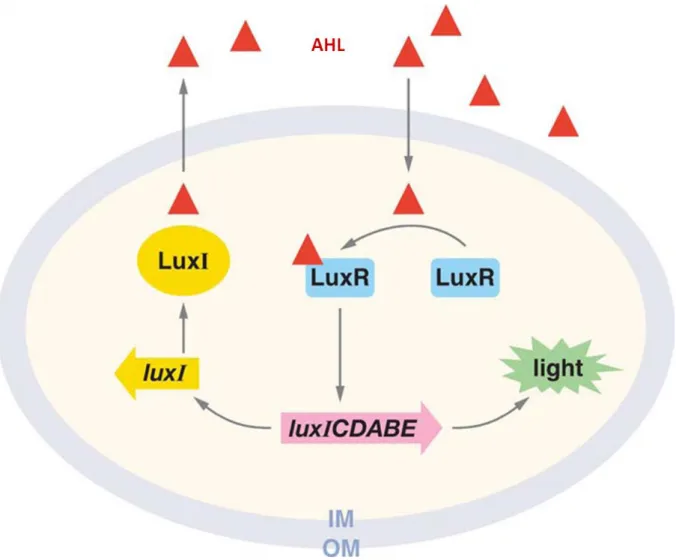
La détection du quorum chez les bactéries à Gram négatif repose principalement sur la production, la sécrétion et la détection des AHL. Le complexe LuxR/3OC6-HSL activé induit l'expression de l'opéron luciférase (Engebrecht et Silverman 1984) (Fuqua, Parsek et al. 2001) (Waters et Bassler 2005).
Le quorum sensing directe chez les bactéries à Gram positif
- Caractéristiques structurales
- Les membres
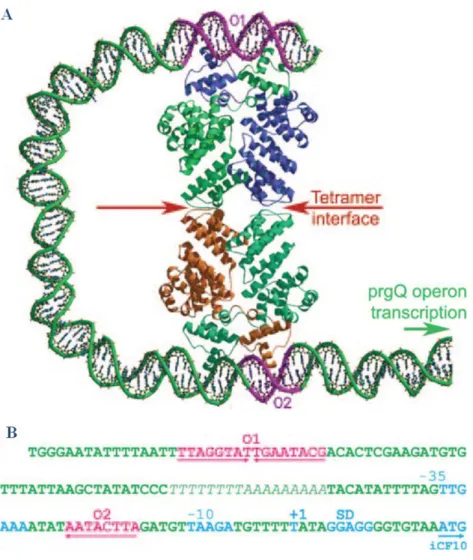
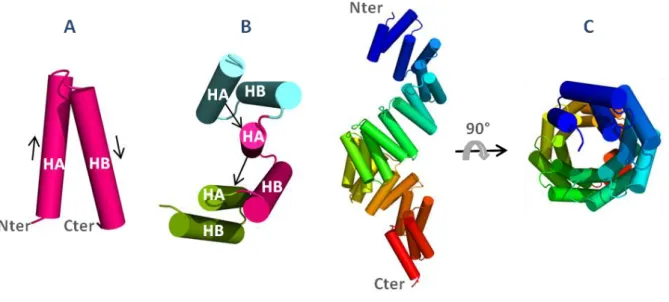
Une étude structurelle de PrgX a révélé une conformation tétramérique d'Apo (code PDB : 2AXU), où deux dimères typiques des facteurs de transcription du motif HTH sont liés via leurs terminaisons Cter (au dernier motif TPR) (Shi, Brown et al. 2005). Cette dernière est constituée de 5 motifs TPR et est fermée par une hélice α en Cter (Declerck, Bouillaut et al. 2007).
Projet
- MATERIELS ET METHODES
Le gène d'intérêt est placé sous le contrôle d'un promoteur inductible fort, le promoteur du phage T5, reconnu par l'ARN polymérase d'E. En présence de glucose (phase de croissance des bactéries en milieu LB), le répresseur LacI se lie à l'opéron lac et empêche ainsi la transcription. L'analogue non hydrolysable du lactose, l'IPTG, se lie à Lac1, libérant l'opéron et permettant ainsi à l'ARN polymérase de se lier au promoteur T5 et donc d'induire l'expression du gène d'intérêt.
Expression des protéines
- NprR et NprRΔHTH natives
- NprRΔHTH marquée au sélénium
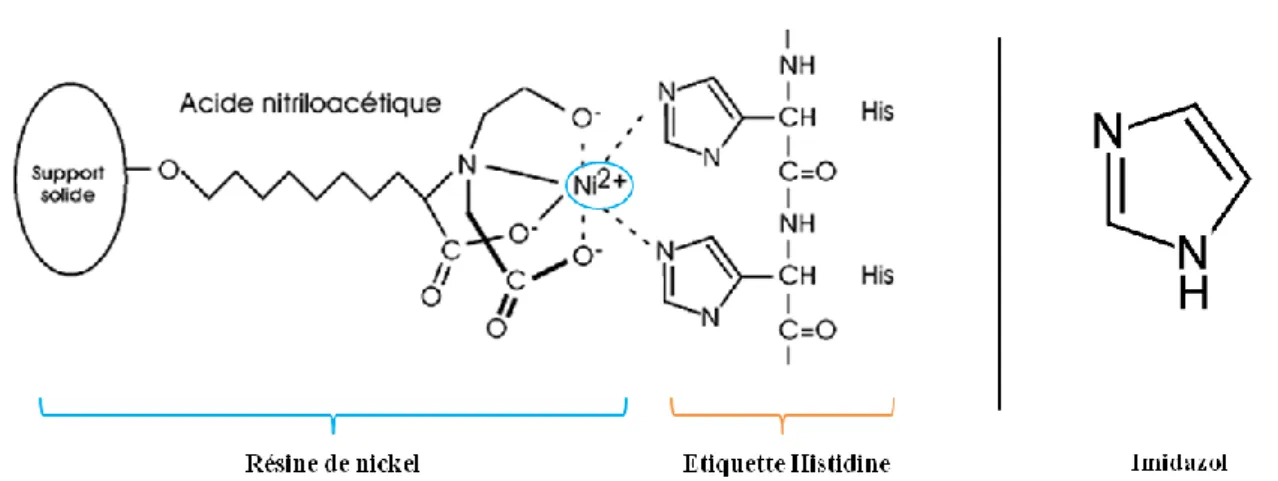
La culture est centrifugée à 5000 tr/min pendant 10 minutes à température ambiante et le culot bactérien est remis en suspension dans un litre de milieu M9-Met. Le culot bactérien est ensuite remis en suspension dans le milieu M9-SeMet dans lequel la méthionine est substituée par la sélénométhionine (SeMet). Il possède donc 2 composés pour interagir avec le cycle imidazole des résidus histidine sur l'étiquette protéique à purifier.
L'utilisation d'une solution concentrée d'imidazole (concurrent des résidus histidine) permet d'éluer les protéines restant sur la colonne.
Purification
- Extraction
- Chromatographie d’affinité IMAC
- Chromatographie d’exclusion par la taille
- Analyse par SDS-PAGE
Pour déterminer la courbe d'étalonnage de la colonne utilisée (Superdex S200-26/60), des kits d'étalonnage LMW (Low Molecular Weight) et HMW (High Molecular Weight) distribués par GE-Healthcare ont été utilisés. Les échantillons ont été injectés dans un volume de 100 µL sur colonne analytique Superdex S préalablement équilibrée dans du tampon E pH 7,5. Les échantillons lyophilisés ont été resolubilisés dans le tampon de conservation des protéines (tampon E) à des concentrations moyennes de 25 mM, puis aliquotés avec 30 µl et congelés à -20°C.
Des études de stabilité thermique par calorimétrie différentielle à balayage (FIGURE 35) du NprR ont été réalisées par Magali Nicaise, à l'IBBMC, sur un microcalorimètre VP-DSC (MicroCal). Les mesures DLS ont été réalisées sur des protéines diluées dans du tampon E, jusqu'à une concentration finale de 1 mg/ml (environ 20 µM).
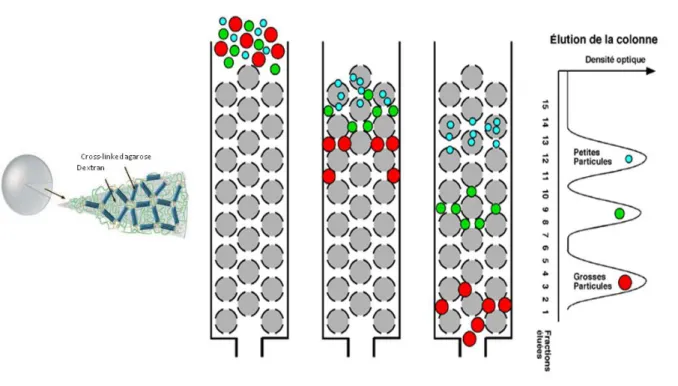
Cristallisation
- Criblage robotisé
- Reproduction manuelle et optimisation
- Cryoprotection et collecte des données
Il s'agit de placer, à proximité de la gouttelette où se sont formés les cristaux, 1 µl d'une solution de glutaraldéhyde (volatil) à une concentration de 0,2% (p/v dans l'eau) et d'incuber pendant 20 minutes à 2 heures. Enfin, tout en collectant des données de diffraction, j'ai testé la technique de recuit des cristaux. Pour limiter les dommages causés par les rayons X, les cristaux ont été congelés dans de l'azote liquide, puis maintenus sous un courant d'azote gazeux pendant la collecte des données de diffraction.
La distance cristal-détecteur est ajustée en fonction de la résolution à laquelle les cristaux se plient. Cette valeur est déterminée par un balayage de fluorescence des cristaux effectué sur la ligne de lumière avant la mise en place de la collection (FIGURE 41).

Résolution des structures cristallographiques
- Traitement des données
- Phasage
- Affinement et validation
Cette dernière a été améliorée par la technique d'aplatissement au solvant du programme RESOLVE (Terwilliger 2000) et utilisée par le programme de construction automatique Buccanner (Cowtan 2006) pour construire un premier modèle (partiel) de la protéine. Le modèle initial est orienté par une fonction de rotation puis positionné dans l'espace par une fonction de translation jusqu'à trouver les coordonnées qui reflètent le mieux l'ensemble des données expérimentales. Les calculs sont effectués en espace réciproque, par autocorrélation des fonctions de Patterson calculées à partir des modules de facteurs structurels observés d'une part et des coordonnées atomiques du modèle (dans différentes conformations) d'autre part.
Le programme Matthews (CCP4suite) a été utilisé pour déterminer le nombre de molécules (protéine seule ou en complexe) à placer dans l'unité asymétrique. Les solutions ont été recherchées dans tous les groupes spatiaux appartenant au groupe de symétrie ponctuelle identifié lors du traitement des données.
SAXS
- Etudes bioinformatiques
- Analyses des séquences
- Analyses structurales
- RESULTATS
Le logiciel en ligne ProtParam (site Expasy) a été utilisé pour déterminer les paramètres physico-chimiques des protéines, comme le point isoélectrique (pI) ou le coefficient d'extinction massique nécessaire au calcul de leur concentration (ANNEXE C). De multiples alignements de séquences ont été réalisés via le logiciel en ligne ClustalW (Chenna, Sugawara et al. 2003), qui ont été optimisés manuellement à l'aide du programme Seaview (Gouy, Guindon et al. 2010) avant d'être convertis dans le logiciel en ligne Esprit a été injecté (Gouet, Robert et al. . 2003) pour la superposition d'éléments de structure secondaires. Les recherches de séquences homologues ont été réalisées à l'aide du logiciel psiBLAST avec les paramètres par défaut (site Expasy).
Les superpositions structurelles ont été réalisées à l'aide du programme en ligne PDBeFold (Krissinel et Henrick 2004). Des études d'interaction ont été réalisées à l'aide des programmes en ligne PDBePISA et PDBsum (Krissinel et Henrick 2007) (Laskowski 2009).
Production et caractérisations biochimiques de l’effecteur
- Généralités
- Purification des échantillons pour la cristallisation
- Analyse par SEC-MALS
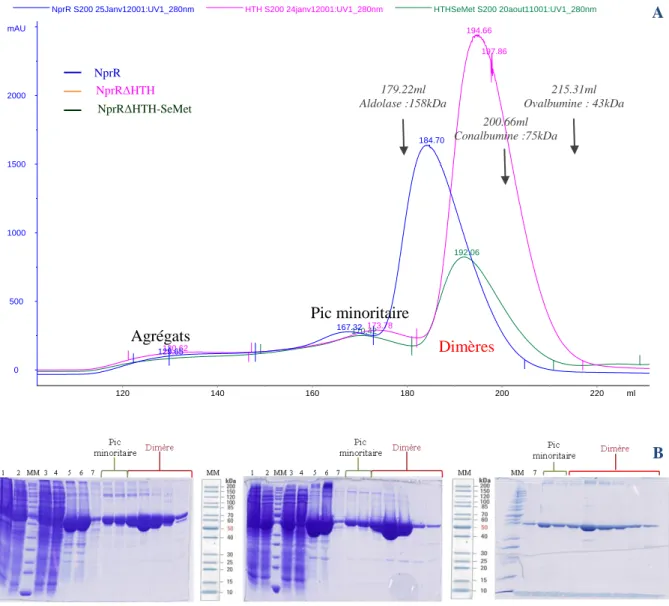
Les protéines NprR, NprRΔHTH et NprRΔHTH-SeMet sont purifiées en deux étapes selon le protocole décrit dans le chapitre Matériels et Méthodes : chromatographie d'affinité sur résine de nickel suivie d'une chromatographie d'exclusion de taille. Dans les deux cas, les profils d'élution des colonnes d'exclusion de taille présentent un pic majoritaire à des volumes d'élution de 184,70 mL et 194,66 mL pour NprR et NprRΔHTH, respectivement, précédé d'un pic minoritaire à des volumes d'élution de 167,32 mL et 173 mL NprR 73 et NrpR 73, respectivement (FIGURE 46-A). L'analyse électrophorétique sur gel SDS-PAGE des fractions d'élution montre que les 2 pics contiennent une bande largement majoritaire de masse correspondant à la protéine purifiée, NprR ou NprRAHTH (FIGURE 46-B).
Dans le tampon de purification, les échantillons de protéines NprR entières peuvent être concentrés jusqu'à 45 mg/ml (876 µM) tandis que la forme NprRΔHTH tronquée est plus soluble et supporte une concentration allant jusqu'à 60 mg/ml (1,33 mM). Nous avons analysé les deux formes de l'effecteur à la concentration de 2mg/ml soit 40µM et 45µM pour NprR et NprRΔHTH respectivement.
Le switch moléculaire
- Effet du peptide sur la structure secondaire de NprR : mesures en dichroïsme circulaire
- Effet du peptide sur NprR : électrophorèse en conditions non dénaturantes
- Effet du peptide sur l’état oligomérique de NprR : mesures en diffusion dynamique de
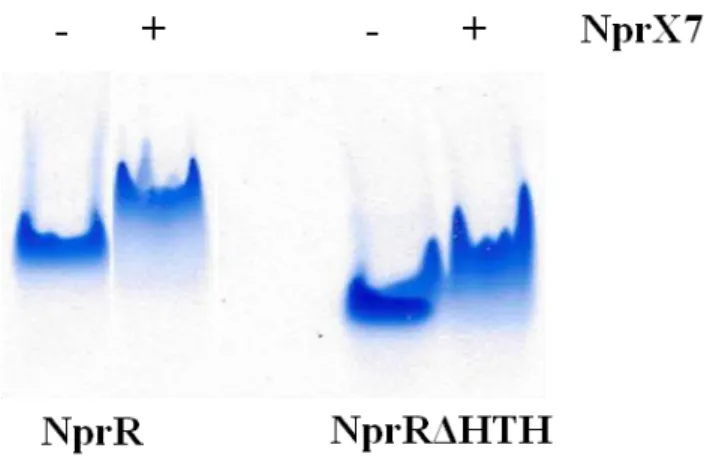
De même, les complexes NprR/NprX7 tels que NprRΔHTH/NprX7 présentent moins de migration électrophorétique que les formes Apo (FIGURE 49). Les formes Apo NprR et NprRΔHTH ont des rayons hydrodynamiques de 4,4 nm et 4,2 nm respectivement qui correspondent à des poids moléculaires moyens de 100 kDa pour une protéine globulaire, comme observé précédemment dans SEC-MALS. En présence de peptide, les complexes NprR/NprX7 et NprRΔHTH/NprX7 ont des rayons hydrodynamiques respectifs de 5,8 nm et 5,7 nm, qui correspondent ici à des masses moléculaires moyennes de 200 kDa (FIGURE 50).
Ces résultats confirment donc que la liaison du peptide induit une modification de l'oligomérisation de l'effecteur, le faisant passer d'une forme Apo dimère à une forme complexée tétramère. Valeurs de la constante de dissociation (Kd), de la stœchiométrie (n) et de l'enthalpie (∆H) calculées pour les expériences ITC décrites dans la figure 50 avec NprR∆HTH et différentes formes du peptide NprX.
Caractérisation des interactions entre NprR et ses ligands
- Interaction entre NprR et son peptide signal NprX
- Spécificité de reconnaissance NprR/ADN et NprR/NprX/ADN
Les mesures ont été réalisées avec le complexe NprR/NprX7 préformé dans la cellule de mesure à 10 ou 30 µM. Les oligonucléotides de la seringue (120 ou 330 µM) ont été injectés en une série de 20 injections de 2 µl espacées de 180 secondes. Pour analyser plus en détail l'effet du peptide sur l'interaction de NprR avec l'ADN, nous avons effectué des mesures de calorimétrie différentielle à balayage (DSC) avec l'oligonucléotide de 22 pb inclus dans la région B (Matériels et méthodes p 90).
Le Kd, la stœchiométrie (n) et l'enthalpie ∆H de l'interaction entre NprR∆HTH et les heptapeptides NprX7 des 7 ferogroupes ont été déterminés par des mesures ITC réalisées dans les conditions suivantes : protéine à 50 µM dans la cellule de mesure et 500 µM Peptide µM dans la seringue. Les peptides ont été ajoutés à la cellule de mesure par une injection initiale de 0,4 µl suivie de 36 injections consécutives de 1 µl, espacées de 180 s.

Activation croisée
- Spécificité d’interaction
- Le Switch moléculaire
Pour vérifier cette hypothèse, j'ai testé l'effet de différents peptides NprX des groupes II à VII sur la migration de la protéine dans le gel natif. Bien que moins prononcé qu'avec NprX7-I, une évolution vers des poids moléculaires plus élevés est également observée avec le complexe effecteur/NprX-II (FIGURE 56). En revanche, la fixation de NprX7-III n'a aucun effet sur la migration de la protéine qui présente un profil électrophorétique similaire à la forme Apo.
Les peptides de plus faible affinité des groupes IV à VII ne semblent pas non plus induire le déplacement indiquant une tétramérisation.

Analyse structurale de la conformation facteur de transcription
- Les complexes NprRΔHTH/NprX
- Le complexe NprR/NprX/ADN
Schéma expliquant le codage couleur des 9 motifs TPR le long de la séquence NprR∆HTH. Zoom sur la poche de liaison du peptide NprX8n dans NprRΔHTH : le peptide est représenté en barres de couleurs atomiques (C en gris, N en bleu et O en rouge). Les résidus de NprRΔHTH impliqués dans l'interaction avec le peptide sont représentés en barres.
Les protomères NprRΔHTH sont tracés et colorés selon le code couleur de la FIGURE 58 p.141. Les protomères NprRΔHTH sont représentés sous forme de dessin animé sur un fond de surface transparent et colorés par chaîne.

Conformation inhibiteur de la sporulation
- Cristallisation et données de diffraction
- Expérience de SAXS
- Analyse par mutagénèse dirigée
- DISCUSSION ET PERSPECTIVES
Analysis of the crystal structure of the NprR(∆HTH)/NprX complex showed that the peptide binds in a deep cleft in the middle of the TPR domain of the protein ( Figure 4A ). Crystallization conditions of the 1/10 NprR(∆HTH)/NprX complex were determined at 18°C by screening commercial crystallization kits using a nanodrop crystallization Cartesian robot (Genomics Solutions, UK). An initial model covering approximately 80 % of the NprR(∆HTH) sequence was built automatically using BUCCANEER [47].
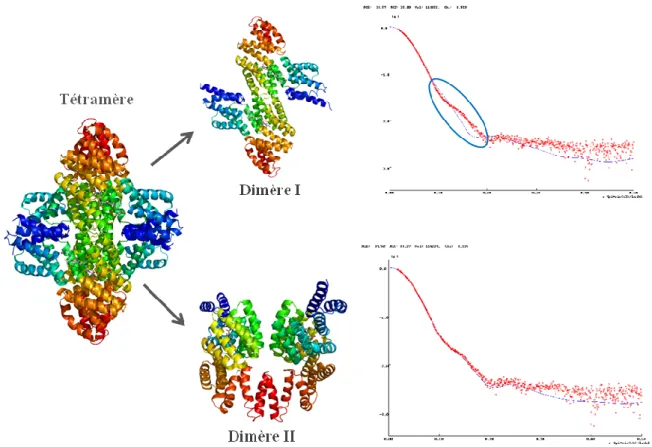
C – Superposition of the type II NprR(∆HTH) dimer shown as a chain-colored plot according to Figure 2D with the 3 best apo NprR(∆HTH) models produced by Dadimodo (see M&M) shown as a gray plot and area . D – Fit of the experimental SAXS curve of apo NprR(∆HTH) (in blue) to the calculated curve obtained with the best model generated by Dadimodo (in red).
La fonction facteur de transcription
- Mode de fixation du peptide de signalisation
- Mode de régulation transcriptionnelle
La fonction Inhibiteur de la sporulation
- Un mode de dimérisation commun aux Rap
- Le domaine RAP
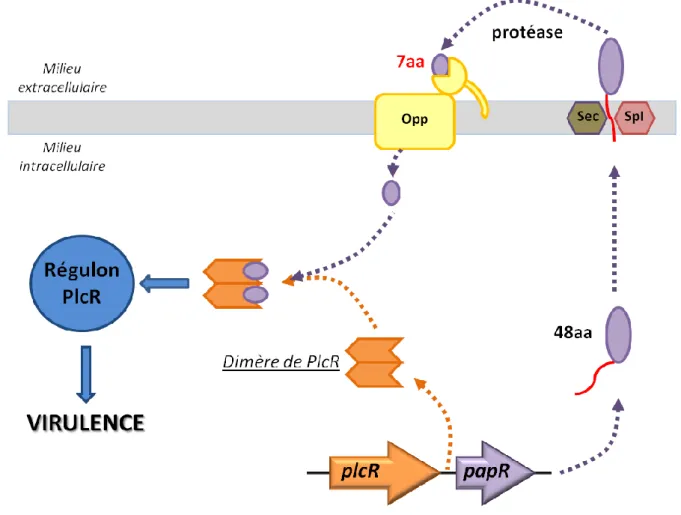
La famille RNPP : évolution et classification
Reconnaissance et spécificité du système NprR/NprX
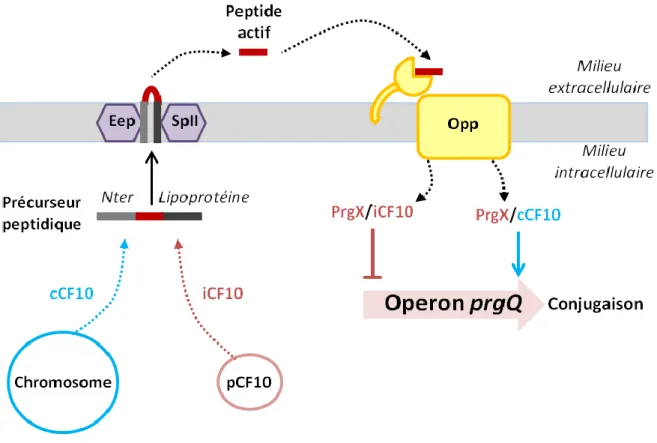
- Export et maturation du peptide NprX
- Phérogroupes et activation croisée
- CONCLUSION
- ANNEXES
- BIBLIOGRAPHIE