Universidade Federal de Minas Gerais Instituto de Ciências Biológicas Departamento de Bioquímica e Imunologia
Laboratório de Genética Bioquímica
Caracterização funcional da proteína SmRbx: uma proteína
de
Schistosoma mansoni
similar à proteína RING box
envolvida no processo de ubiquitinação
Débora Naves Santos
Universidade Federal de Minas Gerais Instituto de Ciências Biológicas Departamento de Bioquímica e Imunologia
Laboratório de Genética Bioquímica
TESE DE DOUTORADO
Caracterização funcional da proteína SmRbx: uma proteína
de
Schistosoma mansoni
similar à proteína RING box
envolvida no processo de ubiquitinação
Débora Naves Santos
Orientadora: Glória Regina Franco
Co-orientador: Carlos Renato Machado
Tese apresentada ao Departamento de Bioquímica e Imunologia do Instituto de Ciências Biológicas da Universidade Federal de Minas Gerais para a obtenção da titulação de Doutora em Bioquímica.
Agradecimentos
À Glória, pela oportunidade de desenvolver este trabalho e pela minha formação científica.
Ao Professor Carlos Renato pela orientação nos trabalhos com leveduras.
Aos Professores Sérgio Pena e Andréa Macedo pelas críticas e sugestões nas reuniões de laboratório.
Ao Professor Francisco Nóbrega e ao seu aluno José Humberto Machado Tambor pela grande ajuda nos experimentos de esporulação e micromanipulação de leveduras.
Aos professores Carlos Rosa, Elida Rabelo e Carlos Salas pelo enriquecimento do trabalho no processo de Qualificação.
Ao Professor Carlos Rosa também pela ajuda nas várias tentativas de esporulação de leveduras.
Ao Professor Marcelo Bemquerer e Jamil pelo sequenciamento da proteína produzida.
Ao aluno de Doutorado Mário Giusta pela fundamental contribuição na manipulação dos camundongos.
Aos amigos do Laboratório de Genética Bioquímica: cada um sabe o quanto foi fundamental no desenvolvimento do trabalho, mas agradeço especialmente pelo carinho, pelas conversas, pelo apoio moral, pelos almoços em boa companhia, enfim, por todos os momentos que tornaram estes anos de LGB muito prazerosos. Adoro vocês!
A todos os amigos do Departamento de Bioquímica e Imunologia, companheiros de trabalho e lazer.
Aos Amigos de Bases por compartilharem os conhecimentos teóricos e estarem sempre disponíveis para ajudar na prática.
Aos eternos e insubstituíveis amigos da PUC: Simone, Maciel, Leomil e Raul.
Ao Pedro, inicialmente meu aluno de Iniciação Científica, que contribuiu enormemente no desenvolvimento deste trabalho. Agradeço por todo o apoio, pela ajuda a qualquer momento, pelo carinho, mas principalmente por ter me tornado uma pessoa melhor.
À minha família, especialmente minha mãe, meu pai e meu irmão, por tudo.
5
ÍNDICE
Lista de figuras 08
Lista de abreviaturas 10
Resumo 12
Abstract 14
1. Introdução
1.1. A esquistossomose
1.2. O ciclo de vida de S. mansoni
1.3. Genoma do parasito
1.4. Projetos Genoma e Transcriptoma de S. mansoni 1.5. Estudos pós-genômicos em S. mansoni
1.6. O sistema de complementação funcional de leveduras nocaute 1.7. O sistema de duplo-híbrido
1.8. A ubiquitinação de proteínas 1.9. Enzimas E3 ubiquitina ligases
1.10. Domínios RING 1.11. A proteína SmRbx
16 16 18 21 23 25 27 28 30 36 40 43 2. Objetivos
2.1. Objetivo geral
2.2. Objetivos específicos
45 45 45
3. Materiais e Métodos
3.1. O vetor pYEDP 60-2 3.2. O vetor pYTS
3.3. O vetor pAS2.1 3.4. O vetor pGAD424
3.5. O vetor pMAL-c2G
3.6. Sequenciamento do cDNA de SmRbx e caracterização gênica e protéica in silico
3.7. Disrupção do alelo do gene HRT1 de levedura por recombinação homóloga
3.8. Clonagem do cDNA de SmRbx no vetor pYEDP 60-2
3.9. Transformação de S. cerevisiae HRT1+/- com pYEDP?SmRbx
6 (cepa DHY)
3.10. Crescimento de S. cerevisiae DHY em meio mínimo contendo galactose
3.11. Esporulação de S. cerevisiae DHY
3.12. Obtenção de leveduras haplóides HRT1 complementadas por SmRbx (cepa NHY)
3.13. Análise morfológica das leveduras complementadas 3.14. Sensibilidade às variações de temperatura das leveduras
complementadas
3.15. Curva de crescimento de leveduras NHY
3.16. Mapeamento das regiões essenciais para a função de SmRbx 3.17. Interação in vivo entre SmRbx e yCul1 utilizando o sistema de
duplo-híbrido em leveduras
3.18. Expressão e purificação da proteína SmRbx
3.19. Digestão da proteína de fusão MBP-SmRbx com enzima Genenase I
3.20. Produção de anticorpos policlonais anti-SmRbx 3.21. Western blot
59 59 60 61 61 64 64 67 71 75 75 76 4. Resultados
4.1. Caracterização in silico da proteína SmRbx
4.2. Obtenção da cepa de S. cerevisiae HRT1+/- contendo o plasmídio recombinante SmRbx/pYEDP (cepa DHY) 4.3. Superexpressão de SmRbx em leveduras DHY
4.4. Obtenção de leveduras haplóides deletadas para HRT1 e complementadas por SmRbx (cepa NHY)
4.5. Caracterização da cepa NHY
4.6. Identificação de regiões essenciais para a função de SmRbx por complementação funcinal da cepa NHY
4.7. Verificação da interação de SmRbx e Cul1 pelo sistema do duplo híbrido de leveduras
4.8. Expressão, purificação e digestão da proteína de fusão MBP-SmRbx
4.9. Produção de anticorpos policlonais anti-SmRbx
7
5. Discussão 111
6. Conclusão 120
7. Perspectivas 122
8. Referências bibliográficas 123
8
Lista de Figuras
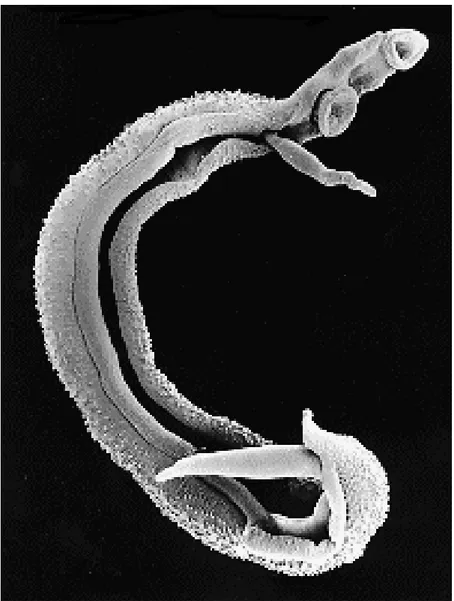
Figura 1: Casal de vermes adultos de S. mansoni 17
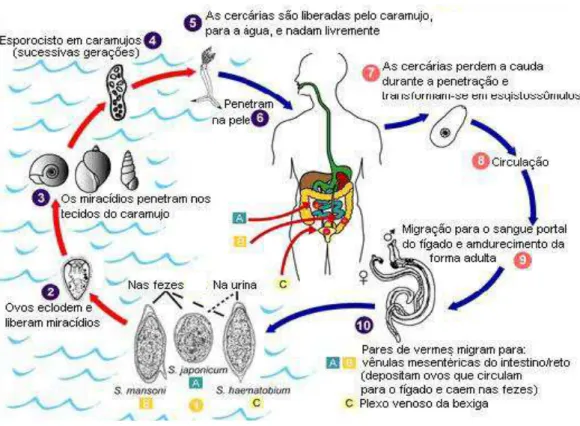
Figura 2: Ciclo de vida dos parasitos do gênero Schistosoma 19
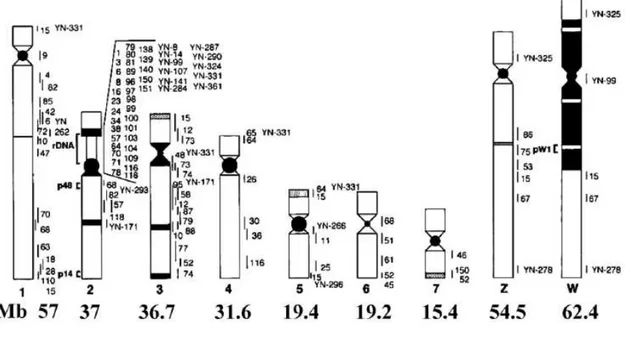
Figura 3: Desenho esquemático dos cromossomos de S. mansoni 22
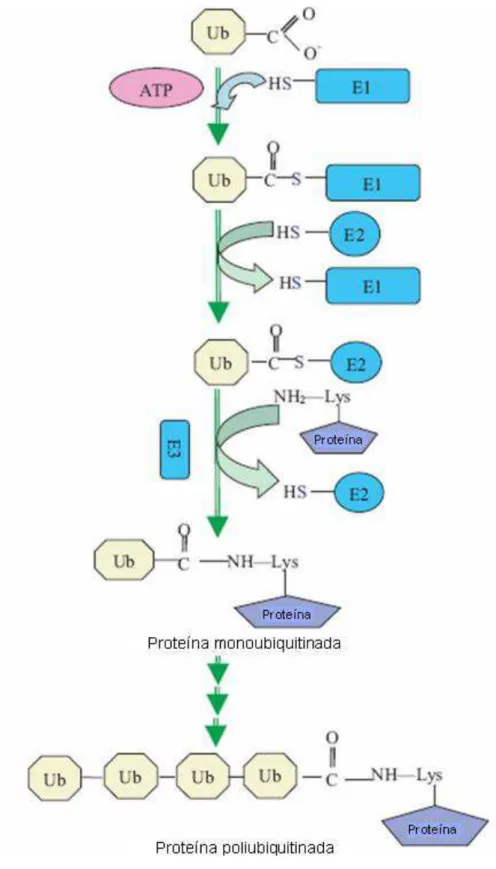
Figura 4: Esquema da via de ubiquitinação 33
Figura 5: Estrutura 3D obtida a partir do cristal do complexo SCF de
humanos 39
Figura 6: Regulação da atividade ligase do complexo SCF através de
CAND1/NEDD8 41
Figura 7: Sequência nucleotídica do gene SmRbx com sua seqüência de
aminoácidos deduzida 80
Figura 8: Alinhamento entre as seqüências de aminoácidos das
proteínas SmRbx, Rbx1 e HRT1 82
Figura 9: Modelo estrutural da proteína SmRbx 83
Figura 10: Estratégia para confirmação da deleção de um alelo de HRT1
de levedura pela inserção do cassete de His3 86
Figura 11: Verificação da toxicidade de pYEDP/SmRbx para a levedura
DHY 89
Figura 12: Crescimento dos esporos após micromanipulação das tétrades obtidas por esporulação da cepa DHY: obtenção da cepa NHY 91
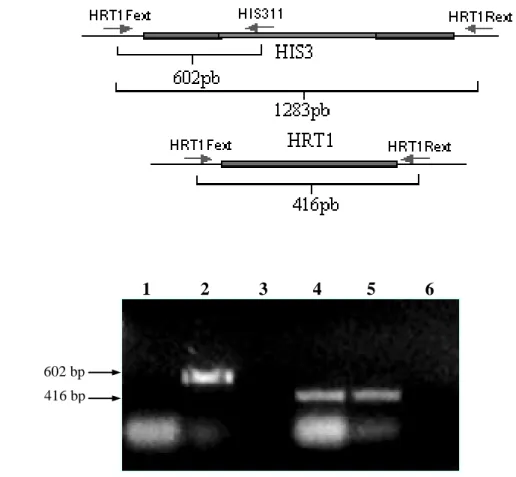
Figura 13: PCR de colônias de leveduras haplóides, obtidas após crescimento dos esporos em meio contendo glicose, utilizando iniciadores para o gene HRT1
92
Figura 14: PCR de colônias de leveduras haplóides, obtidas após crescimento dos esporos em meio contendo galactose, utilizando iniciadores para o gene HRT1
93
Figura 15: PCR de colônias de leveduras haplóides, obtidas após crescimento dos esporos em meio contendo galactose, utilizando iniciadores para o gene HRT1 com a inserção do cassete de HIS3
94
Figura 16: PCR de colônias de leveduras haplóides, obtidas após crescimento dos esporos em meio contendo galactose, utilizando iniciadores para o gene SmRbx
9
Figura 17: Análise morfológica de leveduras NHY 96
Figura 18: Análise da sensibilidade da cepa NHY às variações de
temperatura 98
Figura 19: Curva de crescimento de leveduras NHY e leveduras
selvagens 99
Figura 20: Efeitos da deleção de SmRbx na complementação funcional
de leveduras NHY 101
Figura 21: Interação de SmRbx com yCul1 pelo ensaio de duplo-híbrido 103
Figura 22: Ensaio da atividade da enzima -galactosidase 104
Figura 23: Expressão da proteína SmRbx em fusão com MBP (seta) em grande escala com o sistema de expressão pMAL-c2G 106
Figura 24: Purificação da proteína MBP-SmRbx por cromatografia de
afinidade 107
Figura 25: Digestão da proteína MBP-SmRbx com enzima Genenase I 109
Lista de Tabela
10
Lista de abreviaturas
3 AT: 3-amino-1,2,4-triazole
APC: anaphase-promoting complex
ATP: adenosine trifosfato
BCIP: 5-bromo-4-chloro-3-indolyl phosphate
BLAST: Basic Local Alignment Search Tool
CAND1: cullin-associated e neddylation-dissociated-1
CDK: cinases dependentes de ciclinas
cDNA: Seqüência de DNA complementar
dbEST: banco de dados de ESTs
DNA: Ácido desoxirribonucléico
dNTP:Desoxirribonucleotídeo 5‟ fosfato DTT: dithiothreitol
DUB: proteína desubiquitinadora
E1: enzima ativadadora da ubiquitina
E2: enzima conjugadora de ubiquitina
E3: enzima ubiquitina ligase
EDTA: Ácido etilenodiamino tetra-acético (EthyleneDiamineTetrAcetic acid) EST: Etiquetas de Seqüências Transcritas (Expressed Sequence Tags)
GAL4: Ativador transcricional da levedura S. cerevisiae requerido para a expressão de genes que codificam enzimas utilizadas para o catabolismo da galactose
HECT: Homologous to E6-AP COOH Terminal
HRT1: Proteína de S. cerevisiae homóloga à SmRbx
IPTG: Isopropil -D-tiogalactopiranosídeo
MBP: Proteína que se liga à maltose
NBT: nitro blue tetrazolium chloride
NEED8: neuronal precursor cell expressed developmentally downregulated protein-8
OD: Densidade óptica
OMS: Organização Mundial de Saúde
ONSA: Organisation for Nucleotide Sequencing and Analysis
11
ORF: Janela aberta de leitura (Open Reading Frame)
PCR: Reação em cadeia da polimerase
PEG: Polietilenoglicol
PVDF: Polifluoreto de vinilideno
Rbx1: Proteína humana homóloga à SmRbx
RING: Really Interesting New Gene
RNAi: RNA interferência
RT-PCR: Reação em cadeia da polimerase por transcrição reversa
SCF: Skp1-Cullin-F-box protein SD: Synthetic Dropout
SDS-PAGE: Eletroforese em gel de poliacrilamida dodecil sulfato de sódio
SUMO: small ubiquitin-like modifier
TIGR: The Institute for Genomic Research
UAS: promotor Upstream Activation Sequence
UBL: polipeptídeo análogo à ubiqitina (ubiquitin like)
VCB: pVHL, elongin B/C
12
RESUMO
14
ABSTRACT
16
1. Introdução
1.1. A esquistossomose
A esquistossomose é uma doença causada por trematódeos digenéticos do gênero Schistosoma da família Schistosomatidae, que compreende espécies que vivem como parasitas no homem e em outros vertebrados. A sintomatologia desencadeada pela doença depende do parasito infectante: Schistosoma haematobium é o causador da esquistossomose urinária, S. mansoni (Figura 1), S. japonicum, S. intercalarum e S. mekongi são os responsáveis pela esquistossomose intestinal e hepatoesplênica (Neves, 1995).
A esquistossomose endêmica é determinada principalmente pela distribuição do hospedeiro intermediário: caramujos aquáticos pulmonados do gênero Biomphalaria. Novos focos da doença surgem principalmente em locais onde foram realizadas a construção de barragens, seguido de um aumento da concentração de pessoas nestas áreas (Stelma et al, 1993).
17
Figura 1: Casal de vermes adultos de S. mansoni
18 Na América Latina, a esquistossomose é causada pela espécie S. mansoni, originária da África, trazida pelo tráfico de escravos. A maior
prevalência da doença é principalmente no Brasil, Suriname, Venezuela e Ilhas do Caribe. Porém, houve uma redução nas prevalências de infecção desde o início da década de 50 no Brasil, sendo que nas décadas de 80 e 90 houve uma redução significativa na mortalidade causada por esquistossomose (Savioli et al, 1997).
1.2. O ciclo de vida do S. mansoni
O ciclo de vida do S. mansoni caracteriza-se pela presença de alternância de fase sexuada no hospedeiro vertebrado, principalmente o homem, e fase assexuada no hospedeiro invertebrado, Biomphalaria, como mostra a Figura 2.
O ovo maduro, eliminado junto com as fezes, eclode ao entrar em contato com a água, liberando uma larva livre natante denominada miracídio. No ambiente aquático, a larva nada ativamente até encontrar o hospedeiro, no qual penetra e se aloja. No Brasil, B. glabrata, B. tenagophila e B. straminea são as três espécies de caramujos susceptíveis ao S. mansoni (Paraense, 2001).
19
Figura 2: Ciclo de vida dos parasitos do gênero Schistosoma. As setas vermelhas indicam os estágios de vida do parasito fora do hospedeiro vertebrado, e as setas azuis os estágios internos. (1) Ovos eliminados nas fezes (ou urina para S. haematobium) liberam miracídios (2)
que penetram o caramujo (3). Miracídios se transformam em esporocistos (4) que dão origem a várias cercarias (5), essas penetram no homem pela pele (6) e transformam-se em esquistossômulos (7). Nessa fase, o parasito atinge a circulação (8) e chega ao sistema porta-hepático onde atinge maturidade sexual (9). Os vermes adultos acasalados (10) migram para veias mesentéricas onde realizam a oviposição.
20 Sob influência da luz e temperatura nas horas mais quentes do dia, centenas de cercarias deixam o corpo do molusco, nadando ativamente até encontrar o hospedeiro vertebrado (no caso, o homem). Para isso, as cercarias apresentam alguns comportamentos típicos, tais como fototropismo positivo (de
modo a se posicionar próximo à lâmina d‟água) e a capacidade de se orientar
positivamente em um gradiente de temperatura (McKerrow & Salter, 2002). Ambos os processos maximizam a chance de encontro com o hospedeiro vertebrado. Uma vez tendo se aproximado o suficiente, as cercarias são estimuladas por ácidos graxos de cadeia média livres (como o ácido linoléico) presentes na pele. Nessa etapa, a cercaria secreta o conteúdo da glândula acetabular que permite tanto a adesão da cercaria, através de uma substância glicoprotéica mucosa, quanto a posterior invasão da pele, através de uma protease homóloga à elastase (McKerrow & Salter, 2002).
Após penetrar na pele do hospedeiro, a cercária perde a sua cauda e sofre uma série de alterações (tais como perda do glicocálix, atrofia das glândulas acetabulares, mudança de um metabolismo aeróbio para anaeróbio dentre outras) (Wilson et al, 1978). A partir desse ponto, a cercária passa a ser chamada de esquistossômulo. O parasito parece se orientar para alcançar os vasos sanguíneos, provavelmente através de um gradiente de L-arginina produzido pelo hospedeiro (Haas et al, 2002).
21 mesentéricas posteriores para a postura dos ovos, que variam de 200 a 2000 por dia. Parte dos ovos do parasito atravessa a parede intestinal e são eliminados junto com as fezes, completando-se assim o seu ciclo de vida. Alguns ovos ficam alojados na mucosa intestinal ou nos capilares do sistema porta, sendo circundados por células características de resposta inflamatória granulomatosa do hospedeiro (revisado por Neves, 1995).
1.3. Genoma do parasito
22
Figura 3: Desenho esquemático dos cromossomos de S. mansoni. Abaixo de cada
23
1.4. Projetos Genoma e Transcriptoma de S. mansoni
O entendimento da biologia do organismo e os mecanismos de resistência a drogas e variação antigênica que determina a fuga do sistema imune do hospedeiro são as principais razões para o estudo do genoma de parasitos. Sendo assim, projetos de descoberta gênica de S. mansoni contribuíram com a descoberta de milhares de novos genes do mesmo.
24 aplicadas na construção de mapas físicos, assim como na caracterização de grandes seqüências genômicas (Zweiger & Scott, 1997).
O trabalho de sequenciamento do genoma expresso de S. mansoni que começou em 1992 foi uma iniciativa do Laboratório de Genética Bioquímica, do Departamento de Bioquímica e Imunologia do Instituto de Ciências Biológicas da Universidade Federal de Minas Gerais (ICB-UFMG), e do Centro de Pesquisas René Rachou (FIOCRUZ). Em 1994, este projeto se tornou um consórcio internacional, incluindo laboratórios na Europa, Ásia, África, Austrália, América do Sul e América do Norte, sendo parcialmente financiado pela Organização Mundial de Saúde OMS/UNDP/World Bank Special Program for Research and Training in Tropical Diseases (TDR) (Franco et al, 2000). Em 1995, esse programa gerou a primeira publicação com 607 ESTs (Franco et al, 1995).
25 Centro de Pesquisas René Rachou - FIOCRUZ; Universidade Federal de Ouro Preto; Universidade Federal de Viçosa; Universidade Federal de Lavras; Universidade Federal de Uberlândia; EMBRAPA Milho e Sorgo.
O projeto de São Paulo é parte de uma rede de pesquisas envolvendo vários laboratórios, com financiamento da FAPESP, denominado projeto ONSA (Organization for Nucleotide Sequencing and Analysis). Em Outubro de 2003 o grupo publicou aproximadamente 163.000 seqüências parciais de transcritos geradas pela técnica ORESTES (Open Reading Frame ESTs), através de amplificação por RT-PCR de baixo rigor do RNA mensageiro de seis estágios do ciclo de vida de S. mansoni (Verjovski-Almeida et al, 2003). Essas seqüências foram agrupadas em 30.980 contigs e estima-se que representem 92% do trasncriptoma do parasito.
Já o projeto de sequenciamento do genoma do parasito propriamente dito está sendo desenvolvido nos EUA e na Inglaterra, em colaboração com a Rede do Projeto Genoma de S. mansoni da Organização Mundial de Saúde/UNDP/World Bank. O projeto é realizado pelo The Institute for Genome Research (TIGR; http://www.tigr.org) em Maryland, EUA, e pelo Sanger
Institute em Cambridge, Inglaterra. Este projeto já está em fase final de montagem do genoma e espera-se sua primeira versão para breve.
1.5- Estudos pós-genômicos em S. mansoni
26 Enquanto dados de seqüência de DNA fornecem a informação necessária para o conhecimento dos genes de um organismo, eles são, na maioria dos casos, insuficientes para o entendimento da função biológica de proteínas codificadas por estes genes, ou para o entendimento da relação dessas proteínas com outras moléculas na célula (Tucker et al, 2001). Um completo entendimento do funcionamento de uma proteína requer informação em vários níveis: sua estrutura, modificações pós-traducionais, atividade bioquímica, interações com outras proteínas, sua localização no organismo e seu papel celular.
27 interesse em sistema heterólogo para análise funcional in vitro e in vivo, permitindo esclarecer o papel biológico e molecular dessas proteínas.
Acredita-se que o estudo funcional de proteínas de S. mansoni levará a um melhor entendimento da biologia do parasito, acarretando em novas possibilidades na pesquisa para o controle e tratamento da esquistossomose. Dessa forma, torna-se evidente a necessidade e importância da análise dos possíveis papéis biológicos de proteínas do verme.
1.6- O sistema de complementação funcional de leveduras nocaute
O sistema de complementação funcional heteróloga de leveduras é utilizado para verificar se proteínas de outros organismos estão envolvidas no mesmo processo biológico que a proteína de levedura, possuindo supostamente a mesma função. Para isto, é necessário fazer a disrupção do gene alvo na levedura, transformar estas leveduras com o cDNA codificador da proteína ortóloga de interesse e, posteriormente verificar a reativação da função defectiva.
28 Esta é chamada recombinação sítio-específica (Brown, 1999). A disrupção de um gene pode ser feita inserindo em seu lugar o cassete de um gene repórter. Dessa forma obtém-se uma marcação para leveduras nocaute, permitindo sua seleção.
Uma linhagem de levedura contendo um gene inativado pode ter seu fenótipo examinado para assinalar uma função para o gene. Uma vez identificada a função gênica associada a algum fenótipo específico, linhagens de levedura nocaute podem ser utilizadas em trabalhos de complementariedade funcional. Se um determinado organismo possui um gene ortólogo ao da levedura, este pode ser introduzido na levedura nocaute para verificação da complementação. Se a levedura reverter ao fenótipo selvagem, pode-se dizer que as duas proteínas apresentam a mesma função biológica.
Os efeitos de algumas inativações gênicas são muito sutis e podem não ser reconhecidos quando o fenótipo é examinado. Talvez este gene seja dispensável. Seu produto gênico pode estar envolvido em alguma função que não seja essencial, ou ter uma função que é duplicada por um segundo gene (Brown, 1999). Por outras vezes, a inativação gênica é muito drástica, podendo ser letal.
1.7 - O sistema de duplo-híbrido
29 enzimas utilizadas para o catabolismo da galactose. A GAL4 consiste de dois domínios distintos e funcionalmente essenciais: um domínio N-terminal que se liga a sequências específicas de DNA (promotor Upstream Activation Sequence- UAS) e um domínio C-terminal contendo regiões ácidas que são necessárias para ativar a transcrição. O sistema se baseia em duas proteínas híbridas contendo partes de GAL4: o domínio de ligação ao DNA de GAL4
(vetor pGBT9 ou pAS2.1) fusionado a uma proteína “X” e o domínio de ativação
de GAL4 (vetor pGAD424 ou pAD) fusionado a uma proteína “Y”. No interior da levedura, se X e Y formarem um complexo proteína-proteína, ou seja, um dímero, será reconstituída a proximidade dos domínios de GAL4 e ocorrerá a transcrição de um gene repórter.
30 são conhecidas por estar envolvidas em um processo biológico comum (Auerbach et al, 2002).
1.8 - A ubiquitinação de proteínas
O Prêmio Nobel de 2004 em Química pela descoberta da ubiquitinação de proteínas levou ao reconhecimento da proteólise celular como uma área central de pesquisa. Durante os primeiros estudos no campo da degradação de proteínas, pesquisadores estavam intrigados pela necessidade de ATP neste processo, pois é sabido que a hidrólise da ligação peptídica não requer energia metabólica. A estratégia usada por Hershko e colaboradores em 1979 envolveu o fracionamento de reticulócitos (hemácias diferenciadas sem lisossomos), purificação, identificação de componentes e reconstituição in vitro da degradação protéica citossólica dependente de ATP. Esses estudos foram confirmados por estudos genéticos e dados de outros grupos sobre a função da degradação protéica citossólica in vivo em diferentes processos biológicos (Nandi, 2006).
A ubiquitinação de proteínas é uma importante forma de modificação covalente que regula vários processos biológicos. Além da função de degradação de proteínas via proteassomo 26S, a ubiquitina apresenta outras funções. Dependendo do tipo de ubiquitinação, surgem funções não
tradicionais como: regulação do reparo de DNA (Huang & D‟Andrea, 2006);
31 Staub & Rotin, 2006); controle do ciclo celular (Reed, 2003); sinalização celular (Haglund & Dikic, 2005) e ainda processamento de antígenos (Ciechanover & Iwai, 2004; Varshavsky, 2005).
O processo de ubiquitinação consiste na marcação de proteínas no citosol de células eucarióticas com uma proteína pequena de 76 aa denominada ubiquitina para posterior degradação pelo proteassomo 26S das proteínas poliubiquitinadas (Hershko et al, 1980). O processo de ubiquitinação é composto por uma cascata de três enzimas, envolvendo as enzimas ativadadoras da ubiquitina (E1), as enzimas conjugadoras de ubiquitina (E2), e as enzimas E3 ubiquitina ligases. As enzimas E1 e E2 são responsáveis por ativar e conjugar as moléculas de ubiquitina, respectivamente, enquanto as enzimas E3 são importantes para transferir as moléculas de ubiquitina da enzima E2 para o substrato reconhecido para degradação (Pickart, 2001; Petroski & Deshaies, 2005). Uma nova classe de enzimas tem sido sugerida para estar envolvida no processo de poliubiquitinação: a enzima E4 (Ufd2 em S. cerevisiae), contendo um motivo U-box (Koegl et al, 1999; Hoppe, 2005). Interessantemente, leveduras S. cerevisiae nocaute em Ufd2 falham na proteólise de substratos (Koegl et al, 1999).
32 similar é formada entre a região C-terminal da ubiquitina com o grupo -amino da lisina de outra molécula de ubiquitina para formar uma cadeia poliubiquitinada (Pickart, 2001; Weissman, 2001; Ciechanover & Iwai, 2004). Em algumas proteínas, por exemplo, que não contenham lisina, a poliubiquitinação deve ocorrer no resíduo N-terminal (Ciechanover & Ben-Saadon, 2004). O resíduo de lisina no qual a poliubiquitinação ocorre é importante. Por exemplo, formação de cadeia de poliubiquitina por ligação em Lys-48 e Lys-29 da ubiquitina deve funcionar como um sinal para degradação mediada pelo proteassomo, enquanto ubiquitinação em outros resíduos de lisina (Lys-63) deve funcionar como sinal para reparo de DNA, ativação de fatores de transcrição, etc. (Weissman, 2001). Por outro lado, mono-ubiquitinação de proteínas tem outras funções como endocitose, regulação de histonas, etc. (Hicke, 2001). Também foi demonstrado recentemente que o resíduo N-terminal da proteína alvo serve como um motivo de reconhecimento e ligação para a ubiquitina, entretanto, ubiquitinação subseqüente ocorre no resíduo de lisina interno (Ciechanover & Ben-Saadon, 2004).
33
34 São conhecidas poucas enzimas E1 envolvidas na ubiquitinação em células de mamíferos (10 em humanos) (Hicke et al, 2005). Entretanto, pelo menos três outras enzimas E1 estão envolvidas na conjugação de proteínas análogas à ubiquitina (Schwartz & Hochstrasser, 2003; Staub, 2004). Existem 11 enzimas E2 em leveduras e, pelo menos, 100 em humanos. Enzimas E3 possuem uma variabilidade ainda maior: 54 em leveduras e cerca de 1.000 em humanos (Hicke et al, 2005). Isto indica que a ubiquitinação apresenta um importante papel na regulação celular, assim como a fosforilação. Através da especificidade mediada por E2 e E3, a ubiquitinação regula e elimina proteínas específicas enquanto deixa outras intactas. Essa regulação específica da
degradação protéica permite as células regularem “decisões” celulares de
forma dinâmica (Varshavsky, 2005).
A regulação da ubiquitinação conta também com proteínas desubiquitinadoras (DUBs) que revertem a ubiquitinação. Enzimas DUBs têm um papel negativo na regulação do nível de proteínas ubiquitinadas dentro da célula. Não se sabe se todos os substratos podem ser alvos destas enzimas. É mais provável que funções não proteolíticas de ubiquitinação sejam reversíveis requerendo a função de DUBs (Kim, 2003; Amerik & Hochstrasser, 2004).
Mudanças no sistema de ubiquitinação relacionadas à idade afetam vários tecidos diferentes. Muitos tecidos são objetos de intensas investigações sobre a atividade e sobre componentes do sistema de ubiquitinação (Grillari et al, 2006).
35 muito similares à cascata de ubiquitinação, incluindo enzimas E1, E2, E3 e desubiquitinadoras (Schwartz & Hochstrasser, 2003; Staub, 2004). A conjugação de UBLs às proteínas alvo está envolvida em várias atividades celulares, a saber reparo de DNA, controle do ciclo celular, transporte nuclear e resposta a infecções virais (Gill, 2004). NEED8 (Rub1 em S. cerevisiae) não é um sinal direto para a degradação, mas na regulação de ligases E3 da via de ubiquitinação (Welchman et al, 2005).
Uma reviravolta no conceito do processo de degradação protéica surgiu durante um estudo sobre a degradação do supressor tumoral p53. Foi visto que p53 também está propenso à degradação independente de ubiquitina pelo proteassomo 20S, sugerindo que algumas proteínas apresentam instabilidade inerente. Além disso, foi relatado que este processo de degradação é mediado pelo proteassomo 20S e inibido pela NADH ubiquinona oxidorredutase 1 (enzima mitocondrial da cadeia de transporte de elétrons). Essas descobertas recentes e outras observações de degradação independente de ubiquitina sugerem a existência de mecanismos não relacionados à ubiquitina para a degradação de proteínas via proteassomo nas células (Asher & Shaul, 2006).
1.8.1 - A ubiquitinação de proteínas em S. mansoni
36 Processos como a síntese e degradação protéica, modificações pós-traducionais e processamento de proteínas, bem conhecidas em organismos eucarióticos, provavelmente ocorrem da mesma maneira nesse parasito. No entanto, a investigação de semelhanças e diferenças nesses processos é crucial para o possível desenvolvimento de drogas mais eficientes para o controle da esquistossomose.
Com relação ao processo de ubiquitinação e degradação protéica, alguns avanços foram feitos para o S. mansoni, como relatado a seguir. Um clone de cDNA de S. mansoni codificando para uma proteína homóloga a proteína ligante de multiubiquitina S5a foi caracterizado (Harrop et al, 1999). Outro gene, SmSINA, sugerido como sendo um regulador da ubiquitinação e degradação de receptores nucleares induzida por ubiquitina em S. mansoni também foi caracterizado (Fantappie et al, 2003). Recentemente, a presença do proteassomo 26S em S. mansoni foi inferida baseada na existência de um sistema proteolítico cuja atividade é estimulada por ATP e ubiquitina e reduzida por inibidores de proteassomo altamente específicos (Guerra-Sa et al, 2005).
1.9- Enzimas E3 ubiquitina ligases
37 antes de alcançar o proteassomo, devido à ação das enzimas deubiquitinadoras (Pickart, 2000).
Existem duas classes principais de ligases E3: RING e HECT. Nas ligases E3 que possuem domínio RING (Really Interesting New Gene) a ligase liga-se ao substrato e interage com E2 promovendo a transferência direta da ubiquitina para a proteína alvo (Ho et al, 2006). Ligases HECT (Homologous to E6-AP COOH Terminal) também interagem com enzimas E2, entretanto, a
ubiquitina é primeiramente transferida para uma cisteína conservada no domínio HECT e então a enzima E3 ubiquitina a proteína alvo (Huibregtse, 1995).
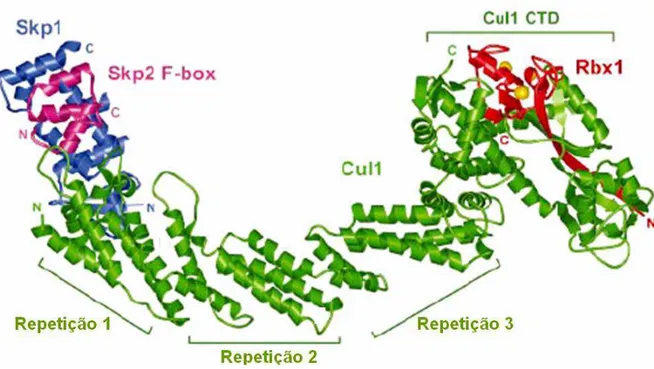
Três tipos de multisubunidades E3 são conhecidos, nas quais uma pequena proteína RING é um componente essencial: SCF (Skp1-Cullin-F-box protein), APC (anaphase-promoting complex) e VCB (pVHL, elongin B/C). Os complexos SCF e VCB possuem uma proteína RING conhecida como Rbx1 (ou Hrt1/Roc1) que interage fortemente com uma proteína da família culina (Cul1/Cdc53 em SCF E3s e Cul2 em VCB) (Ohta et al, 1999). Rbx1 tem um papel importante no recrutamento da enzima E2 (Tan et al, 1999; Seol et al, 1999). O complexo APC também tem uma proteína RING, a Apc11 (Page & Hieter, 1999).
38 leveduras também é parte de um complexo protéico envolvido no processo de ubiquitinação (Kamura et al, 1999; Seol et al, 1999; Skowyra et al, 1999).
A subunidade de VCB que reconhece o substrato é pVHL, o produto do gene supressor de tumor Von Hippel-Lindau (Maxwell et al, 1999). pVHL é recrutado para o complexo através de interações com o adaptador heterodimérico elonguina B/C.
O complexo SCF é assim denominado pela presença das proteínas
Skp1 (163 aminoácidos), Cul1 (776 aminoácidos), um membro da família F-box (~430 a > 1000 aminoácidos) e Rbx1 (108 aminoácidos). Rbx1 e Cul1 formam o centro catalítico do complexo que recruta uma E2. A subunidade variável do complexo, a proteína F-box, liga o substrato. Skp1 funciona como um adaptador que liga a proteína F-box à Cul1 (Zheng et al, 2002). A estrutura tridimensional determinada a partir do cristal do complexo pode ser observada na Figura 5.
Células humanas expressam 7 diferentes culinas (Cul1, 2, 3, 4A, 4B, 5 e 7) cada uma compondo uma multisubunidade de ubiquitina ligase. O complexo SCF é formado pela Cul1, mas outros complexos possuem subunidades distintas (Petroski & Deshaies, 2005).
O componente F-box é responsável pelo reconhecimento de diferentes substratos para ubiquitinação, sendo esta subunidade a mais diversa do complexo SCF. Assim, é possível que existam centenas de diferentes ubiquitinas ligases SCF em células eucarióticas, desenvolvendo um papel de regulação pós-traducional (Ho et al, 2006).
39
Figura 5: Estrutura 3D obtida a partir do cristal do complexo SCF de humanos.
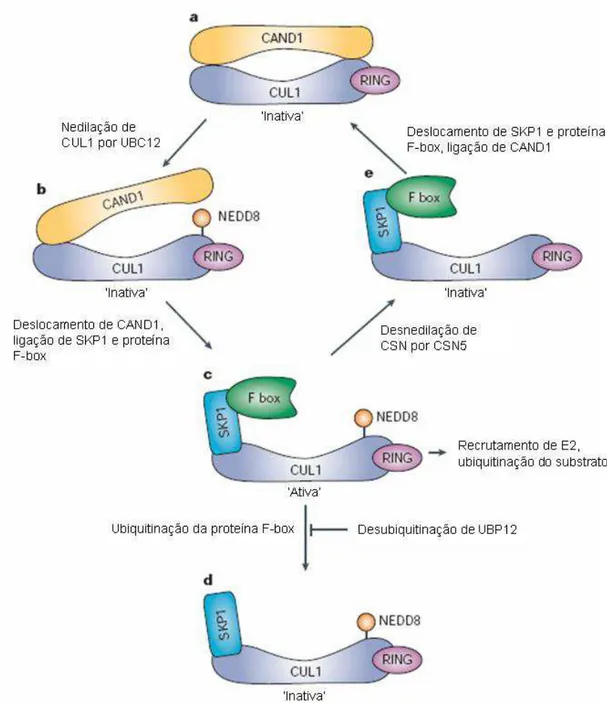
40 SCF através de sua ligação à Cul1, evitando a associação de Cul1 com um inibidor da atividade de SCF, conhecido como CAND1 (cullin-associated e neddylation-dissociated-1) (Cope & Deshaies, 2003; Liu et al, 2002). A regulação das E3 ligases formadas por culina e proteína RING está esquematizada na Figura 6.
O complexo SCF é responsável pela ubiquitinação de uma vasta gama de proteínas envolvidas na progressão do ciclo celular, transdução de sinal e transcrição (Zheng et al, 2002). O complexo é requerido para a transição G1/S do ciclo celular (Tyers & Jorgensen, 2000) porque inibidores da transição do ciclo celular, ciclinas e CDK são substratos do complexo SCF (Feldman et al, 1997). Desregulação do sistema proteolítico deve resultar em proliferação descontrolada, instabilidade genômica e câncer. Por esse motivo, mutações em proteínas do complexo SCF estão relacionadas com várias manifestações de neoplasias (Minella & Clurman, 2005; Nakayama & Nakayama, 2006).
Kus e colaboradores, 2004, reconstituíram e purificaram 12 novos complexos SCF e exploraram a capacidade destes complexos de se ligarem a 5 diferentes enzimas E2: Ubc1, Cdc34, Ubc4, Ubc8 e Ubc11. Foi observado que cada enzima E2 tem especificidade por um tipo de E3. A interação entre SCF e componentes E2 da maquinaria de ubiquitinação pode permitir uma maior diversificação do papel de SCF in vivo.
1.10- Domínios RING
41
Figura 6: Regulação da atividade ligase do complexo SCF através de CAND1/NEDD8.
Ciclos de ligação e remoção de NEDD8 podem ser importantes na regulação da atividade de enzimas Culina-RING-ligases (CRL). (a) Quando o cerne enzimático de SCF está acoplado a CAND1, este fica retido no estado inativo. (b) O fator de conjugação NEDD8, que possivelmente atua em conjunto com fatores desconhecidos, resulta na dissociação de CAND1. (c) Isso pode, por sua vez, causar a montagem de um módulo de reconhecimento de substrato SKP1–F-box no cerne enzimático de SCF. O complexo ativo resultante pode ubiquitinar substratos ligados. (d) A interrelação entre a enzima desubiquitinadora UBP12 ligada a CSN e a maquinaria de ubiquitinação pode modular a abundância de substrato regulando a estabilidade da proteína F-box. Assim que uma proteína F-box é ubiquitinada e degradada, uma nova proteína F-box pode se re-associar com SCF. (e) Alternativamente, NEDD8 pode ser destacada da culina-1 (CUL1) através da atividade isopeptidase da subunidade CSN5 de CSN, que por sua vez, pode levar à dissociação de SKP1 e seqüestro de CUL1 por CAND1.
42 se ligam a dois átomos de zinco usando um arranjo cruzado único. A ligação ao zinco é necessária para o enovelamento do domínio e, consequentemente para sua ação biológica (Borden, 2000).
Não há características comuns aparentes entre membros da família RING em termos de funções, distribuição subcelular ou organismos nos quais estas proteínas são encontradas (Borden, 1998). Estas proteínas são encontradas mediando diversos processos celulares. Dentre eles pode-se citar: progressão do ciclo celular, oncogênese, apoptose, desenvolvimento e infecção viral, repressão da transcrição e ubiquitinação. Várias proteínas RING estão associadas a doenças humanas. Em geral, mutações que alteram a integridade de RING resultam na disrupção dos complexos macromoleculares formados por RING e conseqüentemente, os processos nos quais estes complexos estão envolvidos. Freqüentemente estas disrupções têm efeitos desastrosos no destino da célula (Borden, 2000). A família de proteínas RING é definida pela presença da seqüência padrão C1-x2-C2-x(9-39)-C3-x(1-3)-H1-x(2-3)
-C4-x2-C5-x(4-48)-C6-x2-C7 onde x pode ser qualquer aminoácido, embora
análises de seqüências indiquem que há preferência por certos resíduos (Freemont, 1993).
Algumas proteínas RING contêm, além do domínio RING, um ou mais domínios que se ligam a zinco conhecidos por B-box seguido por um domínio coiled coil de leucina (BCC). Este subconjunto de RINGs é conhecido como RING B-box coiled coil (RBCC) ou simplesmente RING box. O domínio B-box contém um único motivo de ligação a zinco, definido pela seqüência consenso C1-x2-H1-x7-C2-x7-C3-x2-C4-x5-H2-x2-H3. Embora existam sete resíduos
43 proteína RING box pode conter um ou dois domínios B-box adjacentes. A separação entre os motivos de ligação a zinco entre 2 domínios B-box é altamente conservada entre membros da família, sugerindo que as posições destes motivos possuem uma importância funcional. O espaçamento entre RING e o primeiro B-box varia entre 38 e 40 aminoácidos. A conservação da distância entre os domínios RING e B-box sugere que estes possam interagir, formando um enovelamento integrado (Borden, 1998).
1.11- A proteína SmRbx
Através da triagem de uma biblioteca de cDNA de vermes adultos pelo sistema do duplo-híbrido em leveduras, utilizando o fator de transcrição SMYB1 como isca, foi isolado um cDNA que apresentava homologia com uma proteína RING box de vários organismos. Posteriormente, foi demonstrado que este se tratava de um resultado de interação falso-positivo (Valadão et al, 2002). Apesar disso, houve interesse em estudar esta proteína, tentando caracterizá-la funcionalmente.
A proteína de S. mansoni, SmRbx, é codificada por um cDNA que possui uma janela aberta de leitura (Open Reading Frame - ORF) de 324 pb. Ao compararmos a proteína SmRbx com seu ortólogo humano (Rbx1) verificamos que a semelhança entre as duas proteínas é muito grande, sendo que elas apresentam 89% de identidade. Esta grande similaridade pode sugerir então que essas proteínas apresentam um papel biológico relacionado.
45
2- OBJETIVOS
2.1- Objetivo geral
Estudar funcionalmente a proteína SmRbx utilizando o sistema de complementação funcional em leveduras e o sistema de duplo-híbrido em leveduras.
2.2- Objetivos específicos
Caracterizar in silico o gene e a proteína SmRbx.
Construir uma linhagem de levedura nocaute para HRT1
complementada por SmRbx.
Caracterizar leveduras nocaute para HRT1 complementadas com
SmRbx, analisando sua morfologia, sensibilidade a variações de temperatura e curva de crescimento.
Verificar regiões da proteína SmRbx essenciais para sua função por complementação funcional da levedura HRT1 nocaute.
Verificar se SmRbx é capaz de interagir com Cul1 através do sistema de duplo-híbrido em leveduras.
Identificar genes de S. mansoni similares àqueles codificadores das proteínas do complexo SCF (Cul1 e Skp1) nos contigs genômicos de S. mansoni.
Expressar e purificar a proteína SmRbx.
46
3- MATERIAIS E MÉTODOS
3.1- O vetor pYEDP 60-2
O vetor pYEDP 60-2 (9.257 pb), gentilmente cedido pelo Dr. Francisco Nóbrega, UNIVAP, São Paulo, possui um sítio múltiplo de clonagem posicionado na região 3´ de um promotor que é ativado pela presença de galactose, o GAL10/CYC1. O DNA codificador da proteína de interesse deve ser ligado no sítio múltiplo de clonagem na orientação correta do promotor. O vetor pYEDP 60-2 é capaz de se replicar em Escherichia coli e S. cerevisiae. Ele possui o gene bla de resistência a ampicilina e os marcadores URA3 que permite que leveduras auxotróficas carregando pYEDP 60-2 cresçam em meio faltando uracila e ADE2 que permite que leveduras auxotróficas carregando pYEDP 60-2 cresçam em meio faltando adenina. O mapa do vetor pYEDP 60-2 está esquematizado no Anexo 1.
3.2- O vetor pYTS
47
3.3- O vetor pAS2.1
O vetor pAS2.1 (8.393 pb) (Clontech) possui a seqüência de DNA codificadora do domínio de ligação ao DNA da proteína GAL4 (aminoácidos 1-147). Este vetor possui um sítio múltiplo de clonagem (nucleotídeos 5.970-6.015) posicionado na região 3´ da seqüência do domínio de ligação de GAL4. Para a construção de uma proteína híbrida, o cDNA codificador da proteína de interesse é ligado no sítio múltiplo de clonagem na orientação e janela de leitura corretas. A proteína híbrida é expressa em altos níveis na levedura hospedeira e direcionada ao núcleo devido à seqüência de localização nuclear adicionada ao domínio de ativação. O vetor pAS2.1 é capaz de se replicar em E. coli e em S. cerevisiae. Este vetor possui o gene bla (nucleotídeos 7.403-8.263) de resistência à ampicilina em E. coli e o marcador TRP1 (nucleotídeos 1.884-2.258) que permite que leveduras auxotróficas carregando pAS2.1 cresçam em meio faltando triptofano. O mapa do vetor pAS2.1 está esquematizado no Anexo 3.
3.4- O vetor pGAD424
48 sítio múltiplo de clonagem na orientação e janela de leitura corretas. A proteína híbrida é expressa em altos níveis na levedura hospedeira e direcionada ao núcleo devido à seqüência de localização nuclear de fonte heteróloga adicionada ao domínio de ativação de GAL4. O vetor pGAD424 é capaz de se replicar em E. coli e S. cerevisiae. Este vetor possui o gene bla (nucleotídeos 5.108-4.248) de resistência a ampicilina em E. coli e o marcador LEU2 (nucleotídeos 2.640-1.546) que permite que leveduras auxotróficas carregando pGAD424 cresçam em meio faltando leucina. O mapa do vetor pGAD424 está esquematizado no Anexo 4.
3.5- O vetor pMAL-c2G
O vetor pMAL-c2G apresenta forte expressão induzida pelo promotor tac na presença de IPTG e a proteína de interesse é produzida em fusão com uma proteína que se liga a maltose (MBP), o que proporciona fácil purificação por cromatografia de afinidade em coluna de resina de amilose. Além disso, a proteína de interesse purificada pode ser separada da MBP pela clivagem proteolítica com a enzima Genenase I, que age em um sítio de clivagem
específico (His-Tyr-Glu e His-Tyr-Asp)
49
3.6- Sequenciamento do cDNA de SmRbx e caracterização gênica e
protéica in silico.
O cDNA de SmRbx foi obtido pela então aluna de Doutorado Analina Furtado Valadão através de uma triagem de bibliotecas de cDNAs de vermes adultos (Valadão, 2002). Este cDNA estava clonado no vetor pAD e foi seqüenciado usando os iniciadores ADFwd e ADRev (Tabela 1) em aparelho de sequenciamento automático de DNA MegaBACE 1000 (GE HealthCare), segundo o método descrito por Sanger e colaboradores em 1977. Na reação de sequenciamento foram utilizados 200 ng de DNA, 10 pmol de iniciador e 4 L do kit DYEnamicTM ET dye terminator MegaBACETM (GE HealthCare).
A procura de ORF no cDNA foi feita utilizando o programa ORF Finder (http://ncbi.nlm.nih.gov/projects/gorf). A tradução do cDNA em proteína foi feita através do programa Translate (http://expasy.org/tools/dna.html). Para a análise da estrutura gênica de SmRbx, de Cul1 e Skp1 foi feito um BLASTN (Altschul et al, 1990), utilizando as seqüências de cDNAs destes genes como pergunta contra e os contigs gerados pelo Projeto Genoma de S. mansoni do
Wellcome Trust Sanger Institute
(ftp://ftp.sanger.ac.uk/pub/pathogens/Schistosoma/mansoni/genome/Assembly-v3.1), com um e-value cutoff de -10. O contig que apresentou melhor resultado
50
Tabela 1 – Iniciadores usados para amplificação por PCR.
Iniciador Sequência
ADFwd 5’ CAGGGATGTTTAATACCACATC 3’
ADRev 5’ GCACAGTTGAAGTGAACTTGC 3’
RboxF 5´ GACGAATTCATGGAAGTTGATGATACA 3´
RboxR 5´ GAGCTGCAGAATCACCAGGTCCACT 3´
yHRT1F 5´ GACGGATCCATGAGCAACGAAGTTGAC 3´
yHRT1R 5´ GAGGGTACCTTTTCACCTACCGCATCT 3´
yCulF 5’ GAGGAATTCTCGAAGACTTCCGAGATGCTA 3’
yCulR 5’ GAGCTGCAGTTAAGCAAGGTAAGCATACGA 3’
HRT1F 5’TAGTTCAACATGAGCAACGAAGTTGACAGGATGGATGTTCTTCATTCAA
CGTTTCCCAT 3’
HRT1R 5’TCACCTACCGCATCTTGCTAACTGCCAAGGTTGGTTATCTAGTATCATA
CTGTTCGTATA 3’
HRT1Fe 5’ GCAGAGAACTTTAAAGAG 3’
HRT1Re 5’ TTTACGGGCAATTCATTT 3’
HIS311 5´ AACCCTATACCTGTGTGGA 3´
SmboxF1 5’GACGGATCCATGTCAGAAATGGAAGTT 3’
SmboxF2 5’ GACGGATCCAATAAGAAACGTTCCGAG 3’
SmboxF3 5’ GACGGATCCATAAAAAAGTGGTATGCG 3’
SmboxF4 5’ GACGGATCCGTTGCTTTATGGGCCTGG 3’
SmboxF5 5’ GACGGATCCGTTGTTGATAATTGTGCT 3’
SmboxR1 5’ GAGGGTACCCACTCAGTGGCCATATTT 3’
51 Para analisar a expressão gênica em diferentes estágios do ciclo de vida do parasito, foi feito um BLASTN usando a seqüência do cDNA de SmRbx contra as ESTs de S. mansoni do dbEST (http://www.ncbi.nlm.nih.gov/BLAST).
Para a análise teórica do peso molecular e ponto isoelétrico da proteína SmRbx, foi utilizado o programa ProtParam (http://expasy.cbr.nrc.ca/cgi-bin/protparam). A presença do domínio RING foi observada por pesquisas em três bancos de dados distintos para a análise de domínios protéicos e assinaturas em proteínas: SMART (http://www.smart.ebml-heidelberg.de), PROSITE (http://www.expasy.ch/prosite) e Interpro (http://www.ebi.ac.uk/interpro). Para identificação de possíveis sítios de modificações pós-traducinais foi utilizado o programa PROSITE. Para identificar sinais na seqüência primária da proteína capazes de determinar sua localização celular, foi utilizado o programa Psort (http://psort.nibb.ac.jp).
Com o objetivo de comparar a estrutura tridimensional de SmRbx com seu correspondente humano, foi feita uma modelagem computacional por
homologia, utilizando o programa SwissModel
52
3.7- Disrupção do alelo do gene HRT1 de levedura por recombinação
homóloga
3.7.1- Amplificação do DNA codificador da proteína HIS3
Na disrupção de um dos alelos HRT1, primeiramente, foi amplificado o DNA do marcador auxotrófico HIS3 clonado no plasmídio pDIS, gentilmente cedido pelo Professor Dr. Francisco Nóbrega (UNIVAP, São José dos Campos, SP). Os iniciadores utilizados nesta amplificação (HRT1F e HRT1R) continham em suas extremidades 40 nucleotídeos da seqüência de HRT1 de S. cerevisiae (em azul – Tabela 1) e 20 nucleotídeos da seqüência de HIS3 (em preto –
Tabela 1).
Nessa reação foram utilizados 0,2 M de cada iniciador, 200 M de cada dNTP, 2,5 U de Taq DNA polimerase (Phoneutria), tampão de reação 1B (Phoneutria) (Tris-HCl 10mM pH 8,4, KCl 50mM, 0,1% Triton X-100, MgCl2 1,5
mM) e 10 ng do vetor pDIS contendo o DNA de HIS3, em um volume final de 50 L. Para a reação de PCR foi utilizado o seguinte programa:
desnaturação a 96°C por 5 minutos
25 ciclos de: desnaturação a 95°C por 1 minuto
anelamento dos iniciadores a 55°C por 1 minuto extensão a 72°C por 1 minuto
extensão a 72°C por 1 minuto
53 especificação do fabricante, e dosado em gel de agarose 1% corado com brometo de etídio.
3.7.2- Transformação e recombinação homóloga em S. cerevisiae
diplóide
Na disrupção de um dos alelos HRT1 foi utilizada a cepa de S. cerevisiae diplóide construída a partir das linhagens haplóides BY4727 (MAT
his3200 leu20 lys20 met150 trp163 ura30) e BY4736 (MATa ade2::hisG his3200 met150 trp163 ura30) (Brachmann et al, 1998) produzindo uma linhagem BY4727/36 (MATa/MAT his3200 met150 trp163 ura30). Leveduras BY4727/36 foram crescidas por 16 horas a 30°C em meio YPD (peptona 2%, extrato de levedura 1%, glicose 2%, pH 7,0) sob agitação. Foi feita uma diluição das células em meio YPD e o crescimento das leveduras foi acompanhado até atingirem DO600 de 1,0. As leveduras foram centrifugadas
por 5 minutos a 5.000 rpm em centrífuga Hitachi, lavadas com H2O e
centrifugadas novamente por 2 minutos a 5.000 rpm. As células foram ressuspendidas em 100L de H2O. Foram transferidos 50L para um tubo
54 ressuspensão das células foi feita em 1 mL de H2O estéril. Dentro da levedura
ocorre um processo de recombinação gênica entre o produto da PCR contendo o gene de HIS3 e um dos alelos do gene HRT1 da levedura. Dessa forma é feita a disrupção de um dos alelos de HRT1 e a inserção do gene repórter HIS3, interrompendo a fase de leitura deste gene. As leveduras transformadas
foram crescidas em meio SD +Met +Ura +Trp + Gli. Os clones obtidos foram submetidos à PCR de colônia de levedura para confirmar a inserção do cassete de HIS3. Para isso, foram feitas duas amplificações: na primeira foram utilizados iniciadores externos à região codificadora do cDNA de HRT1 (HRT1Fextn e HRT1Rextn – Tabela 1). Na segunda amplificação, foi utilizado um iniciador externo à região codificadora do cDNA de HRT1 (HRT1Fextn) e um iniciador que se anela em uma região específica do gene HIS3 (HIS311 –
Tabela 1).
55
3.8- Clonagem do cDNA de SmRbx no vetor pYEDP 60-2
3.8.1-Amplificação do cDNA de SmRbx
O cDNA de SmRbx foi amplificado do vetor pAD, onde havia sido previamente clonado, utilizando os iniciadores RboxF e RboxR (Tabela I). Estes iniciadores contêm em suas extremidades 5´ sítios para as enzimas de restrição EcoRI e PstI, respectivamente, e amplificam toda a região codificadora do cDNA.
Nessa reação foram utilizados 0,2 M de cada iniciador, 200 M de cada dNTP, 0,5 U de Taq DNA polimerase (Phoneutria), tampão de reação 1B (Phoneutria) e 10 ng do vetor pAD contendo o cDNA de SmRbx, em um volume final de 10 L. Na reação de PCR foi utilizado o mesmo programa citado na seção 3.7.1. A amplificação de um produto de 351 pb foi confirmada por eletroforese em gel de agarose 1% corado com brometo de etídio. O produto amplificado foi purificado utilizando o Kit WizardTM PCR prep (Promega), conforme especificação do fabricante, e dosado em gel de agarose 1% corado com brometo de etídio.
3.8.2- Clonagem de SmRbx no vetor pCR2.1-TOPO
O produto de amplificação de SmRbx, utilizando os iniciadores RboxF e RboxR, foi ligado ao vetor pCR2.1-TOPO (Invitrogen). Este é um T-vector, ou seja, é linearizado com uma enzima que corta o DNA em extremidades cegas
e, posteriormente, é adicionado um nucleotídeo de timina nas extremidades 3‟
deste vetor linearizado. Desta forma, é possível ligar produtos de PCR, pois
56 naturalmente adicionado pela Taq DNA polimerase. A ligação foi feita com 2 L do produto de PCR, utilizando o kit de clonagem TOPO TA Cloning (Invitrogen).
3.8.3- Transformação de bactérias E. coli DH5 eletrocompetentes
com o plasmídio pCR2.1-TOPO/SmRbx, seleção, obtenção e
sequenciamento dos plasmídios recombinantes
O produto de ligação foi utilizado na transformação de bactérias E. coli DH5 eletrocompetentes. Estas bactérias (50 L) foram incubadas com 3 L de cada produto de ligação por 5 minutos, em gelo. Como controle positivo da transformação, foi feita uma incubação com 10 ng do vetor pUC18 intacto. Como controle negativo da transformação, as células DH5 foram incubadas sem plasmídios. Após a incubação, as bactérias foram submetidas a uma descarga elétrica de 3.400 volts por 2,5 milisegundos em eletroporador Micro PulserTM (BIORAD). Após o choque elétrico, foram adicionados 250 L de meio
57 351 pb foi confirmada por eletroforese em gel de agarose 1% corado com brometo de etídio. Os clones positivos foram crescidos em 3 mL de meio 2xYT suplementado com ampicilina. O DNA plasmidial dos clones selecionados foi extraído utilizando o kit WizardTM Mini prep (Promega) e seqüenciado em aparelho de sequenciamento automático de DNA MegaBACE 1000 (GE HealthCare), segundo o método descrito por Sanger e colaboradores em 1977. Na reação de seqüenciamento foram utilizados 200 ng de DNA, 10 pmol de iniciador e 4 L do kit DYEnamicTM ET dye terminator MegaBACETM (GE
HealthCare). Os iniciadores utilizados foram RboxF e RboxR.
3.8.4-Construção do plasmídio recombinante pYEDP/SmRbx
O inserto correspondente a SmRbx foi retirado do vetor pCR2.1-TOPO por digestão com a enzima de restrição EcoR1. Nesta reação, 3 g de DNA plasmidial foram digeridos com a enzima EcoR1 (Invitrogen) em tampão React3 (Invitrogen). O vetor de clonagem pYEDP foi digerido com a mesma enzima para permitir posterior ligação ao inserto. Para as digestões foram feitas incubação por 2 horas a 37°C e, posteriormente, foi confirmada por eletroforese em gel de agarose 1% corado com brometo de etídeo. Os produtos digeridos foram extraídos do gel de agarose e purificados por centrifugação em uma coluna Ultrafree-DA (Millipore). A ligação do cDNA codificador da proteína SmRbx ao vetor pYEDP foi feita seguindo-se uma razão molar fragmento/vetor de 2:1 utilizando 6 unidades da enzima T4 DNA ligase (Invitrogen) em tampão de ligação (250mM Tris-HCL (pH7,6), 50mM MgCl2, 5
58
3.8.5- Transformação de bactérias E. coli DH5 eletrocompetentes
com o plasmídio pYEDP/ SmRbx, seleção, obtenção e sequenciamento
dos plasmídios recombinantes
O produto de ligação (3 L) foi utilizado na transformação de bactérias E. coli DH5 eletrocompetentes seguindo metodologia já descrita (seção 3.8.3).
Para confirmar a presença do cDNA de interesse nos clones de bactérias obtidos foi feita uma PCR de colônia utilizando os iniciadores específicos de SmRbx. As bactérias contendo o plasmídio de interesse foram crescidas em 3 mL de meio 2xYT suplementado com ampicilina. Os plasmídios recombinantes foram extraídos utilizando o kit WizardTM Mini prep (Promega). Como foi utilizada apenas uma enzima de restrição no processo de clonagem de SmRbx ao pYEDP, foi necessário confirmar o sentido da ligação do inserto ao vetor. Para isto, inicialmente foram utilizadas enzimas de restrição para análise do perfil de digestão. Foi feita uma primeira digestão com 3g de DNA utilizando 10 unidades da enzima SmaI (Promega) em tampão J (10 mM Tris-HCl, 7 mM MgCl2, 50 mM KCl, 1mM DTT, pH 7,5) (Promega) em volume final de 20 L.
Esta digestão foi feita a 25°C por 2 horas. Após este período, foram adicionadas 10 unidades da enzima XbaI (Stratagene) e tampão Buffer3 (500mM NaCl, 250 mM Tris-HCl, 100 mM MgCl2, 100 mM -mercapetanol, 100
g/mL BSA, pH 7,7) (Stratagene), aumentando-se o volume para 40 L. Esta
59
3.9- Transformação de S. cerevisiae HRT1+/- com pYEDP/SmRbx (cepa
DHY)
Plasmídios recombinantes pYEDP contendo o cDNA de SmRbx foram utilizados na transformação de leveduras S. cerevisiae com um alelo de HRT1 deletado. Nesta transformação, foram utilizados 2 g de plasmídios, seguindo metodologia já descrita (seção 3.7.2). As leveduras transformadas foram crescidas em meio seletivo SD +Met +Trp +Gli por 5 dias a 30°C. A presença do cDNA codificador da proteína de S. mansoni foi confirmada por PCR de colônia de levedura, segundo procedimento já descrito (seção 3.7.2). A cepa obtida foi denominada DHY.
3.10- Crescimento de S. cerevisiae DHY em meio mínimo contendo
galactose ou glicose
As leveduras DHY foram transferidas para placas contendo meio SD +Met +Trp +Gal, pois a expressão de SmRbx é induzida por galactose, e incubadas a 30°C por 4 dias. Estas leveduras também foram transferidas para placas contendo meio SD +Met +Trp +Gli para comparação do crescimento nas duas condições.
3.11- Esporulação de S. cerevisiae DHY
60 incubadas a 30°C sob agitação por 20 horas. Após incubação, as células foram centrifugadas por 5 minutos a 1500rpm em centrífuga Eppendorf, lavadas em 3 mL de H2O, re-centrifugadas por 5 minutos e plaqueadas em meio KAc (extrato
de levedura 0,5%, acetato de potássio 1%, dextrose 0,05% ágar 2%, pH 5,8). As células foram observadas ao microscópio após 4 dias para verificar a formação das tétrades.
3.12- Obtenção de leveduras haplóides HRT1 complementadas por
SmRbx (cepa NHY)
3.12.1- Micromanipulação dos esporos
61
3.12.2- Genotipagem das leveduras haplóides por PCR
A determinação de quais leveduras haplóides eram nocaute complementadas por SmRbx foi feita por PCR de colônia. Esta cepa foi denominada NHY. A presença de SmRbx foi confirmada utilizando os iniciadores RboxF e RboxR. A presença de HIS3 ou HRT1 foi verificada utilizando 2 pares de iniciadores: HRT1Fext/ HRT1Rext e HRT1Fext/ HIS311. Nessa reação foram utilizados 0,2 M de cada iniciador, 200 M de cada dNTP, 2,5 U de Taq DNA polimerase (Phoneutria), tampão de reação 1B (Phoneutria), em um volume final de 25 L. Nas reações de PCR utilizou-se programa descrito na seção 3.7.1. A amplificação foi confirmada por eletroforese em gel de agarose 1% corado com brometo de etídio.
3.13- Análise morfológica das leveduras complementadas
Para observação de alterações morfológicas, leveduras NHY foram crescidas em meio YPD contendo galactose a 30°C por 16 horas. Após crescimento, as leveduras foram colocadas em lâminas de vidro e observadas em microscópio ótico Olympus CH20, em aumento de 100 vezes.
3.14- Sensibilidade às variações de temperatura das leveduras
complementadas
3.14.1- Amplificação do DNA de HRT1
62 (Tabela 1). Estes iniciadores contêm em suas extremidades 5´ sítios para as enzimas de restrição BamHI e KpnI, respectivamente, e amplificam toda a região codificadora do DNA.
Nessa reação foram utilizados 0,2 M de cada iniciador, 200 M de cada dNTP, 0,5 U de Taq DNA polimerase (Phoneutria), tampão de reação 1B (Phoneutria), em um volume final de 25 L. Na reação de PCR foi utilizado o mesmo programa citado na seção 3.7.1. A amplificação de um produto de 369 pb foi confirmada por eletroforese em gel de agarose 1% corado com brometo de etídio. O produto amplificado foi dosado em gel de agarose 1% corado com brometo de etídio.
3.14.2- Construção e obtenção do plasmídio recombinante
yHRT1/pYTS
63 Ultrafree-DA (Millipore). A ligação do DNA codificador da proteína HRT1 ao vetor pYTS foi feita seguindo-se uma razão molar fragmento/vetor de 2:1 utilizando 6 unidades da enzima T4 DNA ligase (Invitrogen) em tampão de ligação (250mM Tris-HCL (pH7,6), 50mM MgCl2, 5 mM ATP, 5 mM DTT, 25%
(p/v) polietilenoglicol 8000) (Invitrogen) em um volume final de 10 L, a 22°C por 4 horas. O produto de ligação (3 L) foi utilizado na transformação de bactérias E. coli DH5 eletrocompetentes seguindo metodologia já descrita (seção 3.8.3). Para confirmar a presença do cDNA de interesse nos clones de bactérias obtidos foi feita uma PCR de colônia utilizando os iniciadores HRT1F e HRT1R. As bactérias contendo o plasmídio de interesse foram crescidas em 3 mL de meio 2xYT suplementado com ampicilina. Os plasmídios recombinantes foram extraídos utilizando o kit WizardTM Mini prep (Promega) e
seqüenciados no aparelho MegaBACE 1000 (GE HealthCare) (seção 3.8.3).
3.14.3- Transformação de S. cerevisiae NHY com HRT1/pYTS
64
3.14.4- Crescimento em meio YPD a 23°C, 30°C e 37°C
Leveduras NHY foram crescidas em meio YPD contendo galactose a 30°C por 16 horas. Após crescimento, as leveduras foram contadas em câmara hematocitométrica em microscópio ótico e cerca de 105 células foram crescidas em placa contendo meio YPD/galactose por 3 dias em temperaturas de 23°C, 30°C e 37°C. Como controle foram utilizadas leveduras NHY + HRT1/pYTS crescidas em meio YPD contendo glicose. Nestas condições, SmRbx não é expresso e o crescimento das leveduras se deve à expressão de HRT1.
3.15- Curva de crescimento de leveduras NHY
Leveduras NHY e leveduras selvagens BY4736 (Brachmann et al, 1998) foram crescidas em meio YPD galactose por 16 horas a 30°C. Após crescimento, as leveduras foram contadas em câmara citométrica e diluídas a 2,4 X 106 células/mL em YPD-galactose. As células foram incubadas a 30°C sob agitação e contadas a cada hora por 14 horas. Os dados obtidos foram utilizados na construção de um gráfico para comparação do crescimento das duas linhagens.
3.16- Mapeamento das regiões essenciais para a função de SmRbx
3.16.1- Amplificação de porções do cDNA de SmRbx
65 diretos contêm em suas extremidades 5´ sítios para as enzimas de restrição BamHI e os iniciadores reversos contêm em suas extremidades 5´ sítios para
as enzimas de restrição KpnI. Foram feitas amplificações utilizando as combinações de iniciadores: SmboxF1/SmboxR1, SmboxF2/SmboxR1, SmboxF3/SmboxR1, SmboxF4/SmboxR1, SmboxF5/SmboxR1, SmboxF1/SmboxR2 (Tabela 1).
Nas reações foram utilizados 0,2 M de cada iniciador, 200 M de cada dNTP, 0,5 U de Taq DNA polimerase (Phoneutria), tampão de reação 1B (Phoneutria), em um volume final de 25 L e o mesmo programa citado na seção 3.7.1. As amplificações foram confirmadas por eletroforese em gel de agarose 1% corado com brometo de etídio. Os produtos amplificados foram dosados em gel de agarose 1% corado com brometo de etídio.
3.16.2- Construção dos plasmídios pYTS-SmRbx,
pYTS-SmRbx18N, pYTS-SmRbx24N, pYTS-SmRbx29N, pYTS-SmRbx38N,
pYTS-SmRbx11C
66 (Millipore). As ligações dos cDNAs codificadores das proteínas de SmRbx contendo regiões deletadas ao vetor pYTS foram feitas seguindo-se uma razão molar fragmento/vetor de 2:1 utilizando 6 unidades da enzima T4 DNA ligase (Invitrogen) em tampão de ligação (Invitrogen) em um volume final de 10 L, a 22°C por 4 horas, obtendo-se as seguintes construções:
pYTS-SmRbx (contendo os nucleotídeos 1-324),
pYTS-SmRbx18N (contendo os nucleotídeos 46-324), pYTS-SmRbx24N (contendo os nucleotídeos 72-324), pYTS-SmRbx29N (contendo os nucleotídeos 81-324), pYTS-SmRbx38N (contendo os nucleotídeos 114-324), pYTS-SmRbx11C (contendo os nucleotídeos 1-291).
Os produtos de ligação (3 L) foram utilizados na transformação de bactérias E. coli DH5 eletrocompetentes seguindo metodologia já descrita (seção 3.8.3). Para confirmar a presença dos fragmentos de interesse nos clones de bactérias obtidos foram feitas PCR de colônia utilizando os mesmos iniciadores com que foram feitas as amplificações dos cDNAs. As bactérias contendo os plasmídios de interesse foram crescidas em 3 mL de meio 2xYT suplementado com ampicilina. Os plasmídios recombinantes foram extraídos utilizando o kit WizardTM Mini prep (Promega) e seqüenciados no aparelho MegaBACE 1000 (GE HealthCare) (seção 3.8.3).
3.16.3- Transformação de S. cerevisiae NHY com SmRbxT/pYTS