DISSERTAÇÃO DE MESTRADO
Clonagem do cDNA codificante para a toxina madura LTx5 da aranha Lasiodora sp no vetor pYES2.1/V5-His-TOPO® e expressão da proteína recombinante.
Universidade Federal de Ouro Preto Núcleo de Pesquisas em Ciências Biológicas Programa de Pós-Graduação em Ciências Biológicas
ii Universidade Federal de Ouro Preto
Núcleo de Pesquisas em Ciências Biológicas Programa de Pós-Graduação em Ciências Biológicas
Clonagem do cDNA codificante para a toxina madura LTx5 da aranha Lasiodora sp no vetor pYES2.1/V5-His-TOPO® e expressão da proteína recombinante.
Thiago Mafra Batista
ORIENTADOR: PROF. DR. IESO DE MIRANDA CASTRO
Dissertação apresentada ao programa de pós-graduação do Núcleo de Pesquisas em Ciências Biológicas da Universidade Federal de Ouro Preto, como parte integrante dos requisitos para a obtenção do título de Mestre em Ciências Biológicas, área de concentração Biologia Molecular
iii Dedicatória
iv Agradecimentos
À CAPES pelo auxílio financeiro.
À Universidade Federal de Ouro Preto, pelo ensino gratuito e de qualidade. À histórica cidade de Ouro Preto, pelos incríveis momentos vividos e que estarão guardados pra sempre em minha memória.
Ao meu orientador Prof. Dr. Ieso de Miranda Castro, pelo exemplo de responsabilidade e seriedade com a ciência.
Ao Prof. Dr. Rogelio Lopes Brandão, botafoguense, exemplo de profissionalismo e comprometimento com a educação.
Ao amigo/irmão de infância, companheiro em todos os momentos, João Victor, só nós dois sabemos como foram esses dois anos, muita história pra contar! Amigo pra vida toda. Fusca azul!
À minha namorada Juliana, por todo carinho, amor e compreensão durante esse tempo longe um do outro, obrigado por tudo, eu amo você!
Aos amigos pensionistas do LBCM, Filippe “Ariscu” Gadiolli e Anderson “Ploust” pelos ótimos momentos de risadas, café na cantina e boas histórias, valeu “dimais” da conta e lembrem-se sempre: “Dápáfazê”!
Aos colegas de pós graduação que se tornaram amigos, em especial Tiago “Listerine”, Larissa, Ramon “Mamona”, Nilza “Banzai”, Roenick, Matheus, Manoel, Eduardo (cruzeirense) e Auffy.
À Zezé Trópia, por todo carinho e por ser a simpatia do LBCM.
À galera do LBCM, doutoras Anamaria e Gilzeane obrigado por tudo, Prof. Dr. Tótola obrigado pelo incentivo e boas idéias, Igor “Água-móli” valeu demais, Carol, Renata, Natália, Érica, Simone, Wesley, Sr. Bráz, enfim, a todos que passaram por aqui, meu muito obrigado!
v À Cida, alma do NUPEB, obrigado por ser tão prestativa e amiga!
Ao professor Dr. Milton Hércules Guerra de Andrade, pelas valiosas idéias e discussões científicas que muito me ajudaram em minha busca pela LTx5. Obrigado!
À professora Dra. Juliana Lopes Rangel Fietto, pela simpatia e por estar sempre bem disposta a ajudar, mesmo que via e-mails.
Ao professor Dr. William Borges, pelas valorosas discussões científicas.
Ao professor Dr. Luiz Carlos Crocco Afonso, botafoguense, obrigado pela ajuda nos experimentos de Western e Dot Blot.
À professora Dra. Renata Guerra de Sá, obrigado pela amizade e carinho.
Ao amigo professor Dr. Luciano Gonçalves Fernandes, outro botafoguense, futuro pescador! Viva o Nag Champa, Peperone!
Ao professor Dr. Leonardo Máximo Cardoso, obrigado pelo Reference Manager e por toda ajuda com as referências bibliográficas.
Ao Sr. Milton, Dona Iraci e toda família, por me acolherem tão bem durante esses 2 anos e 5 meses em Ouro Preto!
E finalmente, à minha família, minha mãe Florentina, meu paiJoão Batista e meu irmão Phillipe, flamenguistas como eu, minha origem, meu passado, meu presente e meu futuro. Amor não se agradece, se valoriza, e vocês são tudo pra mim! Amo vocês!
vi A luta pela vitória nem sempre é vantajosa aos fortes nem aos espertos. Mais cedo ou mais tarde quem cativa a vitória é aquele que crê plenamente: “EU CONSEGUIREI!”
vii Resumo
viii Abstract
ix Índice
RESUMO vii
ABSTRACT viii
LISTA DE ABREVIATURAS xiii
LISTA DE FIGURAS xv
1. INTRODUÇÃO 1
1.1 Aspectos gerais 1
1.2 Estudo de toxinas animais 5
1.3 Toxinas de aranhas 9
1.3.1 Ação na liberação de neurotransmissores 10
1.3.2 Ação em canais de sódio voltagem-dependentes 11
1.3.3 Ação em canais de cloreto 12
1.3.4 Ação em canais de potássio 13
1.3.5 Ação em canais de cálcio voltagem-sensíveis 14
1.4 Estudos com o veneno da aranha Lasiodora sp 15
2. OBJETIVOS 18
2.1 Objetivo geral. 18
2.2 Objetivos específicos. 18
3. MATERIAL E MÉTODOS. 19
3.1 Microorganismos. 19
3.2 Meios de cultura. 19
x
3.2.2 Meio LB-XIA 19
3.2.3 Meio YP 19
3.2.4 Meio seletivo SD 19
3.2.5 Meio 2XYP 20
3.3 Vetor de clonagem e expressão. 20
3.4 Sub clonagem da sequência codificante para a LTx5 no vetor de
expressão pYES2.1/V5-His-TOPO®. 21
3.4.1 PCR para amplificação da sequência codificante para a toxina madura. 22
3.4.2 Purificação do produto de PCR. 23
3.4.3 Ligação do produto de PCR no vetor de clonagem. 23
3.5 Preparo de células competentes de E. coli TOP10F’. 24
3.6 Transformação de bactérias. 24
3.7 PCR das colônias provenientes da transformação de E. coli TOP10F’
com o produto de ligação pYES2.1/V5-His-TOPO-LTx5. 25
3.8 Eletroforese em gel de agarose. 26
3.9 Extração de DNA plasmidial de bactéria. 26
3.10 Digestão do DNA plasmidial com endonucleases de restrição. 27
3.11 PCR para confirmação da orientação do inserto no vetor
pYES2.1/V5-His-TOPO®. 27
3.12 Sequenciamento do DNA. 30
3.12.1 Mini prep para sequenciamento. 30
3.12.2 PCR para reação de sequenciamento. 31
3.12.3 Precipitação da reação de sequenciamento. 32
3.13 Preparo de células competentes de Saccharomyces cerevisiae. 32
3.14 Transformação de leveduras. 33
xi
3.16 Extração de proteínas totais. 34
3.17 Solubilização da fração insolúvel. 34
3.18 Dosagem de proteínas totais. 35
3.19 SDS-PAGE (Laemmli, 1970). 35
3.20 SDS-Tricina-PAGE (Schagger et al., 1987). 35
3.21 Coloração de gel de poliacrilamida por nitrato de prata. 36
3.22 Dot Blot. 36
3.23 Western Blot. 37
3.24 Cromatografia de filtração molecular. 37
3.25 Cromatografia de troca aniônica. 38
3.26 Purificação da proteína recombinante utilizando resina Ni-NTA. 38
3.27 Fracionamento do extrato protéico utilizando sistema Microcon®
YM-50K Millipore. 39
4. RESULTADOS. 40
4.1 Amplificação da região codificante para a toxina madura LTx5. 40
4.2 Clonagem da sequência codificante para LTx5 no vetor de expressão
pYES2.1/V5-His-TOPO®. 41
4.3 Digestão do DNA plasmidial com as endonucleases PvuII e XbaI. 42
4.4 Sequenciamento do DNA. 43
4.5 Análise da sequencia de aminoácidos da LTx5 recombinante. 43
4.6 Expressão da proteína recombinante LTx5 com 2% de galactose. 44
4.7 Dot Blot. 45
4.8 Expressão da proteína recombinante LTx5 com 4% de galactose. 46
4.9 Dot Blot. 47
xii
4.11 Dot Blot em condições desnaturantes. 49
4.12 Tentativa de purificação da proteína recombinante usando a resina
Ni-NTA. 51
4.13 Cromatografia de filtração molecular e cromatografia de troca aniônica. 52
4.14 Tentativa de fracionamento do extrato protéico utilizando sistema
Microcon® YM-50K Millipore. 52
5. DISCUSSÃO. 53
6. CONCLUSÕES. 60
7. COMENTÁRIOS FINAIS. 61
8. BIBLIOGRAFIA. 62
xiii Lista de Abreviaturas
Abs Absorbância
cDNA DNA complementar
CTAB Cetyl Trimethyl Ammonium Bromide (Brometo de Cetil-Trimetilamônio) dNTP’s Desoxiribonucleotídeos
DTT Ditiotreitol
EDTA Ácido Etilenodiamino Tetra Acético
ELISA Enzyme Linked Immuno Sorbent Assay (Ensaio de Imunoabsorvente ligado à enzima)
Gal Galactose
GET Glicose/EDTA/Tris IgG Imunoglobulina G
IPTG Isopropil- -D-thiogalactopiranoside
Lqh Toxina isolada do veneno do escorpião L. quinquestriatus hebraeus LTx Toxina isolada do veneno da aranha Lasiodora sp.
mRNA RNA mensageiro NEM N-Ethilmaleimide NMDA N-metil-D-aspartato
PCR Polymerase Chain Reaction (Reação em Cadeia da Polimerase) PEG Polietilenoglicol
pI Ponto isoelétrico PLA2 Fosfolipase A2
PMSF Phenylmethylsulphonylfluoride (Fenil-Metil-Sulfonil-Fluoreto) RNAse Ribonuclease
xiv TAE Tampão Tris/Acetato/EDTA
TE Tampão Tris/EDTA
TEMED N,N,N',N'-tetramethil-etano-1,2-diamino THP Tris(hidroxipropil)phosphine
tRNA RNA transportador TTX Tetrodotoxina
xv Lista de figuras
Figura 1 - Aranha do gênero Lasiodora sp ...3
Figura 2 – Dendograma das toxinas presentes no veneno da aranha Lasiodora sp ... 16
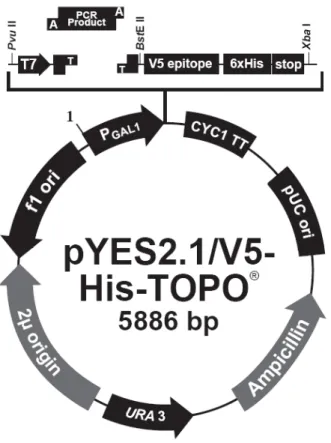
Figura 3 - Mapa do vetor de clonagem e expressão pYES2.1/V5-His-TOPO® ... 20
Figura 4 – Sequencia de nucleotídeos e aminoácidos da LTx5 ... 21
Figura 5 - Desenho esquemático representando a PCR-A ... 28
Figura 6 - Desenho esquemático representando a PCR-B ... 29
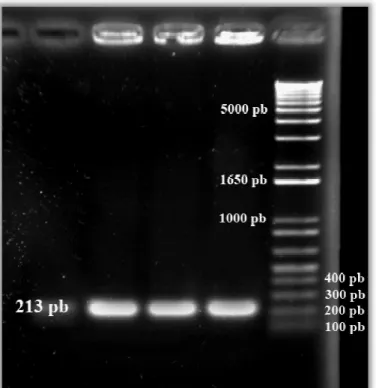
Figura 7 - Amplificação da região codificante para a toxina madura LTx5 ... 40
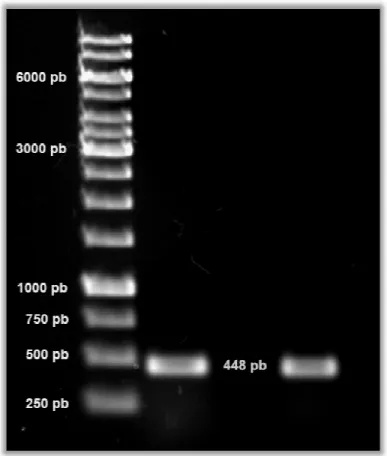
Figura 8 - PCR das colônias transformadas com o produto de ligação pYES2.1/V5-His-TOPO-LTx5 ... 41
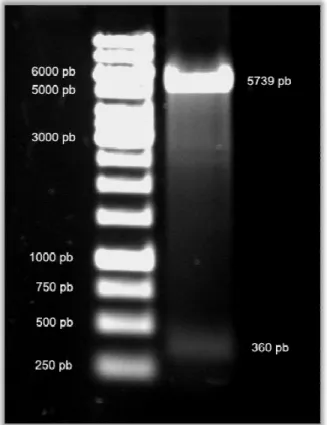
Figura 9 – Digestão da construção pYES2.1/V5-His-TOPO-LTx5 com as endonucleases de restrição PvuII e XbaI ... 42
Figura 10 – Análise da sequencia primária de aminoácidos da LTx5 recombinante. ... 43
Figura 11 - SDS-Tricina-PAGE dos diferentes tempos de indução com 2% de gal. ... 44
Figura 12 - Dot Blotting das amostras induzidas com 2% de galactose ... 45
Figura 13 – SDS-Tricina-PAGE dos diferentes tempos de indução, com 4% de gal ... 46
Figura 14 - SDS-Tricina-PAGE dos diferentes tempos de indução, com 4% de gal ... 47
Figura 15 - Dot Blotting ... 48
Figura 16 - Western Blotting ... 49
Figura 17 - Dot Blotting em condições desnaturantes ... 50
1
1. INTRODUÇÃO.
1.1Aspectos gerais.
As aranhas pertencem à ordem Araneae, classe Arachnida, subfilo Chelicerata e juntamente com a classe Tracheata (lacraias, diplópodes e insetos) compreendem um dos grupos de artrópodes que mais se adaptaram à vida terrestre com aproximadamente 40.000 espécies já descritas em mais de 100 famílias e 3.000 gêneros. Estima-se a existência de 170.000 espécies de aranhas no mundo (Ruppert et al., 2005; King, 2004). As aranhas são animais exclusivamente carnívoros, bem adaptados ao ambiente terrestre e encontrados em todos os continentes com exceção da Antártida. As aranhas podem ser encontradas em diferentes tamanhos, medindo de 0,5 mm até 9 cm de comprimento, e apresentam o corpo segmentado em cefalotórax e abdômen ligados por um pedículo. Na parte posterior do abdômen encontram-se os apêndices tecedores de seda, inovação que confere destaque às aranhas em relação aos demais aracnídeos (King, 2004; Ruppert et al., 2005; Saucier, 2004; Rash et al., 2002).
2 semi tropicais. Estas aranhas se adaptaram bem ao convívio com o homem, e algumas espécies são utilizadas como animais domésticos (King, 2004). A atividade predatória das aranhas terafosídeas conta com uma diversidade de presas vertebrados e invertebrados (Escoubas et al., 2004). Essas aranhas desenvolveram habilidade de captura utilizando suas quelíceras ortognatas dotadas de grandes aguilhões, que se movem no plano longitudinal, capazes de ferir e matar presas como pequenos pássaros e vertebrados de pequeno porte (Ruppert et al., 2005). A principal forma de defesa destas aranhas são os pêlos urticantes localizados em seu abdômen, que são lançados quando elas se sentem ameaçadas, causando irritação na pele e em mucosas (Waggoner et al., 1997; Isbister et al., 2002). Mesmo apresentando uma aparência amedrontadora devido ao seu tamanho, a picada das aranhas caranguejeiras não apresenta risco clínico ao ser humano (Lucas, 1988). Embora seu veneno não cause graves efeitos em humanos, há relatos de picadas de aranhas terafosídeas na América do Sul, África, Ásia e Austrália (Lucas et al., 1994; Schmidt, 1989; Isbister et al., 2003). A picada desta aranha, na maioria dos casos, causa dores locais moderadas, coceiras, edema, ardência e rigidez no local da injúria. Em casos mais graves, as picadas podem provocar espasmos musculares por algumas horas (Escoubas et al., 2004; Isbister et al., 2002), causados principalmente pelo baixo pH do veneno (pH 5.0) e pela ação de aminas biogênicas como serotonina, histaminas, adenosina, poliaminas e ATP (Chan et al., 1975; Schanbacher et al., 1973a; Cabbiness et al., 1980; Schanbacher et al., 1973b).
3 Figura 1 - Aranha do gênero Lasiodora sp
As aranhas do gênero Phoneutria, são popularmente conhecidas no Brasil como aranhas-armadeiras (Ministério da Saúde do Brasil, 2001). Estas aranhas são muito agressivas e quando se sentem ameaçadas armam imediatamente o bote, apoiando-se nas patas traseiras, levantando as patas dianteiras e os palpos, abrindo as quelíceras e deixando à mostra os ferrões. No Brasil, já foram descritas quatro espécies de aranhas Phoneutrias (Phoneutria nigriventer, P. fera, P. keyserfingi, e P. reidyi). Estas espécies podem ser encontradas em todas as regiões brasileiras exceto na região Nordeste. São animais de hábitos errantes, podem medir até 15 cm de envergadura, não constroem grandes teias e caçam preferencialmente à noite. É neste período que concentram a maioria dos acidentes dentro de residências e em suas proximidades (Bucaretchi et al., 2000; Ministério da Saúde do Brasil, 2001; Lucas, 1988).
4 gênero são muito perigosos, devido à ação dermonecrótica de seu veneno. A picada destas aranhas causa lesões cutâneas que se espalham ao redor da ferida e manifestações clínicas como insuficiência renal, coagulação intravascular disseminada e hemólise intravascular (da Silva et al., 2004; Swanson et al., 2006; Wasserman et al., 1983). A evolução do quadro de envenenamento para os demais sistemas é menos comum do que as manifestações cutâneas, mas podem acarretar complicações levando à morte das vítimas (Felicori et al., 2006).
As aranhas do gênero Latrodectus, popularmente conhecidas no Brasil como viúvas-negras, são encontradas em todo o mundo, com incidências das espécies Latrodectus curacaviensis, L. gemetricus e L. mactans principalmente na região litorânea do nordeste e sudeste do Brasil (Ministério da Saúde do Brasil, 2001; Lucas, 1988). Estas aranhas possuem abdômen globular negro, com uma mancha vermelha distinta em seu ventre. As fêmeas chegam a medir 1 cm de comprimento, enquanto os machos medem em média 3 mm de comprimento, o que facilita a ação canibal da fêmea após a cópula, motivo este responsável pelo nome popular desta aranha. A picada desta aranha, causada apenas pelas fêmeas, geralmente é indolor, porém os sintomas são graves e dolorosos, causados pela ação do veneno altamente neurotóxico, levando a dores abdominais e em membros inferiores, náuseas, espasmos musculares, sudorese, elevação da pressão do fluido cérebro-espinhal e paralisia respiratória (Ministério da Saúde do Brasil, 2001; Rash et al., 2002; Lucas, 1988; Ruppert et al., 2005; Timms et al., 1986). O tratamento de acidentes com viúvas-negras é feito com soro e anti-veneno (Heard et al., 1999; Isbister et al., 2003; Peterson, 2006; Timms et al., 1986; Vetter et al., 2008).
5 1.2Estudo de toxinas animais.
Organismos venenosos estão distribuídos em todo o reino animal, com mais de 100.000 espécies distribuídas nos principais filos, como cordata (répteis, peixes, anfíbios, mamíferos), equinoderma (ouriço-do-mar, estrela-do-mar), molusca (caracóis, polvos), anelídea (sanguessugas), artrópoda (aracnídeos, insetos, miriápodes), cnidária (anêmonas, águas vivas, corais) e são encontrados em praticamente todos os ecossistemas. A manutenção da vida em diferentes habitats, a busca por alimentação e a necessidade de defesa possibilitou uma grande diversidade de organismos peçonhentos e venenosos (Calvete et al., 2009; Menez et al., 2006).
A biodiversidade de organismos vegetais e animais resulta em uma grande diversidade de venenos e peçonhas, os quais compreendem um enorme leque bioquímico de moléculas pequenas, aminoácidos e proteínas complexas. As moléculas tóxicas podem apresentar grande variabilidade de estrutura e função entre indivíduos, espécies, gêneros e famílias (Mebs, 2001).
O processo evolutivo permitiu aos animais peçonhentos desenvolverem um complexo arsenal de neurotoxinas que atuam em receptores celulares com alta especificidade e seletividade (Escoubas et al., 2000b), tornando-as importantes para o estudo de mecanismos biológicos complexos (Gwee et al., 2002).
Os venenos são formados por uma biblioteca de peptídeos e proteínas, seletivamente adaptadas para atuarem nos sistemas vitais de suas vítimas e presas, interrompendo a atividade de enzimas vitais, agindo em receptores e canais iônicos, desarranjando os sistemas nervoso central e periférico, cardiovascular e neuromuscular, agindo na coagulação sanguínea e na homeostase celular (Calvete et al., 2009).
6 desenvolvimento de uma melhor proteção contra a ação destas toxinas (Menez et al., 2006).
Atualmente são estimadas cerca de 700 espécies de caracóis, 750 espécies de serpentes peçonhentas, 1.500 espécies de escorpiões e 40.000 espécies de aranhas, abrindo um enorme e inexplorado reservatório de componentes bioativos que podem trazer a cura para diversas doenças que não respondem aos métodos terapêuticos disponíveis. Por outro lado, os envenenamentos são uma questão de saúde pública em escala mundial, uma vez que estes animais são encontrados em todos os continentes e em quase todos os países, com predominância de habitat em regiões de clima tropical e semi-tropical (Calvete et al., 2009).
O primeiro relato científico de estudos de toxinas animais foi descrito em 1949, por Rocha e Silva e colaboradores, que descobriram um peptídeo que pode ser usado no controle da pressão arterial. Ao injetar o veneno da serpente brasileira Bothrops jararaca na corrente sanguínea de mamíferos, os autores observaram um acúmulo de bradicinina no organismo do animal. O acúmulo da bradicinina se dá devido à inibição da enzima ECA (enzima conversora da angiotensina), causada pela ação do veneno da serpente (Rocha e Silva et al., 1949). A bradicinina exerce efeito hipotensor quando acumulada no organismo. Os medicamentos conhecidos como inibidores das ECA’s (Capitopril, Enalapril, Benazepril), são igualmente responsáveis por inibir a degradação da bradicinina, causando assim, vasodilação e consequentemente, queda na pressão arterial (Ferreira et al., 1970).
7 neuroprotetora, antiepiléptica e analgésica em modelos animais (Han et al., 2008; Terlau et al., 2004). Recentemente aprovada pelo US Food and Drug Administration dos EUA, a ziconotida é outro exemplo de toxina animal utilizada para fins médicos. A ziconotida é um forte anestésico utilizado para o tratamento de dor crônica. A forma sintética do peptídeo -conotoxina M-VII-A derivado do caracol Conus magus atua bloqueando canais de cálcio tipo-N (Calvete et al., 2009; McIntosh et al., 1982).
Braud e colaboradores, em 2000, trabalhando com as serpentes das famílias Viperidae (víboras) e Crotalidae, através de experimentos realizados in vitro e in vivo, observaram que várias etapas de complexos processos fisiológicos, tais como coagulação sanguínea, ativação plaquetária e fibrinólise podem ser afetados pela ação das proteínas que compõem o veneno destas serpentes (Braud et al., 2000). Trabalhos relativos à estrutura molecular destas proteínas mostraram uma grande similaridade estrutural entre as diferentes famílias de proteínas que compõem estes venenos (serino proteases, metaloproteases, PLA2, lectinas tipo-C, desintegrinas) embora com diferentes alvos de ação (Markland, Jr., 1997; Ogawa et al., 1992; Nobuhisa et al., 1997; Ogawa et al., 1996; Nakashima et al., 1995; Nakashima et al., 1993).
Lqh-8 8/6 do escorpião L. quinquestriatus hebraeus. Todas estas toxinas são seletivas a canais de K+ e Cl- (Tytgat et al., 1998). O estudo destes peptídeos abre possibilidades para sua utilização como ferramenta bioinseticida, uma vez que são altamente seletivos, biodegradáveis e não tóxicos a mamíferos (du Plessis et al., 2008; Gordon et al., 2007; Gurevitz et al., 2007).
9 1.3Toxinas de aranhas.
Embora seja o grupo de animais peçonhentos com maior numero de espécies já descritas, o estudo de toxinas de aranhas ainda é pouco explorado (Escoubas et al., 2000b). Os estudos de peptídeos tóxicos presentes em venenos animais, principalmente aranhas e escorpiões, com alta especificidade de ação no sistema nervoso de insetos tem crescido a cada ano (De Lima et al., 2007). A utilização de peptídeos tóxicos com ação bioinseticida já vem sendo sugerida desde a década de 90 (Maeda et al., 1991; McCutchen et al., 1991; Stewart et al., 1991).
Em estudos recentes, a especificidade de ação de peptídeos tóxicos de venenos de aranhas vem sendo estudada em diferentes organismos, sugerindo aplicações agroquímicas contra larvas de Spodoptera litura e Helicoverpa armigera (Bloomquist, 2003; de Castro et al., 2004; Rajendra et al., 2006; Corzo et al., 2000) e também contra insetos de importância médica da ordem díptera (como moscas e mosquitos) como relatado em estudos com o veneno das aranhas australianas Atrax e Hadronyche (Maggio et al., 2002).
A atividade inseticida do veneno de aranhas tem sido comparada em experimentos “in vivo” contra insetos e camundongos, onde a alta toxicidade/letalidade do veneno para insetos não ocorre em camundongos (Corzo et al., 2000; Zhang et al., 2003; Lipkin et al., 2002; De Lima et al., 2002). A descoberta de toxinas de aranhas seletivas à ação em insetos tem gerado diversas patentes para aplicação no controle inseticida (De Lima et al., 2007).
Embora os dados farmacológicos sejam limitados, os peptídeos tóxicos atuam principalmente em canais de sódio (Brown et al., 1988; Corzo et al., 2000; De Lima et al., 2007; Sheumack et al., 1985), de cálcio (Liang, 2004; Escoubas et al., 2000a; Dutra et al., 2008; Cassola et al., 1996; Wang et al., 2001) e de potássio (Maggio et al., 2002; Tedford et al., 2004; Wang et al., 2000).
10 2004) e de componentes “inativos”, como ácidos nucléicos, aminoácidos livres, neurotransmissores, íons e sais inorgânicos como Ca2+, Na+, Mg2+, Cl- que atuam, na maioria dos casos, potencializando a ação das neurotoxinas (Cabbiness et al., 1980; Corzo et al., 2003a; Escoubas et al., 2000b; Escoubas et al., 2004; Rash et al., 2002). De acordo com sua especificidade farmacológica, as toxinas de aranhas podem ser classificadas em toxinas que afetam (a) a liberação de neurotransmissores, (b) canais de sódio voltagem-dependentes, (c) canais de cloreto, (d) canais de potássio, (e) bloqueadores de receptores colinérgicos pós-sinápticos e (f) canais de cálcio voltagem-sensível (Rash et al., 2002).
1.3.1 Ação na liberação de neurotransmissores.
Os neurotransmissores são responsáveis pelo envio e propagação de todo impulso nervoso entre os neurônios. O veneno da aranha do gênero Latrodectus possui um peptídeo extremamente neurotóxico, a -latrotoxina ( -LTx), que provoca uma grande liberação de neurotransmissores nas fendas sinápticas de vertebrados e invertebrados (Pinto et al., 1974; Cull-Candy et al., 1973). A liberação destes neurotransmissores resulta no bloqueio da transmissão nervosa, levando à paralisia muscular (Henkel et al., 1999; Harvey, 1990; Tzeng et al., 1978).
Estudos com o veneno da aranha Steatoda paykulliana, comumente confundida com aranhas do gênero Latrodectus devido à grande semelhança física, mostraram que em baixa concentração, o veneno foi capaz de aumentar a condutância da membrana lipídica e em alta concentração estimulou a liberação de neurotransmissores em células neuronais do tipo PC12, mas seu veneno não possui as -latrotoxinas (Cavalieri et al., 1987).
11 K+ (Dingledine et al., 1999), enquanto os receptores metabotrópicos são ligados às proteínas G e atuam através de um segundo mensageiro (Rash et al., 2002).
Figueiredo e colaboradores em 1995, trabalhando com o veneno da aranha Phoneutria nigriventer relataram a ação da toxina Tx4(6-1) no sistema nervoso periférico de insetos. Segundo os autores, a toxina Tx4(6-1) estimula a liberação de glutamato nas junções neuromusculares (Figueiredo et al., 1995).
Estudos in vivo com a toxina Tx4(5-5), isolada e purificada a partir da fração PhTx4 do veneno da aranha P. nigriventer, mostraram que esta toxina possui uma alta ação inseticida contra Musca domestica (mosca doméstica), Periplaneta americana (baratas) e Acheta domesticus (grilos), com efeitos neurotóxicos observados em doses muito baixas. Entretanto, quando administrada no hipocampo do cérebro de ratos, a toxina não apresentou nenhum efeito, sugerindo assim, uma especificidade em canais iônicos de insetos, apresentando portanto, um potencial bioinseticida (de Figueiredo et al., 2001).
Trabalhando com o veneno das aranhas Argiope trifasciata e Argiope florida, Usherwood e colaboradores, na década de 80, identificaram vários antagonistas de receptores de glutamato, chamados de argiotoxinas (Usherwood et al., 1984). Um ano mais tarde, Usmanov e colaboradores descobriram que a inibição pós sináptica na junção neuromuscular é uma propriedade comum do veneno de oito aranhas da família Araneidae (Usmanov et al., 1985).
1.3.2 Ação em canais de sódio voltagem-dependentes.
Os canais de sódio voltagem-dependentes estão presentes na maioria dos neurônios e músculos esqueléticos. Os canais de sódio voltagem-dependentes podem ser classificados em canais sensíveis à TTX (tetrodotoxina), presentes no cérebro e nos músculos esqueléticos, e em canais resistentes à TTX, presentes no coração e em neurônios sensoriais periféricos da glia (Weiss et al., 1986).
12 alteração na homeostase iônica destes canais resulta em consequências significativas para a excitabilidade da membrana, causando a paralisia do organismo (Martin-Moutot et al., 2006).
Os primeiros peptídeos tóxicos com ação em canais de sódio foram descobertos a partir do veneno da aranha Agelenopsis aperta e nomeados µ-agatotoxinas (µ-Aga-I a µ-Aga-VI) (Skinner et al., 1989). Estes peptídeos possuem alta similaridade entre si, são constituídos de 36 e 38 aminoácidos respectivamente, sendo oito resíduos de cisteína, e apresentam quatro pontes dissulfeto. As µ-agatotoxinas apresentam similaridade com as curtatoxinas (Ct-I e Ct-II) do veneno da aranha Hololena curta (Stapleton et al., 1990) e com as toxinas Tx1 e Tx2-9 do veneno da aranha Phoneutria nigriventer (Cordeiro et al., 1992). Essas toxinas promovem o aumento do influxo de sódio através da membrana, por meio dos canais de sódio voltagem-dependentes, despolarizando de forma duradoura as células e induzindo um estimulo neuronal pré sináptico, causando uma intensa liberação de neurotransmissores, o que leva a paralisia da presa (Escoubas et al., 2000b). Estudos eletrofisiológicos realizados em sapos, utilizando o peptídeo tóxico PhTx2, purificado do veneno da aranha Phoneutria nigriventer, mostrou uma ação deste peptídeo em canais de sódio voltagem-dependentes (Araujo et al., 1993).
Todos os peptídeos tóxicos de aranhas que atuam em canais de sódio apresentam o mesmo mecanismo de ação, semelhante àqueles descritos para as -toxinas de escorpiões e para as toxinas de anêmonas-do-mar (Escoubas et al., 2000b).
1.3.3 Ação em canais de cloreto.
13 1.3.4 Ação em canais de potássio.
Os canais de potássio representam o maior e mais diversificado subgrupo de canais iônicos e desempenham um papel central na regulação do potencial de membrana da célula, bem como na regulação da liberação de neurotransmissores, na frequência cardíaca, na secreção de insulina, na excitabilidade neuronal e na contração muscular (Corzo et al., 2003a; Shieh et al., 2000). Os canais de potássio possuem um poro central que determina a seletividade iônica, circundado por quatro domínios voltagem-sensíveis que se movem em resposta às mudanças de voltagem na membrana e promovem a abertura do poro (Milescu et al., 2007).
Os canais de K+ são classificados em três diferentes tipos de canais, sendo (a) canais de potássio retificadores de entrada, (b) canais de potássio voltagem dependente e (c) canais de potássio com domínio de dois poros (Corzo et al., 2003a).
Entre os componentes ativos nos venenos de aranhas, os peptídeos que atuam em canais de potássio são os menos abundantes (Grishin, 1999). Entretanto, algumas toxinas animais já descritas atuam em canais de potássio, como as toxinas homólogas Hanatoxina 1 e 2 (HaTx1 e HaTx2), isoladas do veneno da tarântula chinesa Grammostola spatulata, que bloqueiam canais Kv2.1, em concentrações nano molares (Swartz et al., 1995).
As heteropodatoxinas (HpTx1 e HpTx2), isoladas do veneno da aranha Heteropoda venatoria, apresentam alta similaridade com as toxinas HaTx1 e HaTx2, e em experimentos com miócitos ventriculares isolados do coração de ratos prolongaram o potencial de ação, sugerindo que estes peptídeos bloqueiam canais de K+ (Sanguinetti et al., 1997).
14 1.3.5 Ação em canais de cálcio voltagem-sensíveis.
O cálcio livre que entra na célula através dos canais de Ca2+ voltagem-sensíveis é responsável por diversos processos fisiológicos, incluindo a liberação de neurotransmissores, neurosecreção, excitação neuronal, sobrevivência de neurônios e regulação da expressão gênica (Deng et al., 2008). Atualmente existem pelo menos cinco canais de cálcio descritos em vertebrados, que podem ser divididos em dois grupos, os canais de cálcio de baixa voltagem, compreendendo os canais do tipo-T, e os canais de cálcio de alta voltagem, compreendendo os canais do tipo-L, N, P/Q e R (Wheeler et al., 1994; Zhang et al., 1993; Llinas et al., 1989).
Enquanto os canais do tipo-T são ativados por pequenas despolarizações e possuem atividade modulatória do tipo marcapasso, em células cardíacas e neuronais, os demais canais de cálcio são ativados por despolarizações mais intensas e estão essencialmente ligados à liberação de neurotransmissores (Tsien et al., 1991). Os canais do tipo-L são encontrados em células cardíacas e são alvos das dihidropiridinas usadas no tratamento de doenças cardiovasculares, como a hipertensão. Os canais do tipo-P, tipo-N, tipo-Q e tipo-R estão ligados ao sistema nervoso central e periférico (Escoubas et al., 2000b).
15 1.4Estudos com o veneno da aranha Lasiodora sp.
Estudos realizados com o veneno bruto da aranha Lasiodora sp, mostraram a ação do veneno em canais iônicos de invertebrados e em pequenos vertebrados (Kalapothakis et al., 2003; Kushmerick et al., 2001; de Deus, 2003). Utilizando técnicas de imagens de Ca2+, patch clamp e células GH3, na presença da Tetodrotoxina (TTX), um antagonista específico capaz de bloquear canais de Na+, Kushmerick e colaboradores, em 2001, mostraram que o veneno desta aranha aboliu as oscilações de Ca2+ normalmente presentes em nível basal nestas células e reduziu o nível de Ca2+ intracelular. Na ausência da TTX, o mesmo veneno causou um aumento lento nos níveis de Ca2+ intracelular, sugerindo a participação de outras moléculas presentes no veneno da aranha no bloqueio de canais de cálcio tipo-L (Kushmerick et al., 2001).
Em 2003, Kalapothakis e colaboradores, estudando a ação do veneno da aranha Lasiodora sp em corações isolados de ratos, obtiveram dados que sugerem que o veneno da aranha ocasiona um aumento na liberação vesicular de acetilcolina na extremidade de nervos parassimpáticos através da ativação de canais de Na+ resistentes a TTX (Kalapothakis et al., 2003).
Ainda no ano de 2003, de Deus e colaboradores fracionaram o veneno bruto da aranha Lasiodora sp, trabalhando com cromatografia de filtração molecular (Sephadex G-50 Fine - Pharmacia), e obtiveram três frações que foram denominadas P1, P2 e P3. As três frações foram submetidas a testes para determinar sua atividade em vertebrados e em invertebrados. Os resultados obtidos sugerem que as três frações possuem atividade tóxica em mamíferos, porém apenas as frações P2 e P3 apresentam ação contra organismos invertebrados, sendo que a fração P3 apresentou maior toxicidade nas condições testadas (de Deus, 2003).
16 Simultaneamente, foi realizada uma varredura da biblioteca de cDNA utilizando anticorpos anti fração P3 do veneno da aranha (de Moura, 2004). Neste trabalho foram identificados 7 clones codificantes para a toxina LTx1, que apresenta similaridade com diversas toxinas de aranhas já descritas e com ação inseticida comprovada, como as toxinas HWTX-II da aranha Selenocosmia huwena (Shu et al., 1999), a toxina ESTX da aranha Eurypelma californicum (Savel-Niemann, 1989; Kaiser et al., 1994) e com a toxina TXP1 da aranha Brachypelma smithii (Kaiser et al., 1994). A similaridade entre a LTx1 e as toxinas descritas com ação inseticida, sugere uma possível ação da LTx1 em invertebrados. Nesta varredura, também foram identificados 4 clones codificantes para a toxina LTx4, que apresenta similaridade com toxinas com ação comprovada em organismos vertebrados, como as toxinas Magi 1 e Magi 4 (Corzo et al., 2003b) isoladas da glândula de veneno da aranha Macrothele gigas, com a conotoxina Vx-VIb isolada do molusco marinho Conus vexillum, e com a neurotoxina Tx3-2 da aranha Phoneutria Negriventer (Cordeiro et al., 1993) isolada da glândula de veneno da aranha Phoneutria nigriventer, sugerindo assim uma possível ação da LTx4 em vertebrados.
Um alinhamento baseado na similaridade entre as cinco toxinas identificadas pelas varreduras da biblioteca de cDNA do veneno da aranha Lasiodora sp foi realizado utilizando o programa ClustalW, sugerindo a existência de três famílias de proteínas codificadas pela glândula de veneno desta aranha (figura 2).
17 A sequencia codificante para a toxina LTx2, foi sub-clonada no vetor de expressão pET11a (Novagen). O peptídeo foi expresso em células de E. coli (BL21 DE3) e purificado por cromatografia liquida de fase reversa de alta performance (HPLC) (Dutra, 2006). Experimentos eletrofisiológicos em células musculares BC3H1, mostraram que a toxina recombinante LTx2 bloqueou canais de cálcio tipo-L e inibiu a recarga de Ca2+ dos estoques intracelulares, eliminando as oscilações de cálcio livre promovido pelo receptor InsP3. A alta similaridade da LTx2, uma das cinco toxinas previamente identificadas no veneno total da aranha Lasiodora sp, com as toxinas LTx1 e LTx3 sugere um mecanismo de ação semelhante para estas três toxinas (Dutra et al., 2008).
18 2. OBJETIVOS.
2.1Objetivo geral.
Sub clonar a sequência codificante para a toxina madura LTx5 no vetor pYES2.1/V5-His-TOPO®, induzir a expressão da proteína recombinante e purificá-la.
2.2Objetivos específicos.
2.2.1 Amplificar a sequencia codificante para a toxina madura LTx5 a partir do clone 19 isolado da biblioteca de cDNA;
2.2.2 Proceder a ligação no vetor pYES 2.1/V5 His-TOPO®;
2.2.3 Transformar células de Escherichia coli TOP10F’ com o produto de ligação;
2.2.4 Verificar a presença do fragmento por PCR de colônia e através do uso de endonucleases de restrição;
2.2.5 Verificar por sequenciamento do DNA se o inserto encontra-se no frame correto;
2.2.6 Transformar células de Saccharomyces cerevisiae W303-1A; 2.2.7 Induzir a expressão da proteína recombinante;
19
3. MATERIAL E MÉTODOS.
3.1Microorganismos.
Para amplificação do DNA plasmidial, a cepa utilizada foi Escherichia coli TOP10F’. Genótipo: F’, mcr A, D (mrr-hadRMS – mcrBC) Ø 80DlacZ DM15, Dlacx74, deoR, recD1, araD139,D, 7697, galIU, galK, l-,rs1p,end D1,nupG.
Para expressão do gene codificante para a toxina madura LTx5 foi utilizada a cepa Saccharomyces cerevisiae W303-1A. Genótipo: MATa; ura3-52; trp1 2; leu2-3_112; his3-11; ade2-1; can1-100.
3.2Meios de cultura.
3.2.1 Meio LB (Luria-Bertani).
1% de triptona (p/v), 0.5% de extrato de levedura (p/v), 1% de NaCl (p/v), ajustar pH para 7.5 com NaOH. Para meio sólido adicionar 1.5% de agar (p/v).
3.2.2 Meio LB-XIA.
O meio LB é suplementado com 20 µg/mL de X-GAL, 1mM de IPTG e 20µg/mL de ampicilina.
3.2.3 Meio YP.
1% de extrato de levedura (p/v), 2% de peptona bacteriológica (p/v). A este meio foi adicionado 2% de glicose (p/v) e 2% de galactose (p/v).
3.2.4 Meio seletivo SD.
20 utilizado 2% e 4% de galactose (p/v) ao meio. Para meio sólido adicionar 1.5% (p/v) de ágar e ajustar o pH para 6.5.
3.2.5 Meio 2XYP.
1,6% de bactopeptona (p/v), 1% de extrato de levedura (p/v) e pH 7.5.
3.3Vetor de clonagem e expressão.
O sistema utilizado para clonagem e expressão da proteína recombinante foi o pYES2.1/V5-His-TOPO® TA Expression Kit (Figura 3).
Figura 3 - Mapa do vetor de clonagem e expressão pYES2.1/V5-His-TOPO® (disponível em
3.4Sub clonagem expressão pYES
O procedimento de sequência codificante para clone 19 selecionado da b 2005).
Figura 4 – Sequencia de nucle regiões traduzidas para o pept vermelho, verde e azul, respec minúsculo.
m da sequência codificante para a LTx5 ES2.1/V5-His-TOPO®.
e clonagem foi desenvolvido com o intuito de ins ra a toxina madura LTx5 (figura 4), amplificad biblioteca de cDNA da aranha Lasiodora sp (
leotídeos e aminoácidos da LTx5. Em maiúsculo são re ptídeo sinal, peptídeo intermediário e peptídeo maduro ectivamente. As regiões 5' e 3' não traduzidas estão rep
21 no vetor de
inserir apenas a ada a partir do (Vieira et al.,
22 3.4.1 PCR para amplificação da sequência codificante para a toxina madura.
Os iniciadores desenhados para amplificação da região correspondente à toxina madura da LTx5 estão descritos abaixo:
Iniciadores Sequências
Direto (F1) 5’ – CA ATG GCA GAA AAG GGA AAA GAC – 3’ Reverso (R1) 5’ – CTC ACA CCG AAA TGA AGA – 3’
O DNA extraído do clone 19 da biblioteca de cDNA na forma de fagemídeo foi usado como molde na reação. A mistura de reação usada na PCR está descrita a seguir:
• 2,0 µL de DNA fagemídeo • 2,5 µL de dNTP’s (2,5 mM) • 2,5 µL de tampão Taq 10X • 0,75 µL de MgCl2 (50 mM)
• 0,5 µL do iniciador F1 (40 pmoles/µL) • 0,5 µL do iniciador R1 (40 pmoles/µL) • 0,6 µL de Taq DNA polimerase
• 15,65 µL de água Milli-Q estéril
Procedeu-se a reação em termociclador, conforme o programa descrito: 1. 94°C por 4 minutos
2. 94°C por 1 minuto 3. 54°C por 1 minuto 4. 72°C por 1 minuto 5. Etapa 2 a 4 (29 vezes) 6. 72°C por 5 minutos
23 3.4.2 Purificação do produto de PCR.
Após a amplificação da região correspondente à sequencia codificante para a toxina madura LTx5, o produto de PCR foi submetido à eletroforese em gel de agarose 1% por 50 minutos a 100 volts. O gel foi corado utilizando brometo de etídio em tampão TAE 1X (40 mM de Tris-acetato, 1 mM EDTA, pH 8.0). A banda correspondente à sequência codificante para a toxina madura LTx5 foi extraída com auxílio de um estilete, cortada em fatias, e estas transferidas para tubo de microcentrífuga e adicionada de 500 µL de solução TAE 1X contendo 1M de NaCl. A mistura foi agitada com auxílio de um agitador de tubos e imediatamente incubada a 37°C por 2 horas. Após incubação, as amostras foram centrifugadas a 15.000 g por 10 minutos. O sobrenadante foi transferido para novo tubo de microcentrífuga, adicionado 400 µL de PCI (fenol/clorofórmio/isoamílico 25:24:1) e a mistura agitada novamente. Após agitação as amostras foram centrifugadas a 15.000 g por 10 minutos e a camada aquosa transferida para novo tubo de microcentrífuga, e adicionada de dois volumes de etanol 100%. A mistura foi mantida a -80°C por 15 minutos. Posteriormente, o tubo foi centrifugado a 15.000 g por 10 minutos. O sobrenadante foi descartado e o precipitado (DNA) lavado com 800 µL de etanol 70%. Após nova centrifugação a 15.000 g por 10 minutos, o sobrenadante foi desprezado e o DNA foi seco a 65°C, e posteriormente suspenso em 20 µL de H2O Milli-Q estéril.
3.4.3 Ligação do produto de PCR no vetor de clonagem.
O produto de PCR purificado foi inserido no vetor de expressão pYES2.1/V5-His-TOPO® conforme protocolo a seguir:
• 2,0 µL de DNA purificado (produto de PCR) • 1,0 µL de salt solution
• 0,5 µL do vetor
24 A reação foi mantida à temperatura ambiente por 20 minutos.
3.5Preparo de células competentes de E. coli TOP10F’.
Para o preparo de células competentes foi utilizado o método de cloreto de cálcio. Células de Escherichia coli linhagem TOP10F’ foram inoculadas em 3 mL de meio LB contendo estreptomicina (20 µg/mL) e tetraciclina (15 µg/mL) e crescidas por 16 horas à 37°C, sob agitação constante a 200 RPM. Após crescimento o pré inóculo foi utilizado para inocular em 100 mL de meio LB. A cultura foi incubada nas mesmas condições de crescimento descritas anteriormente até atingir um valor de absorbância a 600 nm entre 0.6 e 0.8. Atingido o crescimento ideal as células foram submetidas à centrifugação a 1.000 g por 8 minutos, resuspensas em 40 mL de CaCl2 0.1 M gelado e incubadas em gelo por 1 hora. Após incubação, as células foram novamente centrifugadas e resuspensas em 1 mL de CaCl2 0.1 M. Alíquotas de 100µL foram estocadas em tubos de microcentrífuga na presença de 30% de glicerol estéril, à -80°C.
3.6Transformação de bactérias.
25 3.7PCR das colônias provenientes da transformação de E. coli TOP10F’
com o produto de ligação pYES2.1/V5-His-TOPO-LTx5.
Colônias de bactérias, provenientes da transformação, foram inoculadas em 3 mL de meio LB suplementado com 100 µg/mL de ampicilina para crescimento por 16 horas. Após crescimento, as colônias foram submetidas à reação em cadeia da polimerase para confirmar a presença do fragmento codificante para a toxina madura LTx5 no vetor de expressão pYES2.1/V5-His-TOPO®. Os iniciadores utilizados nessa reação foram:
Iniciadores Sequências
Direto (F2) 5’ – CGG TTT GTA TTA CTT CTT – 3’
Reverso (R2) 5’ – ACC GGT ACG CGT AGA – 3’
Os iniciadores acima anelam no vetor pYES2.1/V5-His-TOPO® e acompanham o kit pYES 2.1 TOPO® TA usado para expressão da proteína recombinante. Como molde da reação foi utilizado 1,0 µL de cultura dos clones provenientes da transformação. O programa da PCR encontra-se abaixo:
• 1,0 µL do iniciador F2 (20 pmoles/µL) • 1,0 µL do iniciador R2 (20 pmoles/µL) • 2,5 µL de tampão Taq 10X
• 2,5 µL de dNTP’s (2,5 mM) • 0,75 µL de MgCl2 (25 mM)
• 0,6 µL de Taq DNA polimerase • 1,0 µL da cultura
• 10,65 µL de H2O Milli-Q estéril
Procedeu-se a reação em termociclador conforme o programa descrito: 1. 94°C por 4 minutos
26 3. 54°C por 1 minuto
4. 72°C por 1 minuto 5. Etapa 2 a 4 (29 vezes) 6. 72°C por 5 minutos
7. Redução da temperatura a 4°C
3.8Eletroforese em gel de agarose.
Para verificar a presença do inserto, após a PCR o material foi submetido à eletroforese em gel de agarose 1% por 90 minutos a 80 volts. Para visualização das bandas, o gel foi corado com brometo de etídio.
3.9Extração de DNA plasmidial de bactéria.
27 Para secagem do DNA o tubo foi incubado a 65°C. Posteriormente o DNA foi resuspenso em 20 µL de H2O Milli-Q estéril.
3.10 Digestão do DNA plasmidial com endonucleases de restrição.
O DNA plasmidial contendo, a priori, a sequência codificante para a toxina madura LTx5 foi digerido com as endonucleases de restrição PvuII e XbaI para confirmação da presença do inserto. Inicialmente o DNA plasmidial extraído conforme descrito no item 3.8 foi incubado com 1 µL de RNAse (10 mg/mL) por 30 minutos, a 37°C. Posteriormente a RNAse foi inativada a 65°C por 5 minutos, e a digestão procedeu-se utilizando o seguinte protocolo:
• 5,0 µL de H2O Milli-Q estéril
• 4,0 µL de tampão universal Tango (Fermentas) • 1,0 µL de PvuII
• 1,0 µL de XbaI
• 9,0 µL de DNA plasmidial
A mistura de reação foi incubada por 90 minutos a 37°C. Para verificar a presença do inserto, após digestão o material foi submetido à eletroforese em gel de agarose 1% por 90 minutos a 80 volts. Para visualização das bandas o gel foi corado com brometo de etídio como descrito anteriormente.
3.11 PCR para confirmação da orientação do inserto no vetor pYES2.1/V5-His-TOPO®.
(F2 e R2). Na PCR-A (figu (figura 6) foi utilizados os i
PCR-A:
• 1,0 µL do in • 1,0 µL do in • 2,5 µL de tam • 2,5 µL de dN • 0,75 µL de M • 1,0 µL de DN • 0,5 µL de Ta • 10,75 µL de
Figura 5 - PCR-A: Desenho e iniciador direto F2, e a seta em representa a toxina madura representada a sequencia cod representado em vermelho.
igura 5) os iniciadores utilizados foram o F2 e R1 s iniciadores F1 e R2, conforme descrito:
iniciador F2 (20 pmoles/µL) iniciador R1 (20 pmoles/µL)
tampão Taq 10X dNTP’s (2,5 mM) e MgCl2 (50 mM)
DNA plasmidial Taq DNA polimerase de H2O Milli-Q estéril
o esquemático representando a PCR-A. A seta em azu em vermelho representa o iniciador reverso R1. A seq
a LTx5, com o códon de inicialização sublinhado. E codificante para a cauda de histidina, e o códon d
28 R1 e na PCR-B
PCR-B:
• 1,0 µL do in • 1,0 µL do in • 2,5 µL de tam • 2,5 µL de dN • 0,75 µL de M • 1,0 µL de DN • 0,5 µL de Ta • 10,75 µL de
Figura 6 - PCR-B: Desenho e iniciador direto F1, e a seta em representa a toxina madura representada a sequencia cod representado em vermelho.
Procedeu-se a reaçã 1. 94°C por 4 m 2. 94°C por 1 m 3. 54°C por 1 m
iniciador F1 (20 pmoles/µL) iniciador R2 (20 pmoles/µL)
tampão Taq 10X dNTP’s (2,5 mM) e MgCl2 (50 mM)
DNA plasmidial Taq DNA polimerase de H2O Milli-Q estéril
esquemático representando a PCR-B. A seta em azu em vermelho representa o iniciador reverso R2. A seq
a LTx5, com o códon de inicialização sublinhado. E codificante para a cauda de histidina, e o códon d
ção em termociclador, conforme o programa descr 4 minutos
1 minuto 1 minuto
29 zul representa o equencia em azul . Em verde está de parada está
30 4. 72°C por 1 minuto
5. Etapas 2 a 4 (29 vezes) 6. 72°C por 5 minutos
7. Redução da temperatura a 4°C
As amostras amplificadas foram submetidas à eletroforese em gel de agarose 1% por 90 minutos a 80 volts. Para visualização das bandas, o gel foi corado com brometo de etídio como descrito anteriormente.
3.12 Sequenciamento do DNA.
3.12.1 Mini prep para sequenciamento.
31 descartado, por inversão da placa. O DNA foi lavado usando 190 µL de etanol 70%, gelado, e a placa centrifugada por 10 minutos a 1.000 g. Descartado o sobrenadante, a placa foi seca à temperatura ambiente por 1 hora. O DNA foi resuspenso em 12 µL de H2O Milli-Q. A placa contendo o DNA foi novamente selada com adesivo e mantida à 4°C, até o momento do uso.
3.12.2 PCR para reação de sequenciamento.
O DNA plasmidial extraído de células de E. coli TOP10F’ como descrito no item 3.9 foi submetido a sequenciamento de acordo com o método descrito por Sanger et al. (1977), utilizando o Kit de Sequenciamento DYEnamic ET DYE Terminator Cycle Sequencing Kit for MegaBace DNA Analysis System (Amersham Biosciences). Os iniciadores F2 e R2 utilizados de forma independente, para sequenciamento de ambas as fitas do DNA, estão descritos abaixo:
Iniciadores Sequências
Direto (F2) 5’ – CGG TTT GTA TTA CTT CTT – 3’
Reverso (R2) 5’ – ACC GGT ACG CGT AGA – 3’
A reação de amplificação foi preparada como descrito:
• 4,0 µL de Mix (Kit do sequenciamento) • 1,0 µL do iniciador (F2 ou R2) (5 pmoles/µL) • 3,0 µL de DNA plasmidial
• 2,0 µL de H2O deionizada
Procedeu-se as reações em termociclador conforme o programa: 1. Rápida elevação da temperatura a 95°C
32 5. Etapas 2 a 4 (30 vezes)
6. Redução da temperatura a 4°C
3.12.3 Precipitação da reação de sequenciamento.
Após a reação da PCR, adicionou-se ao produto da reação 1 µL de acetato de amônio 7,5 M e 28 µL de etanol 100%. Após homogeneização, a mistura foi incubada por 20 minutos à temperatura ambiente na ausência de luz. Após incubação, a mistura foi centrifugada a 1.000 g por 45 minutos. O sobrenadante foi descartado e o produto da PCR precipitado foi lavado com 100 µL de etanol 70%. A suspensão foi submetida à nova centrifugação a 1.000 g por 10 minutos, o sobrenadante descartado, e o DNA resuspenso em 10 µL de loading buffer (Amersham Biosciences) e mantido a 4°C na ausência de luz até o momento da injeção no sequenciador.
3.13 Preparo de células competentes de Saccharomyces cerevisiae.
33 3.14 Transformação de leveduras.
Cerca de 300 µL de suspensão de células competentes de S. cerevisiae foram transferidas para tubos de microcentrífuga. A esta suspensão de células adicionou-se 20 µL de DNA de esperma de salmão (10 mg/mL), previamente desnaturado a 95°C por 5 minutos, e 3 µL da construção pYES2.1/V5-His-TOPO-LTx5. Após incubação por 30 minutos, a 28°C, foram adicionados 700 µL de PEG 4.000 50% (p/v), e procedeu-se nova incubação, por 1 hora, a 28°C. Em seguida as suspensões foram submetidas a 42°C por 10 minutos, e então centrifugadas a 3.000 g por 4 minutos. O sobrenadante foi descartado assepticamente, as células resuspensas em 1 mL de sorbitol 1 M e novamente centrifugadas a 3.000 g por 4 minutos. Após descarte do sobrenadante, as células foram resuspensas no sobrenadante residual, plaqueadas com auxílio de pérolas de vidro em meio seletivo SD-URA e incubadas a 30°C para crescimento por 72 horas. Após crescimento, as colônias positivas foram estriadas em placa contendo meio seletivo SD-URA.
3.15 Expressão da proteína recombinante.
34 ligação pYES2.1/V5-His-TOPO-LTx5 foram crescidas em meio YPD 2% e submetidas às mesmas condições de lise e análise em SDS-Tricina-PAGE. As soluções estoque de rafinose 10% (p/v) e galactose 40% (p/v) foram esterilizadas por filtração.
3.16 Extração de proteínas totais.
Para extração de proteínas totais foi utilizado o protocolo descrito pelo kit YeastBuster™ Protein Extraction Reagent (Novagen). As células foram descongeladas, e o peso úmido determinado. Para cada 1 grama de peso úmido celular foi utilizado 5 mL de YeastBuster™ e 50 µL do agente redutor THP Solution. Foram adicionados inibidores de serino protease (PMSF 1mM) e cisteíno protease (NEM 20 µM) ao tampão de lise. Após serem incubadas com o tampão de lise por 20 minutos, a 28°C, sob agitação constante, as misturas foram centrifugadas a 15.000 g por 20 minutos. A fração solúvel foi coletada e dialisada utilizando membrana benzoilada, capaz de reter moléculas acima de 2.000 Da, contra tampão contendo 50 mM de Tris HCl pH 7.4 para posterior análise em SDS-Tricina-PAGE.
3.17 Solubilização da fração insolúvel.
35 3.18 Dosagem de proteínas totais.
Para a dosagem de proteínas totais das frações solúveis e insolúveis foi utilizado o método descrito por Bradford (Bradford, 1976) e/ou Lowry (Lowry et al., 1951), utilizando albumina bovina como padrão.
3.19 SDS-PAGE (Laemmli, 1970).
Inicialmente, para verificação da expressão da proteína recombinante foi utilizado o método de eletroforese em gel de poliacrilamida em condições desnaturantes proposto por Laemmli (Laemmli, 1970). As concentrações utilizadas para a confecção do gel foram de 18% para o gel de separação e 4% para gel de concentração. As amostras protéicas foram tratadas com tampão de amostra na presença de -mercaptoetanol, fervidas por 5 minutos e aplicadas no gel. A eletroforese foi conduzida em tampão de corrida 1X (Tris 0.025 M, glicina 0.96 M e 0.1% de SDS), sob corrente constante de 100 V, até a total migração do corante pelo gel de separação.
3.20 SDS-Tricina-PAGE (Schagger et al., 1987).
36 3.21 Coloração de gel de poliacrilamida por nitrato de prata.
Após a separação eletroforética, o gel foi fixado em solução fixadora (50% de metanol e 12% de ácido acético) por 1 hora. Após ser lavado três vezes com H2O por 20 minutos o gel foi sensibilizado com solução de tiossulfato de sódio 0,04% por 1 minuto, novamente lavado três vezes com H2O por 20 minutos e submerso em solução de impregnação contendo 0,1% de nitrato de prata e 0,0075% de formaldeído. Após rinsagem por 1 minuto, o gel foi revelado em solução contendo 6% de carbonato de sódio, 0,01% de formaldeído e 0,2% da solução de tiossulfato de sódio. A revelação foi interrompida com solução de parada (50% de metanol e 12% de ácido acético).
3.22 Dot Blot.
37 3.23 Western Blot.
Após desenvolvimento da eletroforese, as proteínas foram transferidas para uma membrana de nitrocelulose (Amersham Biosciences, poros de 0,45 m). A transferência foi conduzida a 50 V, 65 mA, por 1 hora, em sistema semi seco Hoefer Semiphor (Pharmacia Biotech) usando-se como tampão de transferência uma solução contendo Tris 25 mM, glicina 192 mM, 10% de metanol v/v, SDS 0.1% p/v, pH 8.3. Após transferência, a membrana foi bloqueada com solução de bloqueio PBS-T 0.05% (Na2HPO4 0.05 M, NaCl 0.15 M, 0.05% de Tween-20, pH 7.4), contendo 5% de leite em pó desnatado, por 16 horas, sob agitação. Após bloqueio, a membrana foi lavada com PBS-T 0.05% por 20 minutos e incubada com anticorpo primário anti His-Tag (Novagen) diluído 1:2.000 em solução de bloqueio, por 3 horas, sob agitação constante. Após incubação com o anticorpo primário, a membrana foi novamente lavada com PBS-T 0.05% por 20 minutos e incubada por 2 horas com anticorpo secundário anti IgG de camundongo conjugado à peroxidase (Zymed/Invitrogen) diluído 1:2.000 em solução de bloqueio. Após nova lavagem, a reação imunológica foi revelada utilizando o kit ECL Western Blotting Detection Reagents (Amersham Biosciences). O filme foi exposto por 1 minuto à membrana de nitrocelulose e revelado utilizando as soluções de revelação e fixação comercializadas pela Kodak.
3.24 Cromatografia de filtração molecular.
38 3.25 Cromatografia de troca aniônica.
Na tentativa de excluir as proteínas carregadas negativamente (com ponto isoelétrico abaixo de 7.20), foi realizada uma cromatografia de troca aniônica utilizando resina Q-Sepharose (Sigma). Uma coluna cromatográfica com 4 mL de resina, previamente inchada, foi montada e equilibrada com tampão acetato de amônio 10 mM, pH 7.2. O extrato protéico proveniente da amostra de 60 horas de indução em 4% de galactose foi carregado na coluna Q-Sepharose e frações de 1 mL foram coletadas manualmente. Para eluição das proteínas que se ligaram à resina, foi utilizado tampão acetato de amônio 10 mM contendo 1 M de NaCl. As leituras das frações coletadas foram feitas em espectrofotômetro, à 230 nm e à 280 nm.
3.26 Purificação da proteína recombinante utilizando resina Ni-NTA.
39 3.27 Fracionamento do extrato protéico utilizando sistema Microcon®
YM-50K Millipore.
4. RESULTADOS.
4.1Amplificação d
Para amplificação fagemídeo obtido do clone partir da região codificante em cadeia da polimerase f figura 7 mostra o produt madura LTx5 clonadas no produtos de PCR do frag padrão foi utilizado 1 Kb P
Figura 7 - Amplificação da reg foi amplificada a partir do clo pares de base foi utilizado 10 µ
da região codificante para a toxina madura LT
o do fragmento codificante para a toxina LT ne 19 foi utilizado como molde e os iniciadores
te para a toxina madura. Os produtos obtidos atra e foram submetidos à eletroforese em gel de ag
uto da amplificação do fragmento corresponde o vetor pBK-CMV. Nas canaletas 1, 2 e 3 são ap agmento amplificado que possui 213 pares de
Plus DNA Ladder (Invitrogen).
egião codificante para a toxina madura LTx5. A toxina clone 19 da biblioteca de cDNA e apresenta 213 pb. C
µL de Kb Plus DNA Ladder (Invitrogen).
40 LTx5.
LTx5 o DNA s desenhados a través da reação agarose 1%. A dente à toxina apresentados os e bases. Como
4.2Clonagem da s pYES2.1/V5-Hi
Após a amplificação inserido no vetor pYES2 transformadas com o prod para fornecer o molde d iniciadores utilizados se a nucleotídeo (primer revers correspondentes à toxina m pares de base na reação d alíquota de Generuler™ 1 K
Figura 8 - PCR das colônias LTx5. O produto de amplificaç µL do Generuler™ 1 Kb DNA
sequência codificante para LTx5 no vetor d His-TOPO®.
ção da toxina madura LTx5, o produto da PCR fo S2.1/V5-His-TOPO®. Células de E. coli TOP oduto da ligação. As colônias transformadas for da PCR para verificação da presença do fr anelavam no 360° nucleotídeo (primer direto erso) do vetor, adicionando assim 235 nucleotíd a madura, totalizando um produto de amplificaç da PCR (figura 8). Como padrão de pb foi u 1 Kb DNA Ladder (Fermentas).
as transformadas com o produto de ligação pYES2.1/ cação apresenta 448 pb. Como padrão de pares de base
A Ladder (Fermentas).
41 r de expressão
foi purificado e OP10F’ foram oram utilizadas fragmento. Os eto) e no 593° tídeos aos 213 cação com 448 i utilizado uma
4.3Digestão do DN
Células de E. coli T TOPO-LTx5, foram cresci extraído e então digerido c do tampão Tango. O vetor (T) livres nas extremidade polimerase adiciona uma d facilitando a ligação do endonucleases PvuII e Xb fragmentos, um de 360 pb sequência do vetor e um fr (figura 9).
Figura 9 – Digestão da constru PvuII e XbaI. Dois fragment fragmento de 360 pb contendo Como padrão de pares de base
NA plasmidial com as endonucleases PvuII e X
TOP10F’, transformadas com a construção pYE cidas em meio seletivo por 16 horas. O DNA p com as endonucleases de restrição PvuII e Xba tor pYES2.1/V5-His-TOPO® possui 5886 pb e d des 5’ de ambas as fitas do DNA. Na reação de
deoxiadenosina (A) nas extremidades 3’ do pro do gene de interesse ao vetor. Quando di baI a construção pYES2.1/V5-His-TOPO-LTx pb correspondente à sequência da toxina madu fragmento de 5.739 pb correspondente a sequen
rução pYES2.1/V5-His-TOPO-LTx5 com as endonuclea ntos, um de 5739 pb correspondente à sequencias d do a sequencia codificante para a LTx5 e parte da sequ se foi utilizado 10 µL de Generuler™ 1 Kb DNA Ladder
42 XbaI.
plasmidial foi
aI na presença duas timidinas de PCR, a Taq roduto de PCR digerido pelas x5 libera dois dura e parte da encias do vetor
eases de restrição do vetor, e um quência do vetor.
4.4Sequenciament
Para verificar se a frame correto (janela abe apresentaram a inserção seqüenciados seguindo o m 1977). A análise dos resulta sequencia codificante para identificar a sequencia cod códon de parada (Anexo).
4.5Análise da sequ
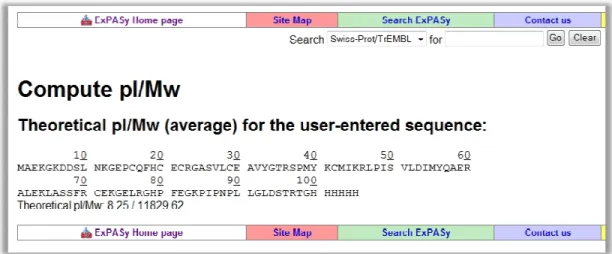
A região codificante V5 epitope e a sequencia co totalizando 105 aminoácid 11.8 kDa e a mesma deve Compute pI/Mw tool (figura
Figura 10 – Análise da seque realizada pela ferramenta Com molecular de 11829.62 Da e um
nto do DNA.
a sequencia codificante para a toxina LTx5 enco berta de leitura), os DNA’s plasmidiais das o do fragmento de 448 pb na orientação c
método descrito por Sanger e colaboradores (S ltados obtidos nos permitiu identificar o códon de ra a toxina madura LTx5 inseridos no frame corre codificante para o V5 epitope, a cauda de
poli-quencia de aminoácidos da LTx5 recombinante
nte para a LTx5 recombinante, mais a região codi codificante para a cauda de histidina possui 315 p
idos. A massa molecular da proteína é de apro ve apresentar um pI 8.25, simulados utilizando ura 10) disponível em www.expasy.org.
uencia primária de aminoácidos da LTx5 recombinan ompute pI/Mw (www.expasy.org) nos mostra uma prote
m pI de 8.25.
43 ncontrava-se no s colônias que correta foram (Sanger et al., de iniciação e a reto, bem como -histidina e o
nte.
dificante para o 5 pares de base, roximadamente o a ferramenta
4.6Expressão da p
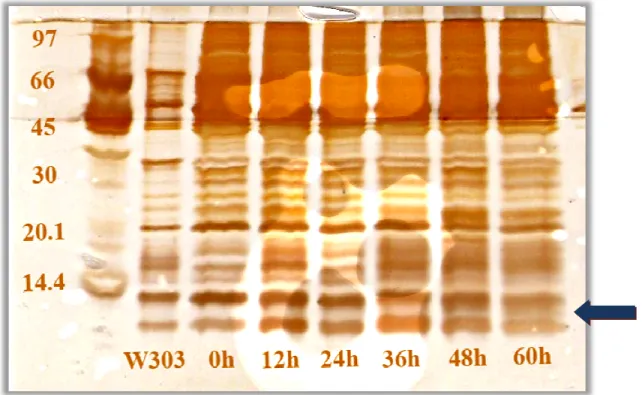
Para análise da expr indução foram submetidas condições desnaturantes. A galactose. Em uma das can (Amersham Biosciences) e total. Nestas condições não recombinante nos diferente
Figura 11 - SDS-Tricina-PAG proteína total foram aplicados µL de Low Molecular Weight utilizado extrato protéico de W da proteína recombinante com
proteína recombinante LTx5 com 2% de galac
pressão da LTx5 as amostras protéicas de diferen as à separação eletroforética em gel de poliac . A figura 11 refere-se ao processo de indução
analetas foram aplicados 2 µL do padrão de mas e nas demais canaletas foram aplicados 30 µg ão foi possível identificar diferença de expressã tes tempos de indução.
GE dos diferentes tempos de indução com 2% de gala os em cada canaleta. Como padrão de massa molecular t (Amersham Biosciences). Como controle da expressão W303-1A não transformada. A seta azul indica a poss m massa molecular de 11.8 kDa.
44 lactose.
entes tempos de liacrilamida em ão com 2% de assa molecular g de proteína são da proteína
4.7Dot Blot.
Com o objetivo de i induzidos com 2% de gala nitrocelulose foi sensibiliza tempos de indução. Como tagged Protein Standard (In extrato protéico de W303-1 expressão da proteína rec galactose.
Figura 12 - Dot Blotting d nitrocelulose foi sensibilizada c a vácuo, em um sistema Bio-Do em solução PBS-T 0,05% con (Novagen) diluído 1:2000 e com 1:2000 (Zymed/Invitrogen). controle positivo foi utilizado Como controle negativo foi uti expressão da proteína recomb em baixo nível de expressão, a
e identificar a expressão da proteína recombinante lactose foi realizado um Dot Blotting no qual a izada com 100 µg de proteínas dos extratos totais d
o controle positivo foi utilizado 1 µL de Bench (Invitrogen), e como controle negativo foi utiliza
1A não transformada. Na figura 12 observamos ecombinante a partir de 48 horas de indução
das amostras induzidas com 2% de galactose. A a com extratos protéicos dos diferentes tempos de induç Dot™ Apparatus (Biorad). Posteriormente, a membran ontendo 5% de leite em pó e incubada com anticorp om anticorpo anti IgG de camundongo conjugado à per Foram aplicados 100 µg de proteína total em cada o 1 µL de Benchmark™ His-tagged Protein Standar utilizado 100 µg de extrato protéico de W303-1A não tr binante contendo a cauda de histidina pode ser identi a partir de 48 horas de indução com 2% de galactose.
45 nte nos extratos a membrana de is dos diferentes chmark™ His-zado 100 µg de os uma possível o com 2% de