DANIELLE ASSIS DE FARIA
Estudos genômicos em Eucalyptus: Mapeamento de genes
candidatos e QTLs, estimativa de desequilíbrio de ligação e
análise de estrutura de populações
Tese apresentada ao Programa de
Pós-Graduação Strictu-Senso em Ciências
Genômicas e Biotecnologia da
Universidade Católica de Brasília, como requisito para obtenção do Título de Doutor em Ciências Genômicas e Biotecnologia
Orientador: Rinaldo Wellerson Pereira
Co-orientador: Dario Grattapaglia
BRASÍLIA
2008
Ficha elaborada pela Sistema de Bibliotecas da Universidade Católica de Brasília ± SIBI//UCB.
!
!
!
F224e Faria, Danielle Assis de
Estudos genômicos em Eucalyptus : mapeamento de genes candidatos e QTLs, estimativa de desequilíbrio de ligação e análise de estrutura de populações / Danielle Assis de Faria. - 2008.
390 f. ; il. ; 30 cm.
Tese (doutorado) ± Universidade C atólica de B rasília,
2008.
Orientação: Rinaldo Wellerson Pereira
1. Genética. 2. Eucalipto. 3. Clonagem molecular. 4. Melhoramento genético. I. Pereira, Rinaldo Wellerson, orient. II. Título.
7HVH GH DXWRULD GH 'DQLHOOH $VVLV GH )DULD LQWLWXODGD ³(678'26
GENÔMICOS EM Eucalyptus: MAPEAMENTO DE GENES CANDIDATOS E QTLs, ESTIMATIVA DE DESEQUILÍBRIO DE LIGAÇÃO E ANÁLISE DE ESTRUTURA
*(1e7,&$'(3238/$d®(6´DSUHVHQWDGDFRPRUHTXLVLWRSDUFLDOSara obtenção
do grau de Doutor em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 03 de outubro de 2008, defendida e aprovada pela banca examinadora abaixo assinada:
____________________________________________________ Prof. Dr. Rinaldo Wellerson Pereira
Orientador
Curso Ciências Genômicas e Biotecnologia/ Universidade Católica de Brasília ± UCB
____________________________________________________ Prof. Dr. Dário Grattapaglia
Co-Orientador
Curso Ciências Genômicas e Biotecnologia/ Universidade Católica de Brasília ± UCB
____________________________________________________
Prof.Dr. Alexandre Siqueira Guedes Coelho
Examinador Externo
Universidade Federal de Goiás ± UFG
____________________________________________________ Prof. Dr. Márcio Elias Ferreira
Examinador Externo
Embrapa Recursos Genéticos e Biotecnologia
____________________________________________________ Prof. Dr. Georgios Joannis Pappas Júnior
Examinador Interno
Curso Ciências Genômicas e Biotecnologia/ Universidade Católica de Brasília ± UCB
!
! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! ! !
Aos meus pais Joanita e Washington e ao meu esposo Samuel, Por acreditarem em mim e no meu trabalho! Por todo amor e compreensão!
AGRADECIMENTO
À Universidade Católica de Brasília e ao programa de Programa de
Pós-*UDGXDomR³Stricto Sensu´HP&LrQFLDV*HQ{PLFDVH%LRWHFQRORJLD, por apoiarem a
realização desse trabalho.
Ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq)
pela concessão da bolsa de estudos durante o curso.
Ao Prof. Rinaldo Wellerson Pereira por concordar em ser meu orientador e
junto comigo investir num campo ainda pouco explorado na genética de Eucalytpus.
Ao Dr. Dario Grattapaglia por acreditar no meu trabalho, pela amizade e pelos
infinitos ensinamentos, paciência e conselhos.
Ao Laboratório de Genética Vegetal ± Embrapa Recursos Genéticos e
Biotecnologia que me acolheu para realização de todos experimentos apresentados
neste trabalho.
Aos membros da banca examinadora: Dr. Alexandre Siqueira Guedes Coelho,
Dr. Márcio Elias Ferreira e Dr. Georgios Joanis Pappas Júnior, que durante a
execução da tese sempre estiveram à disposição para esclarecimentos sobre o
tema.
Aos demais pesquisadores da Embrapa: Dra. Gláucia Buso, Dra. Ana Ciampi,
Dra. Vânia Azevedo, Dr. Márcio Moretzsoh, Marco Antônio Ferreira e Zilneide
Amaral pelo carinho no laboratório e ajuda nos momentos difíceis.
Aos estagiários e estudantes que passaram pelo Laboratório de Genética
Vegetal nesse período, que de uma forma ou de outra se tornaram amigos e
companheiros de dúvidas e alegrias.
$RV PHXV ³ILOKRV´ H DPLJRV /HDQGUR *RPLGH H /HRQDUGR 4XHLUR] TXH PH
ajudaram dentro e fora do laboratório, sempre me acalmando e pegando no pesado
comigo quando necessário.
Aos estagiários do Genolyptus Laís Pinheiro e Mariana Caldas que além de
me ajudarem dentro do laboratório me deram a oportunidade de participar dos seus
projetos e aprender juntamente com elas também.
Aos meus amigos de Genolytpus: Carol, Cesar, Eva, Juliana e Juliano que
apesar dos momentos de aperto e às vezes de correria sempre estiveram ao meu
À pesquisadora Marília Pappas, que ao longo dessa caminhada se tornou
uma amiga especial sempre disposta a ajudar e consolar nos momentos difíceis
dessa jornada.
¬GXDVDPLJDVHVSHFLDLVH³SULPDV´7KDtVDH%UXQDTXHVHPSUHHVWLYHUDP
junto comigo no lab e fora dele para ouvir desabafos e conversar besteiras.
Agradeço muito às duas pois elas demonstraram ao longo da convivência
experiências de vida que me marcaram e mudaram a minha concepção de vida e
trabalho.
Aos meus pais que sempre acreditaram em mim e mesmo depois de casada,
eles sempre incentivaram meus objetivos profissionais e me apoiaram nos
momentos em que tive que decidir por mudanças radicais na minha carreira.
Ao meu amado esposo e melhor amigo do mundo, Samuca, que agüentou
todos os choros, todas as incertezas, ansiedades e dificuldades nessa caminhada.
Por ele sempre estar ao meu lado até altas horas no seqüenciador e/ou no
computador. Por me ajudar a descobrir como funcionam vários programas de
análise que nunca havíamos usado antes. Principalmente por me amar e me deixar
RESUMO
Faria, Danielle Assis de. Estudos genômicos em Eucalyptus: Mapeamento de genes
candidatos e QTLs, estimativa de desequilíbrio de ligação e análise de estrutura de
populações. 2008. 388. Tese de doutorado em Ciências Genômicas e Biotecnologia,
Universidade Católica de Brasília, Brasília ± DF, 2008.
As florestas plantadas com espécies do gênero Eucalyptus e seus híbridos
constituem a base da produção nacional de celulose, papel e carvão vegetal. Estes
setores envolvem empresas que executam melhoramento genético visando o
aumento da produtividade e qualidade produção de matéria prima florestal em nível
altamente competitivo. Neste cenário o uso de tecnologias genômicas associadas às
estratégias de melhoramento genético convencional pode acelerar e aumentar a
eficiência de todo o processo de melhoramento. Este trabalho envolveu um conjunto
de experimentos visando gerar informações para estudos de genética de associação
no gênero Eucalyptus no Brasil. Foram estudadas seis espécies pertencentes ao
gênero Eucalyptus utilizadas no melhoramento florestal no Brasil (E. grandis, E.
camaldulensis, E. urophylla, E. globulus, E. dunnii e E. saligna). Além das espécies puras, foram também avaliados clones híbridos espontâneos utilizados em plantios
operacionais. Trechos de genes candidatos selecionados foram seqüenciados em
amostras populacionais destas espécies. A identificação de genes e regiões
genômicas para futuros estudos de associação foi realizada utilizando mapeamento
de QTLs para diferentes características (volume, densidade básica, altura, DAP,
relação siringil:guiacil, teor de lignina e rendimento depurado) em uma família
envolvendo parentais híbridos (E. dunnii x E. grandis) X (E. urophylla x E. globulus). No mapa genético gerado com microssatélites foram posicionados os genes
candidatos COMT, C4H, SAMS, Celulose Synthase, CCR, F5H, MYB PAL,
foram encontrados 12 QTLs para todas as características. O grupo de ligação com a
maior concentração de QTLs encontrados foi o grupo seis. O mapeamento de genes
candidatos foi uma estratégia válida, pois foram encontrados dois genes (SAMS e
Alfatubulina) co-localizados em um intervalo contendo QTLs para características
correlacionadas. A estrutura genética existente nas amostras populacionais das seis
espécies foi avaliada com o programa STRUCTURE com base em 35 locos
microssatélites nucleares, 11 locos microssatélites cloroplastidiais e SNPs também
pertencentes ao genoma cloroplastidial. Os marcadores moleculares revelaram uma
clara diferenciação entre espécies e procedências dentro de espécies
geograficamente separadas, mas não para procedências próximas. Tanto os dados
moleculares autossômicos, como os dados de cloroplasto mostraram uma alta
diversidade genética nas seis espécies do gênero Eucalyptus. Os clones híbridos avaliados para os mesmos marcadores (microssatélites nucleares e cloroplastidiais;
e SNPs) foram alocados quanto à sua origem híbrida, nas espécies E. grandis e E.
urophylla. Os marcadores no cloroplasto revelaram uma origem materna
predominante de E. grandis. Estimativas de desequilíbrio de ligação (DL) derivadas
de marcadores SNPs revelaram que o DL em regiões gênicas não se estende além
de 1000 pares de base. As estimativas de DL, entretanto variam dependendo do
gene analisado e da espécie. De maneira geral, E. grandis e E. camaldulensis
apresentaram as menores extensões de DL. E. dunnii e E. globulus, apresentam
polimorfismos altamente correlacionados e em DL por até 1000 pb. Para a espécie
E. saligna, a extensão do DL ao longo do gene CAD é intermediária em relação às demais espécies. Já para o gene 4CL, os indivíduos analisados pertencentes a esta
espécie, demonstraram correlações significativas entre os polimorfismos
identificados, estendendo assim o DL por até 900 pares de base. Os clones também
avaliados quanto à extensão do desequilíbrio de ligação, apresentaram extensões
similares por até 400 a 500pb nos dois genes avaliados. Porém no gene 4CL, os
valores da correlação entre os SNPs par a par (r2) foram muito maiores do que os
valores encontrados no gene CAD. Levando em consideração a rápida queda do
desequilíbrio de ligação reportada aqui para o gênero Eucalyptus, a associação
entre genótipos e fenótipos encontrada via mapeamento de QTLs em uma
determinada família não pode ser estendida para outra família. Novas técnicas de
seqüenciamento e genotipagem em larga escala vêm aumentando o número de
PALAVRAS-CHAVE: desequilíbrio de ligação, estrutura genética, mapeamento de
associação, marcadores moleculares, melhoramento florestal, QTL.
ABSTRACT
!
Cellulose and charcoal dependent industries in Brazil are based on
commercial eucalyptus forests developed during the last fifty years in a well
established and highly competitive breeding program looking for improvements in
lignin or cellulose. Genomic based technologies bring to these programs the promise
to improve gains in time and productivity. DNA markers discovery and their
application in linkage mapping and association mapping represent the basic
application of genomics in any organism breeding program. This work involved a
series of experiments in order to generate information for genetic association studies
in Eucalyptus. We studied six species of the genus Eucalyptus used in breeding
programs in Brazil (E. grandis, E. camaldulensis, E. urophylla, E. globulus, E. saligna
and E. dunnii). Besides of pure species, expontaneous hybrid clones, used in
operational plantations, were also evaluated. Fragments of selected candidate genes
were sequenced on these species. The identification of genes and genomic regions
for future association studies were performed using QTL mapping for traits related to
growth and wood quality in a cross involving hybrid parents (E. dunnii x E. grandis) X
(E. urophylla x E. globulus). The genetic map generated with microsatellites was
used to position candidate genes as, COMT, C4H, SAMS, Cellulose Synthase, CCR,
F5H, MYB PAL, Alpha tubulin, among others, using SNPs genotyped by sequencing.
A total of twelve QTLs were found representing all phenotypes investigated with
exception to wood density measured by NIRS. Both parental maps showed a high
number of QTLs mapped to linkage group number six. Among the candidate genes,
two of them (SAMS and alfa-tubulin) were mapped in QTL intervals. Candidate gene
mapping in QTLs intervals showed to be a good strategy to select genes or regions
to be included in association mapping. Marker-assisted selection based on
long-range marker-trait association detected by QTL analysis may only be useful
within-family selection. Population genetic structure was evaluated using 35 autosomal, 11
chloroplast microsatellites loci, besides single nucleotide polymorphisms from
chloroplast genome. Both autosomal and chloroplast markers show that samples
in provenance level. Structured species based on provenance are those from distinct
Australian latitudes. For all kind of markers analyzed, the six species showed high
level of genetic diversity. Hibryd clones evaluated using the same molecular markers
were allocated as having their origin in E. grandis e E. urophylla. Specifically about
the maternal origin, chloroplast markers pointed to E. grandis. The results of genetic
population structure investigated here shows that we have to pay special attention in
hybrids to avoid spurious marked-trait association, since these hybrids are the most
used breeding population in Brazil. The genes CAD and 4CL, both from the
lignification pathway were resequenced in DNA samples from species and also in
hibryd clones. In general, significant linkage disequilibrium (LD) has been found no
longer than 1000 bp. The results were distinct based on gene or specie evaluated.
For CAD gene: E. grandis and E. camaldulensis showed rapidly LD decay. However,
in E. dunnii and E. globulus the decay does not start until 1000 bp. In E. saligna, the
LD showed intermediate values compared to other species. For 4CL gene: E. saligna
presented no LD decay in short distances when compared to the others species. For
the hybrids, the LD decay found was similar to that found in species, in the 4CL gene
analysis, the LD decay was slower than in the CAD gene. Considering the rapid
decay of LD reported here for Eucalytpus, the marked-trait association detected in one segregating population cannot be assumed to hold in a different pedigree. The
great challenge in the application of marker-assisted selection, is the low level of
linkage disequilibrium between genetic markers and QTLs in natural and breeding
populations. Next generation sequencing and highthroughput genotyping strategies
will increase the number of molecular markers available for use in marker-assisted
selection and this study aims to elucidate the genetic basis of association studies in
Eucalyptus.
KEYWORDS: association mapping, forest breeding, genetic structure, linkage
LISTA DE FIGURAS
CAPÍTULO 01
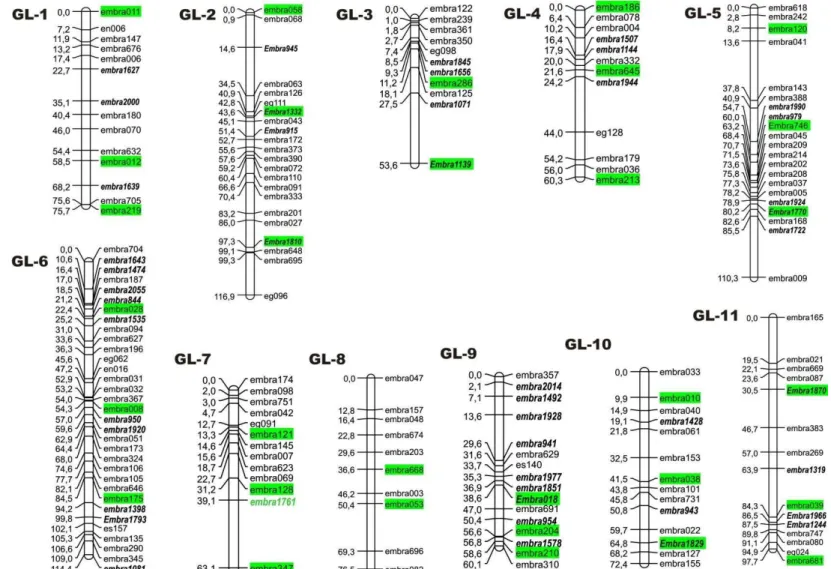
Figura 01± Posição de mapa dos microssatélites utilizados na análise de diversidade e estruturação
genética das populações de Eucalyptus. Em verde encontram-se assinalados os microssatélites no mapa genético construído para a população IP (Mammani & Grattapaglia, 2006a). ... 49!
Figura 02 ± Quantificação de DNA em gel de agarose 1% corado com brometo de etídeo para as
amostras de duas procedências da espécie E. grandis. Canaletas 1 a 3 (pente inferior e superior) ± padrões DNA de fago lambda (Invitrogen), concentração de 20Kg/PL, 50Kg/PL e 100Kg/PL, respectivamente. Canaletas 4 a 27 (pente superior) e 4 26 (pente inferior) ± amostras de DNA concentrado tratados com RNase. ... 54!
Figura 03 ± Eletroforese em gel de agarose 1% das amostras da espécie E. grandis diluídas para aproximadamente 2Kg/PL. Canaletas 1 e 2 (pente inferior e superior) ± padrões de DNA de fago lambda (Invitrogen), concentração de 10 Kg/PL e 20 Kg/PL, respectivamente. Canaletas 3 a 26 ± amostras de DNA diluídas onde foram aplicados 5µl. ... 55!
Figura 04± Dendrograma da relação genética entre as procedências e o conjunto de clones obtido
pela metodologia de agrupamento UPGMA a partir da matriz de distância genética de Nei (Nei, 1972). Valores apresentados entre os nós representam os valores de bootstrap com 1000 replicações. ... 70!
Figura 05± Dendrograma da relação genética entre as espécies e o conjunto de clones obtido pelo
método de agrupamento de UPGMA a partir da matriz de distância genética de Nei (Nei, 1972). Valores apresentados entre os nós representam os valores de bootstrap com 1000 replicações. ... 71!
Figura 06 ± Dendrograma da relação genética entre os indivíduos das espécies analisadas obtido
pela metodologia de agrupamento UPGMA a partir da proporção de alelos compartilhados. Verde ±E.
camaldulensis; Azul ±E. grandis; Vermelho ±E. urophylla; Laranja ±E. globulus; Amarelo ±E. dunnii
e Rosa ±E. saligna. ... 72!
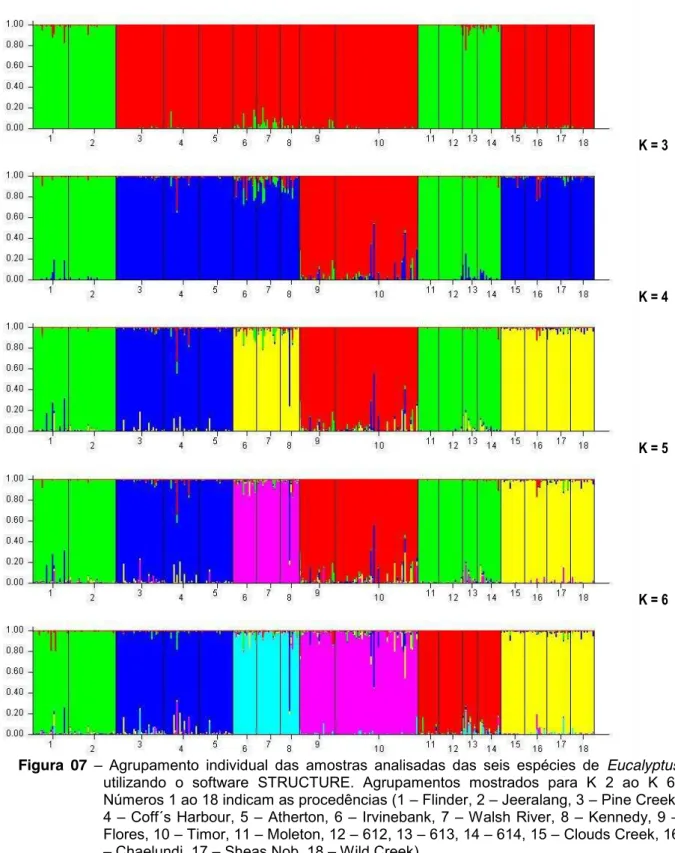
Figura 07 ± Agrupamento individual das amostras analisadas das seis espécies de Eucalyptus
utilizando o software STRUCTURE. Agrupamentos mostrados para K 2 ao K 6. Números 1 ao 18 indicam as procedências (1 ± Flinder, 2 ± Jeeralang, 3 ± Pine Creek, 4 ± Coff´s Harbour, 5 ± Atherton, 6 ± Irvinebank, 7 ± Walsh River, 8 ± Kennedy, 9 ± Flores, 10 ± Timor, 11 ± Moleton, 12 ± 612, 13 ± 613, 14 ± 614, 15 ± Clouds Creek, 16 ± Chaelundi, 17 ± Sheas Nob, 18 ± Wild Creek). ... 74!
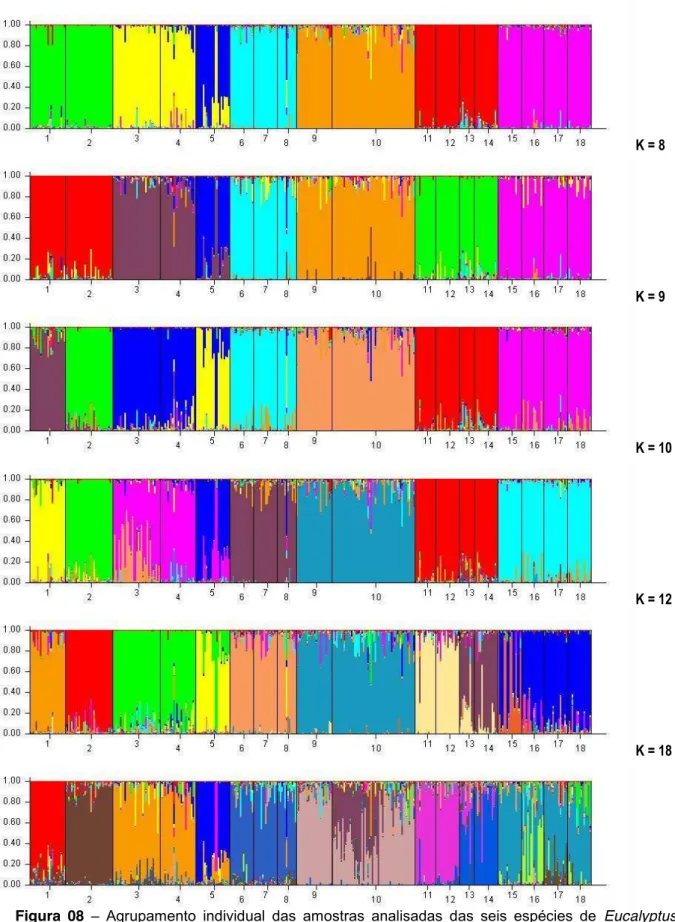
Figura 08 ± Agrupamento individual das amostras analisadas das seis espécies de Eucalyptus
utilizando o software STRUCTURE. Agrupamentos mostrados para K 7 ao K 18. Números um ao 18 indicam as procedências (1 ± Flinder, 2 ± Jeeralang, 3 ± Pine Creek, 4 ± Coff´s Harbour, 5 ± Atherton, 6 ± Irvinebank, 7 ± Walsh River, 8 ± Kennedy, 9 ± Flores, 10 ± Timor, 11 ± Moleton, 12 ± 612, 13 ± 613, 14 ± 614, 15 ± Clouds Creek, 16 ± Chaelundi, 17 ± Sheas Nob, 18 ± Wild Creek). ... 75!
Figura 01 ± Géis de agarose para algumas amostras do gênero Eucalyptus referentes aos três
Figura 02 ± Géis de agarose para algumas amostras do gênero Eucalyptus referentes aos seis fragmentos do gene 4CL amplificados para posterior seqüenciamento. Em cada gel encontra-se o tamanho do fragmento amplificado a partir de comparação com marcador molecular de peso conhecido. ... 114!
Figura 03. Blocos haplotípicos estimados para o gene CAD. 1 - E. camaldulensis; 2 - E-globulus; 3 -
E. saligna; 4 - E. grandis; 5 - E. urophylla; 6 - Clones;7 - E. dunni.i ... 120!
Figura 04. Blocos haplotípicos estimados para o gene 4Cl. 1 - E. camaldulensis; 2 - E-globulus; 3 - E.
saligna; 4 - E. grandis; 5 - E. urophylla; 6 - Clones;7 - E. dunnii. ... 122!
Figura 05. Representação Gráfica DL em Eucalyptus para o gene CAD. 1 - E. camaldulensis; 2 -
E-globulus; 3 - E. saligna; 4 - E. grandis; 5 - E. urophylla; 6 - Clones;7 - E. dunnii. ... 133!
Figura 06. Representação Gráfica DL em Eucalyptus para o gene 4Cl. 1 - E. camaldulensis; 2 -
E-globulus; 3 - E. saligna; 4 - E. grandis; 5 - E. urophylla; 6 - Clones;7 - E. dunnii. ... 140!
Figura 07± Gráficos representando a extensão do Desequilíbrio de ligação no gene CAD versus a
distância em pares de base. O desequilíbrio de ligação foi estimado a partir dos valores de r2. Os pontos vermelhos representam os valores de r2 para cada par de SNPs. ... 142!
Figura 08 - Gráficos representando a extensão do Desequilíbrio de ligação no gene CAD versus a
distância em pares de base. Desequilíbrio de ligação calculado baseado em valores de r2. Os pontos vermelhos representam os valores de r2SDUDFDGDSDUGH613VVLJQLILFDWLYRV/2' ... 143!
Figura 09 - Gráficos representando a extensão do desequilíbrio de ligação no gene 4CL versus a
distância em pares de base. Desequilíbrio de ligação calculado baseado em valores de r2. Os pontos vermelhos representam os valores de r2 para cada par de SNPs. ... 144!
Figura 01. Representação esquemática dos iniciadores desenhados para cada gene. A.
Representação dos quatro iniciadoresdesenhados, sendo dois posicionados externamente com base no consenso de EST (laranja) e dois internos (azul). B. Representação das quatro combinações possíveis de amplificação dos genes ilustrando os segmentos de tamanhos diferentes gerados a partir da referência. ... 159!
Figura 02 ± Representação gráfica em porcentagem do total de locos testados na população de
mapeamento do cruzamento DG x UGL. ... 173!
Figura 03± Triagem de genes candidatos nas famílias de mapeamento (IP e DGxUGL). ... 174!
Figura 04± Resumo da sucesso de mapeamento de ESTs nas famílias de mapeamento (IP e DG x
UGL). ... 175!
Figura 05 ± Alinhamento da seqüência gerada para o EST 981, evidenciando um dos SNPs que
apresentam alelos improváveis na progênie. ... 176!
Figura 06 ± Gráfico representando em porcentagem os locos microssatélites informativos para o
cruzamento DG x UGL. ... 177!
Figura 07. Produto da amplificação do EST 2727 analisado por eletroforese em gel de agarose 3%
Figura 08. Produto da amplificação de segmento do gene 2727 em gel de poliacrilamida 6% mostrando a segregação de alelos para um padrão indel. Segunda e terceira canaletas são relativas ao genitor masculino G38 (E. grandis) e ao feminino U15 (E. urophylla), respectivamente, seguidos por 15 indivíduos F1 e um controle negativo. A e B são codificações para os alelos do parental 1 (P1
± G38) e C e B para os alelos do parental 2 (P2 ± U15). Abaixo de cada indivíduo encontra-se seu genótipo com base nesta codificação. Primeira canaleta corresponde ao marcador DNA ladder 1kb
plus (Invitrogen®) e última ao marcador DNA ladder 10 pb. ... 182!
Figura 09± Mapas genéticos dos genitores masculino (UGL) e feminino (DG) construídos com base
em uma família de 188 irmãos completos de um cruzamento (E. dunnii x E. grandis) x (E. urophylla x
E. globulus) pela estratégia de pseudo cruzamento-teste. As linhas ligam os microssatélites
completamente informativos mapeados em ambos os parentais. Os marcadores destacados em verde são microssatélites originados de seqüências de EST. Os marcadores destacados em vermelho são os genes e ESTs mapeados neste cruzamento. Os grupos de ligação estão numerados de acordo com o mapa de referência para o gênero Eucalyptus (Brondani et al., 2006). ... 186!
Figura 10± Ancoragem dos genes e ESTs no mapa genético E. urophylla x E. grandis (IP) (Mamani
et al. 2006). Marcadores microssatélites genômicos apresentados em preto e aqueles originados de biblioteca de EST em verde; genes candidatos e ESTs apresentados em vermelho. ... 187!
Figura 11± Localização dos marcadores (sublinhados) ligados a QTLs para DAP no mapa de cada
parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. ... 189!
Figura 12 ± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para DAP mapeado por
intervalo composto no grupo de ligação 6 do genoma do genitor UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados pela análise de marca simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,6. ... 190!
Figura 13 ± Localização dos marcadores (sublinhados) ligados a QTLs para altura das árvores no
mapa de cada parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1%, ***Significativo a 0,1% pelo teste F. ... 191!
Figura 14 ± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para altura das árvores
mapeado por intervalo composto no grupo de ligação 6 do genoma do UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados pela análise de marca simples. *Significativo a 5% e **Significativo a 1%. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,6. ... 192!
Figura 15 ± Localização dos marcadores (sublinhados) ligados a QTLs para volume no mapa de
Figura 16 ± Gráficos gerados pelo QTL Cartographer demonstrando o QTL para volume mapeado por Intervalo Simples (linha preta) e Intervalo Composto (linha azul) no grupo de ligação 6 do genoma do UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples. **Significativo a 1% e ***Significativo a 0,1%. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,7. ... 194!
Figura 17 ± Localização dos marcadores (sublinhados) ligados a QTLs para Pylodin no mapa de
cada parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. ... 196!
Figura 18± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para Pylodin mapeado por
intervalo composto no grupo de ligação 8 do genoma do DG. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples. *Significativo a 5% e ***Significativo a 0,1%. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,6. ... 197!
Figura 19 ± Localização dos marcadores (sublinhados) ligados a QTLs para densidade básica no
mapa de cada parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. ... 198!
Figura 20 ± Localização dos marcadores (sublinhados) ligados a QTLs para densidade básica no
mapa de cada parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. ... 200!
Figura 21 ± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para teor total de lignina
mapeado por intervalo composto no grupo de ligação 3 do genoma do UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples. ***Significativo a 0,1%. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,6. ... 201!
Figura 22 ± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para teor total de lignina
mapeado por intervalo composto no grupo de ligação 5 do genoma do UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples. ***Significativo a 0,1%. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,6. ... 202!
Figura 23 ± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para teor total de lignina
Figura 24± Localização dos marcadores (sublinhados) ligados a QTLs para relação siringil/guaiacil no mapa de cada parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. ... 204!
Figura 25± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para relação siringil/guaiacil
mapeado por intervalo composto no grupo de ligação 5 do genoma do DG. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples.***Significativo a 0,1% O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,3. ... 205!
Figura 26± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para relação siringil/guaiacil
mapeado por intervalo composto no grupo de ligação 6 do genoma do DG. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples.***Significativo a 0,1% O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,3. ... 206!
Figura 27± Localização dos marcadores (sublinhados) ligados a QTLs para rendimento depurado de
celulose no mapa de cada parental construído pelo programa Mapmaker. Os locos apresentados na figura foram significativos apenas na análise de Marca Simples. *Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1%. ... 207!
Figura 28± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para rendimento depurado
de celulose mapeado por intervalo composto no grupo de ligação 3 do genoma do UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples.*Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1% O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,5. ... 208!
Figura 29± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para rendimento depurado
de celulose mapeado por intervalo composto no grupo de ligação 6 do genoma do UGL. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. Marcadores assinalados apresentaram-se significativamente correlacionados com o mesmo QTL pela análise de marca simples.*Significativo a 5%, **Significativo a 1% e ***Significativo a 0,1% O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,5. ... 209!
Figura 30± Gráfico gerado pelo QTL Cartographer demonstrando o QTL para rendimento depurado
de celulose mapeado por intervalo composto no grupo de ligação 5 do genoma do DG. Eixo Y: valor de LOD; Eixo X: intervalo entre os marcadores do grupo de ligação. O valor de LOD definido pelo teste de permutação com 5% de significância foi de 2,5. ... 210!
Figura 31± Localização dos QTLs, para todos os caracteres fenotípicos analisados, nos diferentes
Figura 32± Localização dos QTLs, para todos os caracteres fenotípicos analisados, nos diferentes no grupo de ligação do parental feminino DG. São demonstrados apenas os grupos de ligação e as características para as quais foram identificados QTLs por mapeamento de intervalo. As barras demonstram as regiões onde o valor de LOD ultrapassou o limiar definido pelo teste de permutação
FRP QtYHO GH VLJQLILFkQFLD GH DR QtYHO GH WRGR R H[SHULPHQWR ³H[SHULPHQWZLVH´ $V VHWDV
localizam a região mais provável de cada QTL. ... 220!
Figura 01 ± Mapa do continente Australiano ilustrando a localização geográfica das procedências
onde foram coletadas as sementes utilizadas neste trabalho. ... 230!
Figura 02 ± Representação do genoma cloroplastidial de E. globulus evidenciando a posição dos
marcadores microssatélites utilizados neste trabalho. Figura modificada a partir de Steane (2005). 233!
Figura 03 ± Gel de agarose 1% corado com brometo de etídeo, exemplificando a quantificação do
DNA das amostras utilizadas. Primeira e segunda linha ± canaleta 1: O50Kg/Pl; canaleta 2: O100Kg/Pl; demais canaletas 2Pl das amostras de DNA total. ... 242!
Figura 04 ± Eletroferogramas ilustrando a amplificação dos locos de microssatélites cloroplastidiais
em amostras de diferentes espécies ... 243!
Figura 05± Análise de network a partir dos haplótipos obtidos pelos onze locos de microssatélites.
Cada círculo representa um haplótipo e a área do círculo representa a freqüência haplotípica. mv são vetores médios que representam haplótipos hipotéticos não amostrados neste estudo. Em laranja ± E. globulus, vermelho ± E. urophylla, azul ± E. grandis, verde ± E. camaldulensis, rosa ± E. saligna, amarelo ± E. dunnii. ... 259!
Figura 06 - Análise de network a partir dos haplótipos obtidos pelos SNPs e seqüências de bancos
de dados. Cada círculo representa um haplótipo e a área do círculo representa a freqüência haplotípica. mv são vetores médios que representam haplótipos hipotéticos não amostrados neste estudo. Em laranja ± E. globulus, vermelho ± E. urophylla, azul ± E. grandis, verde ± E. camaldulensis, rosa ± E. saligna, amarelo ± E. dunnii. Em vermelho sobre as linhas que interligam os haplótipos estão representados os pontos mutacionais encontrados em pares de base. Em azul claro as espécies cujas seqüências foram obtidas no GenBank; Co: amostras do gênero Corymbia. ... 266!
Figura 07± Gráficos representando a freqüência alélica encontrada para cada loco no conjunto de 51
clones avaliados. ... 268!
Figura 08± Dendrograma baseado na análise de cada amostra individual gerado a partir da matriz
de compartilhamento de alelos dos 11 locos de microssatélites entre os clones híbridos (amarelo) e os indivíduos das espécies E. grandis (azul), E. urophylla (vermelho), E. saligna (roxo) e E. camaldulensis (verde). ... 269!
Figura 09 - Dendrograma baseado na análise de cada amostra individual gerado a partir da matriz de
compartilhamento de alelos dos cinco locos de microssatélites com maiores valores de diversidade gênica entre os clones híbridos (CL) e os indivíduos das espécies E. grandis (GC, GP e GA), E. urophylla (UT, UF). Valores nos ramos correspondem a valores de bootstraping (1000 repetições).270!
Figura 10 - Análise de network a partir dos haplótipos obtidos pelos SNPs entre espécies
± E. saligna, preto ± Clones. Em vermelho, sobre as linhas que interligam os haplótipos estão representados os pontos mutacionais encontrados em pares de base... 272!
Figura 11± Cladograma obtido por neighbor joining a partir do modelo de máxima verossimilhança
LISTA DE TABELAS
CAPÍTULO 01
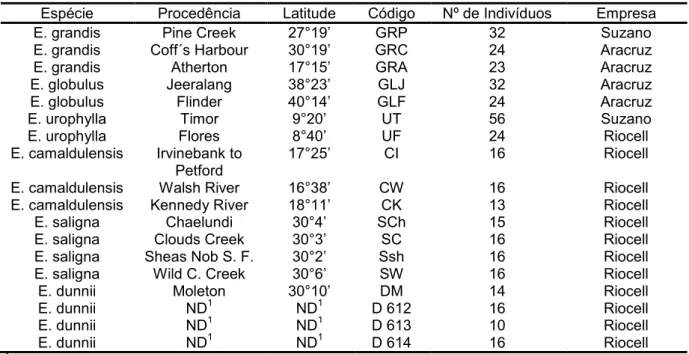
Tabela 01 ± Espécie, procedência, latitude aproximada, código utilizado, número de indivíduos e
empresa fornecedora das árvores utilizadas no estudo. ... 45!
Tabela 02 ± Hibridização, código utilizado, número de indivíduos e empresa fornecedora para as
amostras de clones híbridos utilizadas para o experimento. ... 46!
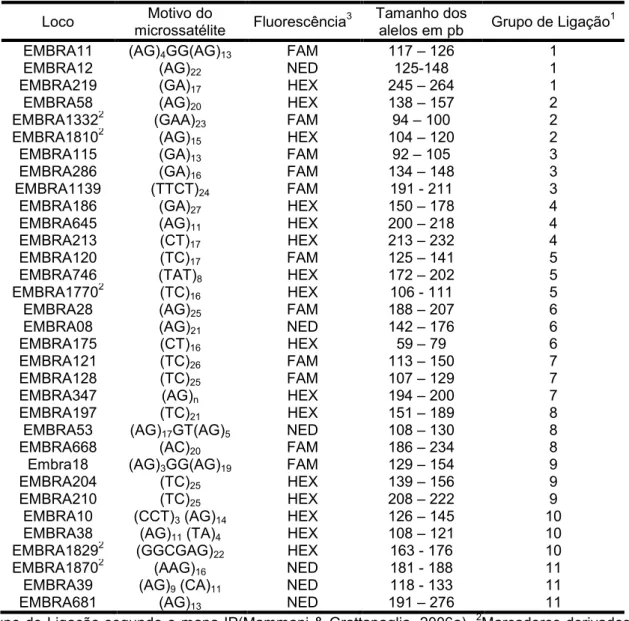
Tabela 03 ± Microssatélites utilizados na análise de diversidade e estruturação genética das
populações de Eucalyptus. ... 48!
Tabela 04± Locos de microssatélites selecionados após a triagem. ... 58!
Tabela 05 ± Sistemas de co-amplificação (multiplex) utilizados para a análise dos 35 locos
microssatélites visando a análise de diversidade e de estrutura genética. ... 59!
Tabela 06 ± Estatísticas descritivas para os 35 locos microssatélites analisados para todas as
procedências estudadas e para o conjunto de clones. Número de alelos (Na), Heterozigosidade Esperada (He), Heterozigosidade Observada (Ho), Conteúdo de informação polimórfica (PIC), Coeficiente de endocruzamento entre indivíduos em relação ao total (FIT), diferença em pares de base entre o menor e o maior alelo encontrado (Amplitude Alélica) e Riqueza alélica (AR). ... 60!
Tabela 07 ± Resumo dos parâmetros genéticos populacionais para as procedências avaliadas
baseado em 35 locos de microssatélites. N ± número de indivíduos; Ne ± média do número efetivo de alelos; Amplitude alélica ± média da diferença em pares de base entre o menor e o maior alelo encontrado em todos os locos; He ± heterozigosidade esperada; Ho ± heterozigosidade observada; EHW ± número de locos que apresentaram desvios no equilíbrio de Hardy-Weinberg; FIS ±
Coeficiente de endocruzamento intrapopulacional e o p-valor do teste de significância; PIC ± Conteúdo de informação polimórfica. ... 64!
Tabela 08± Resumo dos parâmetros genéticos populacionais para as espécies e conjunto de clones
avaliados baseado em 35 locos de microssatélites. N ± número de indivíduos; Ne ± média do número efetivo de alelos; Amplitude alélica ± diferença em pares de base entre o menor e o maior alelo encontrado; He ± heterozigosidade esperada; Ho ± heterozigosidade observada; EHW ± número de locos que apresentaram desvios no equilíbrio de Hardy-Weinberg; FIS ± Coeficiente de
endocruzamento intrapopulacional; PIC ± Conteúdo de informação polimórfica. ... 65!
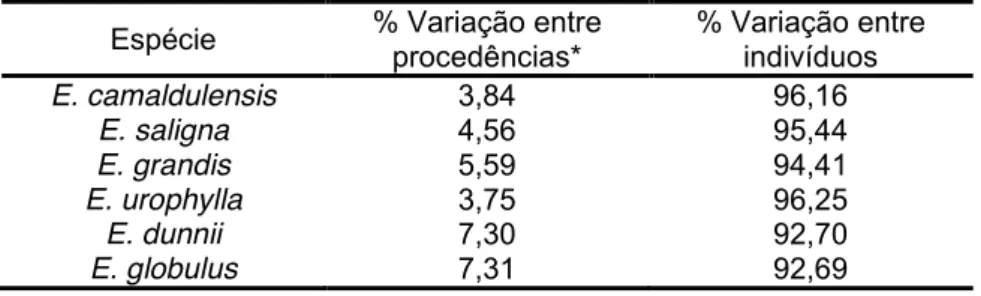
Tabela 09 ± Análise de variância molecular (AMOVA) em diferentes níveis de estruturação entre e
dentro de procedências e espécies de Eucalyptus estudadas. ... 66!
Tabela 10 ± Análise de variância molecular (AMOVA) entre e dentro de procedências para cada
espécie estudada. ... 66!
Tabela 11 ± Estimativa par a par de diferenciação genética (valores de FST ± diagonal superior) e
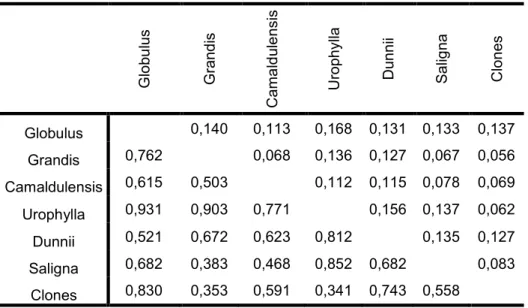
distância genética (distância de Nei ± diagonal inferior) entre todas as procedências e conjunto de clones avaliados. Barras entre as procedências separam as espécies. nsValores de Fst não
Tabela 12± Estimativas par a par de diferenciação genética (valores de FST - acima da diagonal) e
distância genética (distância de Nei - abaixo da diagonal) para o conjunto de espécie e clones avaliados. Todas as estimativas de FSTIRUDPVLJQLILFDWLYDVS ... 69!
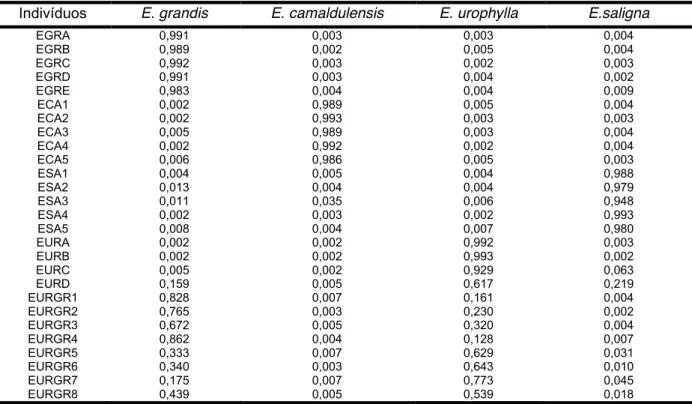
Tabela 13 ± Probabilidades de alocação de indivíduos das espécies puras (E. grandis -EGR, E.
urophylla -EUR, E. saligna ±ESA e E. camaldulensis -ECA) bem como indivíduos híbridos F1
derivados de cruzamentos controlados de E. grandis e E. urophylla (EURGR) com base na análise de 35 locos microssatélites. ... 78!
Tabela 14 ± Probabilidades de alocação dos 54 clones analisados nas espécies E. grandis, E.
urophylla, E. saligna e E. camaldulensis com base na análise de 35 locos microssatélites. ... 79
CAPÍTULO 02
Tabela 01 ± Espécie, procedência, latitude aproximada, código utilizado, número de indivíduos e
empresa fornecedora para as amostras de espécie pura utilizadas para o experimento. ... 101!
Tabela 02 ± Iniciadores desenhados a partir da seqüência completa do gene CAD e 4Cl obtidas
através do seqüenciamento e anotação de BACs contendo os genes. Cada primer seguido pela seqüência 5´- 3´... 104!
Tabela 03 ± Temperatura de anelamento para as combinações de iniciadores utilizados para o
reseqüenciamento dos genes CAD e 4CL. ... 105!
Tabela 04 ± SNPs apresentados pelas espécies estudadas e o conjunto de clones fixados e
diferentes da referência utilizada para montagem e alinhamento do gene CAD. ... 112!
Tabela 05± SNPs identificados nas amostras das seis espécies puras de Eucalyptus seqüenciadas e
um conjunto de clones elite para as regiões do gene CAD. ... 113!
Tabela 06 ± SNPs apresentados pelas espécies estudadas e o conjunto de clones fixados e
diferentes da referência utilizada para montagem e alinhamento do gene 4CL. ... 115!
Tabela 07 ± Resumo dos SNPs encontrados nas amostras das seis espécies seqüenciadas e o
conjunto de clones para as regiões do gene 4CL. ... 116!
Tabela 08± (VWLPDWLYDGDGLYHUVLGDGHKDSORWtSLFDQDVUHJL}HVJrQLFDVVHTHQFLDGDVʌSDUDFDGD
população estudada. ... 117!
Tabela 09 ± Sumário dos SNPs utilizados efetivamente pelo programa para estimativas de
desequilíbrio de ligação nos dois genes seqüenciados nas amostras de espécies puras e clones híbridos. ... 118
Tabela 10. Discriminação dos Tag SNP identificados para cada espécie de Eucalyptus estudada. 125
CAPÍTULO 03
Tabela 01± Nome, seqüência, resultado do BLAST X e temperatura de anelamento dos iniciadores
Tabela 02 ± Nome, seqüência e temperatura de anelamento dos iniciadores disponíveis no laboratório de trabalho anteriores, para genes candidatos da rota de lignificação e formação de parede celular (veja texto). ... 163!
Tabela 03 ± Estatísticas descritivas das características fenotípicas avaliadas em 188
irmãos-completos. ... 171!
Tabela 04 ± Locos de EST informativos para o cruzamento DG x UGL, motivo de repetição e
similaridade encontrada pela ferramenta BLAST com o banco de dados GenBank. ... 178!
Tabela 05 ± Genes e ESTs mapeadas em dois cruzamentos (G38 x U15 ± DG x UGL) a partir da
segregação de haplótipos de SNPs genotipados por re-seqüenciamento da progênie segregante. . 180!
Tabela 06 ± Resumo dos genes e ESTs posicionados nos mapas de ligação de duas famílias do
gênero Eucalyptus. ... 188!
Tabela 07 ± Sumário dos QTLs detectados pelas metodologias de mapeamento de Intervalo pela
estratégia de pseudo-cruzamento teste. Estão descritos os genitores e os grupos de ligação onde os QTLs foram detectados, bem como os seus valores de LOD, porcentagem da variação explicada e os marcadores que os flanqueiam. ... 218
CAPÍTULO 04
Tabela 01 ± Espécie, seção taxonômica, procedência, latitude aproximada, código utilizado e número de indivíduos para as amostras de indivíduos das espécies estudadas do subgênero Symphyomyrtus. ... 231!
Tabela 02 ± Número e origem dos clones elite estudados. ... 231!
Tabela 03 ± Loco, seqüência, posição, motivo de repetição, tamanho esperado do fragmento original seqüenciado e marcação fluorescente utilizada para os iniciadores de microssatélites de cloroplasto genotipados. ... 234!
Tabela 04 ± Iniciadores, seqüência e temperatura de desnaturação utilizadas para obtenção das seqüências do genoma de cloroplasto. ... 234!
Tabela 05 ± Sistemas multiplex, locos e temperaturas de anelamento utilizados na PCR para os locos de microssatélites de cloroplasto. ... 235!
Tabela 06 ± Locos de microssatélites cloroplastidiais e suas características nas seis espécies estudadas. ... 245!
Tabela 07 ± Freqüências alélicas e diversidade gênica dos onze locos de microssatélites cloroplastidiais estudados em seis espécies de Eucalytpus. ... 246!
Tabela 08 ± Verificação da origem materna de haplótipos cloroplastidiais nos trios originados de polinização controlada. ... 249!
Tabela 09 ± Freqüências haplotípicas e haplótipos encontrados nas procedências analisadas de seis espécies de Eucalyptus. ... 251!
Tabela 11- Matriz de diferenciação genética par a par (FST) baseada em freqüências haplotípicas entre as 17 procedências de Eucalyptus estudadas... 256!
Tabela 12 ± Matriz de diferenciação genética (FST) estimada entre as espécies de Eucalyptus estudadas baseada na metodologia da contagem do número de alelos diferentes entre dois haplótipos. ... 257!
Tabela 13 ± Matriz de diferenciação genética (FST) estimada entre as espécies de Eucalyptus estudadas baseada na metodologia de freqüências haplotípicas. ... 257!
Tabela 14 ± Análise de variância molecular (AMOVA) para haplótipos de onze microssatélites cloroplastidiais entre as espécies de Eucalyptus estudadas e procedências dentro de espécies. .... 257!
Tabela 15 ± Análise de variância molecular (AMOVA) para haplótipos de onze microssatélites cloroplastidiais entre e dentro de procedências para cada espécie estudada. ... 258!
Tabela 16 ± Espécies, Gênero, Subgênero e Seção taxonômica das seqüências retiradas do banco de dados GenBank. ... 260!
Tabela 17 ± Haplótipos obtidos a partir do seqüenciamento de 517 pb do genoma cloroplastidial nas 93 amostras analisadas de seis espécies do gênero Eucalyptus. N= número de indivíduos encontrados para cada haplótipo; Freq.=freqüência observada de cada haplótipo. ... 261!
Tabela 18 ± Freqüência haplotípica e diversidade nucleotídica (S) por espécie de Eucalyptus utilizada. ... 262!
Tabela 19 ± Haplótipos encontrados através do alinhamento das amostras utilizadas e as seqüências de outras espécies disponíveis no GenBank. ... 264!
SUMÁRIO
INTRODUÇÃO GERAL ... 26
GENÉTICA E MELHORAMENTO DE EUCALYPTUS ... 26
MARCADORES MOLECULARES,MAPAS GENÉTICOS E QTLS ... 29 GENÉTICA DE ASSOCIAÇÃO... 32
OBJETIVOS ... 35 REFERÊNCIA BIBLIOGRÁFICAS ... 36
CAPÍTULO 1: VARIABILIDADE E ESTRUTURAÇÃO GENÉTICA EM
POPULAÇÕES NATURAIS E CLONES ELITE DE EUCALYPTUS ... 41
1.1RESUMO ... 41 1.2INTRODUÇÃO ... 43 1.3MATERIALEMÉTODOS ... 45
1.3.1 Material Genético ... 45 1.3.2 Extração e Quantificação de DNA ... 46 1.3.3 Escolha dos locos de Microssatélites ... 46 1.3.4 Amplificação por PCR ... 50 1.3.5 Eletroforese Capilar ... 50 1.3.6 Análises Estatísticas... 51
1.4RESULTADOS ... 54
1.4.1 Extração de DNA ... 54 1.4.2 Análises dos locos de microssatélites ... 55 1.4.3 ± Diversidade Genética entre espécies e/ou procedências de Eucalyptus spp. ... 62 1.4.4 ± Estrutura genética das populações de Eucalyptus spp. ... 72 1.4.5 ± Testes de Alocação de Clones híbridos plantados no Brasil. ... 76
1.5DISCUSSÃO ... 80
1.5.1 Análises dos locos de microssatélites ... 80 1.5.2 ± Diversidade Genética entre espécies e/ou procedências de Eucalyptus spp. ... 84 1.5.3 ± Estrutura genética das populações de Eucalyptus spp. ... 87 1.5.4 ± Testes de Alocação de Clones híbridos plantados no Brasil. ... 88
1.6REFERENCIASBIBLIOGRÁFICAS... 92
CAPÍTULO 2: ESTIMATIVAS DE DESEQUILÍBRIO DE LIGAÇÃO EM REGIÕES GÊNICAS DE EUCALYPTUS SPP. ... 96
2.1RESUMO ... 96 2.2INTRODUÇÃO ... 98 2.3MATERIALEMÉTODOS ... 100
2.3.1 Material Vegetal e Extração de DNA ... 100 2.3.2 Regiões Genômicas e Iniciadores escolhidos ... 102 2.3.3 Amplificação dos fragmentos gênicos via PCR ... 105 2.3.4 Purificação e reação de seqüenciamento dos fragmentos gênicos ... 106 2.3.5 Alinhamento das seqüências e análises estatísticas ... 108
2.4RESULTADOS ... 110
2.4.3 Estimativas de Tag SNPs a partir dos Polimorfismos encontrados ... 123 2.4.4 Estimativas de Desequilíbrio de Ligação ... 126
2.5DISCUSSÃO ... 144 2.6REFERÊNCIASBIBLIOGRÁFICAS... 148
CAPÍTULO III: MAPEAMENTO DE QTLS PARA QUALIDADE DA MADEIRA EM UMA POPULAÇÃO SEGREGANTE ENVOLVENDO QUATRO ESPÉCIES DE EUCALYPTUS ... 150
3.1RESUMO ... 150 3.2INTRODUÇÃO ... 152 3.3MATERIALEMÉTODOS ... 154
3.3.1 Material Vegetal e extração de DNA ... 154 3.3.2 Avaliação das características quantitativas ... 155 3.3.3 Triagem e seleção de locos microssatélites ... 157 3.3.4 Triagem e seleção de genes candidatos ... 157 3.3.5 Amplificação dos locos microssatélites e segmentos gênicos via PCR.... 165 3.3.6 Geração dos dados genotípicos dos locos microssatélites ... 166 3.3.7 Geração dos dados genotípicos dos genes e ESTs ... 166 3.3.8 Construção do mapa genético ... 168 3.3.9 Detecção dos QTLs por Marca Simples, Intervalo Simples e Intervalo Composto ... 169
3.4RESULTADOS ... 170
3.4.1 Avaliação das Características Fenotípicas ... 170 3.4.2 Triagem e seleção de microssatélites ... 171 3.4.3 Triagem e seleção de genes candidatos ... 173 3.4.4 Geração dos dados genotípicos dos microssatélites, genes e ESTs ... 176 3.4.5 Construção dos mapas genéticos pela estratégia de pseudo-cruzamento teste ... 182 3.4.6 Análises de QTL ... 188 3.4.7 QTLs para diâmetro das árvores à altura do peito (DAP) ... 189 3.4.8 QTLs para altura das árvores ... 190 3.4.9 QTL para volume ... 192 3.4.10 QTL para densidade da madeira pela profundidade de penetração do Pylodin... 195 3.4.11 QTLs para Densidade Básica da Madeira predita via NIRs ... 197 3.4.12 QTLs para teor de lignina total da madeira ... 199 3.4.13 QTLs para relação Siringil/Guaiacil da madeira ... 203 3.4.14 QTLs para rendimento depurado de celulose ... 206
3.5DISCUSSÃO ... 211
3.5.1 Triagem e seleção de marcadores microssatélites, genes e ESTs ... 211 3.5.2 Mapeamento de QTLs para características de desempenho e qualidade da madeira ... 215 3.5.3 Análise da co-localização de QTLs ... 217
3.6REFERÊNCIASBIBLIOGRÁFICAS... 223
CAPÍTULO 4 VARIABILIDADE HAPLOTÍPICA NO GENOMA DO CLOROPLASTO E SUA UTILIZAÇÃO PARA A CARACTERIZAÇÃO DE POPULAÇÕES E CLONES DE EUCALYPTUS ... 226
4.3.1 Material Genético ... 230 4.3.2 Extração e Quantificação de DNA ... 232 4.3.3 Iniciadores utilizados na PCR ... 233 4.3.4 Amplificação via PCR e visualização dos polimorfismos ... 235 4.3.5 Genotipagem e análise das seqüências ... 237 4.3.6 Análises Estatísticas... 239
4.4RESULTADOS ... 242
4.4.1 Extração e quantificação de DNA ... 242 4.4.2 Transferibilidade e polimorfismo dos locos de microssatélites ... 242 4.4.3 Verificação da origem materna ... 248 4.4.4 Diversidade haplotípica e estrutura genética inter e intra-específica
baseada em locos de microssatélites ... 249 4.4.5 Diversidade haplotípica e estrutura inter e intra-específica baseada em SNPs ... 259 4.4.6 Estimativa da origem materna dos clones híbridos por haplótipos de cpDNA ... 266
4.5DISCUSSÃO ... 274
4.5.1 Transferibilidade e polimorfismo dos locos de microssatélites ... 274 4.5.2 Verificação da Origem Materna ... 275 4.5.3 Diversidade haplotípica e estrutura inter e intra-específica baseada em locos de microssatélites ... 276 4.5.4 Diversidade haplotípica e estrutura inter e intra-específica baseada em SNPs ... 278 4.5.5 Inferência da origem materna dos clones híbridos elite com base nos haplótipos de cpDNA ... 280
4.6REFERÊNCIASBIBLIOGRÁFICAS... 282
ANEXO 1 ± CAPÍTULO I ... 284
INTRODUÇÃO GERAL
!
GENÉTICA E MELHORAMENTO DE EUCALYPTUS
As espécies de Eucalyptus são nativas da Austrália e ilhas ao norte da Oceania (Potts & Pederick, 2000) e estão entre as espécies mais plantadas no
mundo (Doughty, 2000). A linhagem do gênero Eucalyptus é antiga, provavelmente
da era Mezozóica no período Cretáceo a cerca de 70 milhões de anos (Crisp, 2004).
Taxonomicamente, o gênero Eucalyptus é de difícil classificação, apresentando
ambigüidades semelhantes aquelas encontradas para o gênero Pinus (Liston et al.,
1999; Qiao et al., 2007; Syring et al., 2005). Esta dificuldade reside, provavelmente,
no fato de que variações clinais, convergências morfológicas e hibridizações entre
subgêneros sejam comuns. Atualmente, são reconhecidas aproximadamente 700
espécies tendo suas origens em 13 principais linhagens evolutivas (Brooker, 2000).
O gênero abrange desde pequenos arbustos até as árvores mais altas do mundo. As
espécies do subgênero Symphyomyrtus são as mais utilizadas comercialmente.
O gênero é predominantemente polinizado por animais tais como: insetos,
pássaros, marsupiais e algumas espécies de morcegos (Barbour et al., 2005). O tipo
de reprodução é preferencialmente alógamo, sendo descritos até o momento poucos
casos de autofecundação expontânea (Eldridge et al., 1993a). Também não é
comum o intercruzamento entre subgêneros distintitos, porém dentro de cada
subgênero o cruzamento interespecífico é comum (Potts & Dungey, 2004).
Cruzamentos híbridos artificiais têm sido produzidos e em geral a inviabilidade do
híbrido aumenta com o distanciamento taxonômico entre os pais (Potts & Dungey,
2004).
A partir do século 18, diversas espécies do gênero Eucalyptus foram
introduzidas em países como Índia, França, Chile, Brasil, África do Sul e Portugal
onde apresentaram excelente adaptação climática, sucesso reprodutivo e índices
produtivos elevados (Potts et al., 2004). A partir da década de 1960, com o
desenvolvimento de métodos industriais de processamento das fibras curtas do
eucalipto, as plantações de espécies do gênero começaram a apresentar
importância comercial crescente e os programas de melhoramento tiveram início no
posteriormente, testes procedência/progênie foram realizados para a espécie E.
camaldulensis em vários países (Eldridge et al., 1993b). Em 1970 iniciaram-se as
plantações clonais no Congo e no Brasil com destaque para a espécie E. urophylla
principalmente em função da maior facilidade de propagação vegetativa desta
espécie. A partir de 1980, populações base de melhoramento foram formadas,
envolvendo principalmente as espécies E. grandis, E. tereticornis e E. viminalis (Eldridge et al., 1993b).
Segundo Ferreira (1992), depois da Austrália e Indonésia, o Brasil é o país
com maior acervo genético de Eucalyptus spp. Em 1904, Edmundo Navarro Andrade
introduziu oficialmente o gênero no Brasil com a criação do Horto de Rio Claro, onde
diversas espécies nativas da Austrália foram plantadas. As sementes originadas
dessas árvores do Horto de Rio Claro constituíram a base inicial para o início dos
programas de plantio e melhoramento genético de eucalipto no país. As árvores que
apresentaram melhor crescimento, adaptabilidade e tolerância a doenças em
plantios comerciais eram, em sua grande maioria, híbridos espontâneos oriundos de
eventos de cruzamentos interespecíficos não controlados que ocorreram no Horto
de Rio Claro. Estas árvores elite foram as primeiras a serem utilizadas
exaustivamente no processo de clonagem para formação de populações comerciais.
Entre 1970 e 1984, aproximadamente 2.200 lotes de sementes de 55
espécies foram incluídos na pesquisa florestal brasileira. A importação destas
sementes e a execução dos testes de espécie/procedência/progênies dos materiais
genéticos mais adaptados permitiram estabelecer as diretrizes para a instalação de
populações-base com o objetivo principal de recombinação de genótipos superiores
e a produção de sementes (Oda et al., 2007).
Atualmente, os principais produtos obtidos a partir da madeira de Eucalyptus
no Brasil são a celulose, papel e carvão vegetal para a indústria siderúrgica. O país
possui cerca de 3,4 milhões de hectares cobertos por Eucalyptus e em 2007,
segundo dados da Bracelpa (Associação Brasileira de Celulose e Papel), produziu
11,9 milhões de toneladas de celulose e 8,96 milhões de toneladas de papel. Para o
ano de 2008 as projeções são de crescimento de 7,4% na produção de celulose e
3,2% na produção de papel.
O termo clone é comumente utilizado para identificar uma árvore superior
propagada vegetativamente, tanto para fins de plantio operacional quanto para atuar
clonagem é integralmente transmitido à descendência, permitindo assim capturar
toda a superioridade daquela árvore. Esta metodologia representou um grande salto
nos programas de melhoramento especialmente para as características silviculturais
e de qualidade da madeira.
Na produção de celulose e papel, florestas clonais de híbridos de E. grandis e
E. urophylla selecionados durante a década de 1980 permitiram avanços importantes em características de produção e resistência a doenças. Porém,
atualmente, a espécie E. globulus vem sendo intensamente utilizada na produção de
híbridos de segunda geração visto que esta espécie apresenta excelentes
propriedades físicas e químicas da madeira. O emprego de E. globulus e seus
híbridos no Brasil ainda é restrito em razão desta espécie ser adaptada
fisiologicamente a ambientes temperados demandando assim a produção de
cruzamentos absorventes e seleção direcional visando combinar adaptabilidade,
crescimento e qualidade da madeira. O sucesso do seu crescimento como espécie
pura na América do Sul, só é observado em parte da região Sul do país e em países
como o Chile, Uruguai e Argentina.
Discussões, como a exposta anteriormente, foram potencializadas,
principalmente, em razão do desenvolvimento e adaptação de novas estratégias e
metodologias paralelas ao melhoramento tradicional. Desta forma, com o advento
das técnicas de biotecnologia e genômica, procura-se hoje a união do melhoramento
clássico ao melhoramento molecular para acelerar o processo de obtenção de
materiais com características específicas, auxiliando a busca de plantas com alto
desempenho no campo.
Com objetivos GH VH HYLWDU R ³DSDJmR IORUHVWDO´ TXH SRGH YLU D SHUWXUEDU D
economia nacional futuramente, o melhoramento genético e, nele, a utilização de
tecnologias genômicas para a seleção acelerada de genótipos superiores representa
uma ferramenta de grande potencial de utilização. A associação encontrada entre
um marcador de DNA e uma característica quantitativa, permite potencialmente a
redução do tempo para seleção de materiais geneticamente superiores, visto que,
teoricamente, possibilita a seleção em idade precoce antes mesmo de esperar o
crescimento da árvore e a manifestação do fenótipo.
Em termos de genômica, o eucalipto é uma planta diplóide com um número
haplóide de 11 cromossomos (Eldridge et al., 1993b). A poliploidia não acontece de
(Grattapaglia & Bradshaw, 1994) a 644 Mpb (E. globulus) (Pinto, 2005), próximo em
tamanho ao genoma das espécies do gênero Populus (473 Mpb) (Poke et al., 2005)
e significativamente menor do que Pinus com cerca de 23 Gpb (Ahuja, 2001).
MARCADORES MOLECULARES, MAPAS GENÉTICOS E QTLS
Os marcadores moleculares têm auxiliado o melhoramento de espécies
florestais principalmente permitindo a identificação e descriminação de clones elite,
em processos de proteção varietal, caracterização de coleções de germoplasma e
populações de melhoramento, predição de desempenho com base na distância
genética entre os genitores e gerenciamento de variabilidade genética. Os
marcadores utilizados podem se localizar no genoma nuclear ou em organelas
citoplasmáticas como mitocôndrias e cloroplastos.
Várias classes de marcadores moleculares podem ser aplicadas para auxiliar
o melhoramento florestal. Porém, marcadores com alta repetibilidade e
codominantes, como os microssatélites e os polimorfismos de base única (Single
Nucleotide Polymorphisms ± SNPs), tendem a ser mais utilizados em razão de
menor dificuldade de intercâmbio de dados entre diversos núcleos de melhoramento.
Adicionalmente, com o desenvolvimento das teorias de genética quantitativa,
reconheceu-se que caracteres complexos ou quantitativos são controlados pelos
mesmos fatores hereditários descritos por Mendel, com diferença que vários fatores,
cada um contribuindo com uma pequena parcela, estão envolvidos na expressão do
caráter.
Na década de 1980, com o surgimento de diversas classes de marcadores,
baseados tanto em hibridação com sondas marcadas como em Reações da
Polimerase em Cadeia (PCR), tornou-se possível a busca de regiões do genoma
responsáveis por parte da variabilidade fenotípica em qualquer espécie de interesse
(Tanksley, 1993). Entre as várias ferramentas de genética molecular disponíveis
para auxílio ao melhoramento, os mapas genéticos baseados na freqüência de
recombinação entre marcadores polimórficos em uma progênie, tornaram-se
ferramentas fundamentais para acelerar o processo de seleção de marcadores
associados a caracteres quantitativos.
Os mapas genéticos permitem a localização de genes e regiões genômicas
relação estatística entre regiões genômicas e dados fenotípicos de indivíduos de um
dado cruzamento. O mapeamento genético em espécies florestais possui
peculiaridades quando comparado às culturas agrícolas anuais. As espécies
florestais possuem longo ciclo de vida e alta depressão por endogamia o que
dificulta a obtenção de linhagens homozigóticas, e, portanto o emprego de
delineamentos clássicos de mapeamento, tais como populações F2,
retrocruzamentos e linhagens recombinantes endogâmicas (RILs). Dado isso, os
mapas genéticos têm sido construídos utilizando-se famílias F1 já existentes
derivadas do simples cruzamento de duas árvores. A segregação neste caso será
resultado da recombinação meiótica entre os dois parentais e a fase de ligação é
desconhecida (Butcher et al., 2002).
Em gimnospermas, a presença do tecido haplóide nas sementes
(megagametófito) permite a análise direta da co-segregação de pares de
marcadores, a estimativa da freqüência de recombinação além da determinação da
fase de ligação, o que não é possível para as angiospermas (Grattapaglia, 2007b). A
abordagem via megagametófitos foi utilizada na detecção de ligação gênica entre
locos isoenzimáticos em Pinus taeda (Adams & Joly, 1980). Posteriormente, foram
utilizados marcadores dominantes, tais como: RAPD e AFLP, em diversas espécies
de coníferas: Pinus ellioti (Nelson et al., 1993), Picea abies (Norway spruce) (Paglia
et al., 1998), Pinus radiata (Kuang et al., 1999), Pinus taeda (Remington et al., 1999)
e Pinus pinaster (Costa et al., 2000).
Em angiospermas, o mapeamento começou a partir da década de 1990,
quando Grattapaglia & Sederoff (1994) desenvolveram uma estratégia de
mapeamento denominada de pseudocruzamento-teste (pseudo-testcross). A partir
daí os mapas genéticos de diversas espécies alógamas foram construídos: Pinus
(Carlson et al., 1991), cana-de-açúcar (Sobral & Honeycutt, 1993) e Populus
(Cervera et al., 2001). Para o gênero Eucalyptus também não foi diferente, utilizando
também marcadores dominantes foram construídos alguns mapas genéticos: com
marcadores RAPD, E. grandis x E. urophylla (Grattapaglia & Sederoff, 1994), E.
urophylla x E. grandis (Verhaegen & Plomion, 1996), E. grandis x E. globulus
(Myburg et al., 2003), com marcadores AFLP, E. tereticornis x E. globulus (Marques
et al., 1998) e, com uma combinação de marcadores AFLP, RFLP e isoenzimas para
Os mapas genéticos em árvores, posteriormente evoluíram dos marcadores
com menor repetibilidade (marcadores dominantes) para marcadores com maior
repetibilidade e herança codominante (microssatélites) (Brondani et al., 1997). A
conservação dos locos microssatélites entre as espécies do mesmo gênero, como
observado em Eucalytpus e Populus e em menor escala em Pinus, torna possível,
SRUWDQWRDJHUDomRGHPDSDV³FRQVHQVR´SHUPLWLQGRDFRPSDUDomRHLQWHJUDomRGH
informação de mapeamento genético de marcadores, genes e QTLs (Grattapaglia,
2007b). Vários trabalhos foram publicados com microssatélites nas mais diversas
espécies de árvores: Pinus taeda, (Brown et al., 2001; Sewell et al., 1999; Zhou et
al., 2003), Populus (Yin et al., 2004), Picea abies (Achere et al., 2004), Cryptomeria
japonica (Tani et al., 2003).
No gênero Eucalyptus, os mapas utilizando marcadores microssatélites ainda
são poucos e podemos citar como o primeiro mapa consenso construído baseado
somente neste tipo de marcador, o mapa desenvolvido por Brondani et al. (2006). O
mapa contém 240 microssatélites e cobertura genômica estimada de 90%. A
transferibilidade dos microssatélites em diferentes espécies de Eucalyptus facilita a
sua adoção em processos de seleção assistida, uma vez que a hibridação é o
método utilizado no melhoramento desse gênero (Grattapaglia, 2007c).
Posteriormente aos mapas genéticos, foram mapeados QTLs para
características de interesse econômico em várias espécies florestais (Devey et al.,
1995; Emebiri et al., 1998; Plomion et al., 1996; Weng et al., 2002). Não
diferentemente das demais espécies florestais, mapas de QTLs foram construídos
para o gênero Eucalyptus. Os mapas de QTLs foram construídos baseados em
mapas genéticos com os mais diversos tipos de marcadores (dominantes e
codominantes). As características avaliadas nestes mapas são características de
interesse econômico para a indústria. QTLs para características juvenis tais como,
peso da semente, área foliar e tolerância das sementes ao frio já foram mapeados
(Byrne et al., 1997a; Byrne et al., 1997b; Vaillancourt et al., 1995). QTLs para
características de vegetação propagativa (Grattapaglia et al., 1995; Marques et al.,
1999) também já foram identificados. Diversos mapas de QTLs também já foram
construídos até o momento na área de características relacionadas com qualidade
da madeira e desempenho (Grattapaglia et al., 1996; Kirst et al., 2004; Missiaggia,