ESTABILIDADE ALÉLICA DOS MARCADORES FORENSES
NAS LEUCEMIAS
CLÁUDIO VELOSO BATISTA
Dissertação de Mestrado em Medicina Legal
CLÁUDIO VELOSO BATISTA
ESTABILIDADE ALÉLICA DOS MARCADORES FORENSES
NAS LEUCEMIAS
Dissertação de Candidatura ao grau de
Mestre em Medicina Legal submetida ao
Instituto de Ciências Biomédicas de Abel Salazar
da Universidade do Porto
Orientadores: Doutora Henriqueta Alexandra Coimbra Silva
Professora Auxiliar
Instituto de Genética Médica
da Faculdade de Medicina
da Universidade de Coimbra
Doutora Luísa Maria de Sousa Mesquita Pereira
Investigadora do IPATIMUP
Instituto de Patologia e Imunologia
Molecular da Universidade do Porto
I
Este trabalho é dedicado,
em especial e com um sentimento de imensa saudade, ao meu cunhado e amigo Filipe Neves.
II
Dedico ainda à minha irmã e aos meus pais, a quem tudo devo, à minha namorada Sandra, a toda a minha família, aos meus avós, aos meus padrinhos, ao meu primo Pedro, a todos os meus amigos, em especial à Clara, aos amigos das andanças musicais, em particular ao pessoal da Smooth, às colegas de curso Sofia, Marisa, Viviana, Ana Teresa, Joana e Cátia Daniela, aos amigos do Secundário, ...
Às minhas orientadoras, Prof. Luísa e Prof. Henriqueta, agradeço a oportunidade que me proporcionaram de concretizar este trabalho, e além de todo o conhecimento transmitido, agradeço a disponibilidade, simpatia e amizade com que me acolheram.
À Prof. Maria José, coordenadora do curso de mestrado, agradeço a disponibilidade e a colaboração, bem como a consideração demonstrada ao longo de todo o curso.
Às pessoas que contribuíram directamente para este trabalho, em particular ao Dr. Luís e à Dra. Caroline, do Instituto de Genética Médica da Faculdade de Medicina da
Universidade de Coimbra, e à Dra. Verónica, à Dra. Marta e à Dra. Ana Mafalda, do IPATIMUP.
A todos os que estiveram de algum modo envolvidos na concretização deste projecto, em particular ao Marco e à Cristina.
À APLL- Associação Portuguesa de Leucemias e Linfomas, na pessoa no Dr. Herlander Marques, agradeço a bolsa disponibilizada para o financiamento do trabalho.
À Dra. Virgínia Lopes e Dra. Maria João Porto, do Serviço de Genética e Biologia Forense da Delegação do Centro do Instituto Nacional de Medicina Legal.
A todos os professores que contribuíram para a minha formação académica, musical e pessoal.
III
RESUMO
Os marcadores genéticos usados no contexto forense são microssatélites, também denominados STRs, constituídos por unidades de três a cinco pares de bases, que se repetem geralmente entre cinco a trinta vezes.
Os loci STR são altamente polimórficos, com uma extensão inferior a 350 pares de bases, podendo ser facilmente analisados através de sistemas de amplificação simultânea (PCR multiplex). Um dos requisitos essenciais à escolha dos STRs a incluir no painel de marcadores usados para identificação humana é a sua dissociação com qualquer tipo de patologia ou efeito fenotípico. Contudo, existem vários estudos que referem que a estabilidade destes marcadores se encontra afectada em diversas patologias, nomeadamente nas neoplasias.
No presente trabalho efectuou-se um estudo da estabilidade alélica apresentada pelos marcadores genéticos aplicados no âmbito da medicina legal e das ciências forenses para efeitos de identificação humana individual, em amostras de sangue de pacientes com o diagnóstico de leucemia. Para isso, foram analisados dois fenómenos essenciais indicativos de instabilidade genómica, a instabilidade dos microssatélites (MSI) e a perda de heterozigotia (LOH), através da comparação de diversos parâmetros entre amostras de DNA de doentes com leucemia e amostras de dois grupos controlos: um controlo constituído por sujeitos sem nenhuma patologia diagnosticada e um controlo publicado para a população do Centro de Portugal. A comparação com estes grupos controlo resultou da impossibilidade de comparar tecido tumoral e normal do mesmo indivíduo.
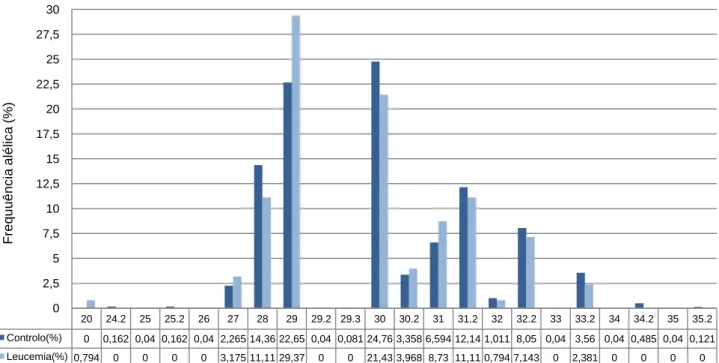
Para o estudo da instabilidade dos microssatélites analisou-se a existência de polialelismo. O estudo da perda de heterozigotia foi avaliado indirectamente pela análise das frequências de heterozigotia para cada locus, das frequências alélicas para cada população e pelo desequilíbrio alélico entre os picos dos alelos dos loci heterozigóticos. As duas populações foram também comparadas relativamente à variação e diferenciação genética, através de diversos testes estatísticos.
A ausência de diferenças estatisticamente significativas entre as duas populações e a concordância dos resultados obtidos neste trabalho através de diferentes métodos de análise das amostras sugerem que, no contexto da Medicina Legal e das Ciências Forenses, a identificação humana realizada através da elaboração de perfis genéticos não parece ser condicionada pela utilização de sangue periférico de indivíduos diagnosticados com leucemia e que os marcadores forenses que integram o kit AmplSTR®Identifiler são estáveis nas células do sangue leucémico.
IV
ABSTRACT
Genetic markers used on forensic context are microsatellites, also named STRs, formed by units that vary between 3 and 5 base pairs, which are usually repeated five to thirty times.
STR loci are highly polymorphic, relatively undersized (with less than 350 base pairs) and can be easily analyzed by simultaneous systems of amplification (multiplex PCR). To be included on the markers panel used on human identification, one of the fundamental requests for STRs choice is their dissociation with any kind of pathology or phenotypic effect. However, several studies propose that the stability of these markers is affected by a variety of diseases, particularly neoplasias.
The study of the allelic stability in leukemic patients, by means of genetic markers currently used for human identification in legal medicine and forensic sciences areas, is the main aim of this work. To address this issue two major characteristics of genomic instability were analyzed by comparison of several parameters in DNA samples from leukemia patients and two control groups: microsatellite instability (MSI) and loss of heterozygosity (LOH). The first control group was composed of healthy unrelated individuals and the second by a published control group for Portuguese population from the Central area. The impossibility of comparing tumoral and normal-matched tissue justifies the use of the mentioned control groups.
With the aim of studying the microsatellite instability, the polyallelism existence has been analyzed. The loss of heterozygosity was evaluated indirectly by the analysis of the heterozygosity frequencies for each locus, by allelic frequencies of both populations and by the allelic imbalance between allelic peaks of heterozygotic loci. Several statistic tests were also applied in order to compare the two populations focusing on the genetic variation and differentiation.
The absence of statistically significant differences between the two studied populations and the consistency of the obtained results using different practical approaches to samples analysis suggest that, in Legal Medicine and Forensic Sciences context, human identification by means of genetic profiles seems not to be conditioned by the use of peripheral blood of leukemic patients. Also, the forensic markers of the AmplSTR®Identifiler kit were stable in leukemic blood cells.
V
RÉSUMÉ
Les marqueurs génétiques utilisés dans le contexte forensique sont des microsatellites, aussi appelés de STR, formés par des unités de trois à cinq paires de bases, qui se répètent cinq à trente fois.
Les loci STR sont très polymorphes, avec une longueur inferieur à 350 paires de bases, qui peuvent être facilement analysées par des systèmes d’amplification simultanée (PCR multiplex). Une des conditions essentielles pour choisir les STRs qui doivent incorporer le group des marqueurs utilisés pour l’identification humaine c’est leur dissociation avec aucune pathologie ou effet phénotypique. Cependant, ils existent plusieurs études qui montrent que la stabilité de ces marqueurs se trouve modifié en certaines pathologies, particulièrement au niveau des néoplasies.
Dans ce projet nous avons effectué une étude de la stabilité allélique présentée par les marqueurs génétiques qui sont appliquées au domaine de la médecine légale et des sciences forensiques pour l’identification humaine individuelle, dans un échantillon de sang périphérique de patients avec le diagnostique clinique de leucémie. À ce propos, nous avons analisé deux phénomènes essentiels indicatifs de l’instabilité génomique, l’instabilité des microsatellites (MSI) et la perte d’hétérozygotie LOH), par la comparaison de plusieurs paramètres parmi l’échantillon d’ADN des patients avec leucémie et l’échantillon de deux groupes contrôles: un contrôle formés par des individus avec aucune pathologie diagnostiquée et un contrôle publié sur la population de la région centre du Portugal.
Pour l’étude de l’instabilité des microsatellites nous avons examinés l’existence de polyallélisme. La perte d’hétérozygotie a été étudié indirectement par l’analyse des fréquences d’hétérozygotie de chaque locus, des fréquences alléliques de chaque population et par le déséquilibre allélique entre les pics des allèles des loci hétérozygotiques. Les deux populations ont été comparées relativement à la variation et différentiation génétique, par des tests statistiques divers.
L’absence de différences statistiquement significatives entre les deux populations et la concordance des résultats obtenus avec différentes méthodes d’analyse des échantillons suggèrent que, au niveau de la Médecine Légale et des Sciences Forensiques, l’identification humaine n’est pas limitée par l’utilisation du sang périphérique des patients avec leucémie et que les marqueurs forensiques du kit AmplSTR®Identifiler sont stables dans les cellules du sang leucémique.
VI
ABREVIATURAS
µg – micrograma AI – Allelic Imbalance
AMPFLPs – Amplified fragment length polymorphisms CHLC – Cooperative Humane Linkage Center
DNA – Ácido desoxirribonucleico FAB – Franco-américo-britânico FSS – Forensic Science Service HLA – Human leukocyte antigen
HNPCC – Cancro colorectal hereditário não-polipótico Homoz. - Homozigótico
kb – kilobases kV - quilovolts
LLA – Leucemia Linfoblástica Aguda LLC – Leucemia Linfóide Crónica LMA – Leucemia Mielóide Aguda LMC – Leucemia Mielóide Crónica LOH – Loss of heterozygosity Mb - megabases
MIN ou MSI – Microsatellite instability ml – mililitro
MLPs – Multi locus probes MMR – Mismatch repair ng – nanograma
OMS – Organização Mundial de Saúde PAR – Peak area ratio
pb – pares de bases
PCR – Polymerase chain reaction PHR – Peak height ratio
RER+ – Replication error-positive
RFLPs – Restricted fragment length polymorphisms SLPs – Single locus probes
SSLPs – Single sequence length polymorphisms STRs – Short tandem repeats
VII
ÍNDICE
Resumo Abstract Résumé Abreviaturas I – INTRODUÇÃO1.1 – SHORT TANDEM REPEATS
1.1.1 – Evolução dos marcadores de aplicação forense 1.1.2 – Definição e classificação dos STRs
1.1.3 – Função e aplicabilidade biológica dos STRs
1.1.4 – Selecção dos STRs forenses utilizados actualmente 1.1.5 – Caracterização dos STRs forenses
1.1.6 – STRs e patologias 1.1.7 – STRs e patologia oncológica 1.2 – LEUCEMIAS 1.3 – OBJECTIVOS II – MATERIAL E MÉTODOS 2.1 – AMOSTRAS 2.1.1 – População de estudo 2.1.2 – População controlo 2.2 – METODOLOGIA 2.2.1 – Reagentes
2.2.2 – Preparação das amostras 2.2.3 – Amplificação
2.2.4 – Leitura e análise da amplificação 2.2.5 – Análise estatística
2.2.6 – Determinação do desequilíbrio alélico
III IV V VI 1 1 1 4 7 8 10 15 17 21 29 30 30 30 30 30 32 32 33 33 34 37
VIII III – RESULTADOS E DISCUSSÃO
3.1 – Avaliação da estabilidade dos STRs pela pesquisa de polialelismo 3.2 – Diversidade genotípica: heterozigotia dos loci e Equilíbrio de
Hardy-Weinberg 3.3 – Distribuição das frequências alélicas
3.4 – Distâncias genéticas, AMOVA e testes de diferenciação 3.5 – Estudo do desequilíbrio alélico
3.5.1 – Definição dos critérios e valores-padrão 3.5.2 – Análise global
3.5.3 – Análise do comportamento dos loci
3.5.4 – Análise das amostras com loci em desequilíbrio alélico
IV – CONCLUSÕES
V – REFERÊNCIAS BIBLIOGRÁFICAS VI – ANEXOS
Anexo 1 – Sistema de classificação FAB das leucemias mielóides agudas Anexo 2 – Sistema de classificação FAB das leucemias linfoblásticas agudas Anexo 3 – Equivalência entre os sistemas de classificação de Rai e Binet
Anexo 4 – Perfis genéticos dos indivíduos que constituem o grupo das leucemias Anexo 5 – Ratio das alturas dos loci heterozigóticos do grupo controlo 2
Anexo 6 – Ratio das áreas dos loci heterozigótico do grupo controlo 2
38 39 41 44 58 59 59 61 67 69 76 78 94 95 95 95 96 98 101
1
I – INTRODUÇÃO
1.1 – SHORT TANDEM REPEATS (STRs)
1.1.1 – Evolução dos marcadores de aplicação forense
A Biologia e a Genética Forense dispõem actualmente dos mais avançados recursos tecnológicos e científicos que permitem alcançar com êxito um dos seus principais objectivos de estudo, a identificação humana. Ao longo da história da ciência houve acontecimentos que se revelaram de extrema importância para o desenvolvimento destas áreas do conhecimento. Antes de referir a descoberta e o estudo pormenorizado do DNA ou a revolução imposta pela invenção da PCR a nível das metodologias laboratoriais, há que, e não apenas por motivos cronológicos, referir o contributo de Mendel, cujas leis permitiram definir as bases da genética actual.
A identificação humana, quer pela longínqua utilização das provas de semelhança fisionómica, quer pelo actual recurso às mais recentes tecnologias usadas para elaboração de perfis genéticos, pressupõe sempre a comparação, usando como medida a “quantidade de semelhanças”. Por perfil genético entende-se, do ponto de vista médico-legal, o conjunto de características hereditárias de um indivíduo relativamente a um determinado número de marcadores genéticos, que se podem detectar em qualquer vestígio biológico que lhe pertença. A identidade genética baseia-se na individualidade biológica de cada baseia-ser humano, baseia-sendo fundamentada pela exclusividade do seu material genético, bem como pela igualdade e imutabilidade do DNA em todas as células do organismo, ao longo da vida.
Antes da aplicação da análise de DNA à resolução de casos de investigação forense, a vinculação genética baseava-se em exames laboratoriais que permitiam uma caracterização individual tão pormenorizada e discriminatória quanto possível, através do estudo de marcadores imunológicos ou proteicos, sendo de destacar a determinação dos grupos sanguíneos do sistema ABO, Rh, M e N, Hr, haptoglobinas, grupos P e o sistema HLA (França, 2005).
Os primeiros caracteres humanos de transmissão mendeliana simples que se estudaram foram os antigénios eritrocitários: o primeiro marcador humano polimórfico (sistema ABO) foi descrito em 1900 por Landsteiner, mas só em 1924 Bernstein conseguiu determinar o processo exacto de transmissão hereditária dos alelos do sistema ABO. Trabalhos posteriores de Landsteiner, Levine, Wiener, Sturli, entre outros, permitiram descrever por completo os sistemas eritrocitários ABO, MNs, P e Rh, que viriam a constituir a base fundamental de todas as investigações biológicas de
2
paternidade. A caracterização dos sistemas Kell, Lutheran, Duffy, Kidd e Xg completou o grupo de marcadores eritrocitários caracterizado pela determinação imunológica dos seus fenótipos através de anti-soros.
Em 1947, com as proteínas plasmáticas, surge o segundo grupo de marcadores polimórficos: Jayle e Gillard reconheceram a existência de vários tipos de haptoglobinas (Hp), sendo este o primeiro passo para a posterior descoberta de múltiplos polimorfismos de proteínas plasmáticas: Gm, Gc, Ag, Tf, Pi, Lp. Mais tarde, começaram a descrever-se os polimorfismos que constituem o terceiro grupo de sistemas polimórficos, que engloba as enzimas eritrocitárias e leucocitárias: ACP, PGM, AK, GPT.
Em 1967, os trabalhos de vários investigadores (Van Rood, Terasaki e Dausset) culminaram na descrição completa do sistema antigénico mais polimórfico descrito até ao momento, o sistema Hu-1, que mais tarde se denominou HLA (Human Leukocyte Antigen) (Calabuig, 1998).
A partir de 1984, a tipagem genética de material biológico tornou-se uma das ferramentas mais utilizadas para efeitos de identificação humana.
A técnica de DNA Fingerprint está incluída nos métodos clássicos de análise de material genético e assenta na variabilidade de determinadas porções do DNA não codificante. Essas porções são denominadas VNTRs (Variable Number of Tandem Repeats) (Jeffreys, 1985; Nakamura, 1987) e são constituídas por sequências de nucleótidos que se repetem sucessivamente, entre 2 a 10000 vezes, dependendo do tipo de polimorfismo, e que podem apresentar um comprimento superior a 10 kilobases (kb) (Benecke, 1997).
Estas estruturas genómicas eram analisadas sob a forma de RFLPs (Restricted Fragment Length Polymorphisms), que se obtinham pelo reconhecimento e delimitação de uma sequência específica através de enzimas de restrição, como por exemplo as enzimas HaeIII, HinfI ou HindIII. Os fragmentos obtidos eram separados em gel por electroforese, sendo posteriormente revelados pela técnica de Southern Blot (Southern, 1975), por hibridização com sondas complementares à sequência, marcadas com fluorescência. Dependendo da especificidade da sonda, poderia identificar-se um único segmento do DNA, para o qual se utilizava SLPs (Single Locus Probes) (Budowle et al., 1991) ou vários segmentos, através de MLPs (Multi Locus Probes) (Jeffreys et al, 1985), com os quais se obtinha um padrão de 20 a 30 bandas - comparado por alguns autores a um código de barras - padrão esse que seria único e exclusivo para cada pessoa, e que contribuiu para a designação DNA Fingerprint. Após um período inicial de bastantes interrogações e dúvidas científicas (Lewin, 1989; Lander 1989; Norman, 1989), esta técnica implementou-se e foi bastante utilizada na resolução de casos forenses, como
3
por exemplo testes de paternidade (Jeffreys et al., 1991), casos de imigração (Jeffreys et al., 1985) ou investigações criminais (Gill et al., 1987).
Uma das principais limitações desta metodologia resultava das deficientes condições de armazenamento e preservação do DNA, do seu elevado estado de degradação e contaminação ou da limitada quantidade de material genético disponível para análise, em consequência da própria natureza das amostras usadas neste tipo de casos. A técnica de DNA Fingerprint permitia obter resultados aceitáveis utilizando uma concentração mínima de 5 a 10 µg de DNA não degradado, valores nem sempre possíveis de obter em amostras forenses. Por exemplo, um 1 ml de ejaculado contém cerca de 150-300 µg de DNA, contudo, a partir de uma zaragatoa vaginal pós-coito só se consegue obter 0.01-3 ng de DNA; um cabelo arrancado com raiz contém cerca de 30 ng de material genético, enquanto que a haste de um cabelo contém no máximo 0.1 ng de DNA (Benecke, 1997).
Ao comparar a utilização de SLPs com MLPs, conclui-se que os primeiros se revelavam mais vantajosos no estudo de casos forenses pelo facto de permitirem obter bandas a partir de 10 ng de DNA e apresentarem maior sensibilidade. Contudo, a análise do comprimento dos alelos não se conseguia fazer com absoluta precisão, principalmente devido aos limites de resolução impostos pelo gel utilizado na electroforese (Tamaki e Jeffreys, 2005). Além destes argumentos, é ainda de referir o facto de os VNTRs terem o inconveniente de se agruparem, formando clusters nas regiões teloméricas dos cromossomas (NIH/CEPH Collaborative Mapping Group, 1992) e de os RFLPs terem a desvantagem de possuírem baixa variabilidade, sendo as técnicas laboratoriais utilizadas no seu processamento demasiado morosas.
Em 1985, Kary Mullis desenvolveu uma técnica, a Polymerase Chain Reaction (PCR), cujo contributo para as ciências forenses, à semelhança de outras áreas de investigação, se veio a revelar determinante. A sua fundamentação baseia-se em dois princípios: o da complementaridade das bases azotadas, ou seja, o processo de emparelhamento das mesmas não é aleatório (a adenina e a timina ligam-se de forma específica, bem como a guanina e a citosina); e o da reversibilidade da desnaturação e hibridização do DNA, que podem ocorrer num ambiente alcalino ou em determinadas condições térmicas, provocando a ruptura das pontes de hidrogénio entre as bases e a separação das duas cadeias de DNA
.
Esta técnica permite amplificar pequenas quantidades de material genético e obter milhões de cópias a partir de um determinado fragmento de DNA. A aplicação desta metodologia à genética forense viabilizou o estudo de amostras em avançado estado de degradação (sendo a fragmentação do material genético o tipo de degradação
4
mais limitante em amostras forenses), bem como de amostras com quantidades mínimas de DNA.
Alguns loci de minissatélites com alelos relativamente pequenos (cerca de 1000 pb) puderam ser amplificados, originando AMPFLPs (Amplified Fragment Length Polymorphisms); um dos minissatélites bastante utilizado em laboratórios forenses denominava-se D1S80 (Nakamura et al., 1988; Kasai et al., 1990). Os fragmentos obtidos a partir da análise deste marcador eram constituídos por 14 a 42 repetições, sendo o comprimento de cada unidade de repetição de 16 pares de bases (pb). A amplificação destes fragmentos permitia obter alelos com um tamanho significativamente inferior aos fragmentos analisados pelas técnicas disponíveis até ao momento. Ao invés do que acontecia com o estudo dos RFLPs, era possível fazer uma análise comparativa dos alelos do D1S80 com um padrão que incluísse todos os alelos conhecidos para este marcador (allelic ladder) no mesmo gel. Este novo procedimento facilitava significativamente a leitura e interpretação dos resultados, de tal modo que viria a ser adoptado pelos sistemas de identificação genética usados posteriormente. Após uma avaliação pormenorizada da frequência alélica deste marcador na população mundial (Peterson et al., 2000) a utilização do D1S80 caiu em desuso, em detrimento de uma metodologia ainda mais vantajosa baseada na amplificação multiplex de STRs.
1.1.2 – Definição e classificação dos STRs
Os microssatélites pertencem à família das sequências de DNA repetitivo em tandem, mais concretamente a uma classe denominada VNTR (Variable Number of Tandem Repeats). Esta classe inclui três tipos de sequências distintas, nomeadamente: satélites, minissatélites e microssatélites.
As sequências satélites são constituídas por repetições cujo comprimento varia entre 5 a 100 pares de bases (pb), caracteristicamente organizados em grupos que na sua generalidade podem atingir um tamanho 100 megabases (Mb). O tamanho deste tipo de sequências é pouco variável dentro de cada população, quando comparado com os tipos de sequências referidos mais adiante (John et al., 1998; Tyler et al., 1993). Este tipo de sequências encontra-se localizado predominantemente na heterocromatina junto dos centrómeros dos cromossomas.
As sequências minissatélites têm um comprimento total que pode variar entre os 0,5 e 30 kb, sendo que a dimensão de cada repetição que as constituem é de 15 a 70 pb. Este tipo de sequências está situado em determinadas regiões da eucromatina junto dos telómeros e o seu tamanho é muito variável dentro de uma população (Shriver et al., 1993; Armour e Jeffreys, 1992).
5
As sequências microssatélites são constituídas por repetições com um comprimento que varia entre 2 a 6 pb, cuja agregação resulta em sequências que se caracterizam por uma elevada variabilidade, apesar do seu tamanho não ultrapassar 350 pb. Os microssatélites localizam-se na eucromatina e os seus alelos são agrupados em múltiplas classes, que variam relativamente ao seu comprimento, e são característicos de cada população (Tautz e Rents, 1984).
Os microssatélites também podem ser designados de SSLPs (Single Sequence Length Polymorphisms) ou STRs (Short Tandem Repeats) (Edwards et al., 1991). Existem autores que fazem distinção entre microssatélites e STRs, considerando que os primeiros são constituídos por repetições de 2 pb enquanto que os segundos são constituídos por repetições de 3 a 6 pb; contudo, e de acordo com a linguagem científica institucionalizada, estes dois termos serão usados com o mesmo significado (Koreth et al., 1996).
Os STRs existem abundantemente tanto em procariontes como eucariontes, representando cerca de 30% do DNA repetitivo e 3% da totalidade do genoma humano. Em média, pode encontrar-se um STR a cada intervalo de 2000 pares de bases (International Human Genome Sequencing Consortium, 2001). Embora geralmente se considere que os STRs estão localizados nas regiões não codificantes do DNA, existe também uma pequena percentagem (cerca de 8%) que se localiza em regiões codificantes (Ellgren, 2000).
A distribuição destes microssatélites pelos cromossomas é heterogénea e irregular, podendo encontrar-se espalhados em diferentes graus de densidade. As regiões subteloméricas são os locais onde se localizam em menor quantidade(Koreth et al.,1996). Além disso, a sua frequência varia entre cromossomas; no homem, o cromossoma 19 é o que apresenta maior quantidade de STRs (Subramaninan et al., 2003).
Os STRs podem ser classificados de acordo com o comprimento da unidade de repetição: as repetições podem distinguir-se em mono, di, tri, tetra penta ou hexanucleotídicas, conforme sejam constituídas por uma, duas, três, quatro, cinco ou seis pares de bases, respectivamente (quanto maior for o tamanho da unidade de repetição, mais informativo se considera o microssatélite) (International Human Genome Sequencing Consortium, 2001). Por outro lado, tendo em conta a estrutura da repetição, existem repetições perfeitas ou simples, quando contêm apenas uma unidade de repetição, e imperfeitas ou compostas, quando são constituídas por mais do que uma unidade de repetição, distintas entre si (Urquhart et al., 1994). Quando os alelos de um STR são constituídos por unidades de repetição incompletas são designados de microvariantes, sendo representados pelo número de unidades de repetição completas,
6
seguido do número de nucleótidos que formam a repetição incompleta. O exemplo mais comum de um microvariante é o alelo 9.3 do locus TH01, que é composto por nove unidades de repetição completas e uma unidade incompleta, constituída por três nucleótidos (Butler, 2005).
Os STRs mais frequentes no genoma humano são constituídos por repetições dinucleotídicas, o que confirma a constatação de que a frequência de cada um dos tipos anteriormente referidos diminui à medida que aumenta o seu tamanho. Além disso, também se verifica que as unidades de repetição mais frequentes apresentam uma predominância de adeninas: A, AC, AAAN, AAN e AG, por ordem decrescente de abundância (International Human Genome Sequencing Consortium, 2001 e Nadir et al., 1996).
Os microssatélites estão entre os marcadores genéticos mais versáteis, sendo utilizados num vasto número de aplicações biológicas. Vinte anos após a sua descoberta, apesar de já haver algumas teorias, existem ainda muitas respostas por obter no que concerne ao conhecimento destes marcadores, mais precisamente em relação à sua origem, aos processos que definem a sua localização e garantem a sua manutenção no genoma, aos mecanismos biológicos responsáveis pela sua enorme variabilidade, e acima de tudo, relativamente à sua importância biológica e funcional (Buschiazzo e Neil, 2006).
O processo que leva à formação dos microssatélites continua a ser alvo de vários estudos (Wilder e Hollocher, 2001). Sabe-se, no entanto, que a sua síntese ocorre aleatoriamente como resultado de um desequilíbrio entre eventos moleculares promotores e inibidores da iniciação de microssatélites (Buschiazzo e Neil, 2006). Existem duas teorias explicativas predominantes: uma refere que os microssatélites surgem espontaneamente a partir de sequências únicas – microssatélites de novo (Messier et al., 1996); outra sugere a existência de receptores genómicos aos quais serão ligados formas primárias obtidos a partir de elementos móveis – microssatélites adoptados (Wilder e Hollocher, 2001). Existem também referências a estruturas anteriores ao microssatélites, denominados proto-microssatélites, cuja existência resulta da substituição ou inserção de bases em determinadas regiões do genoma, levando à formação de 3 ou 4 unidades de repetição sucessivas (Hancock, 1999), que posteriormente estariam altamente sujeitas a mecanismos de expansão (Messier et al., 1996). Nos mamíferos, já há muito que se conhece a associação entre microssatélites e a sequência Alu (Nadir et al., 1996; Arcot et al., 1995; Clark et al., 2004), os elementos L1 (Duffy et al., 1996) e a fracção poli(A), justificada pela elevada susceptibilidade de erros de transcrição a que estão sujeitos, o que resulta na síntese de proto-microssatélites
7
ricos em A e na sua expansão, motivada por um acontecimento mutagénico denominado slippage (deslizamento) do DNA.
Apesar destas hipóteses serem plausíveis, a sua fundamentação em critérios especificamente selectivos poderá ser questionada, visto que não são representativas da totalidade do genoma.
1.1.3 - Função e aplicabilidade biológica dos STRs
Durante muito tempo os STRs foram considerados como uma parte do material genético inútil e sem interesse (Buschiazzo e Neil, 2006) mas a partir da última década do século XX, assistiu-se a um interesse crescente relativamente aos seus efeitos no organismo.
Inicialmente pensava-se que estas sequências repetitivas desempenhavam um papel funcional a nível do genoma, quer directamente pela sua participação na regulação dos genes (Hamada et al., 1984), quer indirectamente, funcionando como hot spots para a recombinação (Slighton et al., 1980). Contudo, se por um lado não é possível atribuir uma função específica a estas porções do DNA, por outro lado sabe-se que em determinadas circunstâncias as repetições trinucleotídicas CAG são transcritas; existem proteínas que se ligam ao DNA, com especificidade para repetições di e trinucleotídicas (Richards et al., 1993) e estudos in vitro revelaram que existem também repetições que podem funcionar como locais indutores da agregação do nucleossoma (Wang et al., 1994).
Estudos posteriores vieram a constatar algumas funções que se podiam atribuir a estas porções de material não codificante, tais como a regulação da transcrição do gene do factor de crescimento da epiderme (Gebhardt et al., 1999), do gene da tirosina-hidroxilase (Meloni et al., 1998), e do gene PIG33 (Contente et al., 2002); em genomas de mamíferos, repetições (CA)n e (CT)n junto de determinados genes podem afectar a expressão desses mesmos genes (Hamada et al., 1984; Naylor et al., 1990); os STRs podem afectar a recombinação (Wahls et al., 1990), a formação de sinais para o posicionamento do nucleossoma (Wang e Griffin, 1995) e a manutenção da organização espacial da cromatina (Heale e Petes, 1995); em 2000, Escher e colaboradores demonstraram que alguns STRs estão envolvidos na regulação do processo de transcrição, visto que as proteínas envolvidas neste mecanismo contêm domínios ricos em glutaminas e repetições trinucleotídicas constituídas por sequências poliglutamínicas (Escher et al., 2000).
Além dos exemplos mencionados é sabido que as repetições trinucleotídicas estão implicadas em pelo menos 19 doenças genéticas no Homem, nas quais se
8
observou uma forte correlação entre o comprimento das repetições, a idade em que se manifestaram os primeiros sintomas e a severidade da doença (Pearson et al., 2005).
Apesar de um crescente número de funções para estes microssatélites estar a ser descoberto, a sua maioria continua ainda desconhecida, sendo imperativa uma maior compreensão da variabilidade e mutabilidade dos STRs para perceber as suas variadas funções biológicas. Mesmo com todas as dúvidas e desconhecimento ainda existentes, a sua abundância e elevada variabilidade tornaram-nos os marcadores genéticos mais utilizados nas últimas décadas (Chambers e MacAvoy, 2000).
Os STRs caracterizam-se por uma transmissão estável e conservativa, sendo herdados por uma geração e transmitidos à geração seguinte de acordo com as Leis de Mendel (Weber e May, 1989); o perfil de STRs é único para cada indivíduo e comum às várias células do organismo (Beckman, 1992). Estas características conferem-lhes uma vasta aplicabilidade no domínio das ciências biológicas a nível individual, familiar, populacional e filogenético, sendo usados frequentemente para identificação humana, testes de paternidade/parentesco, construção de árvores filogenéticas de evolução, estudos genéticos populacionais, elaboração de mapas genéticos, localização de genes e como meio auxiliar no diagnóstico de patologias. Além de tudo isto, pelo facto de se poderem localizar na proximidade de genes importantes, podem ser utilizados como marcadores de determinadas patologias associadas a esses genes e permitir a obtenção de informação relativamente à importância dos genes num estádio específico da doença ou da sua evolução, especialmente no caso de patologias do foro oncológico (Naidoo e Chetty, 1998).
1.1.4 – Selecção dos STRs forenses utilizados actualmente
Edwards e Hammond foram os primeiros investigadores a descrever os STRs como instrumentos essenciais na identificação humana, no início dos anos 90 (Edwards et al., 1991 e 1992). Vários estudos foram realizados por diversas instituições científicas internacionais com o intuito de definir quais os marcadores a aplicar, de acordo com vários critérios que serão especificados mais adiante. Neste sentido, há que destacar a Royal Canadian Mounted Police (RCMP) que contribuiu com vários estudos efectuados em parceria com laboratórios europeus (Frégeau, 1993) e o Forensic Science Service (FSS), que começou por investigar exaustivamente novos loci e a variação populacional de uma série de prováveis STRs (Kimpton et al., 1993).
Os loci FES/FPS, F13A1, vWA e TH01 foram agrupados para constituir o primeiro multiplex aplicado ao estudo de um caso forense pelo FSS (Kimpton et al, 1994). Seguiu-se um multiplex de Seguiu-segunda geração que incluía os Seguiu-seguintes loci: TH01, vWA, FGA,
9
D8S1179, D18S51 e D21S11 (Kimpton et al, 1996). Em Abril de 1995, foi criada a Base de Dados Nacional de DNA do Reino Unido, construída a partir da aplicação do multiplex anteriormente referido, ao qual foi adicionado o estudo da amelogenina para a determinação do género masculino e feminino (Werret, 1997).
Um ano após a criação da Base de Dados Nacional de DNA daquele país surge o “Projecto STR”, nos EUA, que tinha como principal objectivo a criação de uma base de dados genéticos norte-americana. Esta base de dados foi o resultado do trabalho desenvolvido pelos laboratórios do FBI, que, comprovada a eficácia da tecnologia genética associada aos STRs, centraram as suas investigações na definição do conjunto de STRs a aplicar na estruturação do CODIS (Combined DNA Index System) que iria servir de suporte à NDNAD (U.S. National DNA Database). Este projecto envolveu 22 laboratórios que, durante cerca de ano e meio, estudaram e avaliaram 17 loci candidatos que se encontravam provisoriamente disponíveis em kits comerciais. Foram então desenvolvidos vários estudos com o objectivo de avaliar o desempenho e os diversos protocolos associados a estes kits, bem como a estabilidade das bases de dados populacionais e a valorização forense dos vários sistemas de STRs em análise.
Paralelamente ao desenvolvimento dos princípios científicos, também se verificou um progresso acentuado no domínio das tecnologias e técnicas laboratoriais. Enquanto numa fase inicial se empregava um gel de poliacrilamida com revelação de prata para detecção de STRs, numa fase posterior começaram a ser usado métodos fluorescentes associados à electroforese, culminando com o desenvolvimento de analisadores e sequenciadores automáticos (Fregeau et al, 1993).
A 14 de Novembro de 1997, após uma reunião do “Projecto STR”, foram anunciados os 13 loci integrantes de um painel utilizado na criação da base de dados genéticos norte-americana (CSF1PO, FGA, TPOX, TH01, VWA, D3S1358, D5S818, D7S820, D8S1179, D13S317, D16S539, D18S51, D21S11) (Budowle et al., 1998). Foram igualmente providenciados kits comerciais pelas duas maiores empresas que ainda hoje são detentoras dos recursos mais avançados relativamente à tecnologia associada a estes sistemas de análise de DNA repetitivo: a Applied Biosystems lançou no mercado os kits Profiler Plus e COfiler, enquanto que a rival Promega Corporation, completou um kit já existente, o PowerPlex 1.1, pela adição de novos loci, dando origem ao kit PowerPlex 2.1. Após o desenvolvimento destes kits, a comunidade científica centrou os seus esforços no sentido de conseguir que os 13 loci utilizados fossem amplificados numa única reacção, num sistema denominado multiplex (Holt et al., 2002 e Levedakou et al., 2002).
Em Maio de 2000 surge o kit PowerPlex 16 (Promega Corporation), que permitia a amplificação dos 13 loci e incluía também o estudo da amelogenina e dois
10
pentanucleótidos denominados de Penta D e Penta E, que foram descobertos e caracterizados pelos investigadores numa tentativa de definir loci com elevada variabilidade e que demonstrassem uma baixa tendência para a formação de artefactos (Bacher et al., 1998). Em Julho de 2001 a Applied Biosystems apresenta o kit 16plex Identifiler, que incluía a amplificação dos 13 loci referidos, o estudo da amelogenina e dois tetranucleótidos: D2S1338 e D19S433, que aumentavam o poder discriminativo do kit e facilitavam a interpretação dos resultados em caso de misturas (Schumm et al., 2001).
O Reino Unido e a maioria dos países europeus viriam a adoptar o kit comercial disponibilizado em 1999 pela Applied Biosystems, denominado SGM Plus, que era uma versão actualizada do kit SGM já existente, tendo sido introduzidos os loci D3S1358, D16S539, D2S1338, D19S433 e a amelogenina. O locus SE33 define um STR extremamente polimórfico e foi incluído em kits comerciais produzidos pelas duas empresas anteriormente referidas e também por companhias alemãs, visto que a Alemanha usou este STR quando concebeu a sua base de dados genéticos, em 1998, uma vez comprovada a sua elevada utilidade em casos resolvidos anteriormente (Wiegand et al., 1993).
Pode constatar-se, pelos factos supracitados, que a escolha dos loci STRs usados actualmente na maioria dos países da Europa e nos EUA resultou de uma selecção orientada pela aplicação dos STRs aquando da concepção das bases de dados nacionais, o que, em parte, dependia dos marcadores que se encontrassem disponibilizados pelos kits comerciais ou que já se encontrassem a ser utilizados com regularidade e êxito na resolução de casos forenses (Butler, 2006).
1.1.5 - Caracterização dos STRs forenses
Os marcadores genéticos usados no contexto forense são microssatélites constituídos por unidades de repetição que variam entre 3 a 5 pares de bases, que geralmente se repetem entre 5 a 30 vezes, denominados Short Tandem Repeats. Os loci STR são altamente polimórficos, com uma extensão inferior a 350 pb (Brinkmann, 1966), podendo ser analisados através de sistemas de reacções de polimerização em cadeia (PCR multiplex). Esta característica permite a sua utilização em amostras degradadas ou com quantidades mínimas de material genético, o que acontece com muita frequência no tipo de amostras estudadas no âmbito médico-legal.
A primeira aplicação forense destes microssatélites foi a identificação dos restos cadavéricos de uma vítima de assassinato (Hagelberg et al., 1991) e do criminoso Josef Mengele, conhecido como o “Anjo da Morte” de Auschwitz (Jeffreys et al., 1992), sendo
11
que a sua primeira utilização como marcadores polimórficos foi em 1989 (Litt e Luty, 1989; Weber e May, 1989). O elevado grau de polimorfismo destes microssatélites resulta da variação do número de unidades de repetição repetidas sequencialmente (em tandem) e de uma microvariação a nível da sequência ou da estrutura das unidades de repetição.
Estes marcadores podem ser amplificados por PCR e os amplicons obtidos podem ser medidos com precisão através da electroforese em gel de poliacrilamida. Em casos forenses as sequências constituídas por repetições tetranucleotídicas são utilizadas preferencialmente relativamente às que são compostas por repetições di ou trinucleotídicas, visto que a separação electroforética baseada no tamanho das sequências é menos eficaz em alelos que possuam repetições de dois ou três pares de bases e pelo facto de nestas existir uma maior propensão para a formação de bandas stutter (Jeffreys e Tamaki, 2005). De facto, em STRs compostos por repetições de tetranucleótidos, a percentagem de formação de produtos stutter é de 15%, enquanto que nos STRs compostos por repetições de tri ou dinucleótidos essa percentagem eleva-se para os 30% (Butler, 2005). Quando os loci STR são amplificados poderá haver a formação de um artefacto cujo tamanho difere uma unidade de repetição em relação ao alelo correspondente, o que acontece devido ao fenómeno de deslizamento da cadeia de DNA durante a PCR ou a uma desnaturação incompleta dos produtos da PCR, originando produtos stutter (Hauge e Litt, 1993).
Estes artefactos tornavam mais difícil e imprecisa a interpretação dos resultados no gel, visto que se visualizavam na forma de bandas secundárias ou sombras. Isto acontecia principalmente no caso de amostras constituídas por misturas de fluidos biológicos, nas quais se obtém mais do que um perfil genético – muito frequente em vestígios colhidos de vítimas ou cenas de crimes de índole sexual. As repetições penta e hexanucleotídicas, apesar de serem estudadas por alguns investigadores, não são usadas por serem menos frequentes no genoma humano (Bacher et al., 1999).
Assim, pode enumerar-se um conjunto de vantagens na utilização de microssatélites com repetições tetranucleotídicas relativamente aos minissatélites VNTRs ou aos STRs com repetições tri ou dinucleotídicas, entre as quais, a reduzida tendência para formação de produtos stutter, o que permite a interpretação de resultados obtidos no processamento de amostras com misturas de material biológico; a reduzida variação no comprimento dos alelos, permitindo uma análise por multiplex, e a capacidade de obter produtos de PCR de pequenas dimensões, possibilitando a obtenção de informação a partir de amostras de DNA degradado (Butler, 2005).
Actualmente a amplificação dos STRs é feita quase automaticamente, através de um sistema denominado PCR multiplex, que permite a amplificação de vários loci numa
12
reacção que ocorre num só tubo. Este método, além de diminuir significativamente o tempo de processamento das amostras e a quantidade de reagentes e de DNA necessários, mostra-se ainda vantajoso pelo facto de eliminar eventuais fontes de contaminação e de evitar a troca de amostras (Schumm et al., 2001). Além disso, um sistema complementar de detecção fluorescente completa a automatização de todo o processo e facilita a correcta obtenção e interpretação de perfis genéticos.
O perfil genético de um indivíduo pode ser caracterizado a partir de vários STRs. O painel usado neste trabalho inclui os marcadores implementados e mais usados em todo o mundo, estando reunidos num kit desenvolvido por uma empresa comercial pelo facto de serem os STRs escolhidos pelas duas maiores potências mundiais das ciências forenses para construírem as suas bases de dados nacionais de perfis genéticos. Assim, tal como referido na secção anterior, em 1997 os EUA escolheram um conjunto de 13 STRs que viriam a integrar o CODIS usado pelos laboratórios do FBI na criação da NDNAD e o Reino Unido implementou 10 STRs que viriam a ser adoptados por outros países da Europa (Gill, 2002). A tabela 1.1 resume algumas das principais características destes marcadores. A partir dos 13 STRs que integraram o CODIS e dos 10 STRs adoptados pelo Reino Unido, a Applied Biosystems criou um kit denominado AmplfSTR Identifiler, que é vastamente usado não só na resolução de crimes ou investigações de paternidade, mas também na identificação de pessoas desaparecidas ou vítimas de desastres em massa (Ballantyne, 1997).
Os STRs que constituem o painel de marcadores genéticos forenses partilham um conjunto de características que lhes conferem uma elevada taxa de sucesso na resolução de casos de interesse médico-legal: elevada heterozigozidade e elevado grau de polimorfismo; elevado poder de discriminação, geralmente superior a 0,9 numa heterozigotia superior a 70%; localização dispersa no genoma e segregação independente, com um posicionamento em diferentes cromossomas ou pelo menos em braços diferentes, estando a sua localização bem definida no cariótipo humano; localização em regiões não codificantes, não associadas a patologias; robustez e reprodutibilidade dos resultados quando analisados em sistemas multiplex com outros marcadores; baixa propensão para a formação de produtos stutter; alelos de menor comprimento, compreendidos num intervalo de 90 a 500 pares de bases, conferindo maior possibilidade de uma análise eficiente do DNA degradado (Butler, 2005) e estabilidade na maioria dos tecidos, incluindo tecidos post-mortem (Hoff-Olsen et al., 2001).
13
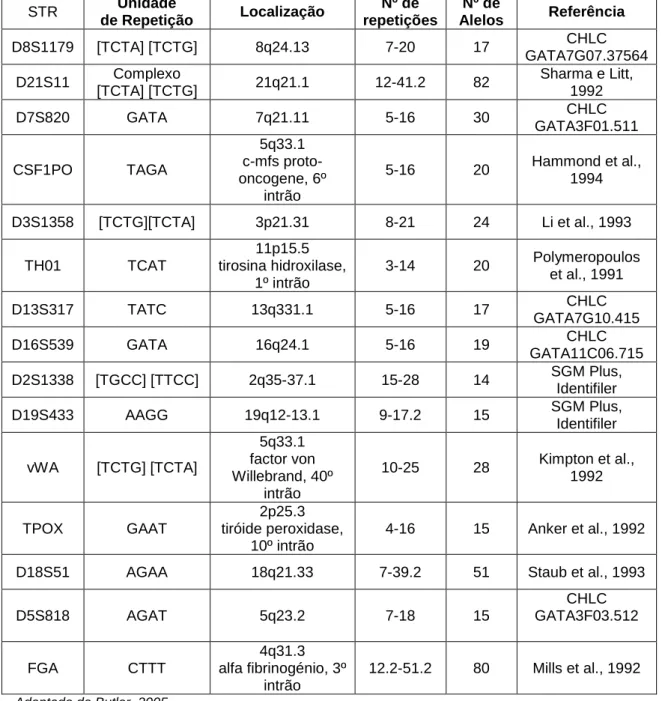
Pela análise da tabela 1.1 pode observar-se que os STRs estão localizados em cromossomas distintos (com a excepção do TPOX e do D2S1338 localizados no cromossoma 2 e do CSF1PO e do D5S818, ambos localizados no cromossoma 5, com uma distância de aproximadamente 26.3 megabases entre si), o que faz com que sejam segregados independentemente uns dos outros, durante a meiose. Esta característica permite a aplicação da regra do produto quando se pretende calcular a “probabilidade de coincidência” (match probability) quando são utilizados perfis genéticos construídos a partir de um painel de vários loci (National Research Council, 1996). De referir ainda que, mesmo no caso de marcadores que se localizem no mesmo cromossoma separados por milhões de pares de bases ou até com distâncias inferiores a um milhão de pares de bases, é possível aplicar a regra do produto. Relativamente aos STRs anteriormente referidos que estão localizados no mesmo cromossoma, apesar de terem sido efectuados inúmeros estudos em indivíduos escolhidos ao acaso da população, não foi encontrada nenhuma associação entre eles.
Quando se obtém uma correspondência entre o perfil genético de um vestígio biológico e a amostra de referência colhida a partir de um suspeito ou de uma vítima é necessário calcular uma probabilidade. A probabilidade de discriminação é uma medida relativa de eficácia de um sistema genético, em criminalística biológica. É um parâmetro estatístico que se define como a probabilidade de dois indivíduos, retirados ao acaso da mesma população, não aparentados entre si, se poderem diferenciar geneticamente, mediante a análise de um sistema ou conjunto de sistemas genéticos. O oposto da probabilidade de discriminação é a probabilidade de coincidência. Relativamente aos STRs forenses, os loci FGA, D18S51, e D21S11 são os mais polimórficos e o loci TPOX o menos polimórfico (Butler, 2005). O poder de discriminação do conjunto de 13 marcadores incluídos no kit é extremamente elevado e a média das probabilidades de coincidência entre indivíduos não relacionados é de um num trilião.
Os STRs constituem hoje em dia uma ferramenta essencial no domínio das ciências médico-legais, mais especificamente a nível da Biologia e Genética Forense, mantendo-se os marcadores genéticos mais utilizados (Edwards et al., 1991; Alford et al., 1994)
14
Tabela 1.1 – Características específicas dos loci STR que integram o kit AmplfSTR Identifiler
Adaptado de Butler, 2005 STR Unidade de Repetição Localização Nº de repetições Nº de Alelos Referência D8S1179 [TCTA] [TCTG] 8q24.13 7-20 17 CHLC GATA7G07.37564 D21S11 Complexo [TCTA] [TCTG] 21q21.1 12-41.2 82 Sharma e Litt, 1992 D7S820 GATA 7q21.11 5-16 30 CHLC GATA3F01.511 CSF1PO TAGA 5q33.1 c-mfs proto-oncogene, 6º intrão 5-16 20 Hammond et al., 1994 D3S1358 [TCTG][TCTA] 3p21.31 8-21 24 Li et al., 1993 TH01 TCAT 11p15.5 tirosina hidroxilase, 1º intrão 3-14 20 Polymeropoulos et al., 1991 D13S317 TATC 13q331.1 5-16 17 CHLC GATA7G10.415 D16S539 GATA 16q24.1 5-16 19 CHLC GATA11C06.715 D2S1338 [TGCC] [TTCC] 2q35-37.1 15-28 14 SGM Plus, Identifiler D19S433 AAGG 19q12-13.1 9-17.2 15 SGM Plus, Identifiler vWA [TCTG] [TCTA] 5q33.1 factor von Willebrand, 40º intrão 10-25 28 Kimpton et al., 1992 TPOX GAAT 2p25.3 tiróide peroxidase, 10º intrão 4-16 15 Anker et al., 1992
D18S51 AGAA 18q21.33 7-39.2 51 Staub et al., 1993
D5S818 AGAT 5q23.2 7-18 15 CHLC GATA3F03.512 FGA CTTT 4q31.3 alfa fibrinogénio, 3º intrão 12.2-51.2 80 Mills et al., 1992
15 1.1.6 - STRs e patologias
As alterações genéticas ocorridas em sequências repetitivas podem ser causa frequente para doenças humanas monogénicas. Inicialmente esta relação era exclusivamente atribuída a repetições trinucleotídicas (Richards e Sutherland, 1994) mas mais tarde viriam a ser descritas associações entre vários STRs instáveis e diferentes tipos de cancro. Além disso, sabe-se agora que a própria repetição poderá apresentar predisposição para mutações, decorrente de uma relação entre a instabilidade e o número de repetições (Richards e Sutherland, 1992).
Pelo facto da caracterização de um perfil genético através de STRs permitir a identificação unívoca de um sujeito, cada vez mais tem vindo a interessar a possibilidade de ser estabelecida algum tipo de relação entre STRs e doenças, o que poderá levantar várias questões nomeadamente a nível da privacidade desse sujeito, se se tiver em causa, por exemplo, aspectos como a inclusão do seu perfil numa base de dados, com todas as questões éticas e morais que daí advêm.
Um dos requisitos essenciais à escolha dos STRs a incluir no painel de marcadores usados para identificação humana é a sua dissociação com qualquer tipo de patologia ou efeito fenotípico. Ao efectuar uma revisão da bibliografia que aborda a relação entre STRs específicos e determinadas patologias é importante ter a noção de que apesar de haver trabalhos publicados que sugiram essa relação, os resultados, face à sua ambiguidade e contradição, não foram considerados determinantes a ponto de impedir a utilização desses microssatélites para fins forenses. Na verdade, houve mesmo associações que mais tarde se vieram a confirmar inexistentes, como é exemplo o locus TH01, que está incluído na maioria dos kits para criação de perfis genéticos (Klinstar et al., 2004 e Klinstar et al., 2005). Este marcador, localizado no primeiro intrão do gene que codifica a enzima tirosina hidroxilase, tem sido bastante controverso, pela possibilidade de estar associado a várias patologias. Thibaut et al. (1997) e Burgert et al. (1998) realizaram estudos em que conseguiram observar uma associação com determinados alelos do locus TH01 em indivíduos diagnosticados com doença bipolar ou esquizofrenia, mas por outro lado McQuillin et al. (1999) obtiveram resultados que viriam a demonstrar que esta associação não era estabelecida; Sharma et al. (1998) e Klintschar et al. (2004) obtiveram resultados contraditórios quando tentaram relacionar os alelos 9.3 e 10 deste marcador com a hipertensão, e trabalhos realizados mais recentemente por Anney et al. (2004) indicaram ainda que indivíduos portadores do alelo 7 do TH01 poderão ser mais resistentes à dependência de nicotina e menos propícios a hábitos tabágicos viciantes, não havendo contudo resultados determinantes.
16
Existe apenas uma excepção que, pelo facto de ter sido provada a sua associação com diversas patologias genéticas, resultou na remoção do STR HumARA do painel usado para identificação humana. Este marcador caracteriza-se pela repetição sequencial de trinucleótidos CAG e encontra-se localizado numa região codificante do cromossoma X - exão 1 do gene que codifica um receptor de androgénio (Szibot et al., 2005). Contudo, verifica-se que este marcador possui características que não são partilhadas pelos restantes STRs usados na identificação humana e que poderão inclusivamente estar na base da explicação da sua relação com as referidas patologias: por um lado a sua localização numa região codificante; por outro, o facto de ser constituído por uma repetição trinucleotídica, estando por isso aumentada a propensão para fenómenos de expansão, o que constitui uma causa de distúrbio genético (Tian et al., 2005).
Alguns dos STRs que constituem o painel para identificação humana têm vindo a ser considerados úteis no diagnóstico de determinadas patologias genéticas, através da perda de heterozigotia ou desequilíbrio alélico. Por exemplo, o locus D8S1179 foi utilizado para localizar um gene relacionado com o Síndrome de Meckel-Gruber, uma das causas mais frequentes de defeitos do tubo neural (Morgan et al., 2005); um outro estudo aplicado a cerca de 400 STRs demonstrou que o marcador anteriormente referido poderia estar associado com o gene responsável pela microalbuminúria, que prejudica a função renal e pode elevar o risco de doenças cardiovasculares (Fox et al., 2005).
Além dos microssatélites anteriormente referidos há ainda que mencionar o locus D21S11, que pelo facto de se localizar no cromossoma 21 pode ser associado à Trissomia 21 ou Síndrome de Down, que é uma patologia que pode ser detectada regularmente pela presença de três alelos em qualquer marcador polimórfico naquele cromossoma (Liou et al., 2004); de igual modo, o D18S51 poderá ser caracterizado em amostras no diagnóstico pré-natal do Síndrome de Edwards ou Trissomia 18 (Yoon et al., 2002).
Além dos exemplos acima enunciados, a perda de heterozigotia ou um acentuado desequilíbrio alélico também podem, nalguns casos, estar associados a patologias neoplásicas.
17 1.1.7 - STRS e patologia oncológica
A patologia oncológica encontra-se frequentemente associada à instabilidade genética, permitindo a acumulação de um conjunto de alterações oncogénicas num curto espaço de tempo, necessárias para transformar uma célula normal numa célula neoplásica. Hartwell (1992) constatou que a perda de funcionalidade dos sistemas responsáveis pela salvaguarda da integridade do genoma era determinante para o desenvolvimento de um processo tumoral multifásico.
Existem duas formas principais de instabilidade genética: uma das formas inclui alterações cromossómicas, como aneuploidia, delecções ou expansões; a outra é mais subtil e manifesta-se por variações no comprimento das porções de DNA constituídas pelas sequências microssatélites. Nas células normais, o número de repetições de uma determinada sequência é herdada de uma forma altamente conservativa; nas células tumorais acredita-se que a instabilidade genética resulta de um aumento de erros de deslizamento (slippage) ocorridos durante a replicação do DNA - uma dissociação momentânea entre a cadeia molde e a cadeia complementar entretanto sintetizada seguida de um re-emparelhamento incorrecto pode resultar em unidades de repetição desemparelhadas, o que explica a ocorrência tanto da contracção como da expansão dos microssatélites (Strand et al., 1993).
Este fenómeno foi inicialmente denominado de fenótipo RER+ (Replication Error-Positive), sendo agora referido como MIN ou MSI (Microsatellite Instability). Apesar das alterações no comprimento dos microssatélites serem fenotipicamente silenciosas, podem ser um reflexo da falta de fidelidade na replicação do DNA. Assim, a transformação oncogénica das células que apresentem MSI pode ser acelerada por uma rápida acumulação de erros em proto-oncogenes e genes supressores tumorais (Loeb, 1994).
Em 1993, Thibodeau e colaboradores observaram MSI pela primeira vez em tumores associados com cacinoma colorectal hereditário não-polipótico (HNPCC). A elevada incidência de MSI neste tipo de cancro sugeria que a predisposição para o desenvolvimento desta patologia podia estar associada a algum tipo de instabilidade genética, provavelmente a mutações dos genes reparadores do DNA, mais tarde identificados como os genes MLH1 e MSH2 do sistema MMR. Após esta descoberta, não demorou muito tempo até que surgissem dados relativos à instabilidade dos microssatélites noutros tipos de tumores, nomeadamente cancros esporádicos, não associados ao HNPCC ou a outro tipo de neoplasias de transmissão hereditária.
O estado de MIS em tumores é determinado facilmente, comparando após amplificação por PCR, o comprimento de determinados microssatélites do DNA do
18
tecido tumoral com o DNA do tecido normal do mesmo indivíduo, sendo necessário que se observem variações a nível do número de repetições e consequentemente do comprimento do microssatélite, em cerca de 25% dos marcadores estudados. Contudo, é exigida a devida precaução na interpretação dos resultados, visto que se pode obter resultados desiguais se forem analisados microssatélites distintos. Além disso, podem surgir falsos negativos nos casos em que a instabilidade dos microssatélites seja investigada usando um reduzido número de microssatélites na totalidade do DNA tumoral (Jacoby et al., 1995).
Os microssatélites forenses utilizados para a elaboração do perfil genético de um indivíduo e para a sua identificação têm sido utilizados para avaliar a instabilidade genética em vários tipos de tumores.
A elevada variabilidade destes marcadores resulta das dificuldades que eles impõem aos processos de replicação do DNA e aos seus mecanismos de reparação, elevando a probabilidade de ocorrência de mutações neste tipo de sequências, quando comparado com regiões do DNA não repetitivo. A sua elevada mutabilidade possibilita a existência de uma grande variedade de alelos para um determinado marcador numa população. Por este motivo, os microssatélites localizados próximo ou em genes associados a tumores têm sido utilizados para localizar esses genes e analisar anomalias genéticas em células tumorais.
Para isso, além da MSI é também pesquisada a perda de heterozigotia ou LOH (Loss of Heterozygosity), que é um processo definido pela mutação de um alelo seguida da delecção do alelo normal, o que acontece quer pela delecção da porção do cromossoma que contém esse locus, quer por mecanismos de recombinação somática, levando à substituição do alelo normal por uma cópia duplicada do alelo mutado (Besnard-Gu’erin et al., 1996). A LOH é uma alteração genética muito frequente que se pode encontrar ao longo de todo o genoma da maioria das neoplasias, sendo usualmente indicativa de instabilidade cromossómica, aneuploidia ou rearranjos cromossómicos (Lasko et al., 1991). As regiões que apresentem LOH podem indicar a existência de mutações em genes supressores tumorais, sendo muito importante para o prognóstico da doença (Fearon et al., 1990 e Sidransky et al., 1997).
Em tecidos tumorais nos quais exista uma mistura de células tumorais e células normais poderão ocorrer falsos negativos e a perda de heterozigotia só poderá ser detectada através da análise do desequilíbrio observado entre cada um dos dois alelos de um locus heterozigótico, denominado desequilíbrio alélico ou AI (Allelic Imbalance) (Slebos et al., 2004). Sabe-se que a existência de desequilíbrio alélico num elevado número de loci de um tecido eleva a probabilidade do comprometimento de diversos
19
genes responsáveis por um fenótipo neoplásico, o que poderá ter implicações a nível do diagnóstico e do prognóstico da doença (Heaphy et al., 2007).
Se por um lado, pela perspectiva da patologia oncológica, o estudo da estabilidade genética através de microssatélites se revela vantajoso para uma melhor compreensão dos mecanismos genéticos associados à carcinogénese e da especificidade das alterações ocorridas no material genético das células tumorais, por outro lado, no contexto das ciências forenses, o estudo dos STRs neste tipo de patologias permite aferir quanto à estabilidade destes marcadores não apenas no tecido normal, mas em diferentes tipos de tecidos geneticamente alterados, de acordo com as características específicas de cada processo tumoral.
Neste sentido, tem sido desenvolvida uma enorme variedade de estudos nos últimos anos, a fim de observar como se comportam os marcadores forenses nas mais diversas patologias neoplásicas.
Ceccardi et al. (2006) analisaram a estabilidade de 15 STRs em 68 amostras correspondentes a três tipos de tecido tumoral distintos (cancro oral, urogenital e gastrointestinal) e observaram alterações alélicas em 54,4% dos casos, sendo os marcadores mais afectados os loci vWA, FGA e D18S51. O autor sugere muito cuidado na leitura dos perfis genéticos obtidos a partir deste tipo de tecidos, especialmente na ausência de uma amostra de tecido normal para comparação.
Edelmann et al. (2004) investigaram a LOH e a MSI em doentes com carcinoma cervical humano e, baseados na análise de 21 amostras, concluíram que estes fenómenos são frequentes em determinados loci e que os STRs são afectados pela instabilidade genética, o que condiciona a aplicação dos tecidos tumorais em procedimentos de interesse forense.
Itoh et al. (2004) concluíram num estudo relativo aos STRs TH01, TPOX e CS1PO realizado com amostras de urina de 52 indivíduos com cancro da bexiga, que é necessária muita precaução no processamento laboratorial deste tipo de amostras para efeitos de identificação e que os marcadores referidos poderão ser uma ferramenta útil no diagnóstico da neoplasia em estudo.
Poetsch et al. (2004) aconselharam a utilização de tecido tumoral apenas quando não exista outro tipo de amostra biológica, após o estudo de 118 amostras de diferentes tipos de tumores sólidos e 62 metástases nas quais se observou um maior número de alterações a nível dos marcadores FGA, D3S1558, D18S51 e D21S11.
Vauhkonen et al. (2004) compararam a estabilidade dos STRs dos kits comercias utilizados com maior frequência, em biópsias de cancro gastrointestinal e concluíram que quando estes kits são aplicados, a obtenção de falsa heterozigotia ou homozigotia
20
compromete a tipagem genética, mesmo no que à determinação do género masculino ou feminino diz respeito.
Peloso et al. (2003) compararam os resultados obtidos em 24 biópsias de cancro do pulmão com amostras de tecido normal extraídas de nódulos linfáticos dos mesmos doentes e observaram instabilidade genética na grande maioria das amostras, com desequilíbrio alélico em todos os STRs analisados. Peloso afirma que este tipo de amostras afecta o processo de identificação humana baseada no estudo destes marcadores.
Pai et al. (2002) realizaram um estudo com 300 sujeitos e concluíram que as zaragatoas bucais de indivíduos com cancro oral não podem ser empregues na identificação genética, sendo que não existe qualquer objecção à utilização deste tipo de amostras em indivíduos que não apresentem sintomas de outro tipo de patologia ou lesão da cavidade bucal.
Além destes, outros investigadores tinham já comprovado a instabilidade genética de vários microssatélites forenses que poderiam estar relacionados com as patologias em estudo: Hoff Olsen et al. (1998) obtiveram alterações nos STRs em 19% das amostras de carcinoma colorectal; Silva et al. (1997 e 1998) observaram instabilidade dos microssatélites em casos de cancro gástrico; Rubocki et al. (2000) demonstraram perda de heterozigotia do locus D13S317 em amostras de cancro da bexiga. Por outro lado, Orlandi et al. (2002) não conseguiram obter alterações significativas quando aplicaram este tipo de estudo ao cancro da mama.