Faculdade de Ciências
Departamento de Biologia Animal
Bactérias de Gram negativo produtoras de β-lactamases e
mecanismos moleculares implicados na expressão da
resistência à tigeciclina
Stephanie Vieira Barbosa
Dissertação
Mestrado em Biologia Humana e Ambiente
2013
Bactérias de Gram negativo produtoras de β-lactamases e
mecanismos moleculares implicados na expressão da
resistência à tigeciclina
Stephanie Vieira Barbosa
Faculdade de Ciências
Departamento de Biologia Animal
Dissertação
Mestrado em Biologia Humana e Ambiente
Orientadores
Professora Doutora Manuela Caniça, Instituto Nacional de Saúde Dr. Ricardo Jorge
Professora Doutora Maria Teresa Rebelo, Departamento de Biologia Animal, Faculdade de
Ciências, Universidade de Lisboa
i
Agradecimentos
À Doutora Manuela Caniça, por me receber no laboratório, proporcionando a realização desta tese de mestrado, assim como pela orientação, disponibilidade e conhecimentos partilhados, que tanto acrescentou na minha formação.
À Doutora Eugénia Ferreira, pela orientação e contribuição no laboratório, bem como pela boa disposição que tornou a rotina mais agradável.
À Doutora Teresa Rebelo, pelo acompanhamento e disponibilidade demonstrada.
À Doutora Vera Manageiro pela paciência e pela enorme ajuda no desenvolvimento das práticas laboratoriais, que tanto contribuiu na conclusão deste trabalho.
À Daniela Dias por toda a ajuda prestada no laboratório e partilha de conhecimentos, principalmente na parte final deste trabalho.
À Constança Simões e Joana Almeida pela amizade, pelos bons momentos e por me ajudarem nos momentos que eu precisei.
Aos meus pais, por todo amor e apoio constante, apesar das centenas de quilómetros que nos separam.
À minha família por todo apoio, principalmente à minha irmã Cássia pelo momentos compartilhados e pela paciência ao longo deste ano.
Aos meus amigos, que sabem quem são, pela amizade, incentivo e por terem acreditado sempre em mim.
À Doutora Manuela Caniça e ao Instituto Nacional da Saúde Dr. Ricardo
Jorge pelas condições de trabalho que me proporcionaram e colaboração prestada sem
ii
Resumo
A emergência de bactérias de Gram negativo com resistência aos antibióticos, particularmente, as produtoras de β-lactamases, tem vindo a comprometer as opções de tratamento disponíveis. Este trabalho teve como objetivo avaliar a suscetibilidade e caracterizar os mecanismos de resistência à tigeciclina, pela ação de bombas de efluxo, em 211 isolados de Gram negativo, assim como esclarecer a atividade in vitro deste antibiótico em bactérias com suscetibilidade diminuída a outras classes de antibióticos, sobretudo β-lactâmicos, e com eventual produção de β-lactamases.
Os testes de suscetibilidade permitiram identificar 73% de isolados suscetíveis à tigeciclina e 89% com multirresistência, dos quais 70% eram suscetíveis à tigeciclina. Verificou-se ainda uma percentagem elevada de suscetibilidade à colistina e amicacina, bem como de suscetibilidade diminuída aos antibióticos β-lactâmicos, com exceção dos carbapenemes.
A caracterização molecular dos mecanismos de resistência aos antibióticos β-lactâmicos, permitiu identificar a produção de β-lactamases: penicilinases, ESBLs da família CTX-M [CTX-M-15-(tipo), CTX-M-1, CTX-M-32 e CTX-M-14], TEM (TEM-4, TEM-10), SHV (SHV-2, SHV-12, SHV-55) e GES (GES-7), bem como carbapenemases (KPC-3) e AmpC (CMY-2, DHA-1, MIR-tipo); 65% destes isolados apresentaram suscetibilidade à tigeciclina.
A caracterização molecular dos mecanismos implicados na resistência à tigeciclina, permitiu identificar no gene ramR de 9 dos 20 isolados de Klebsiella
pneumoniae com suscetibilidade diminuída àquele antibiótico, uma deleção, uma
inserção e de uma a quatro mutações pontuais num mesmo gene, contribuindo para a sobre-expressão da bomba de efluxo AcrAB. No gene marR de 12 isolados de
Escherichia coli (5 com e 6 sem suscetibilidade diminuída à tigeciclina), foram
detetadas duas mutações pontuais, que sugerem estar associadas à resistência a diversos antibióticos, podendo contribuir para fenótipos de multirresistência.
Este estudo evidencia diversidade de β-lactamases, sobretudo ESBL, e emergência de carbapenemases em isolados de Gram negativo. No entanto, estes isolados mantêm uma importante suscetibilidade aos cabapenemes, colistina, amicacina e tigeciclina.
iii
Abstract
The emergence of antibiotic resistant Gram negative bacteria, particularly β-lactamase-producing bacteria, seriously compromises the efficacy of the treatment options available. This study aimed to evaluate the antimicrobial susceptibility, and to characterize the tigecycline resistance due to efflux pump production, in a collection of 211 Gram negative clinical isolates. Moreover, it meant to clarify the in vitro activity of tigecycline against isolates resistant to other antibiotic classes, such as β-lactams, eventually related to β-lactamase production.
Susceptibility testing evaluation identified 73% of tigecycline susceptible isolates, and 89% of multidrug resistance, among which 70% were susceptible to this antibiotic. Although there was a high prevalence of β-lactam resistance, carbapenems were still effective against the majority of the isolates studied, as well as colistin and amikacin.
Globally, molecular characterization allowed the detection of penicillinases, ESBLs from families CTX-M [15-(type), 1, 32 and CTX-M-14], TEM (TEM-4, TEM-10), SHV (SHV-2, SHV-12, SHV-55) and GES (GES-7), as well as carbapenemases (KPC-3) and AmpC β-lactamases ( CMY-2, DHA-1, MIR-tipo); among those, 65% were susceptible to tigecycline.
The molecular analyses of tigecycline resistance mechanisms revealed one deletion, one insertion and one to four point mutations in the ramR gene that might contribute to the overexpression of AcrAB efflux pump, in 9 out of 20 Klebsiella
pneumoniae isolates showing reduced susceptibility. Considering the analyses of the marR gene from 12 Escherichia coli isolates (5 with and 6 without tigecycline
resistance), two point mutations were detected, which might be accountable for the resistance to several antimicrobial classes, contributing to multidrug resistance scenarios.
This study showed a great diversity of β-lactamases, such as ESBL, and the emergence of carbapenemases in Gram negative bacteria. Nevertheless, the studied isolates still showed decisive susceptibility patterns to carbapenems, colistin, amikacin and tigecycline.
iv Índice Agradecimentos ... i Resumo ... ii Abstract ... iii Índice de Figuras ... vi
Índice de Tabelas ... vii
I. Introdução ... 1
1. Antibióticos e Resistências ... 1
1.1.Classes de Antibióticos ... 1
1.2.Mecanismos de resistência aos antibióticos ... 2
1.2.1 Resistência Intrínseca ... 3
1.2.2. Resistência Adquirida ... 3
2. Antibióticos β-lactâmicos ... 4
2.1. Mecanismo de ação dos antibióticos β-lactâmicos ... 7
2.2. Mecanismos de resistência aos antibióticos β-lactâmicos ... 7
2.2.1. β-lactamases ... 8 2.2.1.1 Classificação de β-lactamases ... 9 2.2.1.2. Família TEM ... 10 2.2.1.3. Família SHV ... 11 2.2.1.4. Família CTX-M ... 11 2.2.1.5. Família OXA ... 12 2.2.1.6. β-lactamases AmpC... 13 2.2.1.7. Carbapenemases ... 14 3. Tigeciclina ... 15
3.1. Estrutura química e mecanismo de ação da tigeciclina ... 16
3.2. Espetro de ação da tigeciclina ... 17
3.3. Resistência à tigeciclina ... 18
3.3.1. Bomba de efluxo AcrAB-TolC da família RND ... 19
4. Objetivos ... 23
II. Material e Métodos ... 24
1.Isolados bacterianos ... 24
2. Caracterização fenotípica: suscetibilidade aos antibióticos ... 25
2.1. Método de microdiluição ... 25
2.1.1. Preparação da solução-mãe dos antibióticos ... 25
v
2.1.3. Preparação das diluições dos antibióticos na microplaca ... 26
2.1.4. Preparação do inóculo bacteriano... 27
2.2. Método de difusão por disco ... 28
2.3. Método de E-teste... 29
2.4. Teste de sinergia com meropeneme ... 29
3. Caracterização genotípica: mecanismos de resistência aos antibióticos ... 30
3.1. Extração de DNA ... 30
3.2. Pesquisa de genes que codificam β-lactamases ... 31
3.3. Pesquisa de genes implicados na resistência à tigeciclina... 34
3.4. Eletroforese em gel de agarose e visualização de DNA ... 35
3.5. Purificação dos produtos de PCR ... 35
3.6. Sequenciação nucleotídica ... 36
4. Caracterização bioquímica: β-lactamases ... 36
4.1. Extração de β-lactamases ... 37
4.2. Teste do Nitrocefin ... 37
4.3. Gel de focagem isoelétrica ... 37
4.3.1. Preparação do gel de migração ... 37
4.3.1.1. Migração ... 38
4.3.1.2. Preparação do gel de revelação ... 38
III. Resultados ... 39
1. Avaliação da suscetibilidade de bactérias de Gram negativo aos antibióticos ... 39
1.1. Suscetibilidade a 8 classes de antibióticos ... 39
1.2. Suscetibilidade à tigeciclina ... 42
1.3. Análise da multirresistência ... 47
2. Caracterização genotípica dos isolados produtores de β-lactamases ... 49
2. 1. ESBLs, PMAβ e penicilinases ... 49
2. 2. Carbapenemases ... 56
3. β-lactamases de diferentes famílias produzidas por bactérias de Gram negativo com ou sem suscetibilidade à tigeciclina ... 57
4. Contribuição da bomba de efluxo AcrAB-TolC na atividade da tigeciclina ... 59
4.1. Análise da sequência nucleotídica do gene marR em E. coli ... 59
4.2. Análise da sequência nucleotídica do gene ramR em K. pneumoniae ... 59
IV. Discussão ... 62
vi
Índice de Figuras
Figura 1 - Estrutura química dos antibióticos β-lactâmicos (Adaptado de Martín et al., 2010; Nordmann et al., 2012). ... 5 Figura 2 - Estrutura química dos inibidores de β-lactamases (Adaptado de Nordmann et al., 2012). ... 5 Figura 3 - Classificação das ß-lactamases relacionando o perfil do substrato e dos inibidores de ß-lactamases com a sua estrutura molecular. Cb, carbapenemes; Cf, cefalosporinas de primeira geração; CA, ácido clavulânico; EDTA, ácido etilenodiaminotetracético; Esc, Cefalosporina de espectro alargado; M, monobactamos; Pn, penicilinas (Adaptado de Bush, 2013). ... 10 Figura 4 - Estrutura química da tigeciclina (A) e da minociclina (B) (Dean et al. 2003) ... 17 Figura 5 - Estrutura do sistema de efluxo AcrAB-TolC em E.coli (Adaptado de Alvarez-Ortega et al., 2013). ... 20 Figura 6 - Organização genética do locus marAB em E. coli ; PmarI e PmarII, promotores (Alekshun e Levy, 1999). ... 21 Figura 7 - Via de regulação da bomba de efluxo AcrAB: a proteina RamR reprime a expressão do gene ramA que codifica o activador trancripcional acrAB. (Adaptado de Yamasaki et al., 2013). ... 22 Figura 8 - Distribuição, pelos hospitais de origem, de 211 isolados que constituem a amostra do estudo. ... 24 Figura 9 - Representação da diluição seriada dos antibióticos na microplaca ... 27 Figura 10 - Esquema utilizado para interpretação dos resultados obtidos no teste de sinergia com meropeneme para deteteção de metalo-lactamases, carbapenemases de classe A ou D e β-lactamases AmpC (cromossómicas ou plasmídicas). BOR, ácido borónico; CLOX, cloxacilina. ... 30 Figura 11 - Distribuição de 199 isolados suscetíveis e não suscetíveis à tigeciclina por grupo etário dos doentes. TG, tigeciclina; S, sucetíveis; I/R, suscetibilidade diminuída; (Não contempla 12 (6%) dos isolados do estudo, cuja idade dos doentes se desconhece). ... 43 Figura 12 - Distribuição do (A) número de isolados e (B) percentagem, de acordo com a suscetibilidade à tigeciclina, multirresistência e produção de β-lactamases; OXA: OXA-1-tipo (n=9); ND, mecanismo de resistência não detetado ou sobrexpressão de β-lactamases (n=16). a
Isolados cuja produção de ESBL CTX-M (isoladamente ou com coexpressão de KPC ou PMAβ) não implicou a eventual pesquisa da presença de outros genes blaESBL. TGC, tigeciclina; S, suscetível; I/R, suscetibilidade diminuída; MDR, multirresistente. ... 58 Figura 13 - Alinhamento das sequências do gene ramR com a sequência de referência (Klebsiella pneumoniae subsp. pneumoniae MGH 78578 (CP000647)) do nucleótido G401 ao nucleótido G480, representando a deleção nucleotídica do nucleótido G411 ao nucleótido A421 no isolado TG93. ... 60 Figura 14 - Alinhamento das sequências do gene ramR com a sequência de referência (Klebsiella pneumoniae subsp. pneumoniae MGH 78578 (CP000647)) do nucleótido G1 ao nucleótido G80, representando a inserção de 2 nucleótidos entre o nucleótido 50 e 51 no isolado TG70. ... 60
vii
Índice de Tabelas
Tabela 1 - Representação de antibióticos, mecanismos de ação e alvos moleculares (Chopra et al., 2002; Murray et al., 2007; Tenover, 2006) ... 2 Tabela 2 - Classificação das cefalosporinas e exemplos de antibióticos (Murray et al., 2007). ... 6 Tabela 3 - Origem de genes que codificam β-lactamases AmpC (Adaptado de Livermore e Woodford, 2006) ... 14 Tabela 4 - Antibióticos, concentrações das soluções-mãe, solventes e potência dos antibióticos. ... 25 Tabela 5 - Antibióticos, concentração da solução CIM, diluentes e concentrações testadas na determinação da concentração inibitória mínima. ... 26 Tabela 6 - Sequência de primers para pesquisa de diferentes genes bla utilizados no PCR e na sequênciaçãoa, tamanho dos respetivos fragmentos e temperatura de hibridação. ... 32 Tabela 7 - Sequência e tamanho dos primers ramR e marAB e as condições utilizadas nas reações de amplificação. ... 35 Tabela 8 - Suscetibilidade de 211 isolados de Gram negativo a antibióticos de diferentes classes. ... 41 Tabela 9 - Intervalo de concentrações inibitórias mínimas (CIM) nas quais 50% (CIM50) e 90%
(CIM90) dos isolados foram inibidos por 7 antibióticos de 4 classes diferentes, em relação ao
total de isolados (n=211) e à suscetibilidade ou suscetibilidade diminuída à tigeciclina. ... 44 Tabela 10 - Distribuição de 187 isolados com multirresistência aos antibióticos, em 211 isolados de Gram negativo, realçando os perfis com e sem suscetibilidade diminuída à tigeciclina, por espécie bacteriana. ... 48 Tabela 11 - Genótipo de 177 isolados, de diferentes espécies, com suscetibilidade diminuída à cefotaxima, detetados como possíveis produtores de ESBL por métodos fenotípicos: β-lactamases expressas, perfil de resistência aos antibióticos β-lactâmicos e distribuição por hospital a b. ... 51 Tabela 12 - Genótipo de 2 isolados com suscetibilidade diminuída à cefotaxima, produtores de ESBL, mas sem evidência fenotípica de produção de ESBL: β-lactamases expressas, perfil de resistência aos antibióticos β-lactâmicos e distribuição por hospital. ... 53 Tabela 13 - Genótipo de 32 isolados com ou sem suscetibilidade diminuída à cefotaxima, sem evidência fenotípica e genotípica de produção de ESBL: β-lactamases expressas, perfil de resistência aos antibióticos β-lactâmicos e distribuição por hospital. ... 55 Tabela 14 - Isolados produtores de carbapenemase de classe A (KPC-3) ... 56 Tabela 15.β-lactamases identifcadas nos 211 isolados estudados, com e sem blaCTX-M. ... 57
Tabela 16 - Mutações nucleotídicas no gene marR da estirpe de referência e de 12 isolados clínicos e respetivas substituições aminoacídicas. ... 59 Tabela 17 - Mutações nucleotídicas no gene ramR da estirpe de referência MGH 78578 (CP000647) e de 20 isolados clínicos e respetivas substituições aminoacídicas encontradas. ... 61
1
I. Introdução
1. Antibióticos e Resistências
A descoberta e o desenvolvimento dos antibióticos no início do século XX – com a introdução das sulfonamidas nos anos 30 e da penicilina nos anos 40 – foi um marco importante na história da medicina. Desde então foram descobertas várias classes de antibióticos, quer de origem natural, quer sintética: aminoglicosídeos, glicopeptídeos, macrólidos, fluoroquinolonas, entre outras (Van Hoek et al., 2011). Contudo, as bactérias desenvolveram mecanismos de resistência aos antibióticos, resultante, essencialmente, da utilização inapropriada e indiscriminada dos antibióticos. Isto deve-se à pressão deve-seletiva, que resulta do uso, deve-sem controlo, de antibióticos, tanto a nível clínico, como ao nível comercial, proporcionando a seleção e predominância de microrganismos resistentes (Rice, 2009). A emergência e a prevalência destes microrganismos constitui uma das mais importantes ameaças à saúde pública, a nível mundial, com extensivas implicações na morbilidade e mortalidade, assim como no aumento dos custos em saúde e cuidados médicos(Fraise, 2006).
1.1. Classes de Antibióticos
As classes de antibióticos utilizados no tratamento de infeções por bactérias são determinadas de acordo com os alvos em que atuam e com os mecanismos de ação que determinam (Tabela 1). Os antibióticos podem exercer uma atividade bactericida ou bacteriostática, podendo ser classificados da seguinte forma: anti-parietais, quando inibem a síntese de peptidoglicano da parede celular; anti-membranares, quando alteram a permeabilidade da membrana celular; inibidores da síntese proteica, associando-se a alvos ribossomais; inibidores da síntese dos ácidos nucleicos; e anti-metabólitos, quando alteram o metabolismo energético da célula bacteriana (Murray et al., 2007; Tenover, 2006).
A classe dos β-lactâmicos e glicilciclinas, com maior relevância para o presente trabalho, encontram-se descritas com mais detalhe nos tópicos 2 e 3, respetivamente.
2
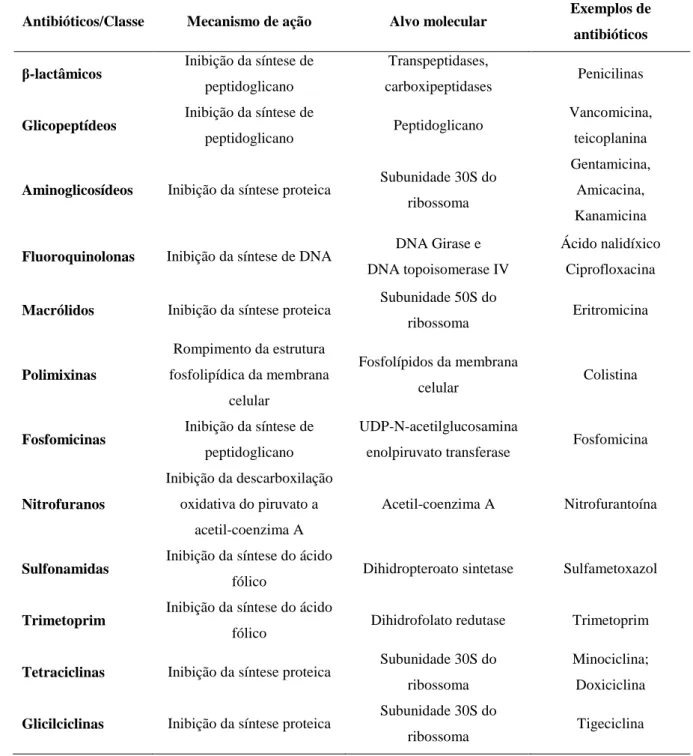
Tabela 1 - Representação de antibióticos, mecanismos de ação e alvos moleculares (Chopra et al., 2002; Murray et al., 2007; Tenover, 2006)
Antibióticos/Classe Mecanismo de ação Alvo molecular Exemplos de antibióticos β-lactâmicos Inibição da síntese de
peptidoglicano
Transpeptidases,
carboxipeptidases Penicilinas
Glicopeptídeos Inibição da síntese de
peptidoglicano Peptidoglicano
Vancomicina, teicoplanina
Aminoglicosídeos Inibição da síntese proteica Subunidade 30S do ribossoma
Gentamicina, Amicacina, Kanamicina
Fluoroquinolonas Inibição da síntese de DNA DNA Girase e DNA topoisomerase IV
Ácido nalidíxico Ciprofloxacina
Macrólidos Inibição da síntese proteica Subunidade 50S do
ribossoma Eritromicina Polimixinas Rompimento da estrutura fosfolipídica da membrana celular Fosfolípidos da membrana celular Colistina
Fosfomicinas Inibição da síntese de
peptidoglicano
UDP-N-acetilglucosamina
enolpiruvato transferase Fosfomicina
Nitrofuranos Inibição da descarboxilação oxidativa do piruvato a acetil-coenzima A Acetil-coenzima A Nitrofurantoína Sulfonamidas
Inibição da síntese do ácido
fólico Dihidropteroato sintetase Sulfametoxazol
Trimetoprim Inibição da síntese do ácido
fólico Dihidrofolato redutase Trimetoprim
Tetraciclinas Inibição da síntese proteica Subunidade 30S do ribossoma
Minociclina; Doxiciclina
Glicilciclinas Inibição da síntese proteica Subunidade 30S do
ribossoma Tigeciclina
1.2. Mecanismos de resistência aos antibióticos
Apesar da diversidade de classes e mecanismos de ação dos antibióticos, as bactérias desenvolveram estratégias de resistência a estes compostos (Barbosa e Levy, 2000a). Os mecanismos de resistência aos antibióticos incluem: alteração do local de
3 ação do antibiótico; alteração da permeabilidade da membrana celular, impedindo a entrada e ação do antibiótico nas células bacterianas; ativação de bombas de efluxo na célula bacteriana, conduzindo à expansão do antibiótico para o exterior antes de alcançar o local alvo; aumento da produção da enzima alvo e inativação do antibiótico por ação enzimática (Van Hoek et al., 2011; Tenover, 2006). De realçar que a resistência das bactérias pode ser intrínseca ou adquirida.
1.2.1 Resistência Intrínseca
A resistência intrínseca, é transmitida apenas verticalmente, de geração em geração, fazendo parte das propriedades inatas, fenotípicas de microrganismos de uma mesma espécie (Alekshun e Levy, 2007). As bactérias de Gram negativo, por exemplo, são intrinsecamente resistentes aos glicopeptídeos. Isto resulta da incapacidade de difusão via canais de porina na membrana externa celular, uma vez que a molécula é muito grande (Woodford e Ellington, 2007).
1.2.2. Resistência Adquirida
Por outro lado, a resistência adquirida ocorre quando populações de bactérias inicialmente suscetíveis se tornam resistentes a um antibiótico. O aumento da prevalência da resistência aos antibióticos está, assim, principalmente associado à aquisição e disseminação de genes de resistência aos antibióticos pela ação seletiva e pela pressão do antibiótico utilizado (Tenover, 2006). As bactérias podem adquirir resistência aos antibióticos, quer modificando o seu genoma por mutação, quer por diferentes sistemas de transferência genética (Levy e Marshall, 2004; Tenover, 2006).
A resistência adquirida por meio de mutação e seleção denomina-se evolução vertical, pois as bactérias com novas mutações, responsáveis pela resistência, são selecionadas pela utilização dos antibióticos e estes eliminam as bactérias suscetíveis, enquanto as resistentes sobrevivem e disseminam (Tenover, 2006).
A resistência adquirida pode ainda resultar da aquisição de elementos genéticos, tais como os plasmídeos, transposões e integrões, que permitem a transferência de DNA no genoma ou ainda entre células bacterianas, facilitando a transmissão e disseminação de genes de resistência aos antibióticos através de mecanismos genéticos (Levy e
4 Marshall, 2004). A transferência genética denominada evolução horizontal constitui o principal meio de difusão de genes de resistência, que pode ocorrer entre isolados da mesma espécie ou entre isolados de diferentes espécies ou géneros (Woodford e Ellington, 2007). A transferência genética entre as bactérias pode ocorrer de diferentes formas: transformação, conjugação e transdução. A transformação é um mecanismo pelo qual o DNA libertado por uma bactéria (dadora) é captado e incorporado por outras bactérias (recetoras). Na conjugação há contacto físico bactéria-bactéria e o material genético é transferido através de pilis de uma célula (dadora) para a outra (recetora). A transdução é um processo que consiste na transferência de genes de resistência por intermédio de um bacteriófago (Alekshun e Levy, 2007; Barbosa e Levy, 2000a; Tenover, 2006). Através destes mecanismos de troca genética muitas bactérias podem tornar-se resistentes a várias classes de antibióticos; se a resistência ocorrer em 3 ou mais classes de antibióticos estruturalmente diferentes, trata-se de bactérias multirresistentes, as quais constituem a causa de maior preocupação, tanto a nível hospitalar como a nível da comunidade (Levy e Marshall, 2004).
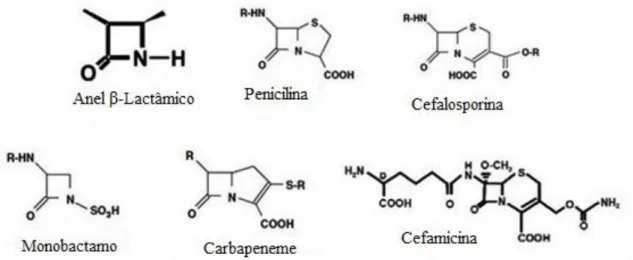
2. Antibióticos β-lactâmicos
Os antibióticos β-lactâmicos são a classe mais variada de antibióticos, sendo amplamente utilizada em todo o mundo devido à sua eficácia terapêutica, baixa toxicidade e propriedades farmacocinéticas (Livermore e Woodford, 2006; Suárez e Gudiol, 2009). É um grupo de antibióticos com a característica comum de exibir um anel ß-lactâmico na sua estrutura molecular (Fig.1). Esta classe divide-se em diferentes grupos: penicilinas, cefalosporinas, cefamicinas, inibidores de ß-lactamases (ácido clavulânico, sulbactam e tazobactam), carbapenemes e monobactamo (Drawz e Bonomo, 2010; Murray et al., 2007).
As penicilinas têm como estrutura básica um núcleo pename bicíclico com um anel tiazolidina (com um átomo de enxofre), um anel β-lactâmico e uma cadeia lateral ligada ao grupo amina (Fig. 1). As penicilinas são frequentemente produzidas por espécies Penicillium e Aspergillus e são classificadas de acordo com o espetro de atividade (Liras e Martín, 2006; Martín et al., 2010).
5
Figura 1 - Estrutura química dos antibióticos β-lactâmicos (Adaptado de Martín et al., 2010; Nordmann et al., 2012).
A penicilina G e a penicilina V pertencem ao grupo das penicilinas naturais, tendo sido a penicilina G (Benzilpenicilina) a primeira a ser desenvolvida. A partir desta produziram-se novas estruturas e, consequentemente, descobriram-se novos antibióticos β-lactâmicos, atualmente utilizados na clínica médica (Drawz e Bonomo 2010). As penicilinas dividem-se em diferentes subgrupos: as penicilinas resistentes às penicilinases (p.e. oxacilina e cloxacilina), aminopenicilinas (p.e. amoxicilina), carboxipenicilinas (p.e. ticarcilina) e ureidopenicilinas (p.e. piperacilina).
Os inibidores de lactamases são compostos associados a alguns antibióticos β-lactâmicos de forma a inativar as β-lactamases e potenciar a ação do antibiótico. O ácido clavulânico, o sulbactam e o tazobactam (Fig. 2) são inibidores de β-lactamases irreversíveis, frequentemente utilizados na prática clínica (Murray et al., 2007).
Figura 2 - Estrutura química dos inibidores de β-lactamases (Adaptado de Nordmann et al., 2012).
As cefalosporinas constituem outro grupo de antibióticos β-lactâmicos, tendo, no entanto, maior atividade sobre as bactérias de Gram negativo em comparação com as penicilinas. O anel β-lactâmico encontra-se associado a um anel di-hidrotiazina (Fig.1).
6 São derivados do ácido 7-aminocefalosporânico, obtidos inicialmente do fungo
Cephalosporium. Os antibióticos deste grupo encontram-se classificados em quatro
gerações, de acordo com o espetro de atividade e período de introdução na prática clínica (Tabela 2) (Murray et al., 2007).
As cefamicinas (cefoxitina, cefotetan) (Fig.1) são semelhantes às cefalosporinas, contudo, apresentam mais estabilidade à hidrólise mediada pelas β-lactamases, devido à substituição de um radical de oxigénio no local do enxofre no anel dihidrotiacina (Murray et al., 2007).
Tabela 2 - Classificação das cefalosporinas e exemplos de antibióticos (Murray et al., 2007).
Classificação de cefalosporinas Exemplos de antibióticos
Cefalosporinas de 1ª geração Cefazolina; Cefalotina; Cefapirina; Cefradina Cefalosporinas de 2ª geração Cefaclor; Cefuroxima
Cefalosporinas de 3ª geração Cefixima; Cefotaxima; Cefriaxona; Ceftazidima; Cefoperazona; Cefpodoxima Cefalosporinas de 4ª geração Cefepima; Cefpiroma
Os carbapenemes (imipeneme, meropeneme, ertapeneme e doripenem) constituem um grupo de antibióticos β-lactâmicos com amplo espetro de ação, frequentemente utilizados para o tratamento de infeções graves e infeções causadas por isolados produtores de β-lactamases de espetro alargado (ESBL - Extended-Spectrum
ß-Lactamase) (Patel e Bonomo, 2013). Como característica comum deste grupo de
antibióticos, o anel tiazolidina contém um átomo de carbono no local do átomo de enxofre (Liras e Martin, 2006). Por fim, os monobactamos (aztreonam) (Fig.1) são compostos que apresentam uma estrutura monocíclica e possuem um espetro de ação reduzido, sendo ativos apenas em bactérias aeróbias de Gram negativo e inativos em bactérias de Gram positivo e anaeróbias (Liras e Martin 2006; Murray et al., 2007).
Deste modo, as modificações nos radicais R e R' da cadeia lateral determinam o espetro de ação dos antibióticos β-lactâmicos, as propriedades farmacocinéticas e a resistência às β- lactamases.
7
2.1. Mecanismo de ação dos antibióticos β-lactâmicos
Os antibióticos ß-lactâmicos atuam na fase parietal da biossíntese do peptidoglicano. O peptidoglicano é o principal constituinte da parede celular, sendo constituído por cadeias de 10 a 65 resíduos de dissacarídeos formados por moléculas de N-acetilglucosamina (NAG) intercaladas com moléculas de ácido N-acetilmurâmico (NAM). As proteínas de ligação à penicilina (PBPs, Penicillin-binding proteins) são enzimas (p.ex., transpeptidases, transglucosidases, carboxipeptidases), da família das serina proteases, que catalisam a síntese dos polímeros de peptidoglicano na parede celular, conferindo rigidez às bactérias. Assim, os antibióticos β-lactâmicos atuam através da ligação às PBPs específicas da parede celular bacteriana, visto que possuem grande afinidade para estas proteínas. Este mecanismo resulta na acilação das PBPs, tornando-as incapazes de realizar a reação de transpeptidação, a principal etapa na síntese do peptidoglicano. Este mecanismo ativa as autolisinas que degradam a parede celular e causam a destruição das células (Murray et al., 2007). Como resultado, a parede celular das bactérias entra em citólise, ou morte celular, devido à pressão osmótica (Van Hoek et al., 2011).
2.2. Mecanismos de resistência aos antibióticos β-lactâmicos
Os mecanismos de resistência aos antibióticos ß-lactâmicos incluem (1) produção de ß-lactamases, que constitui o principal mecanismo de resistência em bactérias de Gram negativo, (2) a alteração no local alvo de ligação, (3) diminuição da permeabilidade da membrana celular, e ainda (4) sistemas de bombas de efluxo (Drawz e Bonomo, 2010).
A resistência mediada pela alteração no local alvo de ligação resulta de alterações no sítio ativo das PBPs, o que pode determinar a diminuição da afinidade e aumento de resistência aos antibióticos β-lactâmicos. Este mecanismo é raro em bactérias de Gram negativo, sendo principalmente encontrado em bactérias de Gram positivo (Livermore e Woodford, 2006).
As porinas, proteínas de membrana externa (OMP, Outer membrane protein), são canais membranares das bactérias de Gram negativo que atuam como canal de
8 entrada dos antibióticos β-lactâmicos. Assim, alterações na expressão ou perda de porinas podem diminuir a permeabilidade dos antibióticos β-lactâmicos na célula. No entanto, este mecanismo não é suficiente para conferir resistência, sendo geralmente encontrado em associação com a produção de β-lactamases (Drawz e Bonomo, 2010).
A resistência mediada por bombas de efluxo é outro mecanismo, que consiste num sistema de transporte ativo intrínseco ou adquirido. Ao serem expressas, as bombas de efluxo são capazes de bombear diversos antibióticos do citoplasma para fora da célula. Desta forma, em muitos casos, estão associados a fenótipo de multirresistência em isolados de Gram negativo (Piddock, 2006). A resistência mediada pelas bombas de efluxo pode derivar de alterações nos genes repressores e/ou hiperexpressão dos seus reguladores transcricionais. Este mecanismo confere, geralmente, baixos níveis de resistência aos antibióticos, pois mesmo possuindo múltiplos antibióticos como substratos, podem não conferir elevado nível de resistência. Contudo, a diminuição da concentração intracelular do antibiótico permite à bactéria sobreviver até que sejam selecionadas bactérias com mutações que podem determinar níveis de resistência clinicamente importantes (Webber e Piddock, 2003).
2.2.1. β-lactamases
A hidrólise dos antibióticos ß-lactâmicos pelas ß-lactamases é o mecanismo de resistência mais comum a esta classe de antibióticos em bactérias de Gram negativo (Bush e Jacoby, 2010). Estas enzimas podem ser classificadas em penicilinases, específicas para as penicilinas, cefalosporinases, que hidrolisam preferencialmente as cefalosporinas, carbapenemases que inativam os carbapenemes, ou ainda ß-lactamases de espetro alargado (ESBLs) capazes de hidrolisar as penicilinas, as cefalosporinas de primeira, segunda e terceira geração e o aztreonam (Van Hoek et al., 2011; Nordmann et al., 2012).
As ß-lactamases desempenham assim um papel crucial na seleção da terapia mais adequada, visto que as penicilinas, as cefalosporinas e os carbapenemes são frequentemente incluídos no tratamento de várias doenças infeciosas (Bush e Jacoby 2010). Estima-se que existem, pelo menos, 1.300 ß-lactamases descritas em isolados clínicos (Bush, 2013).
9 As ESBLs são enzimas de grande relevância, uma vez que se tem verificado uma ampla disseminação a nível mundial nas últimas décadas. Atualmente, existe uma grande variedade de ESBLs, derivando, a maioria, das β-lactamases TEM-1, TEM-2 e SHV-1, após introdução de substituições aminoacídicas no sítio ativo. Os genes bla ESBL são geralmente adquiridos por mecanismos de transferência genética, associados a elementos genéticos móveis, como os plasmídeos (Paterson e Bonomo, 2005). Já se encontram descritas centenas de ESBLs diferentes, sendo a maioria das famílias CTX-M, TEM e SHV.
2.2.1.1 Classificação de β-lactamases
As ß-lactamases são classificadas de acordo com a sua homologia na sequência de aminoácidos (classificação de Ambler) e de acordo com a preferência por um determinado substrato e perfil de inibição (classificação de Bush e Jacoby) (Bush e Jacoby, 2010). Segundo a classificação de Ambler as β-lactamases dividem-se em quatro classes: A, B, C e D (Figura 3). Aquelas que pertencem às classes A, C e D são denominadas β-lactamases-serina, pois possuem um resíduo de serina no centro ativo para facilitar a catálise e aquelas que pertencem à classe B contêm um ião de zinco, sendo designadas de metalo-β-lactamases (Jacoby e Munoz-Price, 2005). A classificação de Bush divide as ß-lactamases em três grupos (1-3). Os grupos 1 e 2 são β-lactamases-serina e o grupo 3 são metalo-β-lactamases. As β-lactamases do grupo 1, associadas à classe C, hidrolisam as cefalosporinas e são resistentes à ação dos inibidores de β-lactamases. O grupo 2, que inclui as classes A e D, representa o grupo com vários subgrupos e com uma ampla variedade de enzimas com diferentes perfis de substratos (Figura 3). O grupo 3 engloba as metalo β-lactamases da classe B de Ambler. Estas enzimas hidrolisam todos os antibióticos β-lactâmicos, à exceção dos monobactamos (Bush e Jacoby, 2010).
10
Figura 3 - Classificação das ß-lactamases relacionando o perfil do substrato e dos inibidores de ß- lactamases com a sua estrutura molecular. Cb, carbapenemes; Cf, cefalosporinas de primeira geração; CA, ácido clavulânico; EDTA, ácido etilenodiaminotetracético; Esc, Cefalosporina de espetro alargado; M, monobactamos; Pn, penicilinas (Adaptado de Bush, 2013).
Atualmente, tem-se verificado uma emergência de ESBLs, sobretudo da família CTX-M (classe A de Ambler), de ß-lactamases AmpC (classe C de Ambler) mediada por plasmídeos e de carbapenemases (das classes A, B e D de Ambler), nomeadamente da família KPC, IMP e VIM, produzidas por Enterobacteriaceae e por outras bactérias de Gram negativo (Bush, 2013; Van Hoek et al., 2011; Jacoby, 2009).
2.2.1.2. Família TEM
As enzimas TEM pertencem à classe A de Ambler e aos grupos funcionais 2b, 2be e 2br segundo a classificação de Bush e Jacoby (Fig.3) (Bush e Jacoby, 2010). No início da década de 60 foi descrita na Grécia a primeira β-lactamase TEM-1 mediada por plasmídeos, num isolado de Escherichia coli. A sua designação advém de Temoniera, nome do paciente a quem foi detetada a enzima pela primeira vez. Esta enzima disseminou-se por todo o mundo, anos depois da sua descrição, sendo a mais encontrada em bactérias de Gram negativo. A enzima TEM-1 é uma penicilinase, que tem como substrato as penicilinas e cefalosporinas de primeira geração, como a cefalotina e cefaloridina (Bradford, 2001).
11 A enzima TEM-2 foi a primeira enzima derivada de TEM-1 a ser descrita. Ambas apresentam o mesmo perfil hidrolítico, contudo TEM-2 tem um promotor mais ativo e um ponto isoelétrico (pI) de 5,6 diferente do TEM-1, com pI de 5,4. A enzima TEM-3, reportada em 1989, foi a primeira enzima derivada de TEM-1 a exibir o fenótipo de ESBL. Desde então, foram descritas outras enzimas derivadas de TEM, entre as quais β-lactamases resistentes aos inibidores (IRT, Inhibitor Resistant TEM) e ESBLs, representando estas últimas a maioria (Bradford, 2001; Paterson e Bonomo, 2005). Atualmente existem mais de 200 β-lactamases TEM, principalmente ESBLs (Jacoby, 2012).
2.2.1.3. Família SHV
As β-lactamases SHV estão incluídas na classe A de Ambler e nos subgrupos funcionais 2b, 2be e 2br de Bush e Jacoby (Bush e Jacoby, 2010). As enzimas desta família derivam da enzima SHV-1. Esta β-lactamase possui 68% de homologia com a sequência aminoacídica de TEM-1 e apresenta uma estrutura semelhante (Jacoby e Munoz-Price, 2005). A enzima SHV-1 é, frequentemente, codificada por genes cromossómicos em Klebsiella pneumoniae (Bradford, 2001).
Ao contrário das β-lactamases TEM, as alterações no gene blaSHV, que dão origem às variantes de SHV ocorrem em menor número de posições aminoacídicas diferentes. A maioria das variantes de SHV, conferindo um fenótipo ESBL, são caracterizadas pela substituição de uma serina por uma glicina na posição 238. Outras variantes de SHV, relacionadas com SHV-5, possuem também a substituição de uma lisina por glutamato na posição 240. A maioria das enzimas derivadas de SHV-1 possui fenótipo de ESBLs, contudo sete variantes estão descritas como β-lactamases resistentes aos inibidores. Globalmente, foram já descritas 182 variantes de β-lactamases SHV (Jacoby, 2012).
2.2.1.4. Família CTX-M
As enzimas da família CTX-M têm origem em genes cromossómicos de espécies de Kluyvera spp. e têm como substratos as cefalosporinas de terceira geração, preferencialmente a cefotaxima. Ao contrário das enzimas da família TEM e SHV,
12 hidrolisam mais a cefotaxima do que a ceftazidima (Bradford, 2001; Paterson e Bonomo, 2005; Jacoby e Munoz-Price, 2005). As β-lactamases CTX-M pertencem à classe A de Ambler e subgrupo funcional 2be de Bush e Jacoby (Bush e Jacoby, 2010).
A enzima CTX-M-1 foi inicialmente descrita na década de 80 na Alemanha, tendo sido identificada num isolado clínico de E. coli (Bauernfeind et al., 1990). Posteriormente, verificou-se uma expansão das enzimas desta família em várias partes do mundo e em vários isolados clínicos, sobretudo em membros da família das Enterobacteriaceae (Bonnet, 2004). Estas β-lactamases são classificadas em diferentes grupos, tendo em conta a homologia nas sequências de aminoácidos: grupo CTX-M-1, CTX-M-2, CTX-M-8, CTX-M-9, CTX-M-25 e o grupo KLUC (Bonnet, 2004; D'Andrea et al., 2013).
As ESBLs da família CTX-M representam β-lactamases importantes, as quais têm sido encontradas em muitos isolados, quer em ambiente hospitalar, quer na comunidade, e, atualmente, têm sido fonte de preocupação dada a sua rápida expansão pelo mundo, inclusive em Portugal (Bush, 2013; Cantón e Coque, 2006; Manageiro et al., 2012a; Mendonça et al., 2007). Considera-se que a emergência destas enzimas em vários locais está associada à aquisição e disseminação de plasmídeos com genes
blaCTX-M (Bonnet, 2004). Atualmente, tem-se verificado um predomínio de ESBLs CTX-M-14 e, sobretudo, CTX-M-15, a nível mundial (Bush e Fisher, 2011).
2.2.1.5. Família OXA
As β-lactamases OXA pertencem à classe D de Ambler e aos grupos funcionais 2de e 2df de Bush e Jacoby. Esta família consiste num grupo heterogéneo, de várias enzimas (Bush, 2013). No subgrupo 2de estão incluídas as oxacilinases, caracterizadas pela elevada atividade hidrolítica à oxacilina e cloxacilina, com exceção dos carbapenemes. Estão incluídas neste grupo enzimas derivadas de OXA-10 (com fenótipo ESBL), entre as quais OXA-10, -11, -14, -15, -16, e -17 (Bradford, 2001; Bush e Jacoby, 2010). A maioria das β-lactamases OXA, embora hidrolisando fracamente a cefotaxima, ceftriaxona e aztreonam, conferem suscetibilidade diminuída a esses antibióticos e fenótipo de ESBL (Paterson e Bonomo, 2005). As ESBL tipo OXA são encontradas, particularmente, em Pseudomonas aeruginosa e ainda em
13 As oxacilinases com atividade hidrolítica aos carbapenemes estão incluídas no subgrupo funcional 2df, capazes de hidrolisar as amino- e as carboxipenicilinas. Estas têm sido, frequentemente, encontradas em Acinetobacter spp., com um aumento em isolados de Enterobacteriaceae, principalmente de OXA-48 (Patel e Bonomo, 2013; Bush e Jacoby, 2010). Inativam eficientemente as penicilinas, cefalosporinas de primeira geração, antibióticos lactâmicos combinados com inibidores de β-lactamases, com exceção das cefalosporinas de espetro alargado. Contudo, a variante OXA-163, que apresenta uma baixa atividade de carbapenemase, hidrolisa estas cefalosporinas. A maioria das enzimas desta família apresenta baixa suscetibilidade aos inibidores de β-lactamases (ácido clavulânico e EDTA), mas são inibidas in vitro pelo cloreto de sódio (NaCl) (Patel e Bonomo, 2013; Nordmann et al., 2012). Já se encontram descritas mais de 250 variantes de oxacilinases, com uma minoria exibindo baixo nível de hidrólise aos carbapenemes (Patel e Bonomo, 2013).
2.2.1.6. β-lactamases AmpC
As β-lactamases AmpC hidrolisam as penicilinas, as oximino-cefalosporinas, aztreonam (variável) e a cefoxitina e não são inibidas pelo ácido clavulânico (Jacoby e Munoz-Price, 2005; Jacoby, 2009). Estas enzimas pertencem à classe C de Ambler e aos subgrupos funcionais 1 e 1e de Bush e Jacoby. No entanto, as enzimas do subgrupo 1e, designadas por AmpC de espetro alargado (ESAC, Extended-spectrum AmpC), apresentam maior atividade hidrolítica às cefalosporinas de terceira geração do que as do subgrupo 1 (Bush, 2013). As β-lactamases AmpC são importantes cefalosporinases codificadas por genes cromossómicos em algumas espécies de Enterobacteriaceae (Enterobacter spp., Citrobacter freundii, M. morganii e E. coli) e P. aeruginosa. Têm sido também muito descritas β-lactamases AmpC mediadas por plasmídeos (PMAβ,
Plasmid-Mediated AmpC β-Lactamases) em Enterobacteriaceae (E. coli, Klebsiella
spp., Salmonella spp. e Proteus mirabilis) (Livermore e Woodford, 2006; Jacoby, 2009). As PMAβ derivam de genes ampC cromossómicos de diferentes espécies (Tabela. 3) (Jacoby e Munoz-Price, 2005).
14
Tabela 3 - Origem de genes que codificam β-lactamases AmpC (Adaptado de Livermore e Woodford, 2006)
Classe Origem Exemplos
CIT Citrobacter freundii CMY-2 a CMY-7; LAT-1,-3,-4
ENT Enterobacter spp. ACT-1; MIR-1
FOX Aeromonas spp. FOX-1 a FOX-5
MOX Aeromonas spp. MOX-1, MOX-2; CMY-1 e CMY-8
DHA Morganella morgani DHA-1, DHA-2
ACC Hafnia alvei ACC-1
A expressão de AmpC pode ser constitutiva, como por exemplo, em E. coli, ou indutiva, como em E. cloacae, C. freundii, M. morganii e P. aeruginosa, sendo os níveis de produção da enzima dependentes do grau de expressão do gene ampC (Jacoby, 2009). A expressão de AmpC cromossómica pode estar desreprimida em isolados com gene ampC cromossómico indutível, devido a mutações nos genes reguladores (p.e.
ampD e ampR), resultando na hiperprodução, estável, de AmpC (Jacoby, 2009; Jacoby
e Munoz-Price, 2005). A expressão das PMAβ pode também ser indutiva, conferindo resistência semelhante à das AmpC cromossómicas, no entanto, o nível de resistência depende do nível de enzima expressa, bem como da presença de outros mecanismos de resistência (Jacoby, 2009).
2.2.1.7. Carbapenemases
As carbapenemases constituem um grupo diverso de enzimas que têm emergido e que se tem tornado num facto preocupante, uma vez que hidrolisam os carbapenemes, último recurso, em muitos casos, no tratamento de infeções. Além de hidrolisarem os carbapenemes, hidrolisam as cefamicinas e as oximino-cefalosporinas (Alekshun e Levy, 2007; Jacoby e Munoz-Price, 2005).
As carbapenemases estão distribuídas em três classes de β-lactamases de Ambler: classe A, B e D (Patel e Bonomo, 2013). Atualmente, as β-lactamases KPC (classe A), as metalo-β-lactamases, como VIM, IMP, e NDM (classe B), e a carbapenemase OXA-48 (classe D), mediada por plasmídeos, representam as carbapenemases com maior importância clínica em Enterobacteriaceae (Tzouvelekis et al., 2012; Nordmann et al., 2012).
As enzimas NmcA/IMI, SME, e KPC são as principais carbapenemases da classe A de Ambler. Hidrolisam as penicilinas, cefalosporinas, carbapenemes e o
15 aztreonam e são inibidas in vitro pelo ácido clavulânico e tazobactam. A enzima GES também pertence à classe A, contudo, foi inicialmente designada como uma β-lactamase ESBL clássica, uma vez que a enzima GES-1 não hidrolisa os carbapenemes. Posteriormente, foram identificadas outras variantes (p.e., 2, 4, 5, GES-6, GES-11 e GES-14), com atividade hidrolítica fraca, mas significativa sobre os carbapenemes (Nordmann et al., 2012). Todas as carbapenemases apresentam capacidade de hidrolisar as cefalosporinas de terceira geração e os carbapenemes devido a uma substituição aminoacídica no sítio ativo (nas posições 104 e 170). Atualmente, as β-lactamases KPC representam as carbapenemases de classe A, com maior importância clínica. Em 1996, foi identificada a primeira KPC (KPC-2) num isolado de K.
pneumoniae, na Carolina do Norte, nos Estados Unidos da América (Patel e Bonomo,
2013; Nordmann et al., 2012). Desde então, já foram identificadas doze variantes de KPC (KPC-2 a KPC-15), verificando-se uma maior predominância das variantes KPC-2 e KPC-3 (Patel e Bonomo, 2013).
As enzimas do tipo IMP, VIM e NDM representam as principais carbapenemases do grupo das metalo-β-lactamases; em Enterobacteriaceae, são as que possuem maior distribuição geográfica. As metalo-β-lactamases conferem resistência a todas as penicilinas, cefalosporinas e carbapenemes, à exceção do aztreonam, não sendo hidrolisadas pelos inibidores de β-lactamases (Patel e Bonomo, 2013; Nordmann et al., 2012).
Como referido anteriormente (capítulo 2.2.1.5), a enzima OXA-48, pertence à classe D de Ambler e tem atividade hidrolítica sobre os carbapenemes. Desde a sua primeira descrição na Turquia, a enzima OXA-48, tem sido identificada em países Africanos e do sul da Europa, o que se torna preocupante devido à sua rápida disseminação (Nordmann et al., 2012).
3. Tigeciclina
A tigeciclina é um antibiótico recente e o primeiro composto sintético da nova classe de antibióticos glicilciclinas disponibilizado para uso clínico (Stein e Craig, 2006). Foi desenvolvida através da pesquisa de compostos mais ativos, a partir da
16 estrutura química das tetraciclinas, de forma a contornar os principais mecanismos de resistência a esta classe de antibióticos (Doan et al., 2006; Seputiene et al., 2010).
Em 2005, a tigeciclina (Tygacil®) foi aprovada, pela FDA (Food and Drug
Administration), para utilização no tratamento de infeções complicadas da pele, tecidos
moles e intra-abdominais. Em 2009, foi também aprovada pela FDA para o tratamento de pneumonia bacteriana adquirida na comunidade (Stein e Babinchak, 2013; Sun et al., 2012; Sader et al., 2013).
A utilização de tigeciclina é aconselhada apenas em adultos, estando a duração do tratamento dependente da gravidade e local da infeção, bem como da forma como o doente reage ao antibiótico, podendo variar entre 5 a 14 dias. O tratamento consiste na administração da tigecilina por via intravenosa, devendo inicialmente ser administrada uma dose de 100 mg e, nos dias seguintes, doses de 50 mg de 12 em 12 horas (Babinchak et al., 2005).
3.1. Estrutura química e mecanismo de ação da tigeciclina
A tigeciclina (Fig.4A) deriva estruturalmente da minociclina (Fig.4B), antibiótico pertencente à classe das tetraciclinas, por uma substituição na posição D-9, conferindo-lhe um amplo espetro de atividade (Peterson, 2008).A cadeia lateral N-alquilo glicilamido, na posição 9 do átomo de carbono da tigeciclina confere-lhe propriedades ao nível da atividade biológica, tal como o aumento da solubilidade lipídica, o bloqueio do efluxo da tigeciclina para o exterior da célula e ainda o aumento da afinidade ao local de ligação no ribossoma (Seputiene et al 2010).
A tigeciclina é um antibiótico bacteriostático, pois inibe a síntese de proteínas bacterianas através da sua união à subunidade 30S do ribossoma bacteriano. Este mecanismo impede a entrada da RNA aminoacil-transferase no local de ligação A do ribossoma (Petersen et al., 1999). Apesar da tigeciclina apresentar um mecanismo de ação semelhante às tetraciclinas, possui uma afinidade cinco vezes superior para o ribossoma (Bauer et al., 2004).
17
Figura 4 - Estrutura química da tigeciclina (A) e da minociclina (B) (Dean et al. 2003)
3.2. Espetro de ação da tigeciclina
A tigeciclina exibe um mecanismo de ação semelhante às tetraciclinas, apresentando, no entanto, atividade em isolados que possuem genes responsáveis por mecanismos de resistência mediados por bombas de efluxo e proteção do ribossoma, que os tornam resistentes às tetraciclinas. A título de exemplo, um estudo de Petersen et al. (1999) demonstrou que a tigeciclina tem a capacidade de contornar a resistência mediada pelos determinantes tet(M) e tet(O), que promovem proteção ribossomal, e pelos determinantes tet(A-E) e tet(K), que promovem a ação de bombas de efluxo sobre as tetraciclinas.
A tigeciclina apresenta um amplo espetro de ação in vitro, sendo ativa sobre uma grande variedade de microrganismos de Gram positivo e de Gram negativo, quer aeróbios, quer anaeróbios, quer da comunidade quer de origem nosocomial (Betriu et al., 2006; Bouchillon et al., 2005; Castanheira et al., 2008; Goldstein et al., 2006).
Assim, a tigeciclina tem ação sobre isolados de Gram positivo, como
Staphylococcus aureus, nomeadamente S. aureus resistente à meticilina (MRSA, Methicillin-resistant Staphylococcus aureus), Streptococcus pneumoniae (incluindo
resistentes à penicilina), Streptococcus agalactiae, Enterococcus faecium e
Enterococcus faecalis, nomeadamente resistentes à vancomicina (Betriu et al., 2006;
Cercenado et al., 2003; Dowzicky e Chmelarová, 2011; Norskov-Lauritsen et al., 2009). Isolados de Gram negativo, tais como E. coli, K. pneumoniae, Enterobacter spp.
Citrobacter spp. e Serratia marcescens têm apresentado suscetibilidade à tigeciclina
(Betriu et al., 2006; Bouchillon et al., 2005; Castanheira et al., 2008; Roy et al., 2013; Souli et al., 2006).
18 Adicionalmente, este antibiótico possui ação antimicrobiana em bactérias com resistência a outros antibióticos, sobretudo aos β-lactâmicos, nomeadamente em Enterobacteriaceae produtoras de ESBL (Bouchillon et al., 2005; Roy et al., 2013), bem como de β-lactamases AmpC (Bouchillon et al., 2005), carbapenemases e metalo-β-lactamases (Castanheira et al., 2008; Souli et al., 2006), ou multirresistentes aos antibióticos (Gupta et al., 2012).
3.3. Resistência à tigeciclina
Apesar do espetro de ação da tigeciclina e da sua capacidade de contornar os mecanismos de resistência a outras classes de antibióticos, já foram descritos casos de microrganismos com suscetibilidade diminuída a este antibiótico. As P. aeruginosa são intrinsecamente resistentes à tigeciclina (Dean et al., 2003; Sun et al., 2012). De igual modo, isolados de Burkholderia cepacia e isolados da família Proteeae, tais como,
Proteus spp., Providencia spp. e Morganella morganii apresentam geralmente
suscetibilidade diminuída a este antibiótico (Ruzin et al., 2005; Sun et al., 2012). Além disso, tem-se verificado a aquisição de resistência à tigeciclina em Acinetobacter
baumannii (Peleg et al., 2007 Ruzin et al., 2007) e em outros isolados da família das Enterobacteriaceae, como E. coli, K. pneumoniae e Enterobacter spp. (Keeney et al.,
2008; Ruzin et al. 2005; Veleba et al., 2013).
Os mecanismos de resistência à tigeciclina não estão bem esclarecidos, mas, de forma geral, a resistência tem sido atribuída à ação de bombas de efluxo da família RND (Resistance-Nodulation-Division), regulada por genes localizados ao nível do cromossoma; estas bombas são, frequentemente, associadas à multirresistência aos antibióticos (Pérez et al., 2012; Sun et al., 2012). O sistema de efluxo mais descrito em
P. aeruginosa é o MexXY-OprM (Dean et al., 2003). Em A. baumannii foi reportado
uma diminuição da suscetibilidade à tigeciclina associada ao sistema de efluxo AdeABC (Peleg et al., 2007). O sistema de efluxo AcrAB está associado à suscetibilidade diminuída à tigeciclina em P. mirabilis, E. coli, K. pneumoniae, M.
morganii e Enterobacter spp. (Keeney et al., 2007; Keeney et al., 2008; Ruzin et al.
2005; Veleba et al., 2013; Visalli et al., 2003). A hiperprodução da bomba de efluxo SdeXY, da família RND, está associada à suscetibilidade diminuída à tigeciclina em S.
19 Além do mecanismo de resistência à tigeciclina associado às bombas de efluxo, foi, recentemente, identificada uma proteína Tet(X) monoxigenase, dependente da flavina, que confere resistência às tetraciclinas e à tigeciclina em bactérias anaeróbias, como Bacteroides, através da hidroxilação da região-seletiva de tetraciclinas para 11a-hidroxi-tetraciclinas, reduzindo a atividade antimicrobiana. Adicionalmente, a tigeciclina representa um substrato da hidroxilação dependente, quer de NADPH (nicotinamida adenina difosfato), quer de oxigénio por parte da proetína Tet(X) (Volkers et al., 2011). É de realçar que, até a atualidade, esta proteína não foi observada noutras bactérias aeróbias e/ou anaeróbias isoladas em meio clínico; no entanto, se os genes que a codificam forem integrados em elementos genéticos móveis, pode disseminar-se para outras bactérias (Linkevicius et al., 2013).
3.3.1. Bomba de efluxo AcrAB-TolC da família RND
A bomba de efluxo AcrAB-TolC da família RND constitui um mecanismo de resistência com relevância clínica, uma vez que confere resistência a múltiplos antibióticos utilizados no tratamento de infeções humanas, incluindo os β-lactâmicos, fluoroquinolonas, macrólidos, tetraciclinas, clorafenicol e trimetoprim, encontrando-se bastante distribuída em bactérias de Gram negativo (Alekshun e Levy, 1999; Piddock, 2006). Em Enterobacteriaceae, conferem ainda resistência a uma variedade de compostos tóxicos, como detergentes, corantes e desinfetantes (Pérez et al., 2012).
A bomba de efluxo AcrAB é formada por três subunidades: AcrB, uma proteína na membrana interna, que atua expelindo substâncias para o exterior; TolC, uma porina da membrana externa, e AcrA, uma proteína que liga a bomba à porina (Fig.5). As três subunidades da bomba de efluxo permitem a expulsão de compostos diretamente a partir do citoplasma para o ambiente externo, contribuindo para o aumento da resistência aos antibióticos. Este sistema de efluxo utiliza um gradiente de protões na membrana como fonte de energia (Pérez et al., 2012).
20
Figura 5 - Estrutura do sistema de efluxo AcrAB-TolC em E.coli (Adaptado de Alvarez-Ortega et al., 2013).
A expressão da bomba de efluxo AcrAB é regulada por proteínas da família AraC, os quais conferem resistência aos antibióticos e desinfetantes, aumentado a expressão daquela bomba. Os reguladores transcricionais da família AraC são: MarA, SoxS, RamA e Rob (Alekshun e Levy, 1999; Rosenblum et al., 2011). Atualmente, tem sido descrito que este sistema AcrAB-TolC está envolvido na suscetibilidade diminuída à tigeciclina, nalgumas espécies de Gram negativo.
A expressão da bomba de efluxo pode ser desreprimida pela sobre-expressão dos reguladores transcricionais, como MarA em E. coli e RamA em K. pneumoniae. Mutações no gene acrR, repressor da bomba de efluxo AcrAB, ou ainda mutações no genes marR e ramR (repressores de marA e ramA, repetivamente) podem também desencadear um aumento da expressão da bomba de efluxo. Por sua vez, o aumento da atividade da bomba de efluxo AcrAB, pode conferir um fenótipo de multirresistência (Keeney et al., 2008; Ruzin et al., 2005; Hentschke et al., 2010).
A suscetibilidade diminuída à tigeciclina, mediada pela bomba de efluxo AcrAB, em E. coli, está associada a mutações no repressor marR, desencadeando a sobre-expressão da proteína MarA e da bomba de efluxo (Keeney et al., 2008). O operão marAB em isolados de E. coli é constituído pelos genes marA, marB e o gene
marR, repressor de MarA, que regulam a expressão da bomba de efluxo AcrAB
(Alekshun e Levy, 1999). A expressão do operão marAB e marC é controlada por promotores independentes, PmarII e PmarI, respetivamente (Fig.6). Os genes marR e
21
marA codificam o repressor e o regulador transcricional MarR (144 resíduos de
aminoácidos) e MarA (127 resíduos de aminoácidos), respetivamente. O gene marB (codifica 72 resíduos de aminoácidos) tem uma função desconhecida, bem como o
marC (codifica 201 resíduos de aminoácidos). Assim, MarR reprime a expressão de
MarAB através da ligação em dois sítios no operador MarO, podendo a sua inativação resultar na expressão constitutiva de MarAB, dependendo dos níveis de expressão dos reguladores transcricionais, como MarA (Alekshun e Levy, 1999).
Figura 6 - Organização genética do locus marAB em E. coli ; PmarI e PmarII, promotores (Alekshun e Levy, 1999).
Em K. pneumoniae, Enterobacter cloacae e Enterobacter aerogenes a diminuição da suscetibilidade à tigeciclina tem sido associada à sobre-expressão da proteína RamA, homóloga da proteína MarA (Hentschke et al., 2010; Keeney et al., 2007; Ruzin et al., 2005; Veleba et al., 2013). A sobre-expressão de MarA e RamA, designadas de reguladores positivos, desencadeiam o aumento da atividade da bomba de efluxo AcrAB, conferindo um fenótipo de multirresitência à outras classes de antibióticos (Keeney et al., 2008 Ruzin et al., 2005;Rosenblum et al., 2011; Hentschke et al., 2010).
Para além disso, mutações no gene ramR (repressor de ramA) têm sido descritas como uma via de aquisição de resistência à tigeciclina em K. pneumoniae, E. cloacae e
E. aerogenes, que leva a sobre-expressão de RamA e da bomba de efluxo AcrAB
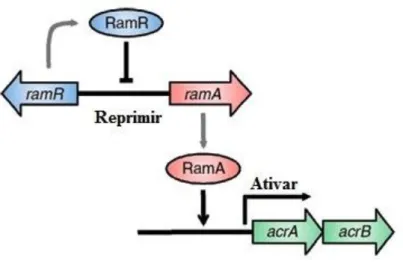
(Hentschke et al., 2010; Veleba et al., 2013). O gene ramR está localizado a montante do gene ramA e codifica a proteína da família de repressores transcricionais TetR (Fig.7). O local de ligação do RamR localiza-se a montante do gene ramA e esta região abrange uma parte do promotor de ramA. A proteína RamR liga-se então à região intergénica, entre o ramR e o ramA, e RamA liga-se na região a montante do acrAB.
22 Deste modo RamR reprime o gene ramA, o qual codifica a proteína RamA que ativa os genes da bomba de efluxo AcrAB (Yamasaki et al., 2013).
Figura 7 - Via de regulação da bomba de efluxo AcrAB: a proteina RamR reprime a expressão do gene ramA que codifica o activador trancripcional, RamA, do acrAB. (Adaptado de Yamasaki et al., 2013).
23
4. Objetivos
Tendo em conta que a tigeciclina é um antibiótico, recentemente, desenvolvido com o intuito de tentar ultrapassar o problema da resistência aos antibióticos, este trabalho teve como objetivo avaliar a suscetibilidade e caracterizar os mecanismos de resistência à tigeciclina em 211 isolados de Gram negativo provenientes de doentes, em Portugal. Pretendeu-se igualmente esclarecer a ação da tigeciclina em bactérias com resistência, sobretudo aos antibióticos β-lactâmicos, ou multirresistentes de forma a compreender a possibilidade da tigeciclina constituir uma alternativa nestes casos. Assim, o presente trabalho teve como objetivos específicos:
i. Avaliar a suscetibilidade da tigeciclina em isolados de Gram negativo
ii. Determinar a suscetibilidade de isolados de Gram negativo a outras classes de antibióticos, nomeadamente β-lactâmicos (aminopenicilinas, carboxipenicilinas, ureidopenicilinas, cefalosporinas da 1ª, 2ª, 3ª e 4ª geração, monobactâmicos, carbapenemes), fluoroquinolonas, aminoglicosídeos, trimetoprim-sulfametoxazol, colistina, fosfomicina e nitrofuranos.
iii. Pesquisar e identificar os genes bla associados à resistência aos antibióticos β-lactâmicos, orientada pela leitura interpretativa dos testes de suscetibilidade.
iv. Caracterizar molecularmente a resistência à tigeciclina, através da pesquisa e identificação de mutações nos genes ramR e marR em isolados estudados de
E. coli e K. pneumoniae, predominantemente, com suscetibilidade diminuída
24
II. Material e Métodos
1. Isolados bacterianos
Para a realização do presente trabalho foram estudados 211 isolados de Gram negativo, reunidos em 2011 e 2013 no Laboratório Nacional de Referência da Resistência aos Antimicrobianos (LNR-RA) do Instituto Nacional de Saúde Dr. Ricardo Jorge, tendo como proveniência diversos hospitais nacionais (Fig. 8). Os isolados estudados foram os seguintes: Escherichia coli (n=101), Klebsiella pneumoniae (n=70),
Citrobacter freundii (n=4), Enterobacter cloacae (n=12), Enterobacter aerogenes
(n=4), Morganella morganii (n=10), Proteus mirabilis (n=7) e Serratia marcescens (n=3). A identificação preliminar dos isolados foi realizada nas instituições hospitalares de proveniência. Sempre que necessário, foi utilizado o método API20E®, com leitura em 24 horas, no LNR-RA para confirmar a identificação dos isolados estudados.
Figura 8 - Distribuição, pelos hospitais de origem, de 211 isolados que constituem a amostra do estudo.
0 10 20 30 40 50 60 A B C D E F G H I J L M N Nú m er o d e is o lad o s Hospitais
25
2. Caracterização fenotípica: suscetibilidade aos antibióticos
2.1. Método de microdiluição
O método de microdiluição em caldo foi realizado para determinar a suscetibilidade de 211 isolados em estudo aos seguintes antibióticos: tigeciclina (Pfizer), colistina (Sigma), ciprofloxacina (Bayer), imipinem (Merck), cefoxitina (Fluka), ceftazidima (GSK), cefotaxima (Sanofi) e cefotaxima (Sanofi) em combinação com ácido clavulânico (Cipan). Assim, a determinação da concentração inibitória mínima (CIM) foi realizada através dos passos descritos a seguir.
2.1.1. Preparação da solução-mãe dos antibióticos
A solução-mãe de cada um dos antibióticos, para a determinação da CIM, foi preparada de acordo com a concentração final descrita na Tabela 4. Para cada antibiótico, a quantidade necessária para a solução-mãe foi calculada de acordo com a potência e pureza do mesmo e dissolvidos em solventes adequados (Tabela 4), utilizando a fórmula descrita a seguir:
Peso (mg) = Volume (ml) x Concentração (μg/ml) Potência (μg/mg)
Os antibióticos em pó foram pesados numa balança analítica (AE200, Mettler). Após a preparação da solução-mãe na concentração estabelecida (Tabela 4), esta foi esterilizada por filtração (Acrodisc® syringe filters, Pall corporation) e acondicionada em eppendorf a -80ºC até o uso.
Tabela 4 - Antibióticos, concentrações das soluções-mãe, solventes e potência dos antibióticos.
Antibiótico Solução-mãe (mg/L) Solvente Potência (%)
Cefotaxima 2048 Água 90,1
Ceftazidima 2048 Carbonato de Sódio 84,6
Cefoxitina 2048 Água 99,4
Imipeneme 2048 Tampão Fosfato [0,01M] pH 7,2 50,0
Ciprofloxacina 1024 Água + hidróxido de sódio [0,1M] 93,6
Tigeciclina 1024 Água 99,7
Colistina 2048 Água 78,6
26
2.1.2. Preparação das concentrações dos antibióticos
Após a obtenção da solução-mãe foi preparada a solução CIM (utilizadas diretamente no teste), que corresponde a uma concentração acima da maior concentração a ser testada. Assim, esta solução CIM (Tabela 5) foi preparada a partir da solução-mãe com um diluente específico para cada antibiótico.
Tabela 5 - Antibióticos, concentração da solução CIM, diluentes e concentrações testadas na determinação da concentração inibitória mínima.
Antibiótico Solução CIM (mg/L) Diluente Concentrações testadas (μg/mL) Cefotaxima 128 Água 1 – 64 Ceftazidima 128 Água 1 – 64 Cefoxitina 256 Água 2 – 128
Imipeneme 1024 Tampão Fosfato [0,01M] pH 7,2 0,25 – 512
Ciprofloxacina 16 Água 0 125 – 8
Tigeciclina 32 Água 0,25 – 16
Colistina 64 Água 0,5 – 32
Ácido Clavulânico 40 mg/mL Tampão Fosfato [0,1M] pH 6
2.1.3. Preparação das diluições dos antibióticos na microplaca
O meio de Mueller-Hinton (INSA) foi distribuído (100 µL) em cada um dos 96 poços da microplaca com o auxílio de um aparelho de microdiluição (Precision TM BioTek, Winooski, USA). Para a tigeciclina utilizou-se meio de Mueller-Hinton fresco (<12 h) (Bradford et al. 2005). De seguida, com aquele aparelho de microdiluição foi realizada a diluição seriada dos antibióticos nas microplacas, que já possuiam meio de Mueller-Hinton (Fig. 9). Assim, foram adicionados 100 µL da solução CIM, do antibiótico a testar, nos poços da primeira fila e diluído de forma seriada, pipetando 100 µL da primeira fila para a segunda e, assim sucessivamente, até à penúltima fila, rejeitando, por fim, os 100 µL. A última fila foi utilizada como controlo negativo.