Faculdade de Medicina de Lisboa
Estudo do gene
diferentes perfis de utilização dos co
Paula Alexandra Alves Cerejo
Mestrado em Microbiologia Clínica
Universidade de Lisboa
Faculdade de Medicina de Lisboa
Estudo do gene env de estirpes de HIV
diferentes perfis de utilização dos co-receptores
Paula Alexandra Alves Cerejo
Mestrado em Microbiologia Clínica
2009
HIV-2 com
receptores
A impressão desta dissertação foi aprovada pela Comissão
Coordenadora do Conselho Científico da Faculdade de Medicina de
Lisboa em reunião de 14 de Julho de 2009.
Faculdade
Estudo do gene
diferentes perfis de utilização dos co
Paula Alexandra Alves Cerejo
Mestrado em Microbiologia Clínica
Dissertação Orientada pelo Professor Doutor José Miguel Azevedo
Todas as afirmações efectuadas no presente documento são da exclusiva responsabilidade da sua autora, não cabendo qualquer responsabilidade à Faculdade de Medicina de Lisboa pelos conteúdos nele apresentados.
Universidade de Lisboa
Faculdade de Medicina de Lisboa
Estudo do gene env de estirpes de HIV
diferentes perfis de utilização dos co-receptores
Paula Alexandra Alves Cerejo
Mestrado em Microbiologia Clínica
Dissertação Orientada pelo Professor Doutor José Miguel Azevedo
Pereira
Todas as afirmações efectuadas no presente documento são da exclusiva responsabilidade da sua autora, não cabendo qualquer responsabilidade à Faculdade de Medicina de Lisboa pelos conteúdos nele apresentados.
HIV-2 com
receptores
Dissertação Orientada pelo Professor Doutor José Miguel Azevedo
Todas as afirmações efectuadas no presente documento são da exclusiva responsabilidade da sua autora, não cabendo qualquer responsabilidade à Faculdade de Medicina de Lisboa pelos conteúdos
i
A
GRADECIMENTOSComeço por agradecer ao meu orientador, o Professor Doutor José Miguel Azevedo Pereira, pela sua orientação científica, e pela paciência que sempre me dedicou.
Obrigado à Unidade de Retrovírus e Infecções Associadas do Centro de Patogénese Molécular, na pessoa do Professor Doutor José Moniz Pereira, por permitir a utilização das instalações para desenvolvimento deste trabalho.
Gostaria de agradecer a todos os coordenadores, professores e colegas da 5ª edição do Mestrado em Microbiologia Clínica. Sem duvida que aprendi muito com todos, principalmente no ano curricular.
Ao Professor Doutor Ricardo Parreira, do Instituto de Higiene e Medicina Tropical, quero deixar um agradecimento muito especial, por toda a ajuda e pelos conhecimentos científicos que amavelmente me transmitiu.
Um muito obrigada a vários professores e colegas dos departamentos de Biologia, Bioquímica, Biotecnologia, Orgânicas e CPM, e a todos os elementos do departamento de Microbiologia, em especial à Quirina, pela sua ajuda nas varias fases do meu trabalho.
Aos meus colegas do laboratório ‘do lado’, Cheila, Inês, Pedro, quero agradecer pelos conselhos que trocaram comigo.
ii Um agradecimento muito especial às minhas colegas e amigas, Fátima Espírito Santo, Marta Calado e Marta Simões pelos bons momentos que passámos juntas, pelas ‘aventuras’, pelas experiências trocadas e até pelas lágrimas vertidas... de ambas, fiquei com a amizade e a confiança, muito obrigada!
Não há palavras suficientes para agradecer a quem está sempre lá! Mãe, Mana, Pai, Eduardo Júnior, Rumbo e Guga... Do fundo do Coração!
Dedico esta tese a todas as pessoas que amávelmente contribuiram para a sua elaboração.
iii
Resumo
A análise dos determinantes genéticos para o uso dos diferentes co-receptores têm sido quase exclusivamente feita usando o HIV-1 como modelo, no entanto alguns dados sugerem que estes determinantes serão diferentes no HIV-2. De forma a dar continuidade à linha de investigação sobre a análise dos determinantes genéticos para o uso dos diferentes co-receptores no HIV-2, procedeu-se à sequenciação do gene env de estirpes primárias de HIV-2, num conjunto de 6 isolados primários, HIV-2TER,
HIV-2MIL, HIV-2MS, HIV-2JAU, HIV-2MLC e HIV-2ETP. Estes vírus foram isolados a partir de
doentes em fases distintas da história natural da infecção e com perfis diferentes na gama de co-receptores usados para a entrada do vírus na célula. O trabalho desenvolvido nesta dissertação teve como principal objectivo, correlacionar as sequências do gene env com o perfil fenotípico apresentado pelas estirpes, na perspectiva de averiguar a existência de algum padrão genotípico subjacente á utilização dos diferentes co-receptores.
Por sequenciação e posterior análise filogenética demonstrou-se que todos os vírus estudados se inseriam no grupo filogenético A do HIV-2. Através da análise do tamanho das regiões variáveis e conservadas, verificou-se que o tamanho da região V1 dos isolados em estudo que se encontram em fase clínica de SIDA é superior ao tamanho da região V1 dos isolados em fase assintomática e em fase sintomática. O número e locais de N-glicosilação das regiões variáveis também foi analisado, tendo-se verificado que independentemente dos co-receptores utilizados e do estado clínico, todos os isolados apresentavam na região V3, um local de glicosilação conservado a seguir à primeira cisteína. Na região V2 o primeiro local de N-glicosilação a seguir à primeira cisteína encontra-se conservado nos isolados que não utilizam o CXCR4 como
iv co-receptor. Estes isolados possuíam ainda um maior número de locais de glicosilação na região V1/V2, relativamente aos isolados que utilizam o CXCR4 como co-receptor. Das 3 regiões variáveis (V1, V2 e V3) a V3 foi a que apresentou sequências mais conservadas tanto em tamanho como na constituição aminoacídica. Não se encontrou nenhuma correlação entre os aa existentes nas posições 17 e/ou 18 e 27 da região V3 (considerados em estudos anteriores determinantes para a utilização dos co-receptores), bem como da carga da mesma região, com a utilização dos diferentes co-receptores. Concluímos, com base neste trabalho, que para o HIV-2 os determinantes genéticos para o uso dos diferentes co-receptores são diferentes dos determinantes do HIV-1. Quando se correlacionou as sequências do gene env, com o perfil fenotípico apresentado pelas estirpes em estudo, não se encontrou nenhum padrão genotípico subjacente à utilização dos diferentes co-receptores, ao contrário do que se tem verificado para o HIV-1.
v
Abstract
The analyses of genetics determinants for different coreceptors usage have been exclusively made for HIV-1 model, however several data suggest that these determinants could be different for HIV-2. In order to continue our ongoing research regarding coreceptors usage by HIV-2, we sequenced the env gene of primary strains of HIV-2, in a group of 6 primary isolates, HIV-2TER, HIV-2MIL, HIV-2MS, HIV-2JAU,
HIV-2MLC, and HIV-2ETP. These viruses have been isolated from patients in different disease
stage and with different coreceptors usage for the viral entry into cells. The main goal of this work was to correlate the env gene sequence with the phenotype presented by the different strains in order to ascertain the possible correlation between env gene sequence and coreceptor usage.
By sequencing and posterior phylogenetic analyses we demonstrated that all the viruses that have been studied belong to the phylogenetic group A of HIV-2. With the analyses of the variable and conserved regions, we verified that the size of the V1 region of the isolates that were obtained from patients with AIDS was larger than the size of V1 region of the isolates in asymptomatic and symptomatic stage. The number and location of N-glycosylation of variable regions (V1, V2 and V3) was also analyzed, showing that regardless the coreceptors that were used and the clinical stage of the patient, all the isolates showed in V3 region, a N-glycosylation location conserved next to the first cystein. In V2 region the first N-glycosylation location, next to first cystein, is conserved in the isolates that do not use CXCR4 as a coreceptor. These isolates have more glycosylation sites in V1/V2 region. Of the three variable regions the V3 was the one that showed more conserved sequences, both in size as in aminoacidic constitution. We did not find any correlation between amino acids in the 17th and/or 18th and 27th
vi position of V3 region (considered in previous studies determinants for the use of coreceptors), as well as the charge of the same region, with the coreceptors usage. In conclusion, based on this work, we showed that for HIV-2 the genetic determinants for the different coreceptors usage are different from the ones that are used for HIV-1. When we correlate the sequences of the env gene, with the phenotypic profile of the HIV-2 strains, we did not find any genetic signature subjacent to the use of the different coreceptors in contrast to what has been demonstrated for HIV-1 model.
vii
Abreviaturas
aa Aminoácido(s)
ADN Ácido desoxirribonucleico
ARN Ácido ribonucleico
CA Proteína da cápside viral
CCR5 Receptor de quimiocina do tipo CC
CMSP Células mononucleadas do sangue periférico
CXCR4 Receptor de quimiocina do tipo CXC
gp Glicoproteína
dNTP Desoxirribonucleótido(s) 5’ -trifosfatados
H Hora
HLA Proteínas apresentadoras de antigénio à superfície das células,
do inglês, Human Leukocyte Antigen
HR Padrões repetidos de sete aminoácidos, do inglês hepetadrepeat
IN Integrase
KDa KiloDaltons
LB Meio de Luria-Bertani
Leucine-zipper Estrutura em fecho de Leucinas
Loop Estrutura em arco
LTNP Progressor Lento, do inglês Long Term Non-Progressors
LTR Repetição terminal longa, do inglês Long Terminal Repeat
M Molaridade
MA Proteína da Matriz viral
viii
mM Milimolar
ml Mililitro
NaOH Hidróxido de sódio
NC Proteína da nucleocápside
Nef Proteína de factor negativo
NSI Não indutor de sincícios, do inglês Non-Syncicium Inducing
Pb Pares de bases
PCR Reacção de amplificação em cadeia pelo enzima Polimerase, do inglês Polymerase Chain Reaction
PR Protease
Primers Oligonucleótidos sintéticos iniciadores
R Região não codificante repetida situada nas extremidades do genoma do HIV, do inglês Repeated Sequence
RANTES Do inglês regulated on activation, normal T- cell expressed and
secreted
Rev Proteina reguladora viral
RT Transcriptase Reversa, do inglês Reverse Transcriptase
SDF-1 do inglês, Stromall Cell-Derived Factor 1
SDS Dodecilsulfato de sódio, do inglês Sodium Dodecyl Sulphate
Seg Segundo(s)
SI Indutor de sincícios, do inglês Syncicium Inducing
SIDA Sindroma da Imunodeficiência Adquirida
SIGN Do inglês, Specific Intercellular adhesion molecule3
(ICAM-3)-Grabbing Nonintegrin
ix
Tat Proteina trans-activadora da transcrição
TE Tampão tris-EDTA
TM Glicoproteína transmembranar
Tris-HCl Tris-Hidrocloreto
U Unidade enzimática
U3 Região situada na extremidade 3’ do genoma do HIV, do inglês
Unique Sequence 3
U5 Região não codificante situada próximo da extremidade 5’ do genoma do HIV, do inglês Unique Sequence 5
UV Radiação ultravioleta
Vif Factor de infecciosidade viral
HIV Vírus da Imunodeficiência Humana
HIV-1 Vírus da Imunodeficiência Humana do tipo 1
HIV-2 Vírus da Imunodeficiência Humana do tipo 2
SIV Vírus da Imunodeficiência Símia
SIVcpz Vírus da Imunodeficiência Símia dos chimpanzés da subespécie
Pan troglodytes troglodytes
SIVsm Vírus da Imunodeficiência Símia dos macacos da subespécie
Cercocebus torgnatus
Vpr Proteína viral R
Vpu Proteína viral U
Vpx Proteína viral X
ß-sheet Estrutura em follha-beta
µg Micrograma
x
xi
Índice Geral
Agradecimentos ... i
Resumo... iii
Abstract... v
Abreviaturas e Símbolos... vii
Índice geral... xi
Índice de figuras... xiv
Índice de tabelas... xvi
1. Introdução... ... 1
1.1. Síndroma da Imunodeficiência Adquirida... 2
1.2. Vírus da Imunodeficiência Humana do Tipo 2... 3
1.2.1 Organização Estrutural... 4
1.2.2. Organização Genómica... 5
1.2.3. Ciclo de Replicação... 7
1.2. Variabilidade Genética... 8
1.4. Patogénese e progressão para doença... 11
1.4.1. Patogenicidade do HIV-2... 15
1.5. Epidemiologia da infecção pelo HIV-2... 15 1.6. O gene env do HIV-2... 16 1.6.1. Glicoproteína de superfície e glicoproteína transmembranar... 17
1.6.2. Evolução molecular durante a infecção... ... 20
1.7. Aspectos biológicos e moleculares relacionados com a utilização de co-receptores... 21
1.7.1. O CD4 como receptores primários do HIV... 21
xii
1.7.2. Receptores das quimiocinas como co-receptores do
HIV... 22
1.7.3. Factores determinantes para a utilização dos co-receptores do HIV... 23 1.7.4. Tropismo e uso de co- receptores... 24
2. Objectivos... 28
3. Materiais e Métodos... 30 3.1. Amostra... 31
3.1.1. Preparação da amostra... 31
3.2. Oligonucleótidos Sintéticos iniciadores Primers... 32
3.3. Reacção de polimerização enzimática em cadeia – PCR... 33
3.4. Purificação de PCR a partir de Gel de Agarose... 35
3.5. Reacção de Clonagem... 36
3.5.1. Produção de extremidades cegas (Blunt ends)... 36
3.5.2. Clonagem no vector de expressão e extracção do ADN Plasmídico... 36
3.6. Digestão com enzimas de restrição – confirmação dos clones... 39
3.7. Purificação do ADN plasmídico ... 39
3.8. Sequenciação de ADN... 40
3.9. Análise Bioinformática das Sequências... 40
4. Resultados... 42
4.1. Amplificação e clonagem do gene env... 43
4.2. Alinhamento das Sequências Nucleotídicas das Amostras... 46
xiii
4.4. Organização Aminoacídica da Região C1-C3 do gene env... 52
4.5. Tamanho das regiões variáveis e conservadas estudadas e a sua relação com a progressão clínica e imunológica... 55
4.6. Análise dos locais de glicosilação dependentes da asparagina (N-glicosilação) e utilização dereceptores... 57 4.7. Região variável V3... 60
4.8. Análise genética e utilização de co-receptores... 61
5. Discussão de Resultados... 65
6. Conclusões... 73
xiv
Índice de Figuras
Introdução
Figura 1.1 Organização estrutural do HIV-2... 5
Figura 1.2 Representação esquemática do genoma do HIV-2 e descrição da função
das proteínas auxíliares eacessórias... 6
Figura 1.3 Ciclo de replicação do HIV-2... 9
Figura 1.4 Evolução da infecção por HIV ao longo do tempo. Relação entre a
sintomatologia, carga viral no sangue e contagem de células T CD4+... 13
Figura 1.5 Interacção entre as glicoproteína do invólucro e o receptor
co-receptor celulares... 18
Materiais e Métodos
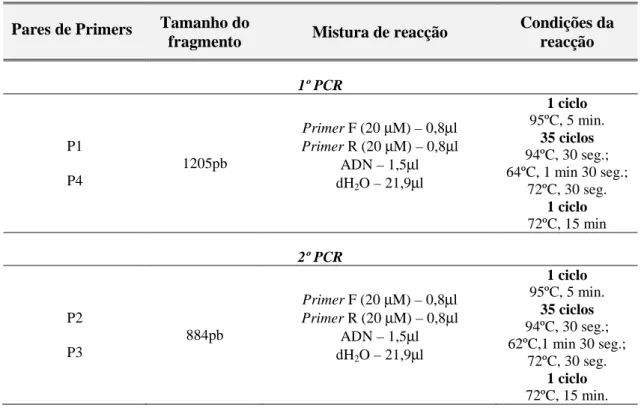
Figura 3.1 Esquema representativo da estratégia de nested-PCR utilizada na
amplificação do fragmento de ADN... 34
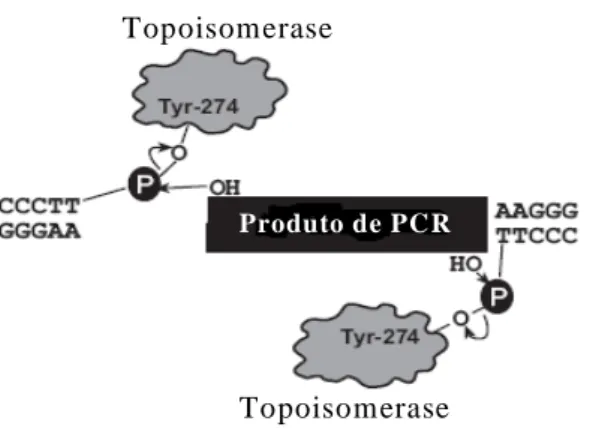
Figura 3.2 Esquema representativo do processo de clonagem em
pCR4Blunt- TOPO, mediado pela enzima topoisomerase I...
37
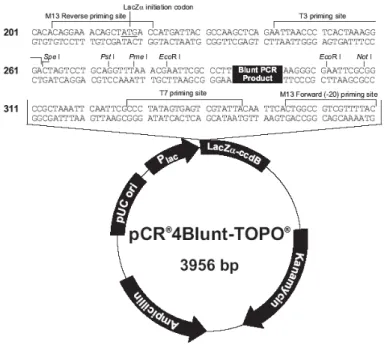
Figura 3.3 Mapa físico do vector pCR4Blunt-TOPO com coordenadas de corte para enzimas de restrição. pUC ori indica a origem de replicação derivada do plasmídio ColE1 e LacZα-CcdB representa o segmento codificante da proteína
de fusãoLacZαCcdB... 38
Resultados
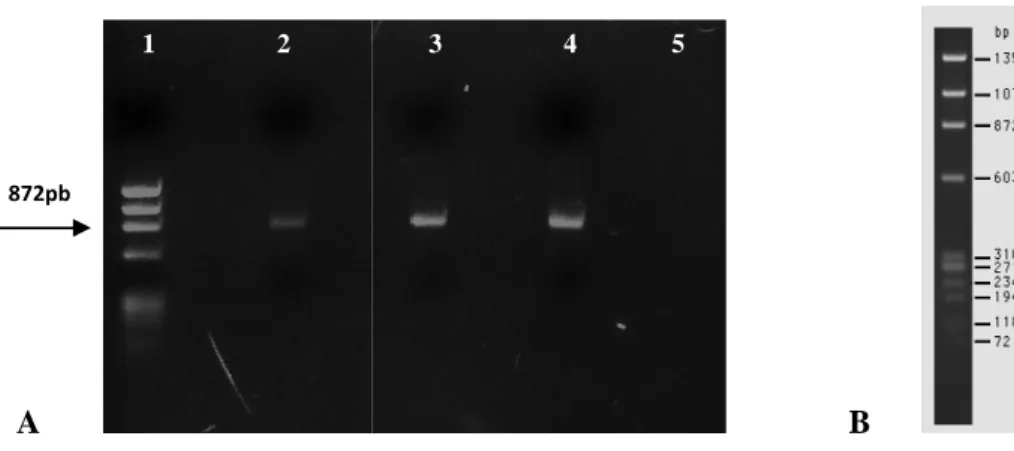
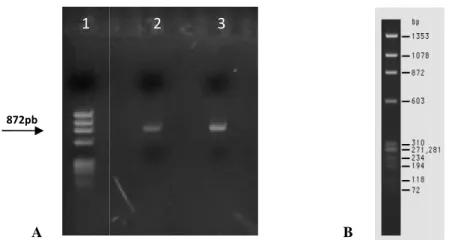
Figura 4.1 Fragmento resultante da amplificação (em duas etapas) da
região que compreende o final da C1 e o início da C3 do gene env... 44
Figura 4.2 Gele de agarose a 0.7% com 1,5 µl de produtos de PCR
purificado, após electroforese coloração com brometo de etídio... 45
Figura 4.3 Clonagem do fragmento amplificado correspondente ao final da C1
e o início da C3 do gene env da amostra HIV-2TER, no vector
plasmídico pCR4Blunt-TOPO... 46
Figura 4.4 Alinhamento das sequências de aminoácidos correspondentes
ao fragmento de 884 pb compreendido entre o final da C1e o início da C3 do gene env de todos os clonesdos vírus HIV-2TER, HIV-2MIL, HIV-2MS,
xv
Figura 4.5 Alinhamento de todas as sequências consenso dos vírus
HIV-2TER, HIV-2MIL, HIV-2MS, HIV-2JAU, HIV-2ETP e HIV-2MLC... 49 Figura 4.6 Árvore filogenética obtida após o alinhamento entre
sequências de referência do gene env de diferentes grupos de HIV-2
e SIV e as sequências obtidas neste estudo... 51
Figura 4.7 Alinhamento de aminoácidos correspondente à região que medeia
o final da C1 e o início da C3, das sequências consenso dos vírus
em estudo... 53
Figura 4.8 Alinhamento de aminoácidos correspondente à região V2... 58 Figura 4.9 Diferenças aminoacídicas na região V3... 61
xvi
Índice de Tabelas
Materiais e Métodos
Tabela 3.1 Informação clínica e co-receptores dos vírus utilizados neste
Trabalho... 31
Tabela 3.2 Primers para amplificação por PCR do gene env do HIV-2... 33
Tabela 3.3 Condições de PCR para amplificação do Fragmento de 884 pb... 35
Resultados
Tabela 4.1 Características dos vírus em estudo... 44
Tabela 4.2 Tamanho de cada região analisada, em termos de número
De aminoácidos e informação clínica... 56
Tabela 4.3 Número de potenciais locais de N-glicosilação em cada
região analisada e co-receptores utilizados... 59
Tabela 4.4 Aminoácidos nas posições 18,19 e 27 da região V3,
carga da região V3, e uso de co-receptores dos vírusem estudo ... 62
Tabela 4.5 Cargas correspondentes a cada região analisada e os
1
2
1.
Introdução
1.1. Síndroma da Imunodeficiência Adquirida
A infecção pelo Vírus da Imunodeficiência Humana (HIV), agente causal da Síndroma de Imunodeficiência Adquirida (SIDA), é uma das epidemias mais devastadoras da história mundial que marcou as últimas décadas do Século XX e início deste Século. Apesar dos investimentos, do desenvolvimento político e do progresso na expansão do acesso ao tratamento para a infecção pelo HIV, a SIDA continua a exigir uma resposta global. A epidemia permanece extremamente dinâmica, em crescimento e a mudar de características à medida que o vírus explora novas oportunidades de transmissão 130.
O HIV foi isolado pela primeira vez em 1983 7 e estabelecido como agente etiológico da SIDA em 1984 37. Estudos filogenéticos demonstram que o HIV tem por ancestral o Vírus da Imunodeficiência Símia (SIV) e que a passagem da barreira da espécie do macaco para o Homem terá ocorrido por volta de 1930 no caso do HIV-1 e na década de 40 para o HIV-2 43,67,72.
A transmissão do HIV para o homem ocorreu a partir de espécies de primatas não humanos diferentes dando origem a dois vírus distintos: o HIV do tipo 1 (HIV-1) e o HIV do tipo 2 (HIV-2). Pensa-se que o HIV-1 tenha origem em SIV que infectam chimpanzés da subespécie Pan troglodytes troglodytes (SIVcpz) e que o HIV-2 provenha
3 Os dois agentes etiológicos da SIDA, o HIV-1 e o HIV-2, pertencem ao género
Lentivirus, à sub-familia Orthoretrovirinae e à família Retroviridae 7,42,59. Distinguem-se pelas suas propriedades antigénicas, moleculares, biológicas e nas relações filogenéticas com os outros lentivírus dos primatas; 5,7,21 diferem entre si em cerca de 55% da sua sequência nucleotídica 74. Em termos biológicos são ambos capazes de degradar progressivamente o sistema imunitário dos indivíduos infectados, aumentando consequentemente a susceptibilidade a infecções oportunistas e a neoplasias, o que conduz ao estado de SIDA e à morte 74.
O HIV-1 é o agente responsável pela pandemia a nível mundial, enquanto o HIV-2 é responsável por epidemias localizadas, sobretudo em países da África Ocidental e por um número reduzido de casos na Europa e noutros continentes 9,31,120,100. Portugal é o país da Europa com maior número de casos de infecção pelo HIV-2, devido às estreitas relações que mantém com diversos países Africanos onde a infecção é endémica 106,110.
1.2. Vírus da Imunodeficiência Humana Tipo 2
O HIV-1 foi isolado pela primeira vez em 1983 pelo grupo liderado por Luc Montagnier
7
, no Instituto Pasteur de Paris. Em 1986 todos os isolados virais associados à SIDA viriam a ser denominados por HIV pela Comissão Internacional de Taxionomia Vírica
73
. Nesse mesmo ano, uma equipa de investigadores portugueses e franceses, isolaram um novo tipo de HIV em indivíduos oriundos da Guiné-Bissau com sinais de imunodeficiência, o qual viria a ser posteriormente denominado de HIV-2 21.
4
1.2.1
. Organização Estrutural
As partículas virais maduras do HIV-2 têm morfologia esférica, com cerca de 110 nm de diâmetro e encontram-se revestidas por um invólucro de natureza lipídica derivado da membrana celular das células infectadas (Figura 1.1). Este invólucro é composto por trímeros da glicoproteína de superfície (SU/gp105) e pela glicoproteína transmembranar (TM/gp36), podendo ainda incorporar diferentes proteínas da célula hospedeira, como por exemplo proteínas HLA (Human Leukocyte Antigens) de classe I e II. Internamente a partícula viral é revestida pela proteína da matriz (MA/p16), essencial para a integridade do virião. A cápside viral (CA), constituída pela proteína p26, possui no seu interior duas moléculas de ácido ribonucleico (ARN) idênticas com polaridade positiva e com cerca de 9200 pares de bases (pb) 117, associadas às proteínas da nucleocápside (NC/p6). Possui também todas as enzimas necessárias à replicação, como a retrotranscriptase (RT/P53), e a protease (PR/p11), a integrase (IN/p34), e as proteínas Nef, Vif, e Vpr 24,37,56.
Figura 1.1
(Adaptado de http://www.wellesley.edu/Chemistry/Chem101/hiv/maturhiv.gif
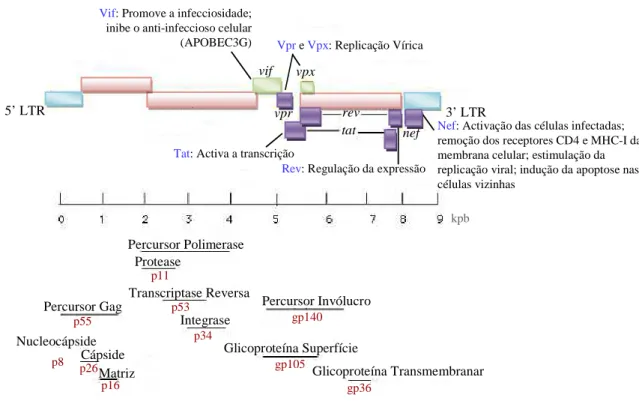
1.2.2. Organização genómica
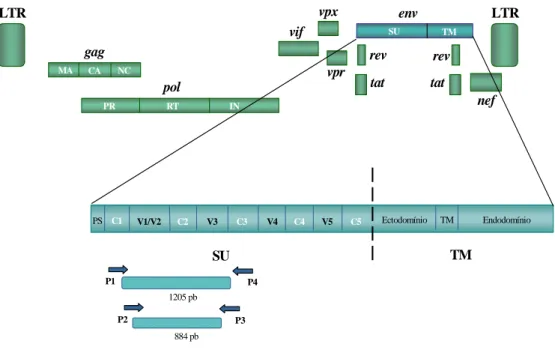
O genoma de HIV-2 é constituído por nove genes, que codificam para proteínas estruturais (Gag e Env), proteína com funções enzimáticas (Pol), proteínas reguladoras (Tat e Rev) e proteínas acessórias (Nef, Vif, Vpr e Vpx), tendo um total de 9,8 Kb (Figura 1.2) 24,74. env glicoproteína transmembranar (TM) gp36 gag proteína da cápside (CA) P26 ARN (2) moléculas pol protease (PR) p11 retrotranscriptase (RT) p53 integrase (IN) p34 env glicoproteína de superfície (SU) gp105 gag proteína da matriz (MA) p16
Figura 1.1 – Organização estrutural do HIV-2
http://www.wellesley.edu/Chemistry/Chem101/hiv/maturhiv.gif
ão genómica
2 é constituído por nove genes, que codificam para proteínas estruturais (Gag e Env), proteína com funções enzimáticas (Pol), proteínas reguladoras (Tat e Rev) e proteínas acessórias (Nef, Vif, Vpr e Vpx), tendo um total de 9,8 Kb
5
http://www.wellesley.edu/Chemistry/Chem101/hiv/maturhiv.gif).
2 é constituído por nove genes, que codificam para proteínas estruturais (Gag e Env), proteína com funções enzimáticas (Pol), proteínas reguladoras (Tat e Rev) e proteínas acessórias (Nef, Vif, Vpr e Vpx), tendo um total de 9,8 Kb
6
Figura 1.2 – Representação esquemática do genoma do HIV-2 e descrição da função das proteínas
auxíliares e acessórias (adaptado de http://uhavax.hartford.edu/bugl/hiv.htm ).
Os genes gag e pol codificam para a poliproteína percursora Gag-Pol de 160 KDa, que é processada em sete proteínas pela PR 24,56,74. O gene gag codifica para quatro proteínas estruturais: MA (p16), CA (p26), NC (p8) e proteína C-terminal (p6) (Figura 1.2). O gene pol codifica para três proteínas essenciais para a replicação viral: PR (p11), RT (p53) e IN (p34). A PR é responsável pelo processamento da poliproteína Gag-Pol durante a maturação do virião; a RT converte o ARN viral em ácido desoxirribonucleico (ADN) proviral de dupla cadeia, a IN cataliza a integração do ADN proviral no genoma da célula hospedeira 24,56,74. 5’ LTR 3’ LTR gag pol env tat vif vpx rev nef vpr kpb Percursor Gag Glicoproteína Superfície Glicoproteína Transmembranar Percursor Invólucro Integrase Transcriptase Reversa Protease Percursor Polimerase p34 gp105 gp36 gp140 p53 p11 p55 Matriz Cápside p16 p26 p56 Nucleocápside
Vif: Promove a infecciosidade; inibe o anti-infeccioso celular (APOBEC3G)
Nef: Activação das células infectadas; remoção dos receptores CD4 e MHC-I da membrana celular; estimulação da replicação viral; indução da apoptose nas células vizinhas
Rev: Regulação da expressão Tat: Activa a transcrição
Vpr eVpx: Replicação Vírica
p8
gag
7 No HIV-2, o gene env codifica para a poliproteína gp140 (Figura 1.2), que é clivada por uma protease celular, originando as glicoproteínas do invólucro gp105 (SU) e gp36 (TM) 108. A gp105 possui os domínios específicos de ligação aos receptores da superfície da célula hospedeira e a gp36 é determinante para a fusão do vírus com a membrana celular. Ambas as glicoproteína são altamente antigénicas 24,56,74.
Nas extremidades do genoma do HIV-2 existem regiões não codificantes – U3, R e U5 – que durante a transcrição reversa são duplicadas para formar a estrutura LTR, (repetições terminais longas). Estas regiões não codificam para qualquer proteína, mas têm um papel importante na integração do vírus no ADN da célula hospedeira e na regulação da expressão de provírus 24,56,74. A região LTR funciona como local de inicio da transcrição. É composta por três regiões: U3 (extremidade 3’ única), R (repetidas em ambas as extremidades), U5 (extremidades 5’ única). A transcrição inicia-se na junção U3/R. A transcrição a partir do provírus é regulada pela actividade do promotor do HIV, a região LTR da extremidade 5’. A região LTR possui locais de ligação para vários factores de transcrição celular, entre os quais se encontram os factores responsáveis pela activação das células T. Este facto poderá explicar o motivo pelo qual a activação destas células promove a expressão viral 140.
1.2.3. Ciclo de replicação
A infecção pelo HIV inicia-se com a ligação do vírus a um receptor específico, o CD4, presente na superfície celular dos linfócitos T auxiliares ou das células de linhas macrofágicas e monocíticas 19,24,56. Para além do CD4, existem outras moléculas expressas à superfície celular que são essenciais para a interacção inicial do vírus e a
8 célula hospedeira. Estas moléculas são os receptores das quimiocinas e funcionam como co-receptores para a entrada do HIV na célula 4,24,42,56. Os principais co-receptores utilizados pelo HIV in vivo parecem ser o CCR5 e o CXCR4 4,42.
A ligação da glicoproteína SU ao receptor CD4 da célula hospedeira provoca alterações na conformação da glicoproteína, expondo o domínio de ligação ao co-receptor 37. Por sua vez, esta ligação induz um rearranjo na glicoproteína TM que permite a fusão do invólucro com a membrana celular e consequentemente a entrada da nucleocápside viral para o interior da célula 37.
Após entrada e descapsidação no citoplasma, a RT efectua a transcrição reversa do ARN viral em ADN de cadeia dupla, originando o complexo de pré-integração 37,49. Este complexo compreende o ADN de cadeia dupla, e as proteínas virais IN, MA, RT e Vpr. O transporte do complexo de pré-integração para o núcleo é mediado por IN, MA e Vpr. Uma vez no núcleo, a IN integra o ADN viral no genoma da célula hospedeira. O ADN proviral integrado pode persistir na célula no estado de latência. O ciclo de replicação viral continua com a transcrição do ADN viral, recorrendo à maquinaria transcripcional da célula hospedeira 37,49.
O ARN genómico e as proteínas virais produzidas são direccionadas para a superfície celular, onde se formam as novas partículas virais imaturas que são libertadas após lise das células (Figura 1.3). A maturação final dá-se com a clivagem da proteína precursora Gag-Pol pela PR e posterior rearranjo estrutural e de posicionamento das proteínas virais, originando as partículas virais maduras e infecciosas 37,49.
Figura
(Adaptado de: http://WWW.rhodes.edu/biology/glindquester/viruses/pagespass/hiv/retrovirus.com
1.3. Variabilidade Genética
A variabilidade genética do HIV está em parte associada ao facto de se tratar de um vírus que utiliza uma polimerase de ácido desoxirribonucleico dependente do ARN transcriptase reversa, com baixa fiabilidade e sem sistema de correcção de erros de transcrição 63. Devido a uma complexidade genética muito limitada, por comparação com os genomas de ADN, os vírus de ARN toleram elevados níveis de mutações. O
Receptor Transcriptase Reversa Integrase Tradução ARN virico Protease
Figura 1.3 – Ciclo de replicação do HIV-2
http://WWW.rhodes.edu/biology/glindquester/viruses/pagespass/hiv/retrovirus.com
Variabilidade Genética
A variabilidade genética do HIV está em parte associada ao facto de se tratar de um vírus que utiliza uma polimerase de ácido desoxirribonucleico dependente do ARN transcriptase reversa, com baixa fiabilidade e sem sistema de correcção de erros de
. Devido a uma complexidade genética muito limitada, por comparação com os genomas de ADN, os vírus de ARN toleram elevados níveis de mutações. O
Membrana
Transcriptase
Reversa ARN virico
ADN Núcleo Integração Integrase Transcrição o Tradução Proteína Virica Citoplasma ARN Virico ARN virico Protease Novos vírus 9 http://WWW.rhodes.edu/biology/glindquester/viruses/pagespass/hiv/retrovirus.com).
A variabilidade genética do HIV está em parte associada ao facto de se tratar de um vírus que utiliza uma polimerase de ácido desoxirribonucleico dependente do ARN – a transcriptase reversa, com baixa fiabilidade e sem sistema de correcção de erros de . Devido a uma complexidade genética muito limitada, por comparação com os genomas de ADN, os vírus de ARN toleram elevados níveis de mutações. O
10 somatório de todos estes factores, levou à estimativa de 3x10-5 mutações por nucleótido incorporado, por ciclo de replicação. Por outro lado, durante a transcrição reversa, dada a natureza diplóide do genoma, existe a possibilidade de ocorrência de eventos de recombinação, homóloga ou heteróloga, entre genomas virais 54,133. Os eventos de recombinação poderão ocorrer, pelo menos, por um de três mecanismos: (1) Recombinação por transferência forçada de cadeia. A descontinuidade da cadeia de ARN dadora leva a paragens forçadas da transcrição reversa dessa cadeia e à continuação da síntese de ADN de polaridade positiva na outra cadeia de ARN; (2) Recombinação por transferência de cadeia, motivada por uma pausa na transcrição reversa. A dissociação do ADN nascente de polaridade positiva do ARN dador e associação com a cadeia de ARN aceitador. (3) Recombinação por transferência de cadeia, independente de pausas na transcrição reversa. Neste caso, a transferência da cadeia depende da formação de uma estrutura secundária no ARN dador, o que permite a aproximação entre o ARN aceitador e o ADN nascente 89.
A elevada taxa de divergência conduz à geração de uma população de partículas virais aparentadas, mas geneticamente heterogéneas nos indivíduos infectados, denominada por Eigen, Schuster e colaboradores por “quasi-espécies” 85.
Quasi-espécies são populações dinâmicas de mutantes e genomas virais diferentes mas
relacionados proximamente. Estão sujeitos a processos contínuos de variação genética, competição e selecção. As implicações biológicas das quasi-espécies estendem-se desde a patogénese viral até à emergência de novos vírus patogénicos 33.
11 A passagem de uma quasi-espécies viral de um indivíduo para outro irá fixar um grupo específico de mutações, cada uma com uma probabilidade igual à sua frequência na população. Se o crescimento continuar sob as mesmas condições ambientais, então o processo evolutivo irá criar uma nova quasi-espécies muito semelhante à inicial. Pelo contrário, se as quasi-espécies forem sujeitas a novas influências selectivas, como por exemplo se um anticorpo aparecer ou se o vírus infectar um novo tecido, então as quasi-espécies permitem uma rápida selecção, a partir do conjunto das mutações preexistentes, conduzindo a uma rápida adaptação às novas condições 85.
.
1.4. Patogénese e progressão para doença
A imunopatogénese da infecção pelo HIV está associada a um paradoxo no sistema imunitário, onde concomitantemente com uma severa supressão do sistema imunitário ocorre uma elevada activação do mesmo. As manifestações clínicas e a gravidade da doença correspondem essencialmente a alterações moleculares, celulares e funcionais, que esta infecção progressivamente origina no sistema imunitário, até à total aniquilação deste na fase terminal da doença 78,136.
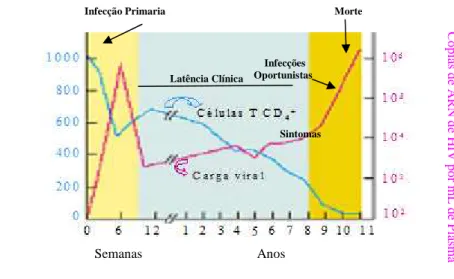
O curso normal da infecção pelo HIV na maioria (80-90%) dos indivíduos infectados pode ser dividido em três fases: infecção primária, latência clínica (fase assintomática) e fase de doença (fase sintomática) 8.
A infecção primária está associada a uma elevada carga viral no sangue e doença aguda febril, caracterizada por sintomatologia não especifica e resolução espontânea. Por
12 acção de uma resposta imunitária, a virémia decresce para níveis relativamente baixos, entrando na fase de latência 8.
A fase de latência clínica é, geralmente, um longo período que dura em média 8 a 10 anos no HIV-1, sendo no entanto mais alargado para o HIV-2 3,84,122,123. Vários factores podem estar implicados no controlo da replicação viral, incluindo anticorpos neutralizantes, células T citotóxicas específicas para o vírus, citocinas, quimiocinas e a disponibilidade dos co-receptores para a entrada do vírus 8.Contudo, durante esta fase verifica-se elevados níveis de replicação no tecido linfóide que persistem durante todo o decurso da infecção. A produção e eliminação de vírus são um processo dinâmico e que se mantém em equilíbrio durante esta fase. Esta replicação, associada à deterioração progressiva da função imunitária, resulta na progressão para a fase clínica (sintomática) ou SIDA. Esta última fase é caracterizada sobretudo por infecções oportunistas e neoplasias. Os indivíduos que apresentam este curso clínico são designados por progressores típicos (Figura 1.4) 114.
13
Figura1.4 - Evolução da infecção por HIV ao longo do tempo. Relação entre a sintomatologia, carga
viral no sangue e contagem de células T CD4+.
(Adaptado de http://www.niaid.nih.gov/puplications/hivaids/fig-5.gif.)
Uma menor percentagem (5-10%) dos indivíduos infectados pelo HIV-1 apresentam um rápido curso da infecção, progredindo para SIDA em 2 ou 3 anos após a seroconversão, são os chamados progressores rápidos 114.
Na última década da epidemia de SIDA tornou-se evidente que uma pequena fracção dos indivíduos infectados não apresenta progressão para doença, sendo designados por progressores lentos (LTNPS- long term nom-progressors), constituindo cerca de 5% dos
indivíduos infectados 114. Infecção Primaria Có p ia s d e A RN d e H IV p o r m L d e P la sm a Morte Co n ta g em d e li n fo ci to s T CD 4 + ( cé lu la s/ m m 3 ) Latência Clínica Semanas Anos Infecções Oportunistas Sintomas
14 Os critérios que definem o estado de não-progressão variam, mas incluem: infecção por HIV por mais de 7 anos; contagens estáveis de células T CD4+ >600/µ L; ausência de sintomas; inexistência de terapêutica antirretroviral 114.
Este facto representará uma resposta atípica do hospedeiro ou será uma resposta normal a um vírus invulgar?
Dada a dinâmica da interacção que envolve as quasi-espécies do HIV e as respostas imunitárias do hospedeiro, a resposta será na maior parte dos casos uma combinação de um vírus “excepcional” e de uma resposta do hospedeiro a esse vírus igualmente invulgar, induzida pela “excepcionalidade” desse vírus 114.
Contudo existem dados que demonstram que em alguns casos a resposta é direccionada para o fenótipo viral e em outros casos para a resistência do hospedeiro. A descoberta de que o HIV-2 é menos patogénico sugere a possibilidade de que algumas estirpes de HIV-1 também possam ser menos virulentas. Para além disso, vírus isolados a partir de doentes de progressão lenta mostram ser menos patogénicos e com replicação mais lenta
in vitro 114.
A infecção pelo HIV-2 in vivo revela várias características únicas quando comparada com a infecção pelo HIV-1, sendo a característica mais marcante o seu longo período assintomático. Neste facto estão envolvidos vários factores e todos eles parecem relacionar-se, directa ou indirectamente, com as características do HIV-2 como um agente patogénico atenuado. Um destes factores parece ser a eficácia com que este tipo de vírus usa os receptores celulares para entrar na célula hospedeira 4.
15
1.4.1. Patogenicidade do HIV-2
Tal como no caso da infecção pelo HIV-1, a infecção pelo HIV-2 provoca falência imunológica e uma série de manifestações clínicas semelhantes. No entanto, a infecção pelo HIV-2 tem um curso clínico e mecanismos infecciosos distintos, revelando um fenótipo menos virulento in vivo 4.
O prognóstico clínico de um indivíduo assintomático infectado pelo HIV-2 é, em geral, melhor do que para um indivíduo com o mesmo quadro clínico infectado pelo HIV-1. Nos indivíduos infectados pelo HIV-2 registam-se valores de mortalidade e morbilidade inferiores aos registados nos infectados pelo HIV-1 10,78. O período de latência clínica é normalmente superior, resultando numa mortalidade muito inferior à registada para o HIV-1, podendo nunca chegar a progredir para SIDA 87.
Como agente patogénico o HIV-2 é menos eficaz do que o HIV-1. Os aspectos moleculares responsáveis por esta menor eficácia estão longe de estar completamente caracterizados ou mesmo identificados, mas são certamente multifactoriais, envolvendo características, directas ou indirectamente, determinadas por este tipo de HIV 4.
1.5. Epidemiologia da infecção pelo HIV-2
A análise filogenética de sequências do HIV-2 permitiu datar a transmissão zoonótica do vírus para a primeira metade do século XX e determinar que esta epidemia teve início na Guiné-Bissau.135 Actualmente, o HIV-2 encontra-se principalmente neste país
16 da África Ocidental e nos países circundantes. Na Guiné-Bissau, a prevalência de HIV-2 é de 8 a 10%, e nos países vizinhos como Gâmbia, Senegal e República da Guiné a prevalência é de 1 a 2% 61,108. Sendo a Guiné-Bissau uma antiga colónia portuguesa, países como Índia, Angola, Moçambique e Brasil, com antigas relações sócio-económicas com Portugal, apresentam igualmente um número significativo de infecções por HIV-2 111,116. Portugal é o país da Europa com maior prevalência de HIV-2 106, 110. Até à data encontraram-se descritos oito grupos filogenéticos de HIV-2, classificados de A a H, que se pensa corresponderem a oito eventos de transmissão distintos do SIVsm
para o Homem 26.Apenas os grupos A e B são considerados epidémicos, uma vez que estão descritas poucas infecções por vírus de cada um dos grupos C, D, E, F, G e H
17,26,44,53,61,108,143
.Foram identificados recombinantes de HIV-2, demonstrando que pode ocorrer co-infecção e recombinação entre vírus do mesmo grupo e de grupos distintos
16,44,53
.
1.6. O gene env do VHI-2
A principal função da proteína Env é mediar a ligação aos receptores celulares e posterior fusão entre as membranas viral e celular, o que permite a entrada do material genético do vírus para o citoplasma da célula hospedeira 108.
17
1.6.1. Glicoproteína de superfície e glicoproteína transmembranar
No HIV, a proteína Env madura é gerada por clivagem proteolítica da poliproteína percursora gp160 ou gp140, no HIV-1 e no HIV-2, respectivamente 108. As glicoproteínas SU (gp120/gp105) e TM (gp41/gp36), resultantes desta clivagem, associam-se de forma não covalente, constituindo complexos tríméricos 136.
A região mais variável no gene env é a que codifica para a glicoproteína SU 18.Apesar desta variabilidade, alguns elementos estruturais e funcionais são conservados, permitindo a divisão da glicoproteína SU em cinco regiões hipervariáveis (V1 a V5) intercaladas com cinco regiões mais conservadas (C1 a C5), e numerosos locais de glicosilação dependentes da asparagina (N-glicosilação) 108. Esta glicoproteína tem uma estrutura secundária complexa, as regiões variáveis V1 a V4 formam estruturas em loop, estabilizadas por pontes dissulfureto. Para além destras regiões é ainda formada por dois domínios, um interno e outro externo, e uma β-sheet (follha-beta), encontrando-se as regiões V1/V2 e V3 expostas à superfície 70,143.
A glicoproteína TM do HIV possui uma região extracelular, uma região transmembranar (inserida na membrana citoplasmática da célula hospedeira) e uma região intracitoplasmática 136,143. Na região extracelular existem domínios comuns a outras proteínas de fusão: uma zona hidrofóbica rica em glicinas na extremidade N-terminal (designada péptido de fusão) e motivos leucine-zipper, também denominados
heptadrepeat (HR1 e HR2) 139.O péptido de fusão, localizado imediatamente após o local de clivagem da gp140, é fundamental para a fusão com a membrana citoplasmática da célula alvo. Os motivos HR apresentam padrões repetidos de sete aminoácidos,
sendo o primeiro e o quarto resíduos h
motivos associam-se formando uma estrutura termoestável super
sêxtupla, constituída por trímeros de HR1 e HR2. Esta estrutura está directamente envolvida na fusão com a célula hospedeira
O processo de entrada do HIV nas células inicia glicoproteína SU ao receptor CD4 (Figura 1.5)
Figura1.5 – Interacção entre as glicoproteína do invólucro e o receptor e co
(Adaptado de
A interacção entre a SU e o CD4 provoca alterações na conformação desta glicoproteína, nomeadamente nas regiões V1/V2 e V3, que levam à maior exposição do local de ligação ao co-receptor. Estas alterações conformacionais conduzem a uma
gp36
gp105
Fusão Ligação ao
CD4
sendo o primeiro e o quarto resíduos hidrofóbicos, sobretudo leucinas. Estes dois se formando uma estrutura termoestável super-enrolada em hélice sêxtupla, constituída por trímeros de HR1 e HR2. Esta estrutura está directamente envolvida na fusão com a célula hospedeira 15,136.
O processo de entrada do HIV nas células inicia-se geralmente com a ligação da glicoproteína SU ao receptor CD4 (Figura 1.5) 136,141.
Interacção entre as glicoproteína do invólucro e o receptor e co-receptor celulares (Adaptado de: www.roche.com/pages/facets/16/hiv_3_e.jpg.com).
A interacção entre a SU e o CD4 provoca alterações na conformação desta glicoproteína, nomeadamente nas regiões V1/V2 e V3, que levam à maior exposição do receptor. Estas alterações conformacionais conduzem a uma
Célula Interacção com Co-receptor gp36 Interacção HR1 18 idrofóbicos, sobretudo leucinas. Estes dois enrolada em hélice sêxtupla, constituída por trímeros de HR1 e HR2. Esta estrutura está directamente
se geralmente com a ligação da
receptor celulares
).
A interacção entre a SU e o CD4 provoca alterações na conformação desta glicoproteína, nomeadamente nas regiões V1/V2 e V3, que levam à maior exposição do receptor. Estas alterações conformacionais conduzem a uma
Interacção HR1-HR2
Ancoragem
19 aproximação entre a SU e a membrana celular, facilitando a ligação ao co-receptor
136,141
. Esta ligação, por sua vez, induz modificações na glicoproteína TM: as regiões HR1 e HR2 ficam mais expostas e associam-se, formando uma estrutura constituída por seis hélices alfa emparelhadas. Este rearranjo traduz-se na retracção da glicoproteína TM com exposição do péptido da fusão. O péptido de fusão insere-se na membrana citoplasmática da célula alvo originando a fusão entre o invólucro viral e a membrana celular, e a consequente entrada do vírus para o citoplasma da célula hospedeira 136.
Durante o processo de fusão, existem alguns aspectos que distinguem o 2 do HIV-1, nomeadamente na ligação ao CD4 e ao co-receptor 40,121. No VHI-1, a ligação da glicoproteína SU ao CD4 induz alterações conformacionais das regiões V1/V2 e V3. Esta interacção aumenta a acessibilidade da região V3 para posterior ligação aos co-receptores 108,121. Sourial e colaboradores propuseram que a região V3 do HIV-2 está totalmente acessível, podendo não ser necessária a ligação ao CD4 para induzir a alteração conformacional no loop V1/V2 durante a fusão 121. Por outro lado, Gallo e colaboradores, através de estudos de cinética da fusão, concluíram que a alteração conformacional da glicoproteína SU que permite a ligação ao co-receptor é mais rápida no HIV-2 do que no HIV-1 40.
Estas diferenças observadas entre as glicoproteínas SU dos dois vírus podem justificar o uso alargado de co-receptores alternativos, a entrada nas células independentes do CD4 e a maior sensibilidade aos anticorpos neutralizantes por parte do HIV-2 4,121.
20
1.6.2 . Evolução molecular durante a infecção
O estudo da evolução molecular permite descrever detalhadamente a taxa a que ocorrem alterações nas sequências de ADN e a forma como essas sequências evoluem 71,90. Os modelos de evolução aceites actualmente dividem-se em duas categorias: os métodos baseados na genética populacional e os métodos baseados na análise filogenética. A genética populacional analisa a frequência e distribuição das alterações nucleotídicas num conjunto de sequências, e a forma como estas alterações contribuem para a evolução. A análise filogenética estuda o padrão de alterações nucleotídicas ao longo do tempo, geralmente a partir de um ancestral. Embora distintos, os modelos de evolução encontram-se interligados e a informação gerada por ambos pode complementar-se 71.
As variações no tamanho das regiões variáveis do gene env foram associadas à progressão clínica e imunológica dos doentes infectados com HIV 80,115. Alguns estudos sugerem que o aumento do tamanho das regiões V1 e V2 está associado a progressão mais lenta para a doença, tanto no VHI-1 como no HIV-2 80,115. Masciotra e colaboradores observaram que ao longo da infecção HIV-1 em progressores rápidos ocorreu diminuição da região V1 sem alteração da V2, ao passo que nos progressores lentos detectaram o aumento de ambas as regiões 80. Na infecção pelo HIV-2 , Shi e colaboradores descreveram que não existiam alterações do tamanho nas regiões V1 e V2 nos progressores rápidos, e que o aumento de ambas as regiões estava associado a uma progressão mais lenta para SIDA 115.
Alguns autores concluíram que a compactação da região V1-V4 da gp120 do HIV-1 implica uma maior exposição do local de ligação da glicoproteína SU ao co-receptor,
21 que se traduz numa maior sensibilidade viral à neutralização 29,35,65. Geralmente esta compactação está associada a uma menor glicosilação da gp120, o que contribui igualmente para a maior sensibilidade à acção dos anticorpos neutralizantes 29,35,65 .Os locais de glicosilação em N (NXT/S) permitem a ligação de hidratos de carbono ao invólucro do HIV, o que poderá proteger os epítopos neutralizantes da exposição ao sistema imunitário12,60,101,102. Além disso, a glicosilação confere estabilidade e é fundamental para a conformação terciária do invólucro, sendo também importante na utilização de co-receptores 12,22,60,101,102,115.
1.7. Aspectos biológicos e moleculares relacionados com a utilização
de co-receptores
Um dos principais factores que condicionam o fitness viral é a entrada na célula 65. Neste contexto, os receptores celulares usados são os principais determinantes da patogénese e tropismo celular 19.
1.7.1. O CD4 como receptor primário do HIV
Um ano depois da identificação do HIV-1 como agente etiológico da SIDA foi conhecida a susceptibilidade dos linfócitos T auxiliares CD4+ à infecção; esta susceptibilidade teve como base a descoberta do CD4 como o receptor com maior afinidade para a gp120 134. Este receptor encontra-se presente na membrana citoplasmática dos linfócitos T, que têm funções auxiliadoras ou indutivas 92.
22 De facto, o papel crucial dos linfócitos T CD4+ numa vasta gama de funções do sistema imunitário, parecia ser suficiente para explicar os efeitos clínicos da infecção pelo HIV-1, no entanto, verificou-se uma discrepância entre o número de linfócitos T CD4+ infectados detectados no sangue e a extensão da disfunção imune 124. Outra inconsistência foi o facto da transfecção de linhas celulares com o gene CD4 humano não as tornar susceptíveis á infecção por HIV, o que implica que a presença deste receptor não é o único factor determinante da infecção 8,92124.
Uma década mais tarde foi identificado um segundo grupo de receptores- os receptores das quimiocinas 124.
1.7.2 . Receptores das quimiocinas como co-receptores do HIV
Os receptores das quimiocinas consistem em receptores da membrana celular ao qual se ligam as quimiocinas, uma família de citocinas que funciona como mediadores da quimiotaxia (um grupo de proteínas imuno-regulatórias) 8,76,124. A maioria das quimiocinas humanas conhecidas está classificada em quimiocinas CXC e quimiocinas CC com base na posição dos resíduos conservados de cisteína nos seus domínios N-terminal. As quimiocinas mostram tipicamente um elevado grau de semelhança estrutural umas com as outras, possuindo motivos conservados de quatro resíduos de cisteínas, que tornam mais fácil a identificação dos numerosos membros desta família, podendo partilhar os receptores a que se ligam. As quimiocinas, especialmente as quimiocinas CC, podem interagir com múltiplos receptores de quimiocinas e estas interações cruzadas são estritamente limitadas dentro das sub-famílias CC ou CXC 132.
23 O primeiro estudo a sugerir o possível envolvimento das quimiocinas e dos seus receptores na patogénese do HIV foi feito por Walker e seus colaboradores no final dos anos 80. Estes autores demonstraram que a infecção pelo HIV era inibida por factores solúveis, produzidos pelos linfócitos T CD8+ 131. Posteriormente as β-quimiocinas RANTES, MIP-1α e MIP-1β foram identificadas como parte destes factores solúveis 23
. Em alguns indivíduos infectados pelo HIV a presença destas moléculas, em elevadas concentrações no plasma, foi considerada responsável pela não progressão para a fase sintomática da doença, bem como pela protecção relativa à infecção em indivíduos não infectados e frequentemente expostos 76.
Actualmente conhecem-se 23 receptores de quimiocinas, ou moléculas estruturalmente relacionadas, que funcionam como receptores para a entrada do HIV-1, HIV-2 e SIV in
vitro (CCR1, CCR2b, CCR3, CCR4, CCR5, CCR6, CCR7, CCR8, CCR9B, CCR10,
CCR11, CXCR1, CXCR2, CXCR3, CXCR4, CXCR5, CXCR6, CX3CR1, XCR1, D6, e DARC, FPRL1, GPR1 4,5,116.
1.7.3. Factores determinantes para a utilização dos co-receptores do
HIV
Os co-receptores estão topologicamente organizados numa região N-terminal, sete segmentos transmembranares, três loops extracelulares, três loops intracelulares e uma região C-terminal. As regiões extracelulares dos co-receptores têm sido o alvo de maior estudo, uma vez que está estabelecido que a proteína Env efectua o contacto directo
24 inicial com estas regiões. No entanto, as regiões transmembranares e/ou citoplasmáticas dos co-receptores também influenciam a sua actividade, possivelmente afectando o funcionamento das regiões extracelulares 8.
1.7.4. Tropismo e uso de co-receptores
O tropismo é um termo utilizado para descrever a “especificidade” dos vírus relativamente à sua capacidade para infectar diversos tipos de células. Esta “especificidade”, e portanto a capacidade viral para infectar um tipo celular e não outro, reside na codificação do gene env do HIV para a glicoproteína de superfície do invólucro viral. Estirpes diferentes de HIV e SIV revelam tropismo para diferentes tipos celulares 105.Esta característica do HIV é igualmente determinada pelos parâmetros de expressão dos co-receptores apropriados, nomeadamente CCR5 e/ou CXCR4 e tem profundas implicações na transmissão e patogénese viral 8,76. Os estudos relativos á progressão da doença, efectuados tanto em primatas humanos como não humanos, têm revelado que o tropismo dos isolados virais para CXCR4 ou CCR5 define relativamente bem os alvos celulares do HIV in vivo 124.
O tropismo do HIV-1, HIV-2 e SIV para os co-receptores CCR5 e/ou CXCR4 está
correlacionado com a capacidade de replicação dos vírus nas diferentes linhas celulares
9,66,118
.Assim, vírus que infectam preferencialmente células da linhagem macrofágica, sem capacidade de indução de sincícios (células gigantes multinucleadas) e com taxa de replicação lenta/baixa, utilizam tipicamente o co-receptor CCR5 e são designados vírus
25 R5 9,66,118. Por outro lado, vírus que infectam principalmente linhas celulares linfocíticas, com capacidade de indução de sincícios e com taxa de replicação rápida/alta, denominam-se vírus X4 e usam o co-receptor CXCR4. Por último, vírus com capacidade de replicação idêntica em ambas as linhas celulares linfocíticas e macrofágicas, utilizam indiferenciadamente o CCR5 e o CXCR4, designando-se vírus R5X4 9,118,123.
O padrão bem definido de utilização de co-receptores para o HIV-1 não se observa no HIV-2. De facto, uma das características mais marcantes das estirpes de HIV-2 é a enorme variabilidade no modo como os vírus interagem com os receptores das quimiocinas 4,5.
Tal como o HIV-1, a maioria das estirpesde HIV-2 usa o CCR5 e/ou o CXCR4 como principais co-receptores para a entrada nos linfócitos T CD4+. No entanto, muitas estirpes de HIV-2 conseguem utilizar uma gama mais alargada de co-receptores comparativamente ao HIV-1 82 em alguns casos de uma forma tão eficaz como usam o CCR5 ou o CXCR4 5,4,10,95. Estes dados sugerem que a estrutura oligomérica das glicoproteínas do invólucro do HIV-2 seja mais flexível do que a do HIV-1 5,4,10,19,95. Por outro lado, a capacidade das estirpes de HIV-2 para interagir e usar eficazmente os diferentes receptores das quimiocinas como factores de entrada viral, pode levar à infecção inapropriada de células não-activadas ou não-permissivas, nas quais o vírus é incapaz de completar o seu ciclo replicativo devido a bloqueios intracelulares 4.
A promiscuidade no uso de co-receptores parece não estar relacionada com a patogenicidade do HIV-2, e a relevância do uso de co-receptores alternativos in vivo não está esclarecida 4,10,86,108.
26 Algumas estirpes de HIV-2 e SIV, contrariamente ao HIV-1, são capazes de infectar células mononucleadas do sangue periférico (CMSP) independentemente do CCR5 ou do CXCR4, o que implica que alguns co-receptores alternativos possam ser importantes na infecção 108,148.
Na infecção pelo HIV-1, os vírus R5 são responsáveis pelas novas infecções e predominam na fase assintomática da doença 12,32,123.Em cerca de 50% dos doentes, os vírus evoluem para X4, seguindo-se um rápido declínio dos linfócitos T CD4 e progressão para SIDA 66.
Na infecção pelo HIV-2, os vírus R5 são isolados de indivíduos assintomáticos ou no inicio da doença, e os vírus X4 são apenas encontrados em indivíduos em estado avançado de SIDA 4,20 Contudo, a evolução de vírus R5 para X4 ao longo da progressão da doença não é evidente, uma vez que muitos isolados primários utilizam vários co-receptores incluindo o CCR5 e o CXCR4independentemente do seu estado clínico 4.
No HIV-1, os aminoácidos (aa) determinantes para a utilização dos co-receptores encontram-se na extremidade C-terminal da região V3 da gp120 25,142. O aumento da carga nesta região, por alteração básica de um ou dois aminoácidos na posição 11 e/ou 25, é habitualmente responsável pela alteração do fenótipo R5 para X4 25,142.No HIV-2, a utilização dos co-receptores CCR5 e/ou CXCR4 poderá estar igualmente ligada à carga da região V3 1,60,115.Isaka e colaboradores, através de estudos in vitro, concluíram que a presença de pelo menos um resíduo básico na posição 18 ou 27 desta região permite prever o tropismo viral 60. SegundoShi e colaboradores, na região V3, os aa que
27 se encontram nas posições 19 e 27 115 são determinantes para a utilização de co-receptores.
Apesar de existirem poucas estirpes HIV-2 geneticamente caracterizadas, é de referir que, ao contrário do observado para o HIV-1, a região V3 do HIV-2 é altamente conservada 2,4,60. Para explicar esta quase ausência de variabilidade foi colocada como hipótese, a ocorrência de uma menor resposta imunitária anti-V3 2,4,60 Por outro lado, a composição aminoacídica da região V1 e V2, entre várias estirpes de HIV-2, é altamente variável. Perante esta observação, sugeriu-se que a pressão selectiva exercida pelo sistema imunitário, seria direccionada para as regiões V1 e V2. No seu conjunto, estas observações parecem indicar que outras regiões variáveis, para além da V3, poderão influenciar a ligação ao co-receptor. Em alternativa a esta hipótese, uma conformação mais relaxada das glicoproteínas do invólucro do HIV-2 poderá ser um factor determinante na utilização indiferenciada dos co-receptores 2,4,60.
A utilização de co-receptores depende também do grau de glicosilação da região V3
22,101,102,115
.Na infecção pelo HIV-1, a ausência de um local de glicosilação na região V3, associada a uma forte carga positiva, tem um papel muito importante na alteração dos vírus R5 para X4 22,101,102. A falta do local de glicosilação a seguir à primeira cisteína é importante para um uso mais eficiente de CXCR4 102. O grau de glicosilação do HIV-2 é inferior ao do HIV-1 e ainda não está estabelecida a sua influência no uso de co-receptores 115.
2
29
2. Objectivos
Este trabalho tem por objectivo a caracterização do gene env de estirpes primárias de HIV-2, isoladas a partir de doentes em fases distintas da história natural da infecção pelo HIV-2 e, em particular, com perfis diferentes na gama de co-receptores usados para a entrada do vírus na célula. Pretende-se correlacionar as sequências desse gene com o perfil fenotípico apresentado pelas estirpes, de forma a estabelecer um padrão genotípico subjacente à utilização dos diferentes co-receptores.
Os estudos que têm sido feitos relativos aos determinantes genénicos subjacentes á utilização dos diferentes co-receptores, tem sido quase exclusivamente feitos usando o HIV-1 como modelo. No entanto alguns dados sugerem que estes determinantes poderão ser diferentes no HIV-2 11,34,36,60,62,83,97,115. Para além disso, a maior parte dos estudos feitos no HIV-2, usou um número limitado de amostras e/ou utilizou estirpes adaptadas ao crescimento em linhas celulares. Em qualquer dos casos, a relevância e o significado prático dessas observações, é obviamente limitado, daí a importância deste trabalho.
3
31
3. Materiais e Métodos
3.1. Amostra
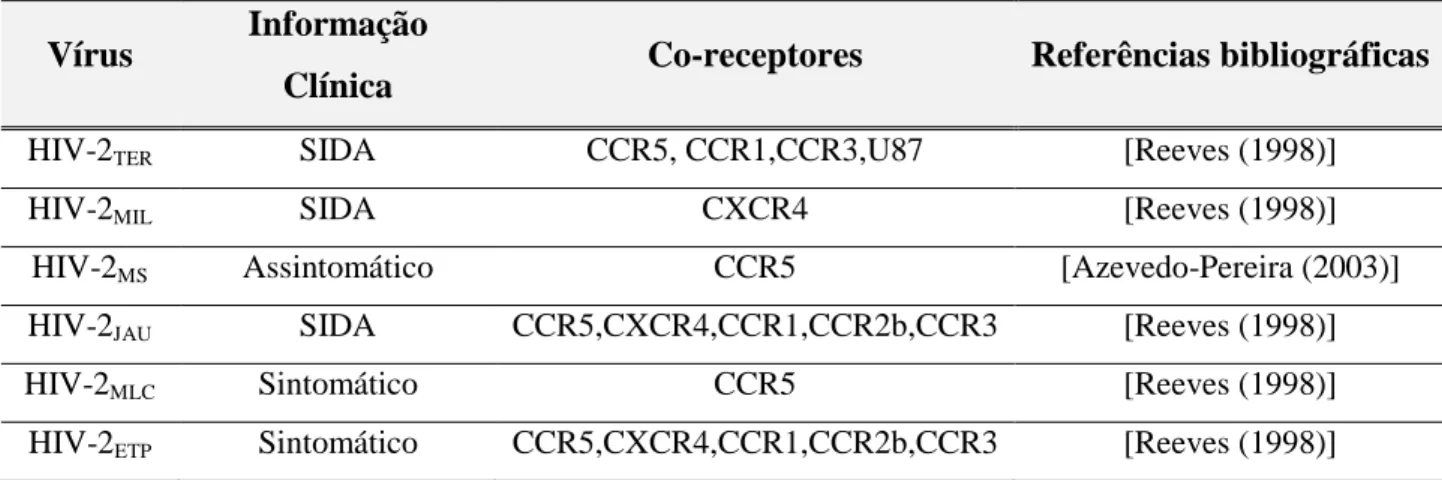
Neste estudo foi analisado um conjunto de 6 vírus, HIV-2TER, HIV-2MIL, HIV-2MS,
HIV-2JAU, HIV-2MLC e HIV-2ETP, em fases distintas da história natural da infecção pelo
HIV-2 e, com diferentes perfiz de utilização dos co-receptores (Tabela 3.1).
As estirpes primárias de HIV-2 utilizadas neste trabalho foram obtidas a partir das CMSP dos indivíduos infectados por co-cultura com CMSP de dadores não infectados pelo VIH.
Tabela 3.1 – Informação clínica e co-receptores dos vírus utilizados neste trabalho.
Vírus Informação
Clínica Co-receptores Referências bibliográficas
HIV-2TER SIDA CCR5, CCR1,CCR3,U87 [Reeves (1998)]
HIV-2MIL SIDA CXCR4 [Reeves (1998)]
HIV-2MS Assintomático CCR5 [Azevedo-Pereira (2003)]
HIV-2JAU SIDA CCR5,CXCR4,CCR1,CCR2b,CCR3 [Reeves (1998)]
HIV-2MLC Sintomático CCR5 [Reeves (1998)]
HIV-2ETP Sintomático CCR5,CXCR4,CCR1,CCR2b,CCR3 [Reeves (1998)]
3.1.1. Preparação das Amostras
Inicialmente utilizou-se o método da Spinoculation 156 para a produção de stock viral. O processamento das amostras foi realizado de forma a extrair o ADN genómico celular. Para a extracção do ADN genómico utilizou-se 10-20 milhões de CMSP. A extracção do ADN foi efectuada com o kit comercial DNA Isolation Kit Puregene
32 (Puregene, Gentra Systems), com algumas alterações às recomendações do fabricante. Resumidamente, fez-se uma centrifugação a 500 xg durante 3 minutos. Ao centrifugado resultante foi adicionado 3 ml de solução de lise. Seguindo-se uma incubação a 37ºC durante 40 minutos. Após este período de incubação, transferiu-se a amostra para o gelo durante 3 minutos e adiciona-se 1 ml de solução de precipitação de proteínas, centrifugou-se a amostra durante 10 minutos a 2000 xg. Ao sobrenadante resultante contendo o ADN adicionou-se 3 ml de isopropanol a 100% e centrifugou-se durante 3 minutos a 2000 xg. Para eliminar restos de isopropanol, seguiu-se uma lavagem com etanol a 70% (3 ml), o qual foi removido após centrifugação durante 3 minutos a 2000 xg. Deixou-se os tubos a secar à temperatura ambiente durante 10 minutos. Após os 10 minutos, procedeu-se à hidratação do ADN com 250 µl de solução de hidratação e incubou-se durante 1 hora a 65ºC. Por fim colocou-se a amostra com o ADN genómico extraído, a 4ºC.
3.2. Oligonucleótidos Sintéticos iniciadores – Primers
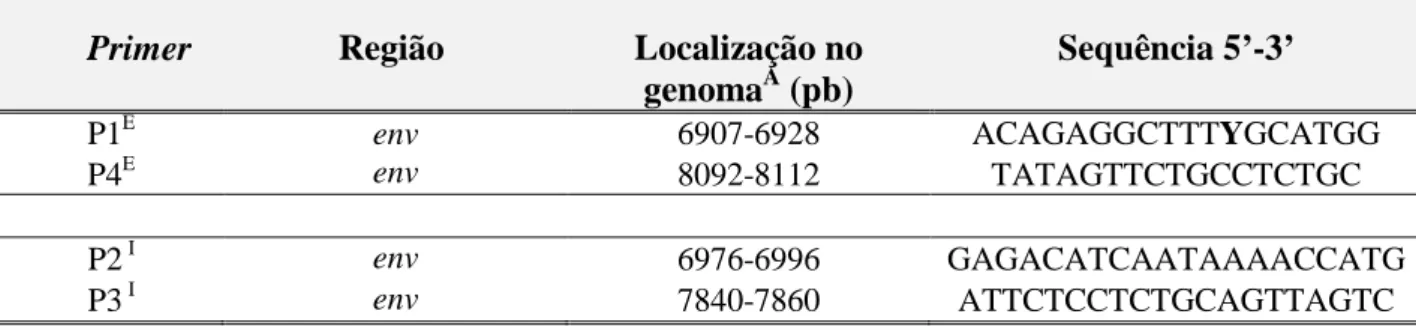
O delineamento dos primers foi efectuado a partir das regiões mais conservadas do gene
env, os primers foram desenhados a partir da análise de alinhamentos de multiplicas
sequências nucleotídicas de estirpes de referência dos diferentes grupos do HIV-2 53. Os
primers foram desenhados de forma a contemplar o final da região C1 e o início da C3,
uma vez que se pretendia analisar as regiões V1,V2, V3.
O programa OligoTech foi utilizado para optimizar os primers desenhados (este permite verificar a formação de estruturas secundárias e emparelhamento dos primers entre si) e para determinar a temperatura óptima de hibridação 58. Um dos primers apresenta