Um sensor de peróxido de hidrogénio numa bacteria anaeróbia: a PerR de Desulfovobrio vulgaris vulgaris Hildenborough
Texto
(2) nº de arquivo “Copyright”. ii.
(3) UNIVERSIDADE NOVA DE LISBOA FACULDADE DE CIÊNCIAS E TECNOLOGIA DEPARTAMENTO DE QUÍMICA . . Sara Cristina Matias . Um Sensor de Peróxido de Hidrogénio numa Bactéria anaeróbia: A PerR de Desulfovibrio vulgaris vulgaris Hildenborough Dissertação apresentada para a obtenção do Grau de Mestre em Bioquímica Estrutural e Funcional, pela Universidade Nova de Lisboa, Faculdade de Ciências e Tecnologia Orientador: Doutora Isabel Cristina Timóteo (FCT/UNL) Co‐orientadora: Profª. Doutora Maria Alice Pereira (FCT/UNL) LISBOA 2010 . iii.
(4) iv.
(5) Agradecimentos Antes de mais gostaria de dedicar todo o meu trabalho bem como esta tese, ao meu Pai. Pois sempre me apoiou bastante e nunca me deixou desistir de nada e sempre teve um papel muito importante na minha vida. Mas, infelizmente não me pode ver a concluir esta etapa, e é a ele que eu dedico todo o meu trabalho. Não posso deixar de agradecer também à minha mãe e ao meu irmão que sempre me apoiaram e sempre estiveram disponíveis para me dar uma palavra de incentivo para que eu progredisse. A minha família é o meu pilar, e sem eles nunca teria alcançado o que alcancei. Às minhas orientadoras, Doutora Cristina Timóteo e Professora Doutora Alice Pereira, um muito obrigada pela disponibilidade ao longo de todo o meu trabalho, quer experimental quer ao longo da escrita desta tese. Agradeço‐lhes ainda a paciência que tiveram comigo ao longo de todo este processo. Ao Professor Doutor Pedro Tavares, pois sem a sua ajuda nem sequer teria sido possível desenvolver esta tese nesta instituição. Aos meus amigos, Joana, Renato e Susana, agradeço o apoio incondicional e a ajuda preciosa para me animar e aliviar o stress quando os resultados do trabalho eram menos favoráveis. À Mafalda que me apoiou durante a escrita da tese enquanto fazia longas noites de escrita e ela de estudo, e à sua mãe que sempre me deu força para continuar. Ao Américo, pela sua ajuda, e pela sua sempre boa disposição. Aos meus companheiros de laboratório, Filipe Folgosa, Cristina Cordas e Márcia, que sempre se mostraram disponíveis para me ajudar bem como para me explicar técnicas com as quais não estava familiarizada. Agradeço‐lhes ainda o bom ambiente de trabalho que me proporcionaram. À Margarida, pelas longas noites de trabalho que atravessei no laboratório, e ela sempre presente para me ajudar a passar o tempo mais depressa. Gostaria ainda de agradecer ao Aldino e ao Rui Almeida pela sua ajuda no RMN. . Ao meu Pai . v.
(6) . vi.
(7) Resumo As espécies reactivas derivadas de oxigénio (ROS), apesar de desempenharem um papel importante nos processos de oxidação e sinalização nos organismos superiores, apresentam um elevado grau de toxicidade. Em Bacillus subtillis a resposta adaptativa a H2O2 está sob controlo de uma metaloproteína denominada Repressor do regulão do peróxido (PerR), membro da super família do Repressor de absorção de Ferro (Fur). A PerR é um homodímero que liga 2 iões metálicos por subunidade: um Zn2+ coordenado por 4 cisteínas num centro com função estrutural, e um Fe2+ ou Mn2+coordenados por 3 histidinas (His) e 2 aspartatos (Asp), no que se pensa ser o centro regulatório, necessário para a função de sensor de H2O2. O objectivo deste trabalho foi a clonagem, sobre‐expressão e caracterização bioquímica básica da PerR de Desulfovibrio (D.) vulgaris vulgaris Hildenborough. O gene perR foi clonado em dois vectores de expressão diferentes tendo‐se conseguido sobre‐expressar a proteína, no entanto, nunca numa forma solúvel. Foi necessário recorrer a um protocolo de solubilização de proteínas a partir de corpos de inclusão tendo‐se obtido a PerR recombinante (rPerR) de D. vulgaris vulgaris Hildenborough parcialmente estruturada, quando reconstituída com Fe2+ e Zn2+, e parte sem estrutura conforme foi possível verificar por RMN. Procedeu‐se à caracterização bioquímica básica da proteína por espectroscopia de UV‐vísivel e determinação da quantidade total de proteína na amostra. Os ensaios de ligação ao pADN permitiram concluir que a proteína após reconstituição com Fe2+ e Zn2+ consegue ligar‐se ao promotor do gene ahpC. . . . vii.
(8) . viii. .
(9) Abreviaturas Abs ADN Amp ARN Asp ATCC B. bp BCA BSA C‐terminal Cys D. Da d’NTP D.O.600 nm DTT E. EDTA Glu His HSQC IPTG Kan mAU N‐terminal PCR PMSF RMN SDS SDS‐PAGE TEMED Tm Tris UV UV‐vis X‐gal . Absorvância Ácido desoxirribonucleico Ampicilina Ácido ribonucleico Aspartato American Type Culture Collection Bacillus Pares de bases Ácido bicinconínico Albumina do soro de bovino Zona carboxilo terminal da cadeia polipeptídica Cisteína Desulfovibrio Dalton Nucleótidos Densidade Óptica a 600 nm Ditiotreitol Escherichia Ácido etilenodiamino tetracético Glutamato Histidina Correlação heteronuclear de quantum simples Isopropil‐β‐D‐1‐tiogalactósido Canamicina Miliunidades de absorvância Zona amino terminal da cadeia polipeptídica Reacção em cadeia da polimerase Fluoreto de fenilmetilsulfonilo Ressônancia Magnética Nuclear Dodecil‐sulfato de sódio Electroforese em gel de poliacrilamida em condições desnaturantes N,N,N’,N’‐tetrametiletilenodiamina Temperatua de fusão Tris‐hidroxi‐metil‐aminometano Ultravioleta Ultravioleta‐vísivel 5‐bromo‐4‐cloro‐3‐indol‐β‐D‐galactopiranosido . . ix.
(10) . x.
(11) Índice Índice de Figuras .................................................................................................................. xv Índice de Tabelas ................................................................................................................. xix Capítulo I Introdução ............................................................................................................................ 1 Stress Oxidativo .................................................................................................................... 3 A Super‐Família Fur ............................................................................................................... 5 A PerR de Bacillus subtilis ...................................................................................................... 7 Estrutura .................................................................................................................................... 8 A ligação ao ADN ..................................................................................................................... 11 Bibliografia .......................................................................................................................... 17 Capítulo II Clonagem da PerR de Desulfovibrio vulgaris vulgaris Hildenborough no vector pET21c e Sobre‐expressão em Escherichia coli .................................................................................... 19 II.1. Desenho de oligonucleótidos iniciadores ........................................................................ 21 II.2. Sub‐clonagem no vector pNZY28® .................................................................................... 25 II.3. Clonagem no vector de expressão pET21c....................................................................... 33 II.4. Sobre‐expressão de PerR ................................................................................................. 42 Bibliografia .......................................................................................................................... 53 Capítulo III Clonagem da PerR de Desulfovibrio vulgaris vulgaris Hildenborough no vector pET30a e Sobre‐expressão em Escherichia coli .................................................................................... 55 III.1. Clonagem no vector de expressão pET30a ..................................................................... 57 III.2. Sobre‐expressão de PerR ................................................................................................ 62 Bibliografia .......................................................................................................................... 72 Capitulo IV Solubilização da PerR recombinante de Desulfovibrio vulgaris vulgaris Hildenborough a partir de Corpos de Inclusão .......................................................................................................... 73 IV.1. Teste com vários agentes de solubilização ..................................................................... 76 IV.2. Solubilização da rPerR de D. vulgaris vulgaris Hildenborough recorrendo a Ureia ........ 82 IV.3. Renaturação da proteína ................................................................................................ 84 Bibliografia .......................................................................................................................... 87 . xi .
(12) Capitulo V ............................................................................................................................ 89 Purificação de PerR recombinante de Desulfovibrio vulgaris vulgaris Hildenborough solubilizada ......................................................................................................................... 89 Purificação da PerR recombinante de Desulfovibrio vulgaris vulgaris Hildenborough .......... 91 Bibliografia .......................................................................................................................... 95 Capitulo VI ........................................................................................................................... 97 Caracterização Bioquímica e Espectroscópica da PerR recombinante de Desulfovibrio vulgaris vulgaris Hildenborough ....................................................................................................... 97 VI.1. Espectroscopia de UV‐visível .......................................................................................... 99 VI.3. Determinação da quantidade total de Proteína ........................................................... 100 VI.4. Verificação da estrutura terciária da PerR por RMN .................................................... 102 VI.5 Obtenção de um modelo estrutural por homologia ...................................................... 108 Bibliografia ........................................................................................................................ 113 Capitulo VII ........................................................................................................................ 115 Ensaios de ligação do repressor, PerR, ao ADN .................................................................. 115 VII.1. Construção de um plasmídeo contendo o promotor do gene ahpC ........................... 117 VII.2. Teste de hidrólise do vector p_SM_ahpc_pMA com a enzima de restrição TfiI ......... 120 VII.3. Ensaio de ligação da PerR ao vector p_SM_ahpc_pMA............................................... 122 Bibliografia ........................................................................................................................ 125 Capitulo VIII ....................................................................................................................... 127 Conclusão .......................................................................................................................... 127 Apêndice ........................................................................................................................... 131 A.Meios de Cultura e Condições de Crescimento ..................................................................... 133 A.1. Antibióticos .................................................................................................................... 133 A.2. Meios de cultura ............................................................................................................ 133 B.Protocolos de Biologia Molecular .......................................................................................... 136 B.1. Obtenção de ADN Genómico ......................................................................................... 136 B.2. Reacções de PCR ............................................................................................................ 136 B.3. Electroforese em gel de agarose .................................................................................... 137 B.4. Purificação de fragmento de ADN através de sistema de electroforese E‐gel® ............. 137 B.5. Quantificação de ADN .................................................................................................... 138 B.6. Ligação do fragmento, perr, ao vector pNZY28® e vectores de expressão ..................... 140 B.7. Selecção de colónias ...................................................................................................... 142 B.8. Transformação em células competentes ....................................................................... 143 B.9. Isolamento de ADN plasmídico ...................................................................................... 144 xii .
(13) B.10. Mapas de vectores utilizados ....................................................................................... 146 C.Solubilização e Renaturação da rPerR ................................................................................... 149 C.1. Solubilização com Benzonase® e DNAse I ...................................................................... 149 C.2. Solubilização com agentes redutores e desnaturantes ................................................. 149 C.3. Solubilização com Deoxicolato de sódio e Lauril sarcosinato de sódio ......................... 150 C.4. Solubilização com Ureia e renaturação .......................................................................... 151 D.Electroforese em gel de Poliacrilamida e Coloração de géis ................................................. 151 D.1. Electroforese em Gel de Poliacrilamida em Condições Desnaturantes (SDS‐PAGE) ..... 151 D.2. Coloração ....................................................................................................................... 153 E.Caracterização Bioquímica ..................................................................................................... 155 E.1. Determinação da quantidade total de Proteína. Método do BCA ................................. 155 E.2. Espectroscopia, NMR ..................................................................................................... 157 F.Ensaios de ligação ao pADN ................................................................................................... 158 F.1. Teste à enzima Tfi I ......................................................................................................... 158 F.2. Ensaio de ligação ............................................................................................................ 159 Bibliografia ........................................................................................................................ 160 . . xiii .
(14) . xiv . .
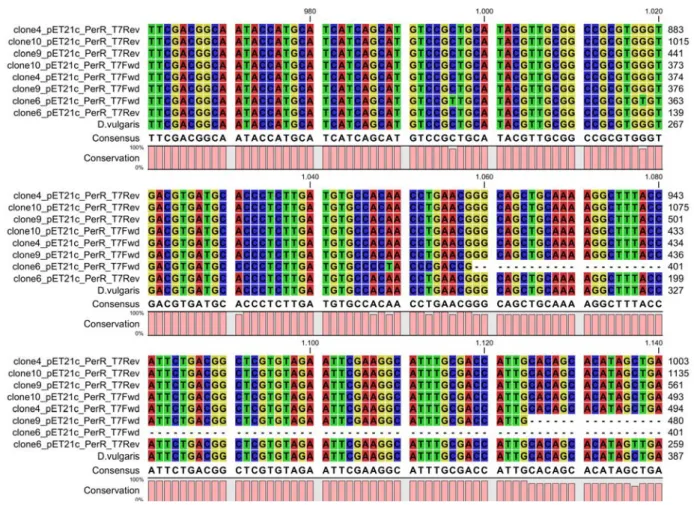
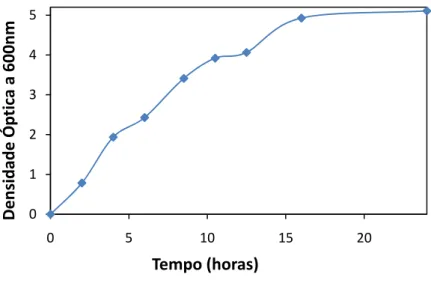
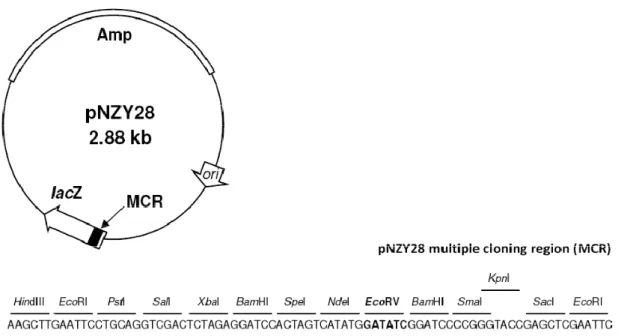
(15) Índice de Figuras Figura I.1 ‐ Representação da estrutura do monómero de Fur‐Zn2 de Pseudomonas aeruginosa (PDB id: IMZB pH 7) ............................................................................................................................................... 7 Figura I.2 – Representação da estrutura de PerR‐Zn de B. subtilis (PDB id: 3F8N). .............................. 9 Figura I.3 – Representação do local regulatório e de ligação ao manganês/ferro .............................. 10 Figura I.4 – Oxidação da PerR (Histidinas 37 ou 91) mediada por ferro adaptada (18, 19) ................ 10 Figura I.5 ‐ Representação da estrutura de PerR‐Zn‐ox de B. subtilis (PDB id: 2RGV). ....................... 11 Figura I.6 – Sequências de ADN de para os locais de reconhecimento das proteínas Fur, PerR e Zur de B. subtilis.(21).. ...................................................................................................................................... 12 Figura I.7 – Diferentes estados conformacionais da PerR de B.subtilis em resultado da adição de Fe2+ ou Mn2+ (círculos a verde) adaptada(15).. .................................................................................................. 13 Figura I.8 ‐ Comparação de sequências de aminoácidos da PerR de Bacillus subtilis e Desulfovibrio vulgaris Hildenborough. ............................................................................................................................. 14 Figura II.1 – Sequência de ADN codificante da PerR (387bp) de D.vulgaris, com a respectiva tradução para sequência de aminoácidos (128 aminoácidos) .................................................................................. 22 Figura II.2 – Comparação de sequências de aminoácidos da PerR de diferentes microorganismos. . 23 Figura II.3 – Mapa de restrição da sequência que codifica gene da PerR de D.vulgaris ..................... 25 Figura II.4 – Curva de Crescimento de D. vulgaris em meio Postgate ao longo de 53 horas .............. 26 Figura II.5 – Fotografias de microscópico da cultura de D. vulgaris vulgaris Hildenborough em diferentes fases do crescimento. ............................................................................................................... 27 Figura II.6 – Análise de PCR por electroforese de em gel de agarose. ................................................ 28 Figura II.7 – Desenho do vector de subclonagem, pNZY28 ® .............................................................. 30 Figura II.8 –Resultado da amplificação do gene de PerR de D. vulgaris de por PCR de colónia. ........ 30 Figura II.9 – Alinhamento das sequências de nucleótidos dos clones 2,3 e 8 no vector pNZY28® com a sequência de PerR de D. vulgaris ............................................................................................................ 32 Figura II.10 – Mapa do vector de expressão pET21‐a ......................................................................... 34 Figura II.11 – Análise da reacção de hidrólise do clone PerR_pNZY28_clone_2 e do vector pET21c com a enzima XhoI ..................................................................................................................................... 35 Figura II.12 – Análise da reacção de dupla hidrólise do clone PerR_pNZY28_clone_2 e do vector pET21c com as enzima NdeI e XhoI ............................................................................................................ 36 Figura II.13 – PCR das colónias resultantes da ligação do pET21c e do fragmento perr. .................... 38 Figura II.14 ‐ Alinhamento das sequências de nucleótidos dos clones 4,6,9 e 10 no vector pET21c com a sequência de PerR de D. vulgaris ..................................................................................................... 40 Figura II.15 – Resumo do processo de sub‐clonagem e clonagem realizado ...................................... 41 Figura II.16 – Curva de crescimento a 37 ºC de células E.coli BL21 (DE3) com o vector pET21c em meio 2xYT ................................................................................................................................................... 43 Figura II.17 – Curva de crescimento a 37 ºC de culturas de E.coli BL21 (DE3) em meio LB/Amp e 2xYT/Amp com diferentes concentrações de indutor, IPTG. ..................................................................... 44 Figura II.18 – Análise de sobre‐expressão de PerR em células E.coli BL21 (DE3) em meio LB e 2xYT . 44 . xv .
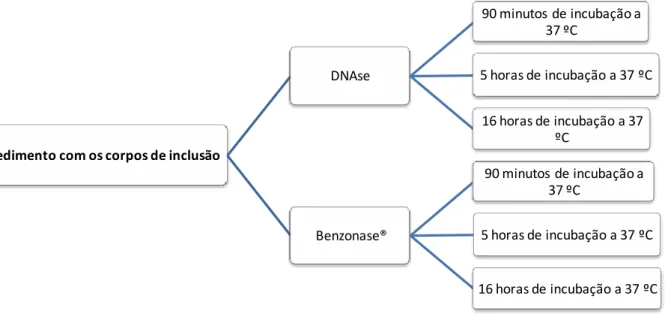
(16) Figura II.19 – (A) Fraccionamento celular(6). (B) Análise da sobre‐expressão da PerR de D. vulgaris em células E.coli BL21 (DE3) ....................................................................................................................... 46 Figura II.20 – Curvas de crescimento de E. coli BL21 (DE3) em meio M9 a 37 ºC: .............................. 48 Figura II.21 – Análise de sobre‐expressão de PerR em células E.coli BL21 (DE3) em meio M9 .......... 49 Figura II.22 – Análise de sobre‐expressão de PerR em células E.coli BL21 (DE3) em meio M9 e posterior fraccionamento ........................................................................................................................... 50 Figura III.1 – Mapa do vector de expressão pET30a ............................................................................ 57 Figura III.2 – Análise da reacção de hidrólise do clone PerR_pNZY28_clone_2 e do vector pET30a com a enzima XhoI ..................................................................................................................................... 58 Figura III.3 ‐ Análise da reacção de hidrólise do clone PerR_pNZY28_clone_2 e do vector pET21c com a enzima NdeI ............................................................................................................................................. 59 Figura III.4 ‐ Análise da PCR de colónia resultante da ligação do fragmento perr ao vector pET30a .. 60 Figura III.5 – Análise da reacção de hidrólise dos clones escolhidos para verificação da presença do fragmento correspondente ao gene perr. .................................................................................................. 60 Figura III.6 ‐ Alinhamento das sequências de nucleótidos dos clones 1, 2 e 5 no vector pNZY28® com a sequência de PerR de D.vulgaris ............................................................................................................. 61 Figura III.7 – Curva de crescimento de células E. coli BL21 (DE3), C41 (DE3) e C43 (DE3) com o vector pET30a em meio M9 .................................................................................................................................. 62 Figura III.8 – Curva de crescimento de culturas de E.coli BL21 (DE3), C41 (DE3) e C43 (DE3) em meio M9 com diferentes concentrações de indutor ........................................................................................... 63 Figura III.9 – Análise de sobre‐expressão de PerR em células E.coli BL21 (DE3), C43 (DE3) e C41 (DE3) em meio M9 a 37ºC induzidas a uma densidade óptica a 600 nm de 0.6. ................................................. 64 Figura III.10 – Análise de sobre‐expressão de PerR em células E.coli BL21 (DE3) em meio M9 com 0.1mM a 20ºC e 0.5mM de IPTG a 37ºC respectivamente e posterior fraccionamento ........................... 65 Figura III.11 – Curvas de crescimento de E.coli BL21 (DE3) em meio M9 com diferentes concentrações de agente indutor e momentos de indução (alta ou baixa densidade óptica, D.O 2 e 0.5 respectivamente) ....................................................................................................................................... 66 Figura III.12 – Análise de sobre‐expressão de PerR em células E.coli BL21 (DE3) em meio M9 a 5ºC induzidas a diferentes densidades ópticas a 600 nm. A ............................................................................. 67 Figura III.13 – Painel A – Análise da sobre expressão de PerR em células E.coli BL21 (DE3), E.coli C41 (DE3), E.coli C43(DE3) em meio M9 a baixa densidade óptica, crescimento de 6 dias (144horas) após indução ....................................................................................................................................................... 68 Figura III.13 ‐ Painel B – Curva de crescimento E.coli BL21 (DE3) e E.coli C43(DE3) e E.coli C41(DE3) em meio M9 com diferentes concentrações de agente indutor ................................................................ 68 Figura III.14 – Análise do fraccionamento celular de E.coli BL21 (DE3), E.coli C41 (DE3) e E.coli C43 (DE3) em meio M9 ..................................................................................................................................... 69 Figura IV.1 – Análise dos corpos de inclusão por electroforese em gel de agarose (0.8 %), tampão TAE 1 X, 100 V durante 50 minutos. ........................................................................................................... 77 Figura IV.2 – Planeamento dos testes de solubilização com diferentes agentes. ............................... 78 Figura IV.3 ‐ Géis de solubilização da proteína.. .................................................................................. 80 Figura IV.4 – Esquema representativo do protocolo de solubilização seguido para a rPerR de D. vulgaris. ...................................................................................................................................................... 81 . xvi .
(17) Figura IV.5 ‐ Gel de solubilização da proteína segundo o protocolo presente no apêndice C.3. ........ 82 Figura IV.6 – Esquema representativo do protocolo de solubilização ................................................ 83 Figura IV.7 – Análise da solubilização da proteína segundo o protocolo presente no apêndice C.4. . 83 Figura IV.8 – Esquema do processo inerente à solubilização e renaturação de proteínas. Figura adaptada (6) ............................................................................................................................................... 84 Figura IV.9 – Esquema do processo de renaturação adaptado (6). .................................................... 85 Figura IV.10 – Análise do processo de renaturação através de diálises sucessivas com redução da concentração de agente desnaturante e adição de endonuclease. ........................................................... 86 Figura V.1 – Perfil cromatográfico da purificação da rPerR de D. vulgaris Hildenborough sobre‐ expressa em E.coli BL21 (DE3), na coluna de afinidade (HiTrap ™ Heparin H.P. (GE Healthcare, 1.6x2.5 cm). ............................................................................................................................................................. 92 Figura V.2 – Análise das fracções obtidas aquando da purificação ..................................................... 93 (A) Gel corado com Azul de Coomassie. (B) Gel corado com Nitrato de prata. ....................... 93 Figura V.3 – Esquema da purificação da PerR de D.vulgaris Hildenborough ...................................... 94 Figura VI.1 – Espectro de UV‐visível da PerR após a purificação. ...................................................... 100 Figura VI.2 ‐ Recta de calibração para quantificação de proteína total pelo método do BCA .......... 101 Figura VI.3 – Espectro 1H RMN típico de uma proteína (A) estruturada, (B) desnaturada, adaptado(6) .................................................................................................................................................................. 103 Figura VI.4 ‐ Espectro 1H‐ 15N HSQC típico de uma proteína (A) estruturada, (B) desnaturada adaptado (6). ............................................................................................................................................ 104 Figura VI.5 – Espectro de 1H RMN da amostra de rPerR de D. vulgaris vulgaris Hildenbourgh obtida a partir de corpos de inclusão, incubada com Zn2+ ..................................................................................... 105 Figura VI.6 – Espectro de 1H RMN da amostra de rPerR de D. vulgaris vulgaris Hildenbourgh obtida a partir de corpos de inclusão e incubada com Zn2+ e Fe2+l. ....................................................................... 106 15. Figura VI.8 – Espectros da rPerR de D. vulgaris vulgaris Hildenbourgh marcada isotópicamente com N, incubada com Zn2+. ............................................................................................................................ 107 . Figura VI.9 – Representação por ordem crescente do score e decrescente do E‐value de uma pesquisa nas bases de dados disponíveis no SwissModel, com a sequência de aminoácidos da PerR de Desulfovibrio vulgaris vulgaris Hildenborough ......................................................................................... 108 Figura VI.10 ‐ Estrutura da PerR (A) de D.vulgaris, obtida através do programa Swiss Model, usando como molde a estrutura da PerR de B .subtilis (com Zn2+ e Mn2+) (PDBid: 3f8n) (B).. ............................. 110 Figura VI.11 ‐ Alinhamento múltiplo das sequências com maior homologia com a sequência da PerR de D.vulgaris ............................................................................................................................................. 111 Figura VII.1 – Possíveis sequências do motivo de ligação da PerR ao ADN(3) .................................. 118 Figura VII.2 – Sequência da região promotora e início da sequência codificante para o gene da ahpc de D. vulgaris vulgaris Hildenbourgh seleccionada para ser utilizada nos ensaios de ligação da PerR. .................................................................................................................................................................. 118 Figura VII.3 – Mapa de restrição para a sequência do protomor do gene ahpC que contem a PerR box evidenciada, mostrando as enzimas que hidrolisam a referida sequência de ADN. ......................... 119 Figura VII.4 – Mapa do vector pSM_ahpc_pMA construído para os ensaios de ligação com a PerR. .................................................................................................................................................................. 120 . xvii .
(18) Figura VII.5 – Verificação da reacção de hidrólise do plasmídeo p_SM_ahpc_pMA com a enzima de restrição TfiI .............................................................................................................................................. 121 Figura VII.6 – Verificação dos resultados do ensaio de ligação da PerR recombinante de D. vulgaris Hildenborough ao vector pSM_ahpc_pMA, na presença da enzima TfiI,. ............................................... 123 Figura B1 – Utilização do sistema E gel ® da Invitrogen .................................................................... 138 Figura B2 – Marcadores de pesos moleculares NZYTech® ................................................................ 139 Tabela B6 ‐ Reacção de ligação do fragmento contendo o gene perr aos vectores de expressão pET21c e pET30a ...................................................................................................................................... 141 Figura B3 – Resumo da selecção de clones positivos ........................................................................ 142 Figura D1 – Marcador de Pesos Moleculares para proteínas – LMW Low Molecular Weight (NZYTech) ................................................................................................................................................. 153 . . xviii . .
(19) Índice de Tabelas Tabela I.1 – Membros da super família Fur, tabela adaptada (9) ......................................................... 5 Tabela II.1 – Enzimas de restrição escolhidas e respectiva sequência de reconhecimento................ 25 Tabela II.2 – Oligonucleótidos iniciadores ........................................................................................... 25 Tabela II.3 – Resumo dos testes (erlenmeyer com 100mL de cultura) e crescimentos (erlenmeyer com 1L de cultura) feitos com o vector pET21c contendo o gene perr ..................................................... 52 Tabela III.1 – Resumo dos testes (erlenmeyer com 100mL de cultura) e crescimentos (erlenmeyer com 1L de cultura) feitos com o vector pET30a contendo o gene perr ..................................................... 71 Tabela VI.1 – Determinação da concentração de proteína total existente na amostra purificada, FI .................................................................................................................................................................. 101 Tabela VII.1 ‐ Enzima de restrição escolhida e respectiva sequência de reconhecimento. .............. 119 Tabela A1 – Concentração dos antibióticos utilizados na manutenção das estirpes e do agente utilizado na selecção das colónias ............................................................................................................ 133 Tabela A2 – Composição do meio de cultura Postgate ..................................................................... 133 Tabela A3 – Composição do meio de cultura Luria Broth (LB) líquido e sólido ................................. 134 Tabela A4 ‐ Composição do meio de cultura 2xYT ............................................................................ 134 Tabela A5 ‐ Composição do meio de cultura SOC ............................................................................. 134 Tabela A6 – Composição do meio de cultura definido M9 10x concentrado modificado e M9 10x concentrado modificado isotopicamente marcado com 15N ................................................................... 135 Tabela A7 – Preparação do meio de cultura definido M9 modificado .............................................. 135 Tabela A8 – Soluções a adicionar ao meio M9 aquando do crescimento bacteriano ....................... 136 Tabela B1 – Componentes utilizados nas reacções de PCR para a amplificação de perr de D. vulgaris vulgaris Hildenborough. ........................................................................................................................... 137 Tabela B2 – Componentes e volumes utilizados na reacção de fosforilação do fragmento com o gene perr ........................................................................................................................................................... 140 Tabela B3 – Reacção de ligação do fragmento contendo o gene perr ao vector de sub‐clonagem pNZY28® ................................................................................................................................................... 140 Tabela B4 – Reacções de hidrólise do fragmento que contem a PerR e o vector pET21c ................ 141 Tabela B5 ‐ Reacções de hidrólise do fragmento que contem a PerR e o vector pET30a ................. 141 Tabela C1 ‐ Planeamento dos testes de solubilização da PerR com endonucleases ......................... 149 Tabela C2 ‐ Planeamento dos testes de solubilização da PerR ......................................................... 150 Tabela D1 – Preparação das Soluções stock usadas para electroforese de SDS‐PAGE ..................... 152 Tabela D2 – Volumes necessários de cada solução para preparar um gel de SDS‐PAGE .................. 153 Tabela D3 – Preparação das soluções necessárias para a coloração com Azul de Coomassie ......... 153 Tabela D4 – Soluções necessárias para a coloração com Nitrato de Prata ....................................... 154 Tabela E1 – Preparação da recta de calibração para a determinação da quantidade total de proteína pelo método do BCA ................................................................................................................................ 156 Tabela E2 – Valores de absorvância obtidos para os padrões preparados conforme a tabela E1 .... 156 Tabela E3 ‐ Condições de aquisição dos espectros de 1H‐RMN ........................................................ 157 . xix .
(20) Tabela F1 – Volumes necessários para a preparação das amostras para o teste à enzima Tfi I ....... 158 Tabela F2 ‐ Volumes necessários para a preparação das amostras para o ensaio de ligação ao pADN .................................................................................................................................................................. 159 . . xx .
(21) . CAPITULO I . Introdução . .
(22) Índice Introdução Stress Oxidativo ................................................................................................................................... 3 A Super‐Família Fur ............................................................................................................................. 5 A PerR de Bacillus subtilis .................................................................................................................... 7 Estrutura .................................................................................................................................................................. 8 A ligação ao ADN ................................................................................................................................................... 11 . Bibliografia ........................................................................................................................................ 17 . 2 .
(23) Stress Oxidativo As espécies reactivas de oxigénio, como por exemplo o anião superóxido (O2.‐), o peróxido de hidrogénio (H2O2) e os radicais hidróxilo (OH•) são resultantes da redução incompleta do oxigénio molecular (O2)(1). Devido à presença de oxigénio na atmosfera, os organismos tornaram‐se ou anaeróbios, vivendo em nichos sem oxigénio, ou desenvolveram mecanismos de protecção contra estas espécies reactivas. Estas espécies são produzidas como consequência do metabolismo celular e têm a capacidade de alterar os componentes celulares desde proteínas a ácidos nucleicos, promovendo mutações ou até mesmo a morte celular. O peróxido de hidrogénio por si só não é muito reactivo, no entanto, é o precursor directo do radical hidroxilo, uma espécie altamente tóxica e oxidante. As enzimas responsáveis pela protecção celular contra estas espécies reactivas existem nas células em níveis basais, ou seja, em quantidade suficiente para uma eficiente resposta a baixos níveis de espécies reactivas de oxigénio. Em condições de desequilíbrio, stress oxidativo, quando o nível de espécies reactivas de oxigénio atinge valores considerados críticos, os organismos activam inúmeras funções para eliminar estas espécies e para reparar os danos celulares associados. As inúmeras enzimas responsáveis pela protecção diferem na sua expressão, na localização celular e regulação. Por exemplo, muitas bactérias expressam uma catalase durante a transição da fase do crescimento para a fase estacionária, provavelmente como forma de adaptação, para proteger o seu genoma, bem como outros componentes essenciais contra a oxidação. Esta resposta é regulada por factores de transcrição que funcionam como activadores ou repressores em resposta às espécies reactivas. Neste trabalho decidiu‐se estudar uma proteína associada ao mecanismo de resposta aos níveis peróxidos proveniente de um organismo anaeróbio, Desulfovibrio (D.) vulgaris vulgaris Hildenborough ATCC 29579. Esta bactéria, embora considerada um organismo anaeróbio obrigatório durante muito tempo, foi recentemente identificada em ambientes com exposição a oxigénio, devendo, como tal possuir um mecanismo de defesa ao stress oxidativo. Em D. vulgaris Hildenborough, este envolve as enzimas dismutase do superóxido (Sod) (2, 3) que catalisa a dismutação do superóxido em oxigénio e peróxido de hidrogénio. A catalase (KatA, KatG) e peroxidases como por exemplo a reductase do hidroperóxido de alquilo (AhpC). Para além destas enzimas existe ainda um mecanismo de protecção alternativo em D. vulgaris Hildenborough que inclui a rubredoxina oxidoreductase (Rbo) que possui actividade semelhante à reductase do superóxido (Sor). Estas enzimas catalisam a conversão do anião superóxido, altamente tóxico, em peróxido de hidrogénio e oxigénio. Existem ainda outras proteínas semelhantes à rubredoxina que funcionam normalmente como dadores de electrões(4, 5). A análise da . 3 .
(24) sequência do genoma de D. vulgaris Hildenborough permitiu a identificação de regulões, conjuntos de genes sob regulação de uma proteína reguladora, entre eles a PerR (Repressor do regulão do peróxido), membro de uma super família de metaloproteínas reguladoras da absorção de ferro designada por super família Fur.(6) Tal como em D. vulgaris em Bacillus (B.) subtilis, o gene perR(5), é regulado concomitantemente com um conjunto de genes redutores de peróxidos como os mencionados acima ahpC, a rbr (rubreritrina da família da Sor), a katA (catalase), zosA (7) (sistema de absorção de zinco) e mrgA (proteína que liga ao ADN semelhante à Dps) entre outros. De forma a avaliar a expressão dos genes responsáveis pela resposta a peróxidos na bactéria D. vulgaris Hildenborough, Mukhopadhyay e seus colaboradores(5) fizeram um ensaio que consistia na exposição de uma cultura de D. vulgaris a 0.1% de O2 e na exposição directa ao ar atmosférico (21% O2). A primeira conclusão extraída desse ensaio foi a de que com exposição a baixa percentagem de oxigénio verificou‐se uma diminuição na taxa de crescimento das bactérias, embora estas permanecessem viáveis. Este ensaio fez reforçar a ideia de que esta bactéria em condições normais de crescimento possui um nível adequado de enzimas requeridas à resposta a exposição a oxigénio. No entanto, relativamente à expressão génica verificou‐se que ocorria uma maior expressão dos genes associados ao regulão da PerR, como o gene da rubreritrina ou da AhpC. Para além do regulão da PerR, a bactéria D. vulgaris Hildenborough possui outros genes associados à protecção contra o stress oxidativo cuja expressão não foi alterada. Em contraste com o ensaio com 0.1 % de oxigénio, quando as células foram expostas ao ar atmosférico (21 % O2), os genes associados ao regulão da PerR sofreram um decréscimo na sua expressão sendo que na medida em que o tempo de exposição aumentava, diminuía consideravelmente a expressão dos genes associados ao regulão da PerR, bem como dos restantes genes de resposta ao stress oxidativo. Assim, de acordo com os resultados obtidos quer com a exposição a menor quantidade de O2 concluiu‐se que o fim da inibição da PerR parece ser a resposta primária de D. vulgaris Hildenborough à baixa exposição a Oxigénio. Por outro lado, os resultados com a exposição a 21% de oxigénio demonstraram uma repressão dos genes associados ao regulão da PerR. Este facto vem reforçar a ideia de que a desrepressão da PerR seja a resposta primária de D. vulgaris Hildenborough à baixa exposição a Oxigénio. 4 .
(25) A SuperFamília Fur A evolução promoveu a adaptação da família de repressores Fur para a sensibilidade a outros factores que não iões metálicos, como é o caso da PerR, um Repressor do regulão do peróxido. A família Fur, de metaloproteínas reguladoras apresenta um vasto mecanismo regulatório. Enquanto a maioria dos membros desta família eram inicialmente caracterizados como dependentes de ferro e repressores na ligação ao ADN, nem todos os elementos poderiam ser caracterizados desta forma. Tal como o ferro, o mecanismo de repressão dependente de metais foi descrito para outras proteínas que funcionam como sensores(8) de zinco (Zur), manganês (Mur) e níquel (Nur), tabela I.1. Outros membros utilizam a oxidação de resíduos da proteína para regular a actividade de ligação ao ADN e assim responder fisiologicamente à presença de peróxidos (PerR) ou hemo e oxigénio (Irr). Os membros da família Fur podem ainda actuar directamente para estimular a transcrição nos promotores alvos e podem ainda actuar indirectamente na expressão de genes através de pequenos ARN ou proteínas intermediárias. Tabela I.1 – Membros da super família Fur, tabela adaptada (9) . Sub‐família da proteína . Organismo . Zur . estrutural/regulatório. Função . Zn2+ / Fe2+ (Mn2+) . Absorção de ferro . B. subtilis . Zn2+ / Fe2+ . Absorção de ferro . P. aeruginosa . Zn2+ / Fe2+ . Absorção de ferro . E. coli . Fur . Metal . H. pylori . ? / Fe2+ . B. japonicum . ‐ / Fe2+ . E. coli . Zn2+ / Zn2+ . B. subtilis . Zn2+ / Zn2+ ‐ /Mn2+ (Fe2+) . Absorção de ferro Armazenamento de ferro Proteína Irr Absorção de zinco Absorção de zinco Mobilização de zinco . Mur . R. leguminosarum . Nur . S. coelicolor . PerR . B. subtilis . Zn2+ / Fe2+ (Mn2+) . Stress oxidativo . S. aureus . ?/ Mn2+ (Fe2+?) . Stress oxidativo . Irr . B. japonicum . ?/Ni2+ . ‐ / Fe‐heme . Absorção de manganês Absorção de níquel . Absorção de ferro . A proteína Fur, proteína reguladora da absorção de ferro, é uma proteína dimérica(10) envolvida na regulação da absorção de ferro nas bactérias gram negativas. É a proteína chave no controlo da . 5 .
Imagem



Documentos relacionados
Detectadas as baixas condições socioeconômicas e sanitárias do Município de Cuité, bem como a carência de informação por parte da população de como prevenir
O número de desalentados no 4° trimestre de 2020, pessoas que desistiram de procurar emprego por não acreditarem que vão encontrar uma vaga, alcançou 5,8 milhões
Os dados experimentais de temperatura na secagem da pêra em estufa a 40 o C estão apresentados na Figura 6, assim como os valores calculados pelo modelo
Os interessados em adquirir quaisquer dos animais inscritos nos páreos de claiming deverão comparecer à sala da Diretoria Geral de Turfe, localizada no 4º andar da Arquibancada
Após a queima, para os corpos com a presença do sienito observou-se que os valores de densidade diminuíram (comparados às respectivas densidades a seco), já
O valor da reputação dos pseudônimos é igual a 0,8 devido aos fal- sos positivos do mecanismo auxiliar, que acabam por fazer com que a reputação mesmo dos usuários que enviam
libras ou pedagogia com especialização e proficiência em libras 40h 3 Imediato 0821FLET03 FLET Curso de Letras - Língua e Literatura Portuguesa. Estudos literários
Feitiço do Segredo: deposita um segredo numa pessoa de confiança, essa pessoa fica deposita um segredo numa pessoa de confiança, essa pessoa fica sendo o "Fiel do sendo o