Enfin, je tiens à remercier M. Anizon Fabrice, professeur à l'Université Blaise Pascal, qui a accepté de réviser ces travaux et de présider le jury de cette thèse. Je tiens également à remercier les membres du département de chimie organique de l'École de Chimie : Safia Laid et Gaëlle Framery. Merci à ma grand-tante Mireille, qui m'a certainement transmis une grande partie de mon sens de l'humour.
Introduction
Interactions protéine/protéine
Les IPP sont généralement classés selon la nature du site d'interaction ou du site du ligand. Selon la littérature4, dans la plupart des cas la surface d'interaction entre les deux protéines concernées est de l'ordre de 6 nm². Les deux protéines en interaction ont tendance à former un complexe sur la surface d'interaction, dont la force dépend de plusieurs caractéristiques physicochimiques, telles que l'hydrophobie, la polarité ou les charges ioniques.4, 5.
Les protéines à domaines PDZ
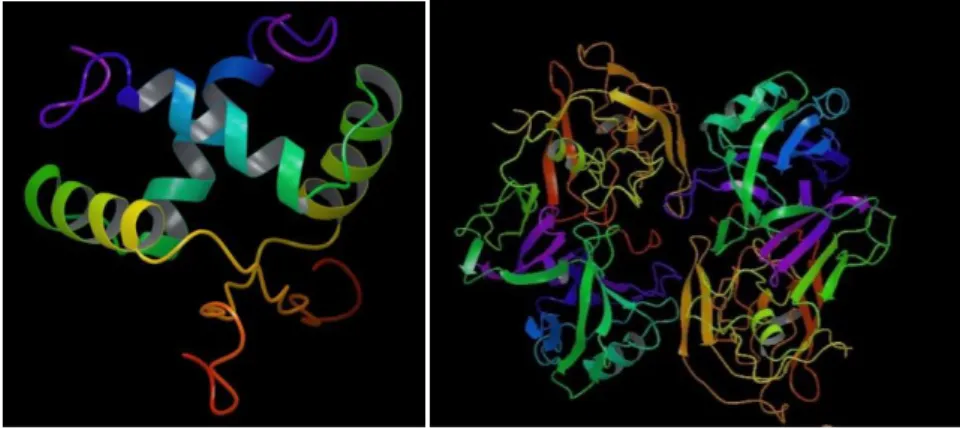
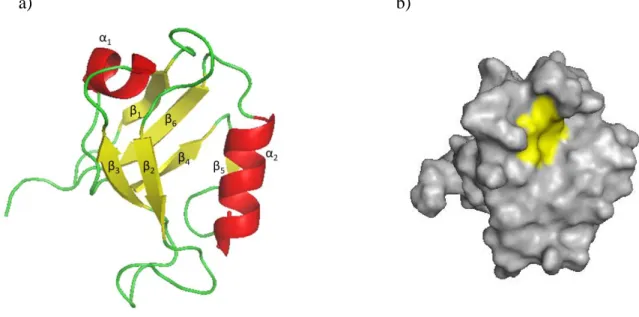
- Structure des domaines PDZ
- Classification des domaines PDZ
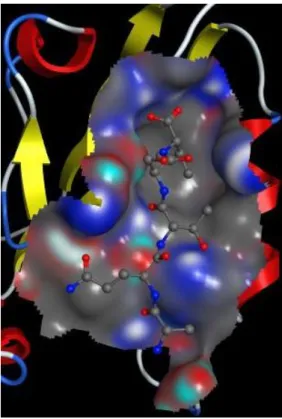
- Sites d’interactions
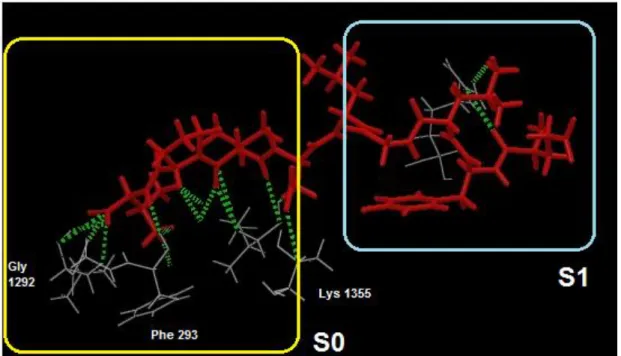
- Le site S0 et la boucle GLGF
- Le site S1
- Domaines PDZ et pathologies
- Le cancer
- La mucoviscidose
- La maladie de Parkinson
- La maladie d’Alzheimer
- La grippe aviaire
- Attaque ischémique
- Inhibition du domaine PDZ
- Les inhibiteurs peptidiques
- Petites molécules organiques
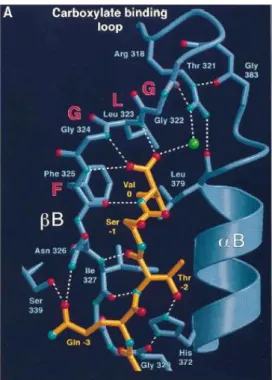
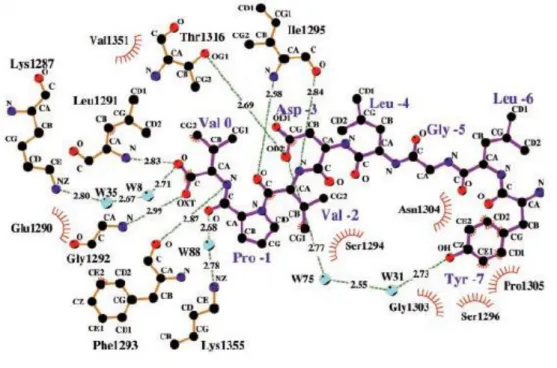
La poche créée entre l'hélice α2 (ou αB) et le feuillet β2 (ou βB) de la protéine forme le site d'interaction des domaines PDZ. La figure 8, tirée des travaux de Doyle et al.15, illustre les interactions entre le domaine PDZ3 de la protéine PSD-95 et le tétrapeptide VSTQ. En 2005, Shan et al.55 ont synthétisé et testé une structure à partir d'un écran virtuel d'une bibliothèque (NCI), le carboxylate 16 (Figure 26), qui a montré des interactions significatives avec le domaine PDZ de la protéine Dvl en utilisant HSQC 1H/15N.
Historique
La douleur fut définitivement traitée thérapeutiquement à partir de la fin du XVIIIe siècle avec l'usage de l'opium, puis au XIXe siècle avec la découverte de la morphine et de l'aspirine. Grâce à Bonica, la gestion de la douleur est devenue un point clé de la médecine ; dans ce but, il ouvre en 1960 la première clinique multidisciplinaire de la douleur dans l'État de Washington, aux États-Unis, et fait appel à des neurologues, des psychiatres, des pharmacologues, entre autres.
La Douleur, un fléau encore mal traité
En matière de recherche fondamentale et clinique, la douleur joue un rôle de plus en plus important. La prise en charge de la douleur est actuellement inadéquate car nous ne disposons pas encore de tous les outils pour répondre à la demande croissante. Structurer le secteur des soins contre la douleur, notamment la prise en charge des douleurs chroniques dites intraitables, pour rendre le système plus efficace.
Neuro-physiologie de la douleur
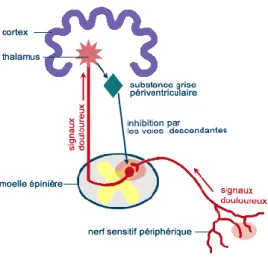
La voie de la douleur implique trois types de neurones : le premier transporte le message des nocicepteurs jusqu'à la moelle épinière, où s'effectue la première transmission synaptique. Ce sont ces régions d'intégration qui sont les cibles de la plupart des analgésiques.73, 74. La troisième étape, la modulation, concerne les neurones chargés de transmettre le message douloureux de la périphérie vers le cortex et leur capacité à adapter l'information.
Différents types de douleur
- La douleur aiguë
- La douleur chronique
Traitements de la douleur
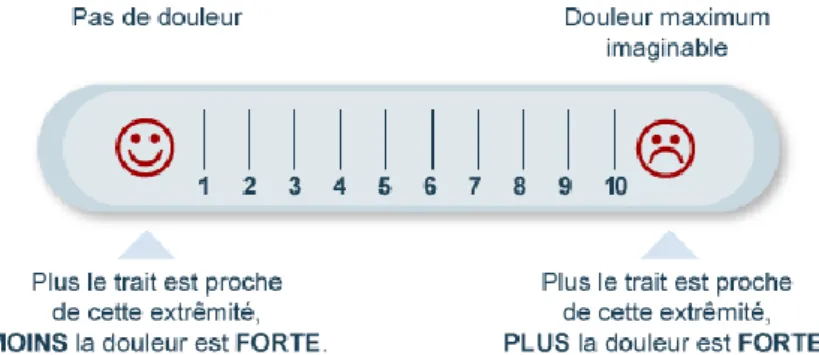
- Evaluation de la douleur
- Antalgiques et Analgésiques
- Antalgiques de palier 1
- Autres médicaments
Ce type de substance agit sur des récepteurs spécifiques du cerveau chargés de réduire la douleur. Il a un effet opioïde avec une sélectivité plus prononcée pour les récepteurs μ-opioïdes et un effet inhibiteur sur la recapture de la noradrénaline et de la sérotonine. L'énantiomère (-) inhibe la recapture de la noradrénaline, tandis que l'énantiomère (+) inhibe la recapture de la sérotonine.
Les domaines PDZ et la douleur
- Récepteurs N-méthyl-D-aspartate
- Récepteur « Acid-sensing ion channels »
Contexte du projet
Objectif du projet
- Les collaborations nécessaires au projet
- Travaux antérieurs
Biologie structurale : Une collaboration avec l'Université de Liverpool, plus précisément avec le laboratoire de biologie structurale, nous permettra de préparer des domaines PDZ et d'étudier par RMN les interactions entre nos molécules et le domaine protéique en question. Biologie moléculaire : Une collaboration avec l'Institut de Génomique Fonctionnelle de Montpellier permettra d'étudier l'activité de nos composés en compétition avec le ligand naturel par chromatographie d'affinité. Deux molécules, l'indole 36 et la chalcone 37, ont montré de bonnes affinités avec le domaine PDZ de TIP-1 au cours de cette expérience (Figure 44).
Les Biligands
Stratégie des biligands
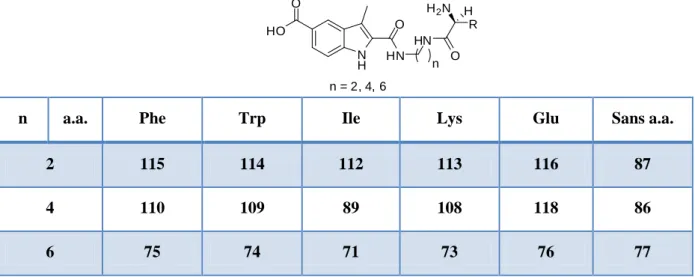
Pour ce faire, nous envisageons de synthétiser des molécules possédant un noyau indole, connu pour interagir avec le site S0, un bras espaceur, puis une entité capable d'interagir avec le site S1, spécifique d'un domaine PDZ donné (Figure 46). . Il comprendra un nombre variable de carbones qui permettront de sonder le site S1 (Figure 48). La troisième partie de la molécule (R7) va se lier à l'amine terminale, nous choisirons différents acides aminés avec des caractéristiques différentes (hydrophobes, hydrophiles, chargés polaires ou non...), afin d'explorer le site S1 et nous étudierons le spécificité. de molécules préparées en association avec diverses protéines du domaine PDZ.
Noyau indolique
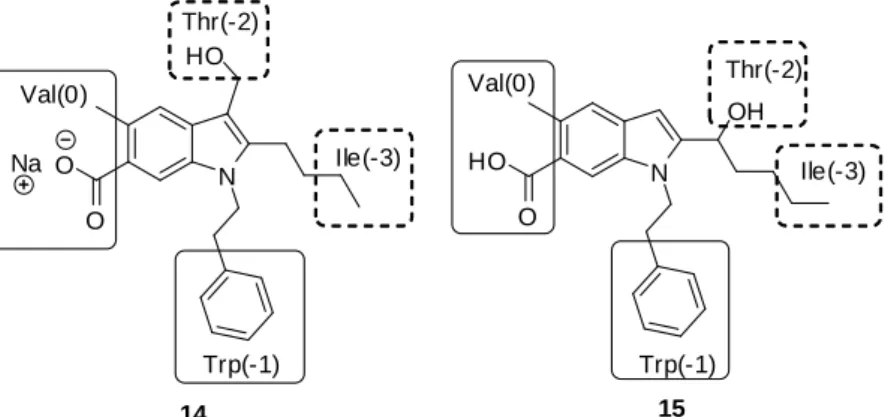
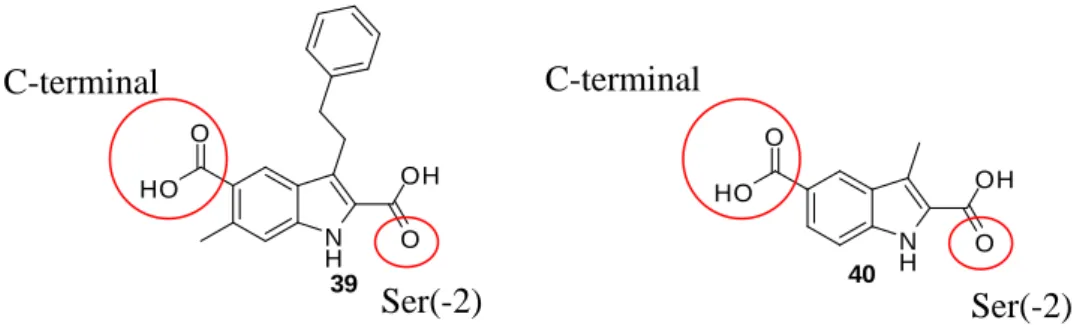
Pour faciliter la compréhension, nous nommerons les biligands de manière simplifiée comme suit : Indole-Cn-AA, signifiant indole attaché à un espaceur contenant n atomes de carbone, attaché à l'acide aminé (Figure 49). Une simulation des interactions moléculaires par docking (Figure 51) entre les indoles 39 et 40 et la protéine PSD-95 PDZ1 a permis de conclure que la molécule 40 adopte une pose similaire à l'indole 39,110. On observe plusieurs interactions entre le ligand et la protéine, notamment avec la boucle GLGF.
Synthèse de l’indole
- Bibliographie
- A partir d’hydrazines (synthon A)
- A partir du synthon B
- A partir du synthon C
- A partir du synthon D
- A partir du synthon E
- Analyse rétrosynthétique
- Travaux antérieurs du laboratoire
- Synthèse en solution
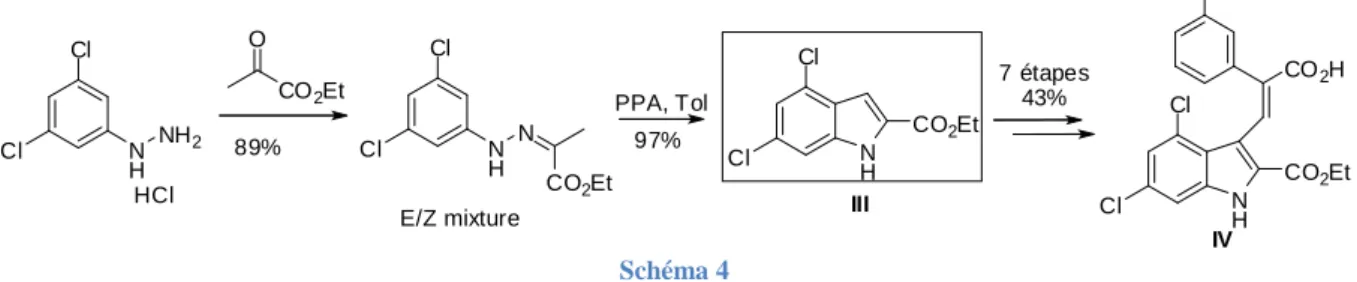
L'hydrazone intermédiaire, formée à partir de 3,3'-dichlorophénylhydrazine commerciale en présence d'éthylpyruvate, est dans ce cas activée par l'acide polyphosphorique (PPA) pour conduire au composé indolique III souhaité avec un rendement de 97 %.114. La réaction « one-pot » du synthon C en présence d'un α-amino ester protégé, catalysée par l'iodure de cuivre, permet la formation du noyau indole avec des rendements moyens de 55 % (Schéma 10).127. L'étape suivante consiste en une syn-addition de la double liaison énamine sur le palladium.
Ce dernier peut être obtenu à partir du composé 41 par protection de l'acide carboxylique avec un groupe R et réduction ultérieure. La réduction du groupe nitro du composé 42 est réalisée en présence de chlorure stanneux.144 La présence de l'amine 43 est confirmée par IR. L'amine 45 est obtenue à partir de l'acide 41 par estérification, réduction du groupe nitro, puis iodation en présence de diiodure et de sulfate d'argent avec un rendement global de 80 % (Schéma 23).
Nous avons donc réalisé la synthèse de l'indole 50 à partir de l'amine commerciale 49, en nous inspirant de la méthode décrite par Fujii et al. La deuxième étape est une condensation/cyclisation intramoléculaire en présence d'acide oxobutyrique et de DABCO, catalysée par l'acétate de palladium. Cette décarboxylation pourrait avoir lieu soit après condensation sur la cétone, au niveau de l'énamine intermédiaire, avant cyclisation, en milieu basique, soit après formation du noyau indole, en présence de palladium (Schéma 26).
Les résultats obtenus lors de l'optimisation de la réaction de formation de l'indole 50 sont résumés dans le tableau 1.
Molécules cibles
Première voie de synthèse
- Couplage peptidique de l’espaceur (n = 6, 8)
- Couplage avec les acides aminés (n = 8)
- Synthèse des biligands (n = 6)
Pour éviter les réactions parasites dues à la double réactivité des diamines, nous avons envisagé de préparer des diamines monoprotégées. Le fragment Boc est ensuite clivé en présence de TFA pour obtenir les produits souhaités. Nous avons donc synthétisé les deux sels de TFA 60 et 61 avec des rendements globaux respectifs de 85% et 78% à partir de l'indole 50.
A partir du sel 60 préalablement préparé, nous avons réalisé des fusions peptidiques avec des acides aminés protégés. En effet, les composés étant totalement insolubles même dans le DMSO, nous avons abandonné la série n = 8. De la même manière que pour la série n = 8, les couplages avec des acides aminés protégés par Fmoc sont réalisés en présence de CDI, TFA 61 traité. saler au préalable dans un réglage basique.
Divers acides aminés protégés, Ile, Lys, Trp, Phe et Glu sont introduits (Schéma 35) en présence d'un agent de couplage. Ainsi, nous avons obtenu cinq intermédiaires 66-70 qui, peu stables, sont inclus directement dans l'étape de saponification. En effet, nous avons observé la dégradation, notamment sur silice neutralisée, du composé final contenant cet acide aminé.
Nous avions initialement envisagé d'appliquer cette stratégie aux deux autres séries de composés (n = 4 et n = 2), mais nous avons observé de faibles rendements lors de la saponification en raison de la rupture de la liaison amide entre l'acide aminé et l'espaceur.
Deuxième voie de synthèse
- Formation de l’ester benzylique de l’indole
- Couplage peptidique de l’espaceur (n = 4, 2)
- Couplage peptidique avec les acides aminés (n= 2, 4)
- Principe
- Résultats
Ce couplage est réalisé dans les conditions développées à l'aide de l'ester éthylique 50 en présence du couple HOBt/EDC et de diamines monoprotégées sous forme du groupe Boc. Ces intermédiaires sont isolés et caractérisés par RMN puis déprotégés en présence de TFA dans le DCM. Le couplage peptidique nécessitant l’utilisation de DIEA pour générer l’amine libre, nous avons choisi d’utiliser des acides aminés protégés par un carbamate de benzyle.
En effet, de cette manière l'ester benzylique et le groupe protecteur des acides aminés peuvent être clivés dans les mêmes conditions d'hydrogénation catalytique et donner les biligands finaux souhaités. Le biligand protégé 88 est obtenu dans les conditions de couplage peptidique décrites précédemment avec un rendement de 52 % (schéma 41). Nous avons également tenté un couplage avec d'autres acides aminés protégés par un benzylcarbamate (Schéma 42), notamment la phénylalanine et le tryptophane, mais malheureusement les rendements obtenus ont été très faibles (inférieurs à 5 %).
Nous avons utilisé des acides aminés protégés par un groupe Boc, stables en milieu basique (DIEA), clivables en conditions acides et permettant l'isolement des sels de TFA correspondants. Le couplage peptidique est réalisé dans les conditions classiques à partir des indoles 84 et 85 pour donner les intermédiaires protégés 90-93 (n = 4) et 94-98 (n = 2) sous forme de Boc dont les structures ont été confirmées par RMN (Figure 43). Ces intermédiaires 90-98 instables sont directement impliqués dans l'étape de clivage du groupe Boc en présence de TFA.
Selon la molécule et la concentration utilisée, une différence d'intensité est observée lors du dépliage du gel, correspondant à la quantité différente de protéine PSD-95 attachée à la résine par interaction avec le peptide.
Criblage RMN
- Synthèse de la protéine PSD-95 PDZ1
- Préparation du plasmide d’expression
- Production de la protéine
- Cartographie
- Evaluation
Par conséquent, pour réaliser la PCR, vous devez trouver la séquence d’ADN de la protéine PSD-95 puis trouver la partie qui code pour PDZ1 (expasy, PDB : 2KA9). Ce médicament provoque la lecture du plasmide et donc la production de la protéine. En effet, baisser la température ralentit la multiplication des bactéries ce qui favorise l'expression de la protéine.
Un dernier contrôle est effectué par RMN 1H en réalisant le spectre de la protéine pour s'assurer que la structure globulaire a été préservée (Figure 63). Pour évaluer l’interaction de nos composés avec le domaine PDZ1 de la protéine PSD-95 par 1H/15N HSQC, il faut d’abord réaliser une cartographie protéique. Cette cartographie est réalisée par RMN en réalisant diverses expériences (NOESY, TOCSY...) qui permettent d'associer chaque signal du spectre aux acides aminés de la protéine.
Nous avons ensuite enregistré le spectre de la protéine produite en laboratoire (Figure 65) afin de le comparer à celui de la littérature dans les mêmes conditions. Ainsi, une affectation correspond à un acide aminé de la protéine (sauf pour les acides aminés qui ont de l'azote dans les chaînes latérales, auquel cas il existe plusieurs affectations de corrélation). Nous avons également montré dans la représentation 3D de la protéine (Figure 67), les acides aminés perturbés par l'interaction du composé 52.
Ces biligands n'ont montré aucune interaction significative avec le domaine PDZ1 de la protéine PSD-95 par HSQC.
Optimisation
Analogues 119 et 121
- Synthèse des analogues
- Interactions des analogues 119 et 121/PDZ par RMN
Relation structure activité autour de 121
- Préparation du composé 133
- Synthèse de 139 et 140
- Synthèse de 149 et 150
- Interactions des analogues 149 et 150 avec le domaine PDZ1 de PSD-95
- Conclusion
Synthèse des amides 153, 154, 155
- Synthèse de l’indole 153
- Synthèse de l’indole 154
- Synthèse de l’indole 155
Interactions des indoles 153, 154, 155 avec le domaine PDZ1 de PSD-95 par
Etude de modélisation moléculaire
- Analyse rétrosynthétique
- Synthèse de l’indole 160 et 161
- Synthèse de l’indole 162
- Synthèse de l’hydroxyindole 178
Analogue substitué en position 2
- Synthèse
- Evaluation de l’interaction de l’indole 179 avec la protéine PSD-95 PDZ1 par
Analogue substitué sur l’azote N-1 de l’indole
- Synthèse
- Evaluation de l’interaction des indoles 185 et 186 avec la protéine PSD-95
- Impact de la molécule 185 sur l’interaction entre le C-terminal de 5-HT 2A et
Synthèse
Evaluation de l’interaction des indoles 187 et 188 avec la protéine PSD-95 PDZ1
Introduction
Structure RMN des complexes 185 et 187 /PSD-95 PDZ1
Evaluation pharmacologique in vivo
Synthèse
- Synthèse du phénylglucopyranosylamine
- Synthèse de la N-acyl hydrazone 192
- Synthèse de la quinoléine 197