La topologie de l'ADN comme cible de la sélection naturelle 90 Article : Evolution expérimentale à long terme chez Escherichia coli. Si la superhélicité de l'ADN est impliquée dans l'adaptation des bactéries à leur environnement, elle doit donc constituer une cible évolutive, c'est-à-dire une cible de sélection naturelle.
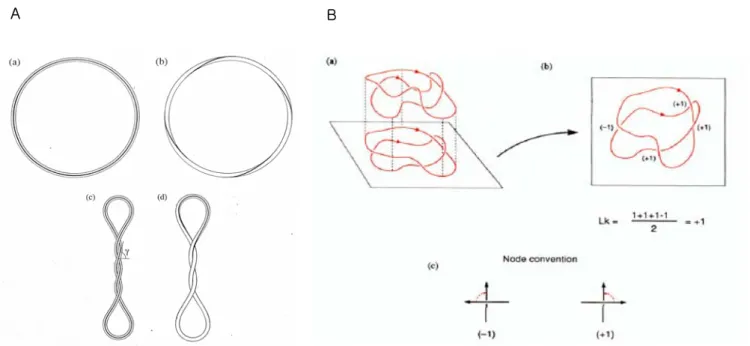
Les acteurs de la topologie de l’ADN
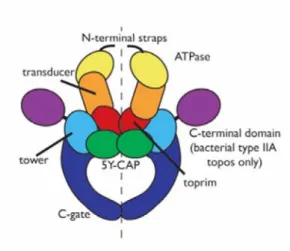
Les topoisomérases
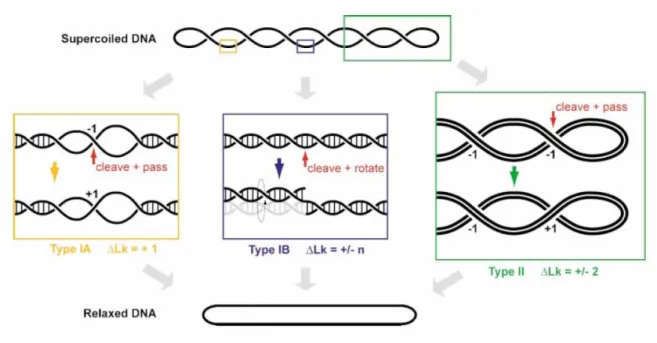
Les Topos de type I
Après la fermeture de la cavité enzymatique, le brin d'ADN clivé est ensuite religaturé par TopoI (Figure 4f). Cela se produit parallèlement à l’activité de la gyrase, qui introduit des superturns négatifs dans l’ADN.
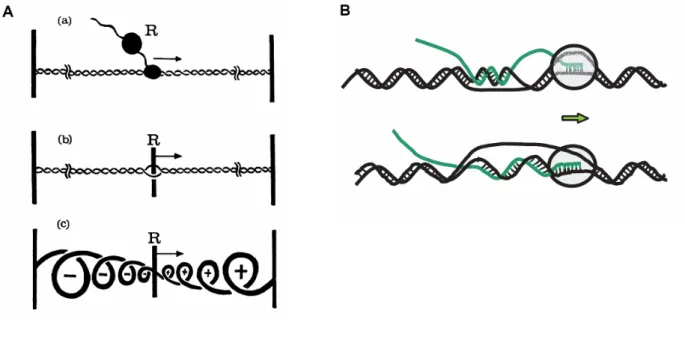
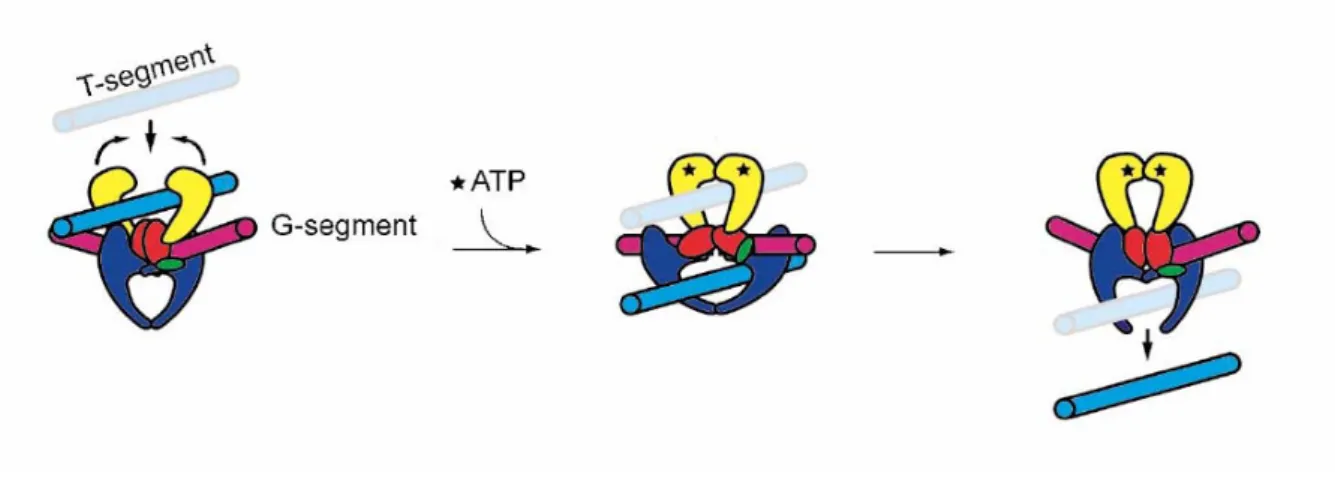
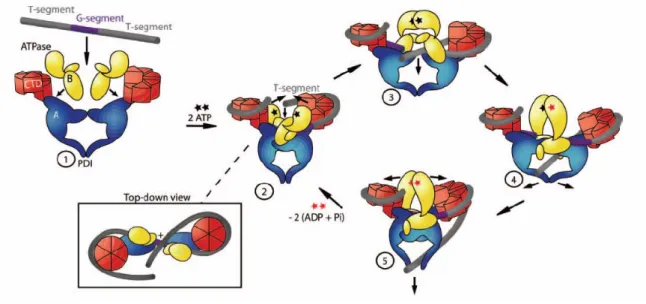
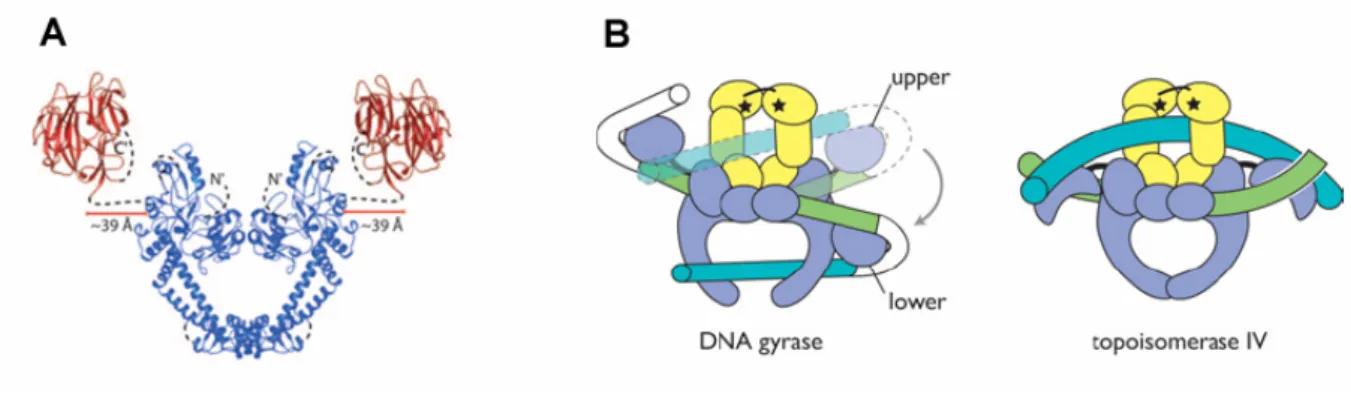
Les Topos de type II
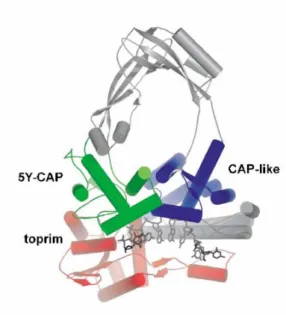
La différence entre les CTD gyrase GyrA (à gauche, bleu) et les CTD TopoIV ParC (à droite, bleu). Dans le cas de la gyrase (à gauche), l'ADN s'enroule autour des CTD de GyrA, et un mouvement de l'un d'eux provoquerait le transfert de la double hélice T dans la cavité formée par les portes C.
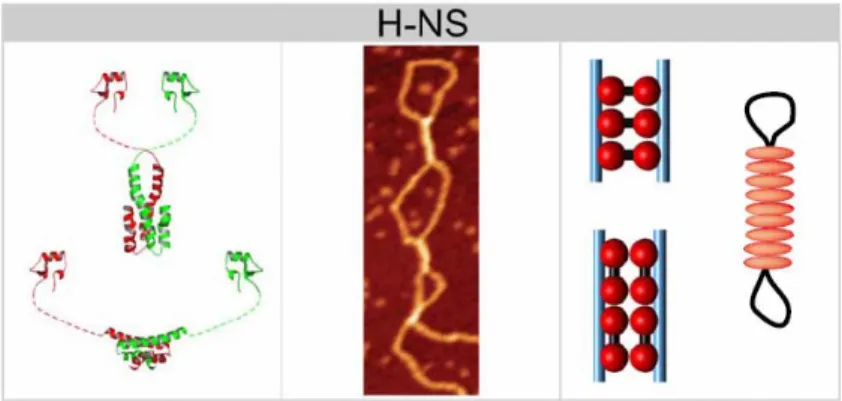
Les protéines de type histone
Les NAPs ne possédant aucune spécificité de fixation
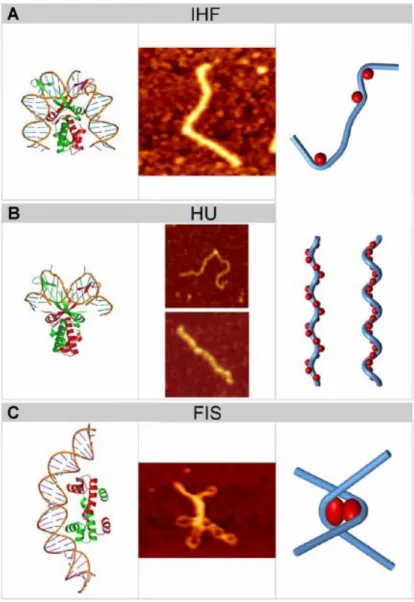
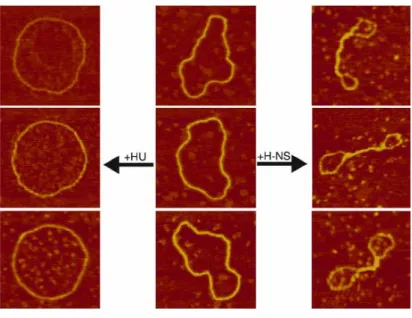
On pense que le mécanisme par lequel H-NS active ces gènes est lié à sa capacité à augmenter le surenroulement de l'ADN (Johansson et al., 2000). À droite, le motif de protection de l'ADN (en rouge) provenant des dodécamères interconnectés (en bleu) (Frenkiel-Krispin et al., 2004).
La protéine IHF
Le Dps est capable de séquestrer les ions Fe2+ et de les oxyder grâce à l'utilisation, et donc à l'élimination, de H2O2, sans libérer de radicaux libres, maintenant ainsi l'intégrité de l'ADN (Zhao et al., 2002). Ainsi, la liaison de l'IHF à l'un de ses sites de liaison en amont du promoteur ilvPG améliore l'ouverture de la bulle de transcription en transférant la tension induite par la courbure de l'ADN des sites riches en AT en amont du promoteur vers ce dernier (Parekh et al., 1996). Sheridan et coll., 1998).
La protéine Fis
En revanche, l'influence de l'IHF sur la structure de l'ADN contribue surtout au contrôle de la transcription. BipA interagit avec les ribosomes et son rôle est de favoriser l'initiation de la traduction de l'ARNm fis.
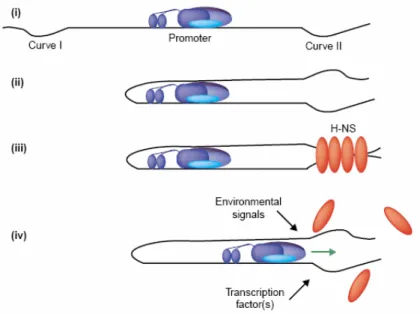
Dynamique d’expression des NAPs
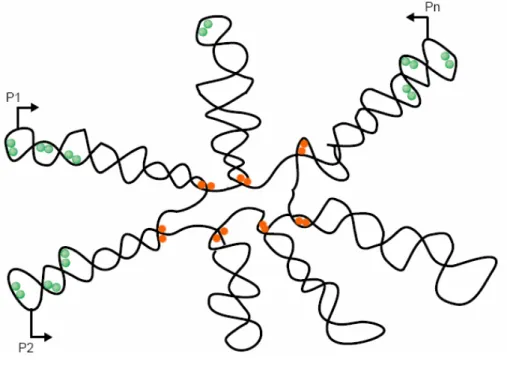
Organisation du chromosome dans la cellule
Les domaines topologiques
- La transcription
- Les complexes SMC
- Les Topos
- Les protéines liées au nucléoïde
- Les autres protéines
Cependant, la transcription des gènes codant pour les protéines cytoplasmiques induit la même diminution de la recombinaison entre sites res (Deng et al., 2004). Plus récemment, Moulin et al. (2005) ont démontré la capacité de la gyrase à former des barrières en utilisant un système expérimental différent.
Les macrodomaines
Impact de la superhélicité de l’ADN sur l’expression des gènes
Les fluctuations du niveau de superhélicité de l'ADN ont des conséquences importantes sur l'expression de nombreux gènes (Steck et al., 1993 ; Peter et al., 2004), notamment sur la transcription des opérons de l'ARN ribosomal (Muskhelishvili et Travers, 2003). En revanche, une augmentation de la superhélicité négative du chromosome provoquée par une mutation du gène topA entraîne une modification de l'expression des gènes, observée par électrophorèse bidimensionnelle des protéines (Steck et al., 1993). Une superhélicité négative élevée supprimera la fuite de l'ARN polymérase au niveau des promoteurs riches en AT ainsi que l'élongation de l'ARN.
Régulation et contrôle homéostatique du niveau de superhélicité de l’ADN
La superhélicité de l'ADN est donc étroitement liée à l'expression des gènes et peut représenter un élément régulateur important dans l'adaptation des bactéries à leur environnement en ajustant la transcription globale des gènes aux variations environnementales (Figure 25). Contrairement à la gyrase, qui est activée par une diminution de la superhélicité de l'ADN, TopoI est activée par une augmentation du niveau de superhélicité (Tse-Dinh, 1985 ; Zechiedrich et al., 2000). Des niveaux supplémentaires de régulation du degré de superhélicité et donc des gènes codant pour Topos doivent donc exister pour détecter et répondre aux signaux environnementaux.
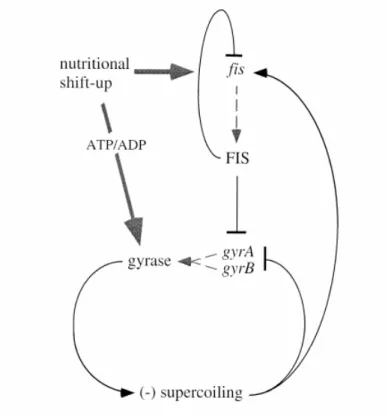
Contrôle homéostatique de la superhélicité de l’ADN en fonction des conditions environnementales
Cette augmentation de la superhélicité de l'ADN et du Fis favorise alors la transcription des gènes nécessaires à la croissance bactérienne, notamment les opérons de l'ARN ribosomal (Muskhelishvili et al., 1997). Étant donné que la transcription de Fis nécessite une superhélicité élevée, cette relaxation de l'ADN entraîne une transcription réduite de Fis (Schneider et al., 2000b). Ainsi, malgré la transcription des gènes codant pour la gyrase lors de la libération de la répression par Fis, le degré de superhélicité de l'ADN diminue en phase stationnaire.
Autres stress environnementaux et topologie de l’ADN
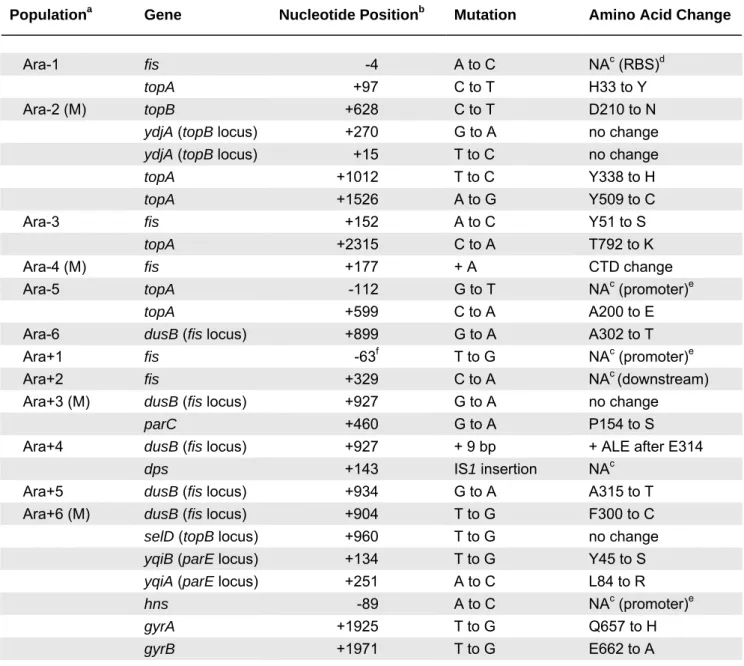
Therefore, a decrease in the expression of the ribose operon and the maltose regulon was detected in both cases (Pelosi et al. 2006). We have previously demonstrated the adaptive loss of ribose consumption capacity under these experimental evolutionary conditions (Cooper et al. 2001). Mutations in malT, the transcriptional activator of the maltose operons, (Danot 2001) were discovered in both focal Ara-1 and Ara+1 populations (Pelosi et al. 2006).
Map of the spot mutations found in eight evolving populations. (Cooper et al. 2003) The amino acid changes are shown along with their locations within the SpoT protein. The SpoT protein is divided into three functional domains: the ppGpp hydrolase domain and the ppGpp synthetase domain (Gentry and Cashel 1996) and the carboxy-terminal domain (CTD) which is proposed to be involved in the regulation of the two previous domains.(Mechold et al. al. 2002).
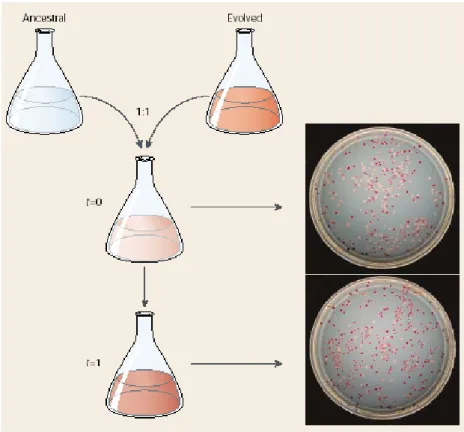
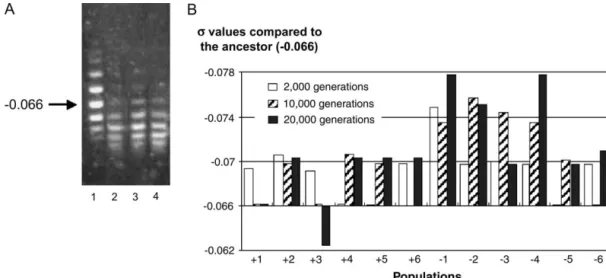
DNA Topology as a Key Target of Selection
Quantitative comparisons between each evolved clone and the ancestor are based on densitometric analyzes of the gels (see Materials and Methods). These data also imply a reduction in DNA attraction and measured DNA topology as before. The level of the control protein explained the observed changes in DNA topology, but did not differ between these strains.
Introduction
Therefore, DNA supercoiling has been shown to be one of the main targets of natural selection. However, and this represents one of the aims of this manuscript, this study was not extended to the other 11 populations to analyze the level of genetic parallelism associated with these parallel increases in DNA supercoiling. Third, only a few of the 12 populations did not show an increase in DNA superhelicity levels [41].
Results
Consistent with our previous results [ 41 ], the Ara-1 evolved allele resulted in a 3-fold decrease in the Fis level. In the case of the Fis progenitor protein, a shift in electrophoretic mobility was seen at the lowest concentration (0.2 nM) of Fis used (Figure 2A). We therefore demonstrated that both mutations in the phys coding region resulted in a reduced activity of the protein.
Discussion
The allele developed by Ara-4 is an insertion of an A residue, which leads to a change in the C-terminal part of the protein. On the other hand, it was shown to lead to a decrease in the number of topological domains in the chromosome [22], which will alter the transcription of at least the genes known to be sensitive to the level of DNA supercoiling [10 ]. For example, endosymbiotic bacteria of the Enterobacteriaceae do not display the dusB-fis operon (Table 5), and this may be related to their specialized living conditions known to lead to gene loss [88].
Acknowledgments
Evolved fis alleles were introduced into the chromosome of the ancestral strain REL606 by homologous recombination using suicide plasmid pKO3 [91], as previously described [41]. This generates plasmid transcriptional fusions with each of the ancestral and evolved promoters that drive transcription of lacZ. Two of the mutations identified in fis in the evolving populations affect its coding sequence.
Matériel et méthodes
Milieux de cultures, souches et plasmides
Electrophorèses bidimensionnelles
Analyse des électrophorèses bidimensionnelles
Fusions transcriptionnelles
Toutes les positions des nucléotides sont données par rapport au site majeur d'initiation de la transcription +1 pour chacun des gènes (Huang et al., 1992 ; Batchelor et al., 2005 ; Ferrario et al., 1995). La séquence de chacun des fragments a ensuite été vérifiée par séquençage, et les fragments contenant les promoteurs des gènes ompF, ompC et micF ont été clonés dans le plasmide pRS551 (Simons et al., 1987). Les fusions transcriptionnelles ont ensuite été réalisées de la même manière que précédemment (voir Résultats, Partie II, Matériels et méthodes) en intégrant le bactériophage λRS45 dans le chromosome de la souche d'intérêt et en sélectionnant et criblant les bactéries lysogènes pour les phénotypes de résistance. kanamycine dont le gène conférant la résistance est porté par pRS550, pRS551 et λRS45, et couleurs bleues sur milieu contenant de la kanamycine et du Xgal.
Retards sur gels
Les fragments contenant les différentes régions promotrices ont été amplifiés par PCR puis clonés dans le plasmide pCRII-TOPO (Invitrogen). La région promotrice ompR a été clonée dans pRS551 et pRS550, dont le site de clonage multiple est inversé par rapport à pRS551, pour obtenir les fusions transcriptionnelles ompR-lacZ et greB-lacZ. Les marqueurs radioactifs, ainsi que les conditions utilisées lors des expériences de retardement du gel, sont les mêmes que celles décrites précédemment (voir Résultats, Partie II, Matériels et méthodes).
Empreintes à la DNase I
- Résultats
Les contrôles permettant de tester la spécificité des retards observés sont également similaires à ceux décrits et utilisés précédemment. Pour déterminer les positions des sites de liaison Fis, une réaction de séquençage G+A de Maxam et Gilbert (Maxam et Gilbert, 1977), réalisée sur le même fragment radiomarqué, est déposée en parallèle sur le gel. L'électrophorèse est réalisée dans du tampon TBE 0,5X et, après un temps de migration approprié, le gel est séché pendant 1h30 à 80°C et coulé de la même manière que les gels retardés.
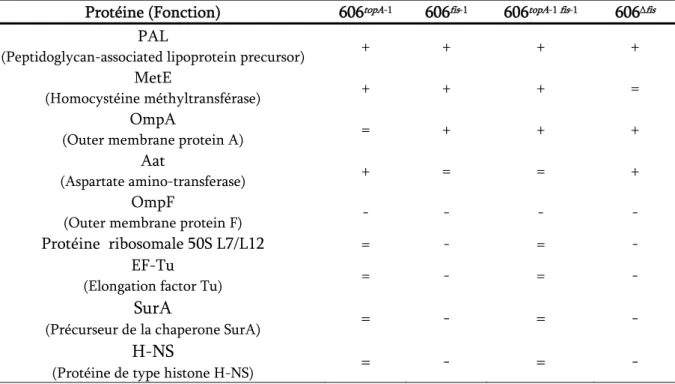
Modification des profils protéiques
Les protéines dont la quantité diffère de manière reproductible dans une ou plusieurs souches isogéniques par rapport au clone ancestral sont présentées ici. Les souches sont toutes isogéniques les unes aux autres à l'exception des allèles évolués de topA et/ou fis et sont comparées au clone ancestral issu de l'évolution expérimentale. Nous avons donc étudié l'expression du gène ompF dans nos souches ancestrales isogéniques portant les allèles ancestraux ou évolués de topA et fis.
Les porines d’ E. coli
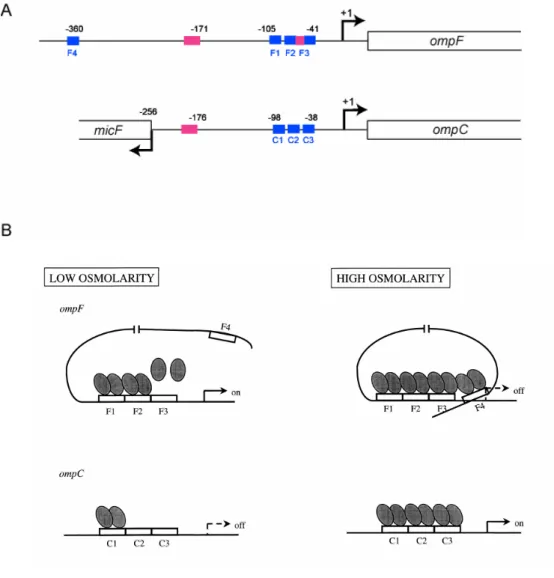
Les gènes sont indiqués en italique, avec des flèches courbes indiquant les sites de début de transcription. La formation d'une boucle conduit alors à l'inhibition de la transcription d'ompF, tandis que la concentration d'OmpR-P est désormais suffisante pour occuper tous les sites C1 à C3, permettant l'activation de la transcription d'ompC (Bergstrom et al., 1998). Cela se traduit par une inhibition très rapide d'OmpF dans ces conditions (Ramani et al., 1994).
Effets des mutations topA et fis sur la transcription de ompF
- La transcription de ompF dépend de la superhélicité de l’ADN et de Fis
- L’activation de ompF par Fis est directe
- Détermination des sites de fixation de Fis sur ompF
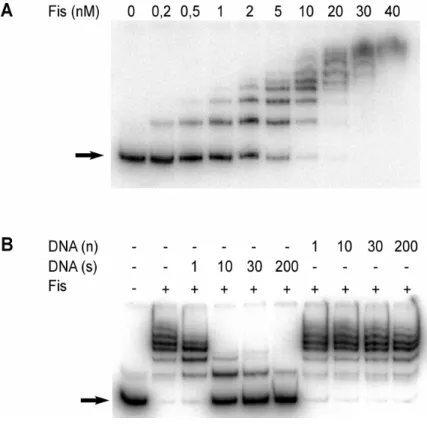
Retard de migration de la région promotrice ompF par différentes concentrations de Fis (en nM). Dans le milieu LB classique (NaCl 0,086 M), l'activation de la transcription de l'ompF B par Fis n'a été trouvée qu'en phase exponentielle. Les positions sont données par rapport au site principal d'initiation de la transcription de l'ompF.
- Régulation du gène ompF K12
La régulation de chacun de ces gènes a été analysée selon la phase de croissance, en phase exponentielle et en phase stationnaire.