Je tiens tout d'abord à remercier Juan Fontecilla, mon directeur de thèse, de m'avoir accueilli dans son laboratoire durant mon année de thèse. Je remercie Patricia Amara d'être toujours là pour m'aider, pour sa bonne humeur, nos échanges, ses encouragements.
Les centres FeS
Nature et fonction des centres FeS
1.1) Différentes formes de centres FeS
Le centre Y [4Fe-4S] lie un autre centre métallique à l'un de leurs atomes de fer. Dans les hydrogénases [Fe-Fe], le centre [4Fe-4S] lie un centre avec 2 atomes de Fe : ce site sera décrit plus en détail ultérieurement.
1.2) Quelques fonctions des centres FeS
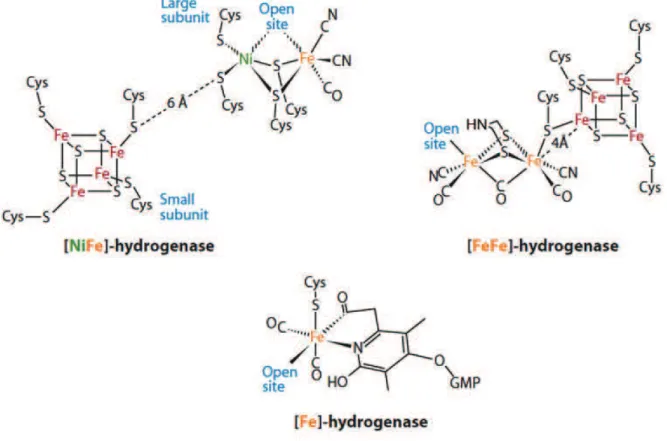
Les hydrogénases
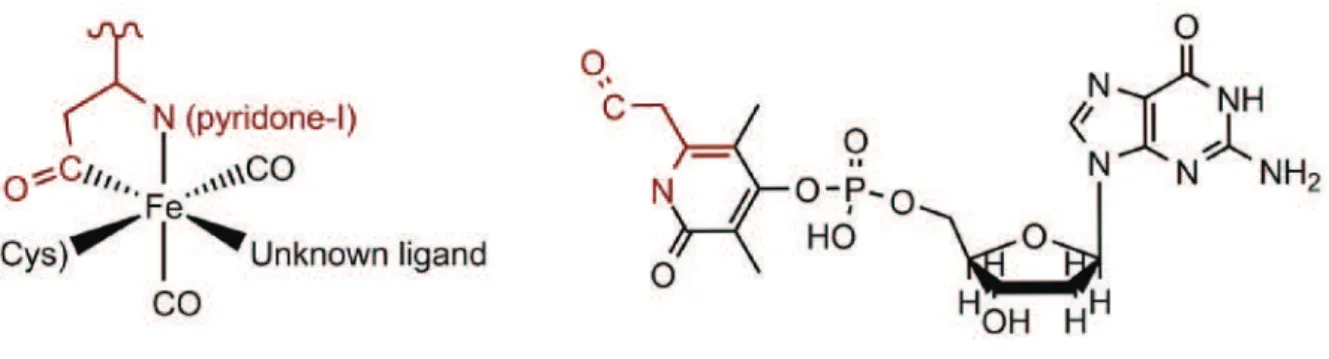
Le site actif de la [Fe-Fe] hydrogénase (appelé « H-cluster ») est composé d'un centre [4Fe-4S] lié par une de ses cystéines à un centre à deux atomes de Fe. Le site actif de la [Fe]-hydrogénase est composé d'un cofacteur appelé FeGP constitué d'un atome de fer lié à deux ligands CO, l'azote d'un dérivé de la 2-pyridone et un groupe acyle attaché à ce dérivé sont liés (Hiromoto, Ataka et al.2009) ).
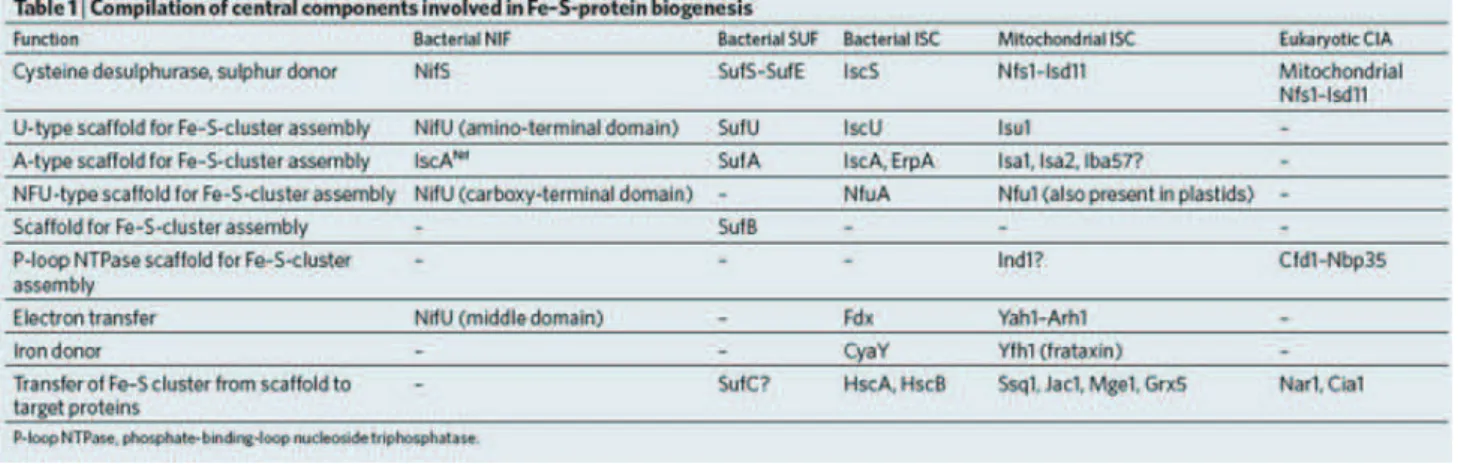
Fabrication des centres FeS
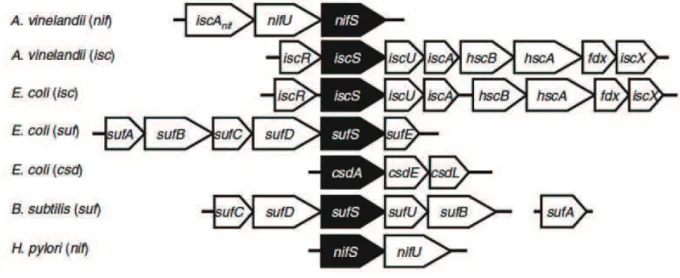
Maturation de la nitrogénase et découverte des machineries ISC et SUF
Il aurait donc un rôle in vivo dans la mobilisation du soufre pour la production de centres FeS pour la nitrogénase. D'autres gènes impliqués dans la production de centres FeS ont été identifiés, ceux de l'opéron SUF codant pour la machinerie SUF (mobilisation du soufre).
La machinerie ISC
En mutant l'opéron isc entier d'E.coli (iscRSUA-hscBA-orf3), nous avons observé une activité résiduelle d'enzymes avec des centres FeS. La suppression de cet opéron en présence de l'opéron ISC n'a montré aucun effet sur l'activité des protéines à centres FeS (Takahashi et Tokumoto 2002), mais a montré son rôle dans des conditions de carence en fer (Outten, Djaman et al. 2004) ou de stress oxydatif. (Nachin, Loiseau et al. et al. 2003).
2.1) Exemples d’opérons codant pour les machineries bactériennes de fabrication des centres FeS
L'activité des protéines contenant un centre FeS est significativement réduite en son absence, tandis que les protéines qui n'en contiennent pas voient leur activité inchangée (Schwartz, Djaman et al. 2000). La machinerie ISC est constituée des protéines IscR, régulatrices de l'opéron isc ; IscS, cystéine désulfurase ; IscU, protéine d'échafaudage ; On pense que IscA est une protéine de transfert de centre FeS ; les protéines chaperons HscB et HscA ; une ferrédoxine et une protéine IscX dont le rôle est encore inconnu.
2.2) Les protéines IscS et IscU
Assemblage des centres FeS chez les eucaryotes
Concernant la production de centres FeS chez les eucaryotes, la mitochondrie possède des composants homologues à ceux de la machinerie bactérienne ISC et les plastes possèdent des composés homologues à ceux de la machinerie SUF. L'assemblage des centres FeS dans le cytosol et dans le noyau des eucaryotes nécessite l'assistance de la machinerie ISC mitochondriale et un système d'exportation des mitochondries vers le cytosol.
Identification d’une machinerie de maturation de l’hydrogénase à [Fe]
Il existe deux PRS dans la machinerie fer-fer hydrogénase (présence des PRS HydE et HydG) (Mulder, Shepard et al. 2011) et la nitrogénase (présence de PRS NifB) (Rubio et Ludden 2008). Le gène MK0014 code pour une protéine avec un domaine de liaison au NADP de type Rossmann (Thauer, Kaster et al. 2010).
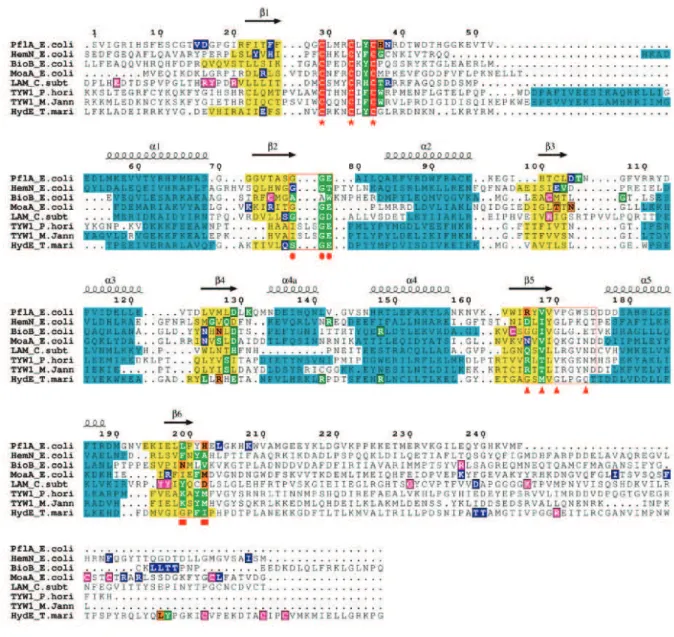
Les protéines à radical SAM
- Le cœur ( ) 6 des PRS
- Des structures spéciales pour des PRS
- Le centre [ 4Fe-4S] et la liaison de la SAM
- Fonction et réactivité des PRS
- Sous-famille de PRS ( ) 8
Il s'agit de protéines impliquées dans la biosynthèse du diphthamide (Dph2) (Zhang, Zhu et al. 2010) (Figure III–. Les autres produits de réaction sont le cyanure (Driesener, Challand et al. 2010) et le CO (Shepard,.
Biologie moléculaire
1.2) Digestion enzymatique de l’insert et du plasmide
1.3) Traitement à la phosphatase
1.4) Ligation de l’insert et du plasmide digérés
1.5) Transformation du produit de ligation
250 µL de milieu de culture LB sont ensuite ajoutés (Sigma) et le mélange est incubé 1 heure à 37°C sous agitation à 200 tr/min. 250 µL de liquide sont retirés après centrifugation et le reste est remis en suspension et étalé sur boîte LB-Agar (Sigma) contenant de l'ampicilline à 50 µg/ml.
1.6) Test PCR sur colonies et extraction de plasmide
Mutant C-terminal
Après une nuit d'incubation à 37 °C, quelques colonies isolées ont été inoculées dans 5 ml de LB contenant 50 µg/ml d'Amp et incubées pendant une nuit à 37 °C, 200 tr/min. 100 µL de cellules BL21 (DE3) à 360 ng de pETMK0016Duet-1 et 500 ng de pRSFIscDuet-1 (voir protocole de transformation au paragraphe I.1.5) et étaler le produit de transformation sur une plaque LB-Agar (Sigma) contenant de l'Amp.
Biochimie
Protéine HmdB II.1.1) Surexpression
1.2) Purification de la protéine HmdB de Methanopyrus kandleri
La protéine est concentrée dans la membrane cellulaire Amicon 30 K. Un test au Rose Bengale a été effectué pour estimer la concentration en protéine, suivi d'un test en fer pour estimer la teneur en fer de la protéine. La protéine est séparée en parties et congelée dans de l'azote liquide. II.1.3) Test de purification de la protéine HmdB de Methanococcus jannaschii.
1.3) Essai de purification de la protéine HmdB de Methanococcus jannaschii
Complexe IscSU
La purification de IscS et du complexe (IscS-D35A-IscU)2 a été réalisée en boîte à gants en conditions anaérobies dans le laboratoire du Professeur. Nous avons obtenu les structures du complexe (IscS-D35A-IscU)2 traitées au DTT (DT ), exposé à l'air (AE) et non traité (UT).
2.2) Calculs théoriques
Dosage au rose bengal et en fer II.3.1) Dosage au rose bengal
La concentration en protéines dans la solution a été déterminée par le test colorimétrique au rose Bengale (Elliott et Brewer 1978), une méthode rarement utilisée. Voici le principe du dosage du Rose Bengale : la concentration en protéines est déterminée à l'aide de
3.2) Dosage en fer
Analyse par électrophorèse sur gel de polyacrylamide
Dans le cas du SDS-PAGE, les échantillons ont été traités avec du dodécylsulfate de sodium (SDS) et un agent réducteur (-mercaptoéthanol) puis chauffés à 98°C pour dénaturer la protéine et réduire ses éventuels ponts disulfure. Dans le cas d'une SDS-PAGE non réductrice, les échantillons ont été traités avec du dodécylsulfate de sodium (SDS), puis chauffés à 98°C pour dénaturer la protéine.
Cristallogenèse et cristallographie des protéines
Cristallogenèse
Dans le cas d’une expérience où le rapport protéine/précipitant était de deux au départ, le volume de la gouttelette à l’équilibre est environ divisé par deux, ce qui implique que la concentration de la protéine double également. On peut décrire un diagramme de phases montrant la solubilité de la protéine en fonction de la concentration du précipitant (Figure III-2).
Cristallographie aux rayons X
2.1) La diffraction des rayons X par un cristal
En fonction de la direction de diffraction par rapport au réseau cristallin, la sphère d'Ewald intercepte différents nœuds du réseau réciproque. Figure III–3 Phénomène de diffraction selon les lois de Laue (a) et de Bragg (b). a) Sphère d'Ewald de rayon 1/ et de réseau inversé d'origine O. Le point P touche la sphère d'Ewald, il y a diffraction.
2.2) L’enregistrement et le traitement des données de diffraction
Cela implique une mise à l'échelle des données, ce qui réduira généralement la différence entre les intensités de réflexions équivalentes. Après mise à l'échelle, les intensités moyennes peuvent être calculées à partir de mesures d'intensités ponctuelles équivalentes.
2.3) Qualité d’un jeu de données
Lorsque des jeux de données sont enregistrés à l'ESRF, les réflexions équivalentes ne recevront pas exactement la même intensité de rayons X si l'expérience dure plusieurs heures, en raison de l'atténuation de l'intensité du faisceau incident, leur diffraction ponctuelle n'aura donc pas nécessairement la même intensité. même intensité. Ce rapport diminue avec l'augmentation de la résolution. permet de comparer l'intensité de chaque réflexion équivalente Ihkl avec la valeur moyenne .
2.4) Estimation du nombre de molécules par unité asymétrique
2.5) Remplacement moléculaire
Fonction de rotation R : la recherche par rotation selon les angles d'Euler est régie par la plus grande probabilité des intensités, donc des amplitudes. Une première recherche rapide est effectuée par la fonction LERF (Likelihood-Enhanced fast Rotation Function), qui est une simplification de la fonction de rotation.
2.6) La diffusion anomale
Le LLG (Log Likelihood Gain) est une indication de la qualité de la solution par rapport à une solution aléatoire. FP : facteur de structure des diffuseurs normaux ; FH : partie réelle (f0+f') du facteur de diffusion du diffuseur déviant.
2.7) La résolution de la structure de la protéine
Le diagramme de Ramachandran (Ramachandran, Ramakrishnan et al. 1963) permet également d'évaluer la qualité stéréochimique du modèle. Lors de la reconstruction graphique de la molécule, comme lors de l'interprétation finale, le modèle est visualisé sous forme de cartes de densité électronique.
Spectroscopies
- Séquençage N-terminal de protéine
- Spectroscopie UV-visible
- Spectroscopie de résonance paramagnétique électronique (RPE)
- Spectroscopie Raman de résonance
La diffusion Raman correspond à l'interaction de la polarisabilité de la molécule avec le rayonnement incident, entraînant un moment dipolaire induit dans la molécule et l'émission d'un photon Raman. Un spectre Raman présente l'intensité Raman diffusée en fonction de la différence de fréquence entre photon incident et photon diffusé : si l'on considère les raies de Stokes, cette différence est positive, et nulle dans le cas de la diffusion Rayleigh.
Etude structurale de la protéine HmdB
- Clonage du gène, expression et purification de HmdB native de Methanopyrus kandleri (MkHmdB)
- Essais de cristallisation
- Résolution de structure de HmdB
- Analyse structurale de la protéine HmdB I.4.1) Résidus non visibles dans sa structure
Le gène de la protéine HmdB de Methanopyrus kandleri a été cloné dans un plasmide pEtDuet-1 pour produire le plasmide pETMK0016Duet-1. Des tests de cristallisation de la protéine HmdB ont été effectués avec la protéine à 5 mg/mL.
4.1) Les limites du modèle de la protéine HmdB
Ils ont une conformation similaire en HydE et BioB. La conformation des résidus visibles de HmdB est similaire à celle de BioB et HydE. La conformation des résidus visibles de HmdB est similaire à celle de HydE et BioB. Tableau I–3 Comparaison de la conformation des résidus situés dans la partie intermédiaire entre le haut et le bas des structures en tonneau de HmdB, BioB et HydE. Afin de définir les résidus dont la conformation serait identique à la structure holoprotéique.
4.2) Agencement spatial des motifs conservés des protéines à radical SAM dans MkHmdB
Empilement cristallin de MkHmdB
Comme vous pouvez le voir sur la figure I-19 (a), à droite, les boucles BioB ou HydE ont de mauvaises interactions les unes avec les autres. ZZa Figure I–19 Empilement cristallin de MkHmdB, boucles non visibles et superposition sur les structures de BioB et HydE.
Elaboration d’un mutant C-terminal et changement d’organisme
Spectres UV-visible de la protéine avant (en rouge, courbe normalisée à 280 nm par rapport à la courbe bleue) et après reconstitution (en bleu). La protéine éluée de la colonne SP-Sepharose et SDS-PAGE a été réalisée sur les différents pics (non représentés).
Conclusion et perspectives
La première tentative d’utilisation de ce protocole a abouti à l’échec de l’élution de la protéine de la colonne SP-Sepharose. Les parties manquantes suivantes de la protéine pourraient être modélisées à l’aide des structures de BioB et HydE.
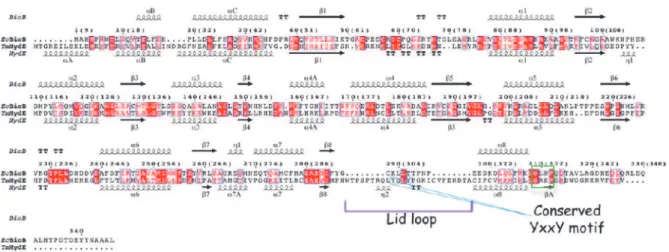
Etude structurale du complexe IscS-IscU
Gènes d’Archaeoglobus fulgidus codant pour IscS et IscU
Les protéines codées par des gènes proches de AfIscS et IscU ont été identifiées à l'aide d'un outil de recherche de domaines protéiques conservés (CDD) (Marchler-Bauer, Lu et al. 2011). -helix elbow (Luscombe, Austin et al. 2000)) est un régulateur transcriptionnel (Wood, Devine et al. 1990).
Structure de AfIscS
2.1) Caractérisation de AfIscS
La solution contenant AfIscS est incolore, alors qu'elle est jaune dans le cas d'une cystéine désulfurase classique. La mesure de l'activité cystéine désulfurase revient à mesurer la formation de produit au cours du temps, c'est à dire de L-alanine ou d'ions sulfure.
2.2) Cristallisation de AfIscS
L'activité de la cystéine désulfurase AfIscS est de 0,00173 nmole d'alanine/min/mg d'enzyme, ce qui est donc très faible (des tests d'activité ont été réalisés par un laboratoire participant). Il a été constaté que la cystéine désulfurase SufS (de la machinerie SUF) multipliait par 30 sa faible activité en présence de son partenaire protéique SufE (Ollagnier-De-Choudens, Lascoux et al. 2003).
2.3) Structure de AfIscS
Structure du complexe Af(IscSU) 2
3.1) Caractérisation de AfIscSU
Le spectre UV-visible du complexe AfIscSU avant traitement (courbes rouges sur la Figure II-13) montre les bandes caractéristiques de la présence de PLP à 330 nm (déjà observées pour IscS seul) et des centres [2Fe-2S]- autour de 400 nm. . De même, l’exposition à l’air après ou non un traitement DTT ne modifie pas le profil du spectre UV-visible même après deux jours (Figure II-13).
3.2) Cristallisation du complexe AfIscSU
Le test de reproductibilité par technique de chute suspendue à l'aide des boîtes EasyXtal (132023, Qiagen, puits à vis) n'a pas abouti. Une troisième forme cristalline a été obtenue dans notre laboratoire de BAG dans les mêmes conditions que les cristaux de la forme DT mais avec la protéine purifiée en conditions anaérobies sans traitement par un agent réducteur.
3.3) Résolution de structure de AfIscSU
La structure du complexe AE a été résolue à une résolution de 2,75 Å par remplacement moléculaire avec le programme PHASER (McCoy, Grosse-Kunstleve et al. 2007), en utilisant la structure haute résolution d'AfIscS précédemment résolue en laboratoire et la structure IscU d'Aquifex. aeolicus (code PDB 2z7e) (Shimomura, Wada et al. 2008) comme modèles de recherche (2 répétitions de chacun, selon le calcul des coefficients de Matthews (Matthews 1968)). La construction a été réalisée avec le logiciel graphique COOT (Emsley, Lohkamp et al. 2010) et le raffinement avec les logiciels AUTOBUSTER (Blanc, Roversi et al. 2004) et REFMAC5 (Murshudov, Vagin et al. 1997).
3.4) Structure de IscSU exposée à l’air (AE)
La position de ce pic de dispersion anormale coïncide avec celle de la carte de densité électronique à coefficients (Fo-Fc) (Figure II-16 A). La coordination µ2-T3 des ions fer avec le ligand persulfure est similaire à celle observée pour un centre fer-molybdène-soufre dans un composé modèle de la nitrogénase FeMoco (Kovacs, Bashkin et al. 1985) (Figure II-17(c)) .
3.5) Structure de IscSU traitée au DTT
Bien que le complexe semble stable dans l'air (d'après nos expériences de spectroscopie UV-visible (II.3.1)) et (Raulfs, O'Carroll et al. 2008), la forme AE semble être deux électrons plus oxydés qu'une forme standard [2Fe- 2S] centre. La présence d'un centre [2Fe-2S] sur le site d'assemblage est évidente à partir de la forme de la densité électronique (Figure II-19).
3.6) Interface entre IscS et IscU
Superposition de la forme DT (rose) sur le complexe IscSU de Ec (vert) (code PDB 3lvl). La forme DT diffère de la forme AE par la liaison hydrogène entre le groupe carbonyle de Cys33 de IscU et le résidu His343 de IscS.
3.7) Forme UT
La flèche rouge indique le pic positif de la différence de Fourier proche de Cys33, indiquant la position du soufre de cette dernière, invisible dans le modèle de la forme AE. De plus, ces changements ont amené du désordre sur le site d’assemblage du centre FeS puisque l’atome de soufre de Cys33 n’est pas visible.
3.9) Calculs théoriques
Figure II–23 Superposition de la forme DT sur le modèle MQ/MM optimisé où Ala35 a été remplacé par Asp. Lors de la dissociation d'IscS d'IscU, le ligand central Cys321 doit être remplacé.
3.10) Spectres RPE et Raman
Les cristaux de la protéine prétraitée avec 5 mM de DTT (Figure II–.27) présentent les bandes caractéristiques d'un centre [2Fe-2S] (Tableau II-3). -12 : cristaux de protéines prétraités dans du DTT (DT) 5 mM ; cris07 : cristaux de protéines sans traitement DTT (UT).
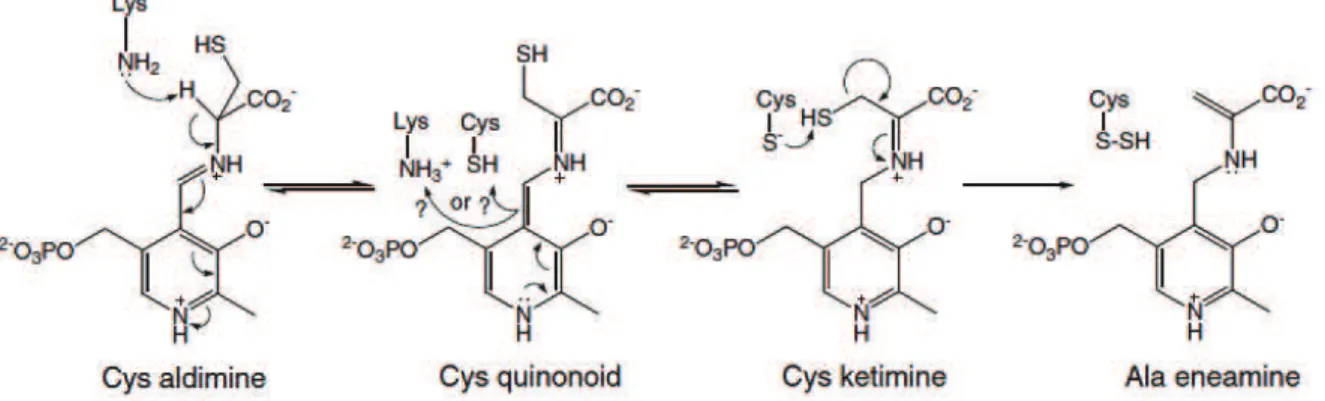
3.11) Proposition d’un mécanisme de formation des centres FeS
Conclusion et perspectives
Le thiolate Cys321 est proche (2,5 Å) de l'un des atomes de soufre du ligand sulfido. Les calculs théoriques confirment la possibilité de remplacement du ligand Cys321 par Asp35 lors de la dissociation du complexe IscS-IscU.