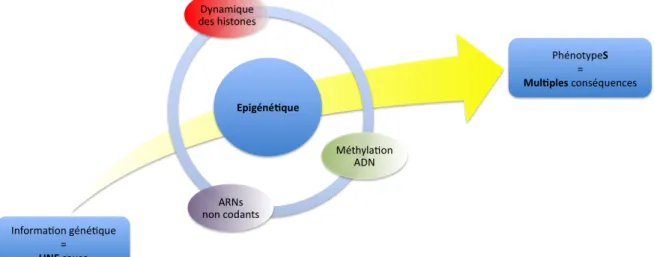
Les mécanismes épigénétiques sont principalement les histones et leur dynamique au sein de la cellule, la méthylation des ARN non codants et de l'ADN. L'épigénétique apporte un niveau supplémentaire de régulation du fonctionnement de notre génome et contribue ainsi à la compréhension de la complexité de l'espèce humaine.

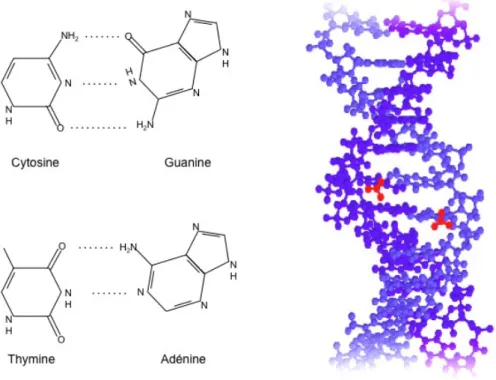
L’ADN support de l’information génétique
Les mécanismes impliqués
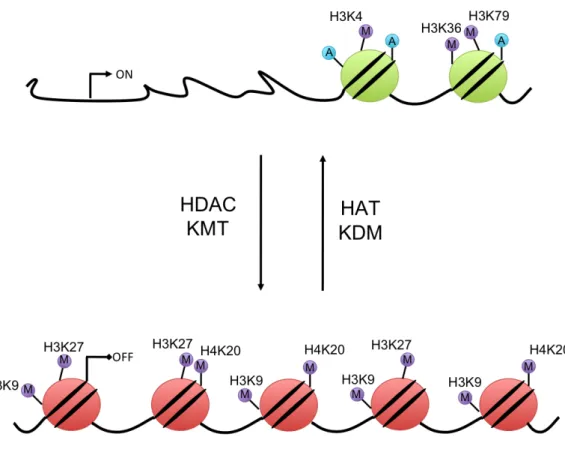
De plus, la méthylation de l'ADN et la variante de l'histone H2A.Z s'excluent mutuellement (Zilberman et al. 2008). De plus, le traitement des lignées cellulaires cancéreuses avec des inhibiteurs d'HDAC tels que le Valproate (VPA) ou la Trichostatine A (TSA) entraîne une diminution de la méthylation de l'ADN (Milutinovic et al.
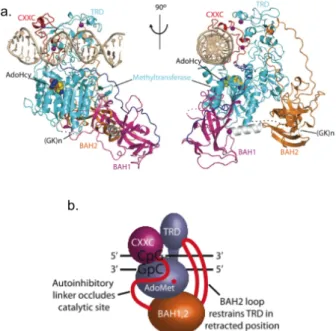
Mise en place et maintien de la méthylation de l’ADN .1'Famille'des'ADNZméthyltransférases'
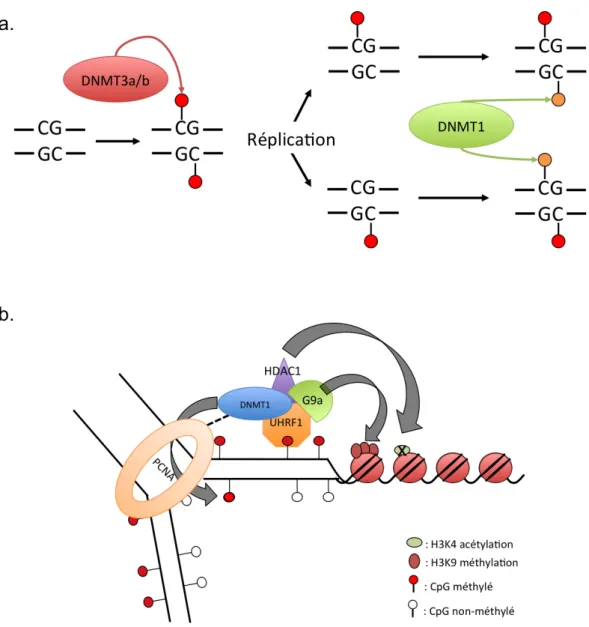
La génération de mutants de souris pour le domaine catalytique de DNMT1 confirme l'importance de la méthylation de l'ADN (Takebayashi et al. 2007). L'interaction entre DNMT3L et DNMT3A et 3B permet l'établissement de la méthylation de l'ADN (Hata et al. 2002).
Interprétation de la méthylation de l’ADN
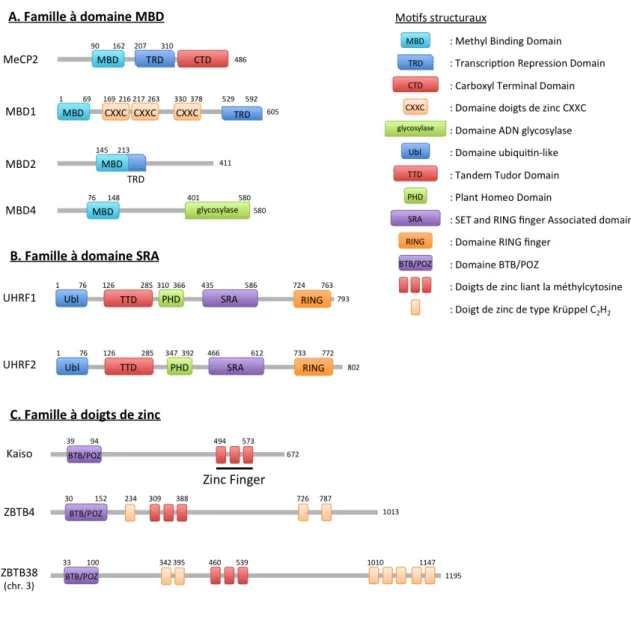
Plus précisément, il intervient dans le système de réparation des mismatch (Mismatch Repair, MMR) via son interaction avec MutL Homolog 1 (MLH1) (Bellacosa et al. 1999). Contrairement aux autres MBP, Kaiso (ainsi que d'autres protéines à doigts de zinc) se lie non seulement aux CpG méthylés, mais également à une séquence spécifique non méthylée appelée KBS (site de liaison Kaiso) (Daniel et al. 2002).
Distribution de la méthylation de l’ADN
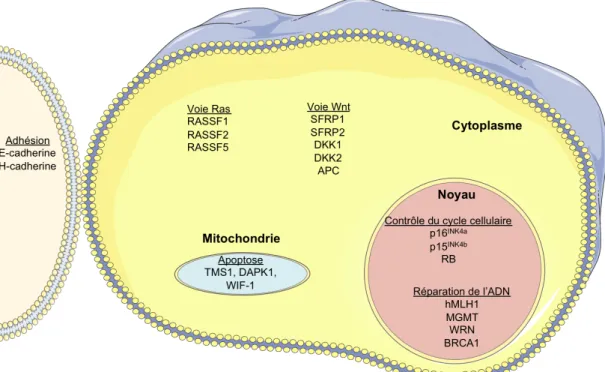
Confirmant également des données connues de longue date, les profils de méthylation montrent une hypométhylation et une hyperméthylation à l'échelle du génome des gènes suppresseurs de tumeurs dans le cancer. Non seulement les gènes imprimés, mais aussi environ 10 % des gènes humains sont régulés de cette manière (Zhang et al. 2009).
La méthylation de l’ADN au cours du développement
La méthylation de l’ADN au cours du développement
C'est notamment le cas des gènes à empreinte (Olek et al. 1997), des régions répétées centro- et péricentromériques (Rougier et al. En effet, l'isoforme DNMT1 présente dans l'ovocyte est séquestrée dans le cytoplasme jusqu'au stade 8 cellules (Carlson et al. 1997). al. .1992) empêche le processus de maintien de la méthylation de l'ADN.
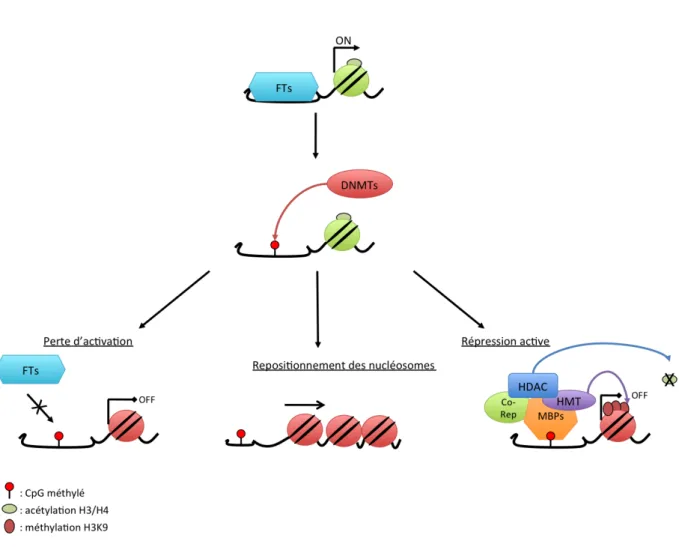
Contrôle de l’expression de certains gènes tissu-spécifiques Les tissus somatiques possèdent des profils d’expression uniques
Contrôle de l’expression des éléments répétés
Cette distribution est conservée au cours de l'évolution et reflète probablement une fonction ancestrale de méthylation de l'ADN (Feng et al. Bien que surprenantes, ces données sont cohérentes avec l'enrichissement de la marque H3K9me3 dans le corps génétique (Vakoc et al. 2005).

L’empreinte parentale
De plus, la méthylation de l’ADN pourrait permettre la modification des histones et changer la position des nucléosomes. Des processus de réparation de l'ADN sont alors nécessaires pour restaurer une cytosine (Popp et al. 2010).
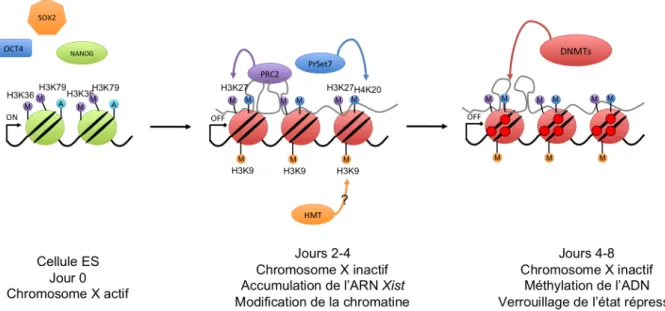
Inactivation du chromosome X
Méthylation de l’ADN et maladies humaines
Les défauts sont essentiellement génétiques mais dans près de 5 % des cas il s'agit d'une perte de méthylation de l'allèle maternel (Lossie et al. 2001). De plus, la réduction du nombre de répétitions est associée à l'hypométhylation des éléments D4Z4 (van Overveld et al. 2003). C'est le cas du syndrome ICF (Immunodéficience, Instabilité Centromérique et Anomalies Faciales) qui est une maladie autosomique extrêmement rare caractérisée par un déficit en immunoglobulines et un dimorphisme facial (Smeets et al. 1994).
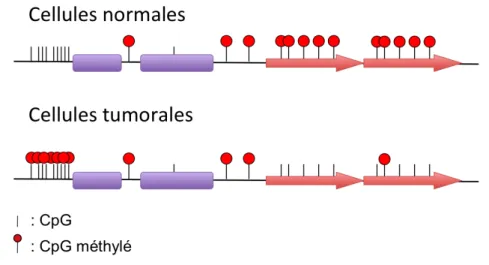
Les cellules cancéreuses ont un niveau de méthylation totale réduit par rapport aux cellules saines (Goelz et al. 1985). En effet, de nombreux îlots CpG (CGI) correspondant aux promoteurs de gènes sont hyperméthylés dans les cellules cancéreuses (Ribieras et al. 1994). D'autre part, les tumeurs sont également caractérisées par une hyperméthylation des miARN, conduisant à une absence d'expression (Saito et al. 2006).
Hydroxyméthylation
Cependant, l’hydroxyméthylation ne permet pas de prédire si la transcription est active ou inactive. D'une part, 5hmC peut être associé à des marques de transcription actives dans les régions hypométhylées. En effet, l'anti-relation entre 5hmC et la méthylcytosine au niveau des promoteurs suggère que l'hydroxyméthylation pourrait renforcer l'inhibition de la méthylation de l'ADN.
La question de la reconnaissance de la 5hmC par des protéines spécialisées s’est rapidement posée, car leur découverte permettrait de mieux comprendre le rôle biologique de la 5hmC. En revanche, il a récemment été démontré que UHRF1 était capable de reconnaître 5hmC via son domaine SRA (Frauer et al. 2011). Tet1 peut convertir la méthylcytosine en 5hmC, qui s'est avéré être un substrat pour la voie BER dans la réparation de l'ADN et conduit à la déméthylation (Guo et al. 2011).
- Un dogme à réévaluer
- Les protéines liant l’ADN méthylé ne sont pas essentielles .1'Redondance'des'MBPs'
- Cibles des MBPs
- MBPs : activateurs transcriptionnels ?
Une approche expérimentale qui pourrait résoudre cette question consisterait à obtenir une carte à l’échelle du génome de tous les MBP dans le même type de cellule. Les profils de méthylation de l'ADN à l'échelle du génome mettent en évidence que dans les cellules de mammifères normales, les promoteurs liés au CGI (HCP) ne sont pas méthylés et ne représentent donc pas un site de liaison pour le MBP. Les MBP semblent être de bons candidats dans le processus de répression de ces gènes, mais cela ne se produit pas.
Les MBP pourraient agir sur d’autres régions du génome et notamment sur les CGI « côtiers » qui représentent différents niveaux de méthylation entre tissus sains. Enfin, les corps génétiques sont fortement méthylés et représentent donc une cible potentielle pour la MBP. Bien que le mécanisme soit indirect, MBD4 est nécessaire au processus d'activation transcriptionnelle grâce à son rôle dans la déméthylation de l'ADN (Rai 2008).
RECHERCHE DE NOUVELLES PROTEINES HUMAINES SE LIANT A L’ADN
METHYLE
- Approche génétique
- Approche basée sur la littérature
Cette approche s'inspire d'anciennes techniques qui ont fait leurs preuves (Singh et al. 1988) et a récemment été utilisée pour identifier des facteurs de transcription qui se lient à une séquence consensus (Cicchini et al. 2002). Ainsi, un article récent a identifié les protéines nécessaires à la méthylation aberrante du promoteur Fas après la transformation de cellules avec H-ras (Gazin et al. 2007). Les expériences d'immunoprécipitation de la chromatine (ChIP) montrent que certaines de ces protéines se lient spécifiquement au promoteur méthylé mais pas au promoteur non méthylé.
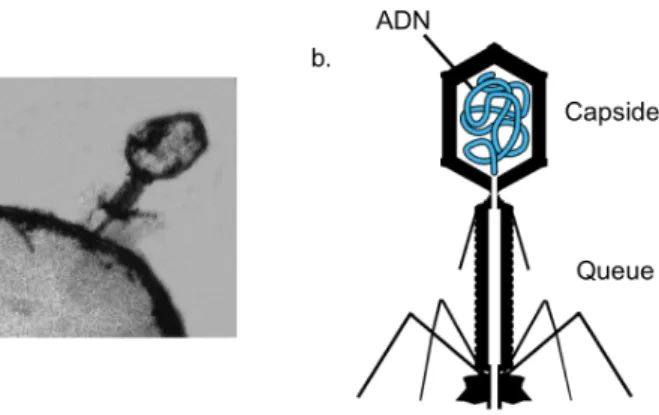
La méthylation de l'ADN contrôle de manière cruciale l'activité du génome dans les cellules normales et sa perturbation contribue à la transformation tumorale. En étudiant les protéines qui se lient à l’ADN méthylé chez l’humain, nous espérons, à terme, mieux comprendre les premières étapes de la transformation afin de pouvoir les détecter et les inhiber. Il possède une queue qui lui permet de s'attacher aux bactéries et d'injecter son ADN comme une seringue.
- Introduction
- Principe du crible par phage display
- Développement du crible par phage display
- Résultats des cribles par phage display
- Discussion
- Matériels et méthodes Hybridation des sondes
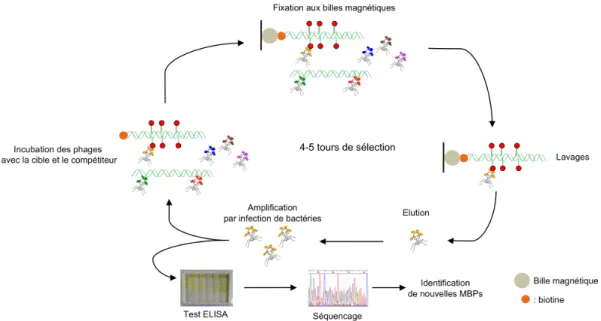
Premièrement, le phage positif a une plus grande affinité pour l’ADN méthylé que pour la matrice seule. Sans concurrent, le phage positif est rapidement sélectionné en présence d'ADN non méthylé et seulement après trois tours avec de l'ADN méthylé. Les phages présentant les interactions les plus fortes spécifiquement avec l’ADN méthylé sont répertoriés ensuite.
Il est donc très peu probable que l’enrichissement en UBTF soit spécifique à la méthylation de l’ADN. Il a été proposé que le complexe NuRD soit recruté par reconnaissance de l'ADN méthylé. Nous avons cherché à développer une présentation sur phage pour identifier les protéines qui se lient à l'ADN méthylé.
- Introduction
- Résultats
- Discussion
- Matériels et méthodes
B : ZBTB4, GFP, Kaiso et les protéines candidates ont été exprimées en fusion avec GFP dans des cellules p53-/-MEF. La GFP est diffuse dans toute la cellule et ne chevauche pas les chromocentres. Cela signifie que la présence de cette protéine au centromère est sans doute indépendante de la méthylation de l’ADN.
Les différents domaines de BAZ2A ont été exprimés en fusion avec la GFP dans des cellules p53-/- MEFs. BAZ2A se localise avec les chromocentres dans les cellules transfectées par lacZ et JMJD2D malgré la perte de H3K9me3. Dix-huit heures après transfection, les cellules sont lavées dans du PBS et fixées avec du PFA 2% pendant 10 minutes à température ambiante.
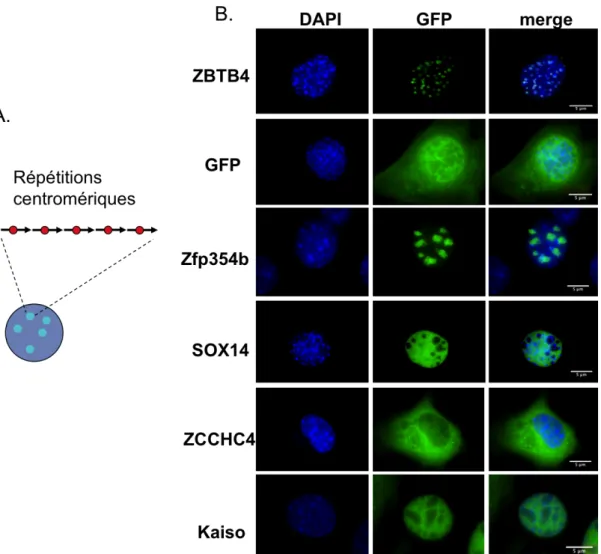
Régulation des éléments répétés par ZBTB4 Chapitre 1 : introduction
- Analyse d’expression des éléments répétés
- Localisation de ZBTB4 in vivo
- Discussion
- Matériels et méthodes Anticorps et plasmide
Chez les souris délétées pour Zbtb4, selon les résultats de PCR semi-quantitative, aucun élément ne semble réexprimé, quel que soit le tissu testé (ex : cœur, muscle et rate). Dans un premier temps, nous avons testé la liaison de la protéine endogène Zbtb4 avec les satellites mineurs et majeurs, à l'aide de l'anticorps #120 (figure 36). Parallèlement, nous avons observé un enrichissement en grands et petits satellites en immunoprécipitations de la marque histone H3K9me3.
De même, l'expression des éléments Charlie, LINE et SINE est limitée au rein. Globalement, nous chercherons à montrer que Zbtb4 contrôle l'expression des petits satellites afin de réguler cette structure essentielle à la ségrégation des chromosomes lors de la mitose et. Sachant cela, nous étudierons la présence éventuelle de défauts de division lors de la mitose chez la souris Zbtb4-/-.
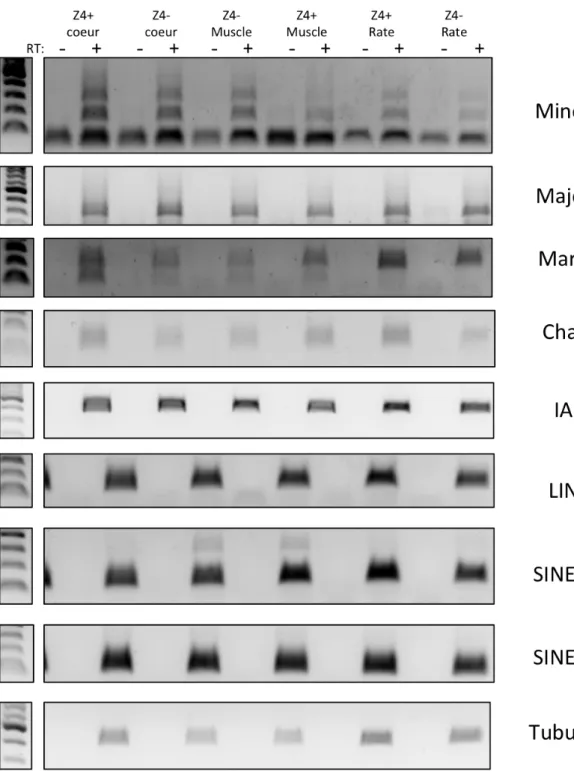
Deux candidats, CHD3 et HMGB1, sélectionnés pour se lier à l'ADN méthylé, semblent intéressants en raison de leurs fonctions respectivement dans la maintenance des centrosomes et dans le remodelage de l'ADN. L'expression de plusieurs éléments répétés a été analysée par PCR quantitative dans le foie de souris sauvages et mutantes ZBTB4. L'expression des petits satellites est respectivement multipliée par 2 et 5 dans le foie des souris mutantes ZBTB4.
L'épigénétique.' '
Avancées et limites de la génétique
Les phénomènes épigénétiques
Conséquences pratiques de l'épigénétique
Conclusion et perspectives
Empreinte parentale
Les souris Agouti
Les mécanismes épigénétiques impliquent les histones, les ARN non codants et la méthylation de l'ADN. Il s’ensuit que nos différentes cellules partagent le même patrimoine génétique, mais pas forcément le même phénotype, et que les individus possédant le même génome peuvent avoir des caractéristiques très différentes. Avec l’avènement de l’épigénétique, les chercheurs se rendent compte que l’environnement peut agir non seulement au niveau génétique, mais aussi au niveau épigénétique.
Les facteurs environnementaux vont perturber l’activité du génome via des mécanismes épigénétiques qui induisent des caractéristiques anormales.
Bibliographie
Methyl-CpG-binding domain 1 (MBD1) interacts with the Suv39h1-HP1 heterochromatic complex for DNA methylation-based transcriptional repression. Involvement of DNA methylation in the control of the expression of an estrogen-induced breast cancer-associated protein (pS2) in human breast cancers. Solution structure of the methyl-CpG binding domain of human MBD1 in complex with methylated DNA.
Genome-wide erasure of DNA methylation in mouse primordial germ cells is affected by AID deficiency. DNMT1, but not its interaction with the replication machinery, is required for the maintenance of DNA methylation in human cells. Analysis of the NuRD subunits reveals a histone deacetylase core complex and a link to DNA methylation.