DETEÇÃO E IDENTIFICAÇÃO RÁPIDAS DOS PRINCIPAIS
CONTAMINANTES MICROBIOLÓGICOS EM FÁRMACOS
POR ESPETROSCOPIA DE INFRAVERMELHO
Joana Anjos Ferreira
Dissertação para obtenção do grau de mestre em Engenharia Farmacêutica
Orientador: Professor Doutor João Almeida Lopes, Faculdade de Farmácia Co-orientadora: Sofia Queirós, Generis Farmacêutica SA
FACULDADE DE FARMÁCIA
UNIVERSIDADE DE LISBOA
DETEÇÃO E IDENTIFICAÇÃO RÁPIDAS DOS PRINCIPAIS
CONTAMINANTES MICROBIOLÓGICOS EM FÁRMACOS
POR ESPETROSCOPIA DE INFRAVERMELHO
Joana Anjos Ferreira
Dissertação para obtenção do grau de mestre em Engenharia Farmacêutica
Orientador: Professor Doutor João Almeida Lopes, Faculdade de Farmácia Co-orientadora: Sofia Queirós, Generis Farmacêutica SA
Resumo
A espetroscopia de infravermelho com transformada de Fourier com refletância total atenuada (FTIR-ATR) combinada com a análise multivariada de dados tem vindo a ser proposta como um método para a discriminação de bactérias patogénicas a diferentes níveis taxonómicos (p.e., espécie, sub-espécie e serotipo). O controlo microbiológico na indústria farmacêutica é um fator extremamente importante para garantir a segurança e eficácia de um medicamento. Durante a análise microbiológica de um produto, pode observar-se crescimento de microrganismos provenientes de várias fontes e é necessário assegurar a qualidade do produto fazendo a sua deteção e identificação. O principal objetivo deste trabalho é avaliar o potencial da espetroscopia FTIR-ATR combinada com técnicas quimiométricas para diminuir o tempo de identificação de microrganismos em medicamentos (formas sólidas). Esta metodologia foi testada com Escherichia coli, Staphylococcus aureus e Pseudomonas
aeruginosa. Foram utilizados 52 isolados destas bactérias recolhidos do ambiente e de
fármacos não estéreis. Estes foram identificados pelo método VITEK implementado na Generis Farmacêutica SA e em seguida analisados por FTIR-ATR. Após o tratamento espectral foram construidos modelos de análise de componentes principais (PCA) e mínimos quadrados parciais de tipologia discriminante (PLS-DA). O método FTIR-ATR permitiu distinguir corretamente bactérias gram-negativas de gram-positivas, assim como discriminar espécies do género Staphylococcus. Quanto ao género Pseudomonas, apesar de se ter considerado um número muito reduzido de isolados, foi possível identificar o correto agrupamento de cada espécie nos modelos PCA e de agrupamento hierárquico. Relativamente ao género Escherichia, não se verificou a presença de microrganismos correspondentes. Dos dois grupos de microrganismos em estudo (Staphylococcus e Pseudomonas) foi possível observar agrupamentos correspondentes aos principais contaminantes patogénicos. Este resultado confirma o potencial do método FTIR-ATR neste campo de utilização mostrando a sua aplicabilidade num laboratório microbiológico de um laboratório farmacêutico.
Palavras-chave: FTIR-ATR; contaminação microbiológica; Staphylococcus; VITEK; análise
multivariada.
Abstract
Attenuated total reflection Fourier transform infrared (ATR-FTIR) spectroscopy combined with multivariate data analysis has been proposed as a taxonomic method used to classify and distinguish pathogenic bacteria at multiple levels (for example, species, subspecies and serotype). The microbial contamination control in the pharmaceutical industry is a key factor to be considered in order to assure the safety and efficacy of a drug. During microbiological analysis of a drug, the growth of several microorganisms can be observed. Therefore it is crucial their identification and classification to safeguard the quality of the product. The aim of this work is to propose and describe a fastest method based on FTIR spectroscopy and chemometric techniques to be used in the microbiological analysis of a drug (solid form). This methodology was applied to distinguish different microorganisms, namely Escherichia
coli, Staphylococcus aureus and Pseudomonas aeruginosa. The fifty-two bacterial isolates
used in this methodology were collected from the environment and from the microbiological analysis of non-sterile drugs. They were identified according to the reference method (VITEK) used in Generis Farmacêutica SA, and the spectral analysis of each isolate was performed by ATR-FTIR spectroscopy. PCA and PLS-DA models were correspondingly constructed, and the two methods were compared. The results obtained revealed that both methods are in agreement. The FTIR method correctly distinguished gram-negative from gram-positive bacteria. It was also able to distinguish species belonging to the Staphylococcus genus. Regarding the Pseudomonas genus although it was considered a reduced number of samples, it was possible to identified accurately group each species when performing the PCA and HCA analysis. Concerning the Escherichia genus, no microorganisms were found. For the two groups of microorganisms considered in this study, well-defined clusters of the main pathogens were identified: S.aureus and P.aerugionosa. Overall, the results of this study demonstrate that ATR-FTIR spectroscopy combined with chemometrics serves as a valid method to be considered in a microbial laboratory of a pharmaceutical company.
Key-words: FTIR-ATR; microbiological contamination, Staphylococcus; VITEK;
Agradecimentos
Gostaria de agradecer a todos aqueles que contribuíram de forma direta ou indireta para a realização desta dissertação.
Ao meu orientador, Professor Doutor João Almeida Lopes pela orientação e conselhos prestados para a realização desta dissertação e por mostrar sempre disponibilidade.
À Dr.ª Sofia Queirós e à Generis por aceitarem este projeto e me disponibilizarem todos os meios para o realizar.
Um agradecimento especial à Ana Paula Grancho cuja ajuda foi imprescindível e por sempre se mostrar disponível para ajudar no que fosse preciso.
À Dr.ª Madalena Pimentel pelo apoio e acompanhamento demonstrado.
À Dr.ª Clara Sousa pelo tempo disponibilizado para esclarecer as dúvidas encontradas desde o início deste projeto.
À Bárbara cujos conselhos e disponibilidade foram uma mais-valia ao longo de todo o trabalho.
Ao meu irmão e aos meus pais cujo apoio foi fundamental a todos os níveis. Sem eles não teria sido possível.
Aos meus amigos Alexandra, Maria e Marcelo por todo apoio e ajuda neste trabalho, como em todos os momentos da minha vida.
Ao Paulo por toda a paciência, pelo apoio incondicional e por nunca me deixar desanimar.
Por fim, resta-me agradecer a todos aqueles que, de alguma forma, contribuíram para a realização desta dissertação e para terminar esta etapa da minha vida.
Índice
1. Introdução ... 9
1.1. Análise microbiológica de produtos não estéreis ... 9
1.2. Microrganismos relevantes ... 10
1.2.1. Enterobacteriaceae ... 10
1.2.2. Pseudomonas ... 11
1.2.3. Staphylococcus ... 11
1.3. Identificação de contaminantes microbiológicos ... 13
1.3.1. Métodos moleculares/biológicos ... 13
1.3.2. Métodos bioquímicos ... 13
1.3.3. Métodos espetroscópicos ... 16
1.3.4. FTIR - ATR para identificação de microrganismos ... 19
1.4. Análise Multivariada ... 22 1.4.1. Pré-processamento espetral ... 22 1.4.2. Técnicas quimiométricas ... 24 2. Revisão da literatura ... 28 3. Objetivos ... 29 4. Materiais e métodos ... 30 4.1. Isolados ... 30 4.2. Análises espetroscópicas ... 33 5. Resultados e discussão ... 35
5.1. Avaliação dos resultados pelo método VITEK ... 35
5.2. Discriminação pelo método FTIR ... 36
5.2.1. Distinção de microrganismos gram-positivos de gram-negativos ... 38
5.2.2. Distinção de microrganismos do género Pseudomonas ... 42
5.2.3. Distinção de microrganismos do género Staphylococcus ... 44
6. Conclusões e perspetivas futuras ... 62
Índice de Figuras
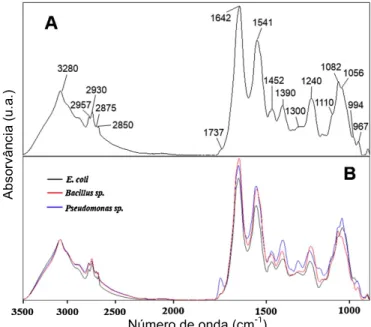
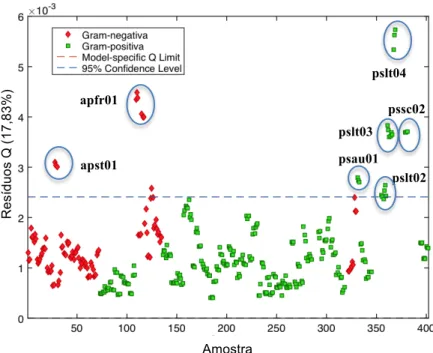
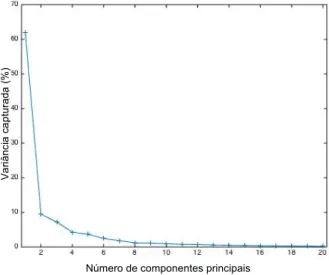
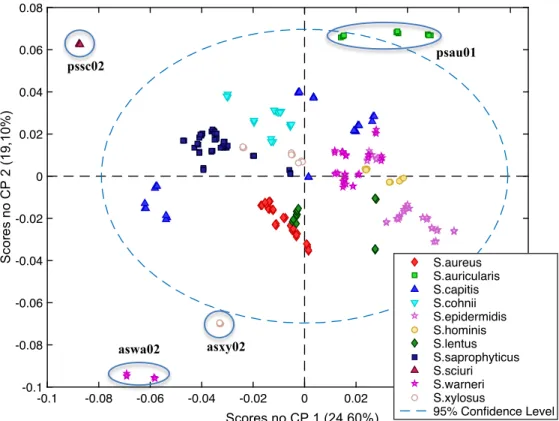
Figura 1. Kits que incluem 20 testes bioquímicos (adaptado de (39)). ... 14 Figura 2. Carta de identificação gram-positiva para o VITEK (adaptado de (41)). ... 15 Figura 3. Sistema VITEK adaptado de (41). ... 15 Figura 4. Esquema de um espetrómetro com interferómetro: F - Fonte de radiação IV; D - Divisor de feixe; E1 - Espelho fixo; E2 - Espelho móvel (Adaptado (52)). ... 17 Figura 5. Esquema de um cristal para refletância total atenuada (ATR) (Adaptado (56)). ... 18 Figura 6. Espetro FTIR representativo de bactérias com respetiva identificação das principais bandas (Adaptado de (64)). ... 22 Figura 7. Ilustração do método de PCA (adaptado de ((74)). ... 24 Figura 8. Ilustração de um modelo de PLS-DA com três classes em que o i é o número de amostras e o j o número de variáveis (Adaptado de (78)). ... 27 Figura 9. Espetros FTIR-ATR dos microrganismos padrão (E. coli, S. aureus e P. aeruginosa) sem pré-processamento. ... 37 Figura 10. Espetros dos microrganismos padrão de E. coli, P. aerugionosa e S. aureus pré-processados com SNV e Sav-Gol (9,2,2) na região 1500-900 cm-1. ... 38 Figura 11. Representação gráfica da variância capturada com o aumento do número de CPs.38 Figura 12. Mapa de scores dos dois componentes principais do modelo PCA de todos os microrganismos em que a vermelho estão representados os negativos e a verde os gram-positivos de acordo com a informação dada pelo VITEK. ... 39 Figura 13.Mapa de scores do modelo de PCA de todos os microrganismos após eliminar aswa02 e asxy02 ( modelo construído com 5 CPs). ... 40 Figura 14. Representação da estatística de Hotelling’s T2 do modelo de PCA de todos os espetros. ... 40 Figura 15. Representação da estatística de resíduos do modelo de PCA de todos os espetros. ... 41 Figura 16. Dendrograma feito com os todos os espetros onde estão representados os
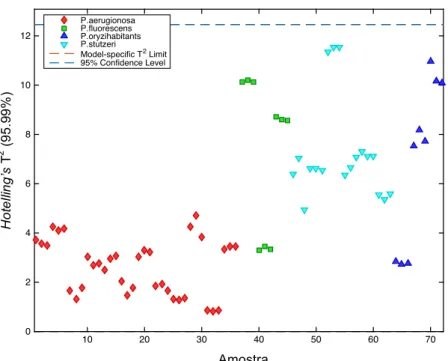
microrganismos gram-positivos a verde e os microrganismos gram-negativos a vermelho. ... 41 Figura 17. Mapa de scores da família Pseudomonas em que estão representados os CPs 1, 2 e 5. ... 42 Figura 18. Representação da estatística de resíduos para o modelo PCA das Pseudomonas. . 42 Figura 19. Representação da estatística de Hotelling’s T2 para o modelo de PCA do género Pseudomonas. ... 43
Figura 20. Dendrograma obtido da análise de HCA de espetros dos isolados pertencentes ao género Pseudomonas (foram usados 6 CPs). ... 43 Figura 21. Variância capturada em função do número de CPs para o modelo PCA dos
espetros de Staphylococcus. ... 44 Figura 22. Mapa de scores de todos os espetros de Staphylococcus com representação do 1º e 2º CPs. ... 45 Figura 23. Mapa de scores do modelo PCA dos Staphylococcus com representação do 1º e 2º CP. ... 46 Figura 24. Mapa de scores do modelo PCA dos Staphylococcus com representação do 1º e 3º CP. ... 46 Figura 25. Média dos espetros FTIR (1500-900 cm-1) de 3 isolados diferentes: S. aureus (vermelho), S. capitis (azul) e S. auricularis (verde). Espetros pré-processados com SNV e Sav-Gol (9,2,2). ... 47 Figura 26. Representação das estatísticas de Hotelling’s T2 e de resíduos para o modelo PCA do género Staphylococcus. ... 48 Figura 27. Dendrograma obtido da análise de HCA de espetros dos isolados pertencentes ao género Staphylococcus (foram usados 10 CPs). ... 48 Figura 28. Representação gráfica da percentagem de erro da validação cruzada em função do número de variáveis latentes. ... 49 Figura 29. Mapa de scores das variáveis latentes 1 e 2 do modelo PLS dos Staphylococcus. 50 Figura 30. Estatísticas de Hotelling’s T2 e de resíduos para o modelo PLS-DA dos
Staphylococcus. ... 51 Figura 31. Média dos espetros FTIR (1500-900 cm-1) de isolados de S. warneri (verde, rosa, preto e azul), comparado com o S. capitis (05)(vermelho). Espetros pré-processados com SNV e Sav-Gol (9,2,2). ... 52 Figura 32. Mapa de scores das variáveis latentes 1 e 2 do modelo PLS-DA de Staphylococcus. ... 53 Figura 33. Mapa tridimensional de scores com as variáveis latentes 1,2 e 3 para o modelo PLS-DA de Staphylococcus. ... 54 Figura 34. Representação das estatísticas de Hotelling’s T2 e de resíduos para o modelo PLS-DA de Staphylococcus. ... 55 Figura 35. Mapa de scores do modelo PLS-DA de previsão com os dados de teste (SA01). .. 55 Figura 36. Previsão da amostra de teste SA01 como membro da classe de S. aureus. ... 56 Figura 37. Previsão da classe da amostra para pssc02. ... 57
Figura 38. Probabilidade de previsão da classe de S. lentus na amostra pssc02. ... 57
Figura 39. Representação das estatísticas de Hotelling’s T2 e dos resíduos com a projeção da amostra desconhecida (pssc02). ... 58
Figura 40. Previsão da classe da amostra para pssc01. ... 58
Figura 41. Previsão da classe da amostra para aslt01. ... 59
Figura 42. Probabilidade de previsão da classe de S. lentus na amostra aslt01. ... 59
Figura 43. Previsão da classe da amostra para psvt02. ... 60
Figura 44. Previsão restrita da classe da amostra para asxy02. ... 60
Figura 45. Probabilidade de previsão da classe de S. xylosus para o isolado asxy02. ... 61
Índice de Tabelas
Tabela 1. Turbidez para preparação dos inóculos. ... 15Tabela 2. Atribuição do número de onda ao grupo funcional existente nas células microbianas (adaptado(61)). ... 20
Tabela 3. Revisão dos estudos publicados recentemente sobre identificação e discriminação de m.o. com o uso de espetroscopia. DA: análise discriminante; CVA: análise canónica variada; PAS: espetroscopia foto-acústica; CA: análise de clusters; SOM: (self-organized maps) mapas auto-organizados; KNN: (K-nearest neighbor); ANN: redes neuronais artificiais. ... 28
Tabela 4: Codificação e origem dos isolados utilizados neste estudo. ... 31
Tabela 5.Resultados das identificações dos isolados utilizados neste estudo pelo método VITEK. ... 36
Tabela 6. Resumo dos resultados das identificações do VITEK para Staphylococcus lentus. 51 Tabela 7. Resumo dos resultados das identificações do VITEK para Staphylococcus capitis. 52 Tabela 8. Estatísticas para cada classe do modelo PLS-DA de Staphylococcus. ... 54
Tabela 9. Resumo dos resultados do VITEK e da previsão pelo modelo PLS-DA das amostras utilizadas para testar o modelo. ... 61
Abreviaturas
ANN - Redes neuronais artificiais API - Princípio ativo
ATR - Refletância total atenuada CA - Análise de agrupamentos CPs - Componentes principais CVA - Análise canónica variada DA - Análise discriminante DNA - Ácido desoxirribonucleico EP - Farmacopeia Europeia FP - Farmacopeia Portuguesa
FTIR - Espetroscopia de infravermelho com transformada de Fourier GMP - Good Manufacturing Practices
HCA - Agrupamento hierárquico IV - Radiação de infravermelho KNN - K-nearest neighbors m.o.- microrganismo
MALDI-TOF - Matrix-assisted laser desorption/ionization-time of flight MRSA - Staphylococcus aureus resistentes à meticilina
MSC - Correção multiplicativa de sinal NID – Não identificado
PAS - Espetroscopia foto-acústica PCA - Análise de componentes principais PCR - Reação em cadeia da polimerase
PFGE – Eletroforese em gel de campo pulsado
PLS-DA - Regressão por mínimos quadrados parciais - discriminante RNA - Ácido ribonucleico
Sav-Gol - Filtro de Savitzky-Golay SNV- Variação de padrão normal SOM - Mapa auto-organizativo
TAMC - Contagem de microrganismos aeróbios TYMC - Contagem de fungos e leveduras UFC - Unidades formadoras de colónias USP - Farmacopeia Americana
1. Introdução
1.1. Análise microbiológica de produtos não estéreis
Na indústria farmacêutica, a análise microbiológica de medicamentos é uma das etapas que garante a qualidade do produto final. Os fabricantes têm de garantir uma baixa carga microbiana no produto acabado, sendo para isso necessária a implementação de boas práticas de fabrico (Good Manufacturing Practices - cGMP). Estas práticas aplicam-se aos procedimentos de produção, embalagem e distribuição dos fármacos. Deste modo, é fundamental a análise microbiológica de matérias-primas, produtos a granel e produto acabado, bem como da água purificada. A monitorização ambiental das áreas de produção e embalagem são também um fator de grande importância na qualidade dos medicamentos(1–3). Os métodos utilizados e os resultados obtidos devem estar em conformidade com as especificações e critérios descritos na Farmacopeia Europeia (EP). As análises realizadas em matérias-primas, produto a granel e produto acabado, envolvem contagem de microrganismos aeróbios totais (TAMC) e contagem de leveduras e fungos totais (TYMC). Estas contagens são expressas em unidades formadoras de colónias (UFC). Para além das análises referidas anteriormente, são feitas pesquisas a microrganismos específicos, que variam consoante a natureza das matérias-primas utilizadas e o método de produção. Normalmente os microrganismos (m.o.) específicos em estudo, na análise de produtos não estéreis, são
Staphylococcus aureus, Pseudomonas aeruginosa, Escherichia coli, Salmonella, e bactérias
gram-negativas tolerantes à bílis(4,5).
A comprovação da qualidade microbiológica de medicamentos existentes no mercado nacional é avaliada pela autoridade reguladora portuguesa (INFARMED). As indústrias farmacêuticas submetem ao INFARMED um dossier de Autorização de Introdução de Mercado (AIM) para cada produto, onde estão estabelecidas as especificações de análise. Estas podem variar consoante a natureza das matérias-primas utilizadas, o método de produção estabelecido, as caraterísticas da substância ativa, a via de administração, a população alvo, a indicação terapêutica e a duração do tratamento(6). Cada laboratório farmacêutico rege-se pela AIM de cada medicamento para proceder à análise dos mesmos, sendo as especificações mais frequentes, para produtos sólidos não estéreis, as seguintes(7):
-‐ TAMC < 103 UFC/g; -‐ TYMC < 102 UFC/g;
-‐ Ausência de Escherichia coli verificada em 1 g; -‐ Ausência de Staphylococcus aureus verificada em 1 g;
-‐ Ausência de Pseudomonas aeruginosa verificada em 1 g; -‐ Ausência de Salmonella verificada em 1 g;
-‐ Bactérias gram-negativas tolerantes à bílis < 100 UFC/g.
Os critérios de aceitação devem ser interpretados da seguinte forma(7): • 101 UFC: Contagem máxima aceitável = 20 UFC
• 102 UFC: Contagem máxima aceitável = 200 UFC • 103 UFC: Contagem máxima aceitável = 2000 UFC
A avaliação microbiológica de produtos não estéreis é particularmente pertinente, uma vez que a contaminação microbiana pode reduzir ou mesmo eliminar o efeito terapêutico dos fármacos ou causar infeções induzidas pelos mesmos. Além destes fatores, a contaminação microbiológica pode alterar as propriedades químicas, físicas e organoléticas dos fármacos, ou alterar o conteúdo em substância ativa(8-10).
1.2. Microrganismos relevantes 1.2.1. Enterobacteriaceae
Enterobacteriaceae é uma família de bactérias gram-negativas muito abundante, com
uma grande variedade de organismos patogénicos. Os indivíduos da família
Enterobacteriaceae são bastante conhecidos, pertencendo alguns à flora intestinal de seres
humanos e animais, outros ao solo ou à água(11,12).
Escherichia coli (E. coli) é uma das bactérias com maior patogenicidade da família Enterobacteriaceae. São bactérias anaeróbias facultativas em forma de bastonete e a maioria
colonizam o trato gastrointestinal dos seres humanos e animais(13). Existem diferentes estirpes patogénicas de E. coli e a sensibilidade destas estirpes a antibióticos varia amplamente. Os organismos gram-negativos por si só são bastante resistentes a antibióticos, quando comparados com os organismos gram-positivos. Os antibióticos que podem ser utilizados no tratamento de infecção por E. coli incluem a amoxicilina, penicilinas semi-sintéticas, cefalosporinas, ciprofloxacina e nitrofurantoína(16,17). O uso excessivo de antibióticos em seres humanos ou como promotores de crescimento em alimentos para animais torna a resistência aos antibióticos um problema crescente. A E. coli, por exemplo tem uma taxa de mutações adaptativas muito elevada o que torna este microrganismo muito preocupante em caso de contaminação(16).
O género Salmonella também faz parte da família das Enterobacteriaceae. Este género é composto por três espécies: Salmonella subterranea, Salmonella bongori e
Salmonella enterica(17,18). Uma infeção por Salmonella enterica pode provocar salmonelose, que varia clinicamente da gastroenterite comum de Salmonella (diarreia, cólicas abdominais e febre) a febres entéricas (incluindo febre tifóide) que são doenças sistémicas febris com risco de vida que requerem administração de antibióticos(19). Um número crescente de estirpes multirresistentes de Salmonella apresenta uma suscetibilidade diminuída a alguns antibióticos como a ciprofloxacina, com falhas de tratamento concomitantes(20).
1.2.2. Pseudomonas
As espécies do género Pseudomonas são bacilos aeróbios gram-negativos contendo mais de 140 espécies, em que cerca de 25 estão associadas a seres humanos. Estas incluem P.
aeruginosa, P. fluorescens, P. putida, P. cepacia, P. stutzeri, P. maltophilia e P. putrefaciens.
As bactérias são encontradas principalmente no ambiente, como no solo, água e plantas. Apenas algumas das muitas espécies causam doenças, sendo a mais comum a Pseudomonas
aeruginosa(21). Infeções por Pseudomonas são frequentemente reconhecidas em hospitais através das mãos dos técnicos de saúde ou por equipamentos hospitalares. As infeções por
Pseudomonas são consideradas infeções oportunistas, o que significa que estas bactérias
provocam doenças quando o sistema imunológico está comprometido. O aumento da resistência a antibióticos de bactérias do género Pseudomonas tornou o tratamento das infeções muito mais desafiante(21,22).
1.2.3. Staphylococcus
O género Staphylococcus é constituído por 39 espécies, incluindo 21 sub-espécies. É caracterizado por m.o. de células esféricas gram-positivas, não esporoladas, com cocos isolados, em pares ou em correntes curtas, formando assim grupos irregulares(23). O metabolismo desta família é respiratório e fermentativo e a parede celular destas bactérias contém peptidoglicano e ácido teicoico(24). Os principais habitats da maioria das espécies de
Staphylococcus são a pele e as membranas mucosas dos mamíferos e aves. S. aureus é
considerado o patogeno humano mais importante entre os Staphylococcus. Outras espécies comuns deste género são: S. epidermidis, S. haemolyticus e S. saprophyticus. S. auricularis,
S. capitis ssp. capitis e urealyticus, S. caprae, S. cohnii ssp. cohnii e S. urealyticus, S. hominis
ssp. hominis, S. lugdunensis, S. pasteuri, S. pettenkoferi, S. saccharolyticus, S. schleiferi
subsp. schleiferi, S. simiae, S. simulans, S. warneri e S. xylosus(25). As espécies do género
Staphylococcus podem ser diferenciadas pela coagulase. A atividade desta enzima é utilizada
para distinguir espécies patogénicas de Staphylococcus de espécies não patogénicas, sendo um bom indicador da patogenicidade do S. aureus. Exemplos de espécies coagulase-positiva
são S. aureus, S. delphini e S. lutrae. A maior parte das restantes espécies de Staphylococcus são coagulase-negativa, como é o caso do S. epidermidis que é a espécie encontrada mais frequentemente em seres humanos. Staphylococcus aureus é um agente patogénico virulento sendo atualmente a causa mais comum de infeções em doentes hospitalizados. Uma infeção por S. aureus pode envolver qualquer sistema de órgãos e tem uma grande capacidade de causar uma vasta gama de infeções(26). Colonizam-se à superfície do corpo, particularmente em áreas húmidas como axilas, área inguinal e narinas. A colonização nasal desempenha um papel crucial como fonte de infeções invasivas desta espécie, uma vez que através das cavidades nasais pode ser transferido para a pele e para outras áreas do corpo. Os profissionais de saúde têm uma elevada taxa de transporte nasal de S. aureus (50% a 90%), assim como os doentes com diabetes, os doentes que recebem hemodiálise a longo prazo e os utilizadores de fármacos por via intravenosa(27). Nas instalações de cuidados de saúde, as estirpes de S. aureus são transmitidas de um paciente para o outro, principalmente pelos profissionais de saúde. Isto é de extrema importância para a transmissão de S. aureus resistentes à meticilina (MRSA). Os profissionais de saúde infetados podem servir como um reservatório(28,29). S. auricularis faz parte da flora do canal auditivo externo humano e coloniza exclusivamente esta região. O S. capitis é encontrado em torno das glândulas sebáceas na testa e couro cabeludo após a puberdade. S. haemolyticus e S. hominis são preferencialmente isolados de axilas e áreas púbicas ricas em glândulas apócrinas. S.
saprophyticus ssp. saprophyticus coloniza frequentemente o reto e o trato genital de mulheres
adolescentes(25,30,31). S. lugdunensis é frequentemente encontrado nas extremidades inferiores e nas virilhas(32). No entanto, estas espécies podem ser encontradas ocasionalmente em outros locais do corpo. As espécies S. sciuri e S. xylosus são comensais da pele e da membrana mucosa de muitos animais e, ocasionalmente, de seres humanos. Ambas as espécies são também encontradas em alimentos; S. kloosii, S. equorum subsp. equorum, e S. galinarum são encontrados em vários mamíferos e produtos alimentares. S. chromogenes, S. hyicus, S. lentus são residentes comuns de animais de casco e, além disso, podem ser isolados a partir dos seus produtos alimentares. S. vitulinus é encontrado preferencialmente em cavalos e baleias. S.
arlettae é encontrado em mamíferos e aves e S. muscae é descrito nas moscas(23).
Os Staphylococcus podem tornar-se patogénicos dependendo da espécie e da estirpe, após quebra na barreira epitelial cutânea, através de trauma ou intervenções médicas. A recuperação de um isolado requer sempre a avaliação do significado clínico para determinar se é um contaminante, colonizador ou patogénico(33).
1.3. Identificação de contaminantes microbiológicos 1.3.1. Métodos moleculares/biológicos
A identificação de m.o. pode ser feita através de vários métodos, sendo os mais precisos as técnicas de genética molecular, tais como impressão digital de DNA, perfis de plasmídeo e eletroforese em gel de campo pulsado (PFGE). Estes métodos de identificação são demorados, dispendiosos, e exigem profissionais especializados, o que pode impedir uma resposta rápida à presença de espécies de bactérias patogénicas em laboratórios da indústria farmacêutica(34). No entanto, a análise comparativa da sequência de DNA dos genes de macromoléculas tornaram-se comuns na microbiologia como uma ferramenta para a classificação de m.o. As moléculas mais úteis e extensivamente investigadas em termos de taxonomia são os rRNAs e os seus genes correspondentes, especialmente 16S rRNAs e 23S rRNAs. A identificação molecular baseada na análise da sequência de DNA tem como principal vantagem uma maior precisão na identificação da espécie do m.o. em estudo e como principal desvantagem, a presença de entradas de sequências defeituosas e/ou redundantes, terminações de sequência irregulares e nomenclatura desatualizada nas bases de dados atualmente disponíveis para esta identificação, podendo induzir a uma identificação incorreta(35).
Por esses motivos, nos últimos anos, os métodos de reação em cadeia da polimerase (PCR) em tempo real foram desenvolvidos e descritos para deteção e identificação rápidas de várias estirpes bacterianas. O PCR em tempo real é uma ferramenta promissora para distinguir sequências específicas a partir de uma mistura complexa de DNA e, portanto, é útil para determinar a presença e quantidade de agentes patogénicos específicos numa amostra. Podem facilmente detetar-se estirpes bacterianas diretamente a partir de amostras clínicas ou de pequenas quantidades de células bacterianas em cultura, melhorando assim a sensibilidade e reduzindo o tempo necessário para a identificação de bactérias. Este método tem sido particularmente útil, facilitando a identificação de bactérias a qualquer nível taxonómico: estirpe, espécie ou género(36–38).
1.3.2. Métodos bioquímicos
Atualmente, na indústria farmacêutica utilizam-se maioritariamente: um método manual que recorre a galerias API (índice analítico de perfil) e um método automatizado que utiliza o sistema VITEK. Recorre-se as estes métodos devido à fácil e rápida execução da técnica, e também porque são facilmente adaptados à rotina de um laboratório de análises microbiológicas de produtos farmacêuticos. Para a velocidade, padronização, redução de
custos e conveniência, os testes clássicos de fermentação, oxidação, degradação e hidrólise de vários substratos são incorporados em sistemas de teste comerciais e testes bioquímicos automatizados. Os resultados com testes convencionais podem ser ligeiramente diferentes daqueles obtidos com sistemas de testes bioquímicos rápidos devido ao uso de outros indicadores mais sensíveis(23).
Galerias API
As galerias API tornaram a identificação microbiana fácil e rápida, baseada no conceito da identificação numérica. Os sistemas API são produtos comerciais. A galeria API 20 E (comercializada pela BioMérieux) é um método de identificação de m.o., que utiliza tiras que contêm até 20 pequenos compartimentos onde ocorrem as reações bioquímicas (Figura 1).
Figura 1. Kits que incluem 20 testes bioquímicos (adaptado de (39)).
Os vinte compartimentos são inoculados com uma suspensão bacteriana de uma cultura idealmente pura. Após incubação, as reações traduzem-se em mudanças de cor que são espontâneas ou manifestadas pela adição de reagentes. As reações são lidas de acordo com uma tabela ou quadro de leitura. A identificação é feita usando um sistema informático de identificação ou um catálogo analítico, onde se obtém a identificação do perfil numérico com sete algarismos, que é denominada de índice analítico de perfil (API). O programa interpreta um grande número de perfis que permite a identificação de cerca de 600 espécies de bactérias e leveduras. O kit de teste API consiste em testes de assimilação de elementos enzimáticos e compostos de carbono, cujo número varia dependendo do tipo de kit API de ensaio utilizado(39,40).
VITEK
O VITEK é um sistema automatizado de identificação direcionado a laboratórios de microbiologia clínica que fornece condições de identificação de colónias através de testes bioquímicos. Este sistema de identificação de bactérias e leveduras, utiliza cartas colorimétricas com 64 poços, contendo substratos para testes individuais, que são incubadas e interpretadas automaticamente(41). No entanto existem requisitos para a preparação adequada do inóculo: meios de cultura adequados, idade da cultura, condições de incubação e turbidez
do inóculo. A turbidez é ajustada em conformidade (Tabela 1) e medida utilizando um medidor de turbidez chamado DensiChek (42).
Tabela 1. Turbidez para preparação dos inóculos.
Produto Gama de turbidez McFarland
Gram-negativa 0,50-0,63
Gram-positiva 0,50-0,63
Levedura 1,80-2,20
Bacilos 1,80-2,20
As cartas de identificação (Figura 2) são inoculadas com suspensões de m.o. utilizando uma cassete. A carta é colocada na ranhura vizinha ao tubo que contém a suspensão com o inóculo. A cassete pode acomodar até 10 testes (ou seja, até 10 cartas de identificação). A cassete é colocada manualmente numa estação de câmara de vácuo. O vácuo que se forma dentro do equipamento faz com que a suspensão do organismo seja forçada, através do tubo de transferência, a preencher os micro-canais que chegam a todos os poços do carta.
Figura 2. Carta de identificação gram-positiva para o VITEK (adaptado de (41)).
Cada um dos poços contém um teste individual a um substrato. Estes medem atividade metabólica como a acidificação, alcalinização, a hidrólise enzimática e crescimento na presença de substâncias inibidoras. Uma película opticamente transparente presente em ambos os lados do cartão permite o nível adequado de transmissão de oxigénio, mantendo o recipiente selado e impedindo o contacto com as misturas organismo-substrato. Os cartões têm códigos de barras que contêm informações sobre o tipo de produto, número de lote, data de validade, e uma identificação exclusiva que pode ser ligada à amostra, quer antes ou depois de carregar a carta no sistema (Figura 3)(42).
1.3.3. Métodos espetroscópicos
Espetroscopia de massa (MALDI-TOF)
A espetrometria de massa MALDI-TOF (matrix-assisted laser
desorption/ionization-time of flight) é outra técnica utilizada para identificação molecular. Através da análise da
massa e da carga dos iões, pode fornecer uma identificação de género e espécie de um microrganismo em apenas alguns minutos. A amostra é depositada numa matriz com um suporte de metal apropriado e é ionizada por um feixe de laser cujo comprimento de onda pode ir desde a gama de ultravioleta (UV) a infravermelho (IV) (impulsos com duração de pico-segundos a alguns nano-segundos)(7).
O MALDI-TOF é um método rápido, preciso e de baixo custo para a identificação de bactérias, em comparação com técnicas convencionais de biologia molecular. Além disso, permite a identificação de bactérias diretamente a partir de amostras clínicas(43,44).
Espetroscopia Raman
A espetroscopia Raman é uma forma de espetroscopia vibracional, assim como a espetroscopia IV. No entanto, enquanto que as bandas do IV surgem a partir de uma alteração no momento dipolar de uma molécula (devido a uma interação da luz com a molécula), as bandas Raman surgem a partir de uma alteração na polarizabilidade da molécula devido à mesma interação. Isto significa que estas bandas observadas (correspondentes a transições de energia específicas) são provenientes de vibrações moleculares específicas. Quando as energias destas transições são representadas como um espetro, proporcionam uma "impressão digital molecular" da molécula que está a ser observada. Certas vibrações que são permitidas no Raman são proibidas no IV, ao passo que outras vibrações podem ser observadas em ambas as técnicas (embora com diferentes intensidades), podendo assim, estas técnicas serem consideradas como complementares(45,46).
A espetroscopia de Raman não requer preparação especial da amostra e os tempos de medição são bastante curtos (a partir de uma fração de segundo a vários minutos)(47,48). Cada m.o. produz um espetro único o que faz com que a especificidade da técnica analítica, em combinação com o processamento quimiométrico apropriado, permita a caracterização, a discriminação e a identificação de bactérias, fungos e leveduras ao nível da espécie e da sub-espécie(49).
Espetroscopia de infravermelho com transformada de Fourier (FTIR)
Os métodos implementados para a identificação de contaminantes microbiológicos na indústria farmacêutica são igualmente métodos dispendiosos e demorados. A espetroscopia de
IV é um método rápido, barato e bastante simples. Esta técnica encontra-se detalhadamente descrita no ponto 1.3.4. e tem características comuns com a técnica de Raman.
A espetroscopia de IV consiste no estudo da interação da radiação infravermelha com a matéria. A luz analisada após ter interatuado com a matéria contém informação sobre a estrutura desta. Um espetro é o registo desta interação(50). Para que uma molécula apresente absorção de radiação de IV deve possuir características específicas: a molécula precisa que o momento dipolar sofra uma variação durante a vibração. A região do espetro eletromagnético correspondente à radiação de infravermelho divide-se em três regiões principais: infravermelho próximo (13000-4000 cm-1), infravermelho médio (4000-400 cm-1) e infravermelho distante (< 400 cm-1). A espetroscopia FTIR baseia-se na absorção de radiação infravermelha média e as principais vantagens dos espetrómetros FTIR com interferómetro em relação aos dispersivos são(51):
• uma relação sinal/ruído muito superior para um espetro acumulado no mesmo intervalo de tempo;
• uma maior exatidão nos números de onda, associada a uma resolução constante em toda a gama espectral.
O método de espetroscopia de IV com transformada de Fourier é baseado na interferência da radiação entre dois feixes resultando um interferograma. Um interferograma é o registo do sinal produzido pela combinação das múltiplas frequências possíveis de obter com a transformada de Fourier.
Figura 4. Esquema de um espetrómetro com interferómetro: F - Fonte de radiação IV; D - Divisor de feixe; E1 - Espelho fixo; E2 - Espelho móvel (Adaptado (52)).
O instrumento transformada de Fourier mede todas as frequências em simultâneo. Isso faz com que o método FTIR seja um método rápido. É utilizado um interferómetro (p.e., de Michelson), que permite uma ampla faixa de frequências de vibração a ser monitorizadas simultaneamente. Este interferómetro é constituído por um divisor de feixe (D) e dois espelhos: E1 que é fixo e E2 que é móvel (Figura 4). O divisor de feixe consiste numa placa que reflete metade da radiação incidente. Assim metade da radiação segue para o espelho E1 e metade para o E2. Após a reflexão em ambos os espelhos, as duas componentes
recombinam-se no divisor de feixe. A única parte que recombinam-se move continuamente no instrumento é o movimento do espelho (E2) no interferómetro. Em suma, o processo instrumental é composto pelas seguintes etapas: a energia infravermelha é emitida por uma fonte (D) de corpo negro (este feixe passa através de uma abertura que controla a quantidade de energia presente na amostra (e consequentemente no detetor); o feixe entra no interferómetro onde é feita a “codificação espectral”, e o sinal resultante do interferograma sai do interferómetro; no compartimento da amostra, esta é atravessada pelo feixe ou reflete-o, dependendo do tipo de análise a ser feita (é aqui que frequências específicas de energia, características de cada amostra, são absorvidas); o feixe passa finalmente para o detector para uma medição final (os detetores utilizados são apropriados para medir o sinal especial do interferograma). O sinal medido é digitalizado e enviado para o computador onde é aplicada a inversa da transformada de Fourier. O espetro final é apresentado ao utilizador para interpretação e posterior processamento dos dados(52–55).
O modo de refletância total atenuada (ATR) pode ser utilizado em amostras sólidas ou líquidas e tem de existir um contato muito próximo entre a amostra e o cristal para que este tipo de refletância ocorra. Uma grande vantagem do ATR é o facto de não ser necessária qualquer preparação da amostra a analisar. O cristal é fabricado com materiais com um elevado nível de refração como por exemplo o germânio, zinco-selénio ou diamante. Um acessório de ATR funciona medindo as alterações que ocorrem num feixe de infravermelho totalmente refletido internamente quando o feixe entra em contato com uma amostra (Figura 4). O componente principal de um acessório de ATR é um prisma de uma substância transparente ao IV, de elevado índice de refração. As faces laterais do prisma são cortadas segundo um ângulo tal que a luz incidente é refletida com um ângulo superior ao ângulo crítico. Nestas condições, há reflexão total e a luz permanece dentro do cristal durante sucessivas reflexões. Se a amostra estiver depositada na face do cristal, com um bom contato ótico, em cada reflexão alguma radiação penetra na amostra e pode ser absorvida, como se esquematiza na Figura 5.
Depois de um certo número de reflexões, a luz deixa o prisma e é focada no detetor. A radiação resultante atenuada é medida e representada graficamente como uma função do comprimento de onda e dá origem a absorções características da amostra(51,56,57).
1.3.4. FTIR - ATR para identificação de microrganismos
A técnica FTIR-ATR pode ser utilizada para a classificação, identificação e deteção de microrganismos. Tem sido aplicada com sucesso na identificação e classificação de m.o. por espécie e sub-espécie(58). Quando aplicada a células microbianas inteiras, o espetro de infravermelho reflete a composição bioquímica qualitativa das células(4,59), permitindo caracterizar e diferenciar células microbianas com base em componentes das células de membrana incluindo os lipopolissacáridos, lipoproteínas, e fosfolípidos(34,54). A base da espetroscopia FTIR é a interação da radiação infravermelha com uma amostra, neste caso específico com o isolado bacteriano, proporcionando uma impressão digital específica que reflete a estrutura e a composição de toda a célula. No modo ATR a magnitude da atenuação depende das bactérias em contato com o feixe. A compreensão dos espetros de IV das células microbianas requer uma perceção geral da sua composição, dos principais tipos de células, das estruturas químicas presentes, e do conhecimento da diferenciação de células e tecidos(60). Ao contrário dos organismos eucariotas (animais, plantas, leveduras ou fungos), os procariotas (bactérias) existem apenas num número limitado de formas morfológicas (p.e. bacilos e cocos). No entanto, a sua composição química e as suas estruturas variam consideravelmente. As estruturas citoplasmáticas de bactérias são menos compartimentadas e mais simples do que as das leveduras e fungos, mas têm estruturas moleculares e supra-moleculares complexas. A maior parte das diferenças estruturais que fornecem a possibilidade de diferenciação entre as bactérias por FTIR residem no envelope celular, que é geralmente definido como a parede celular além da membrana citoplasmática. A parede celular de bactérias gram-positivas é formada por um camada espessa de peptidoglicano, enquanto que a parede celular de bactérias gram-negativas é mais complexa, uma vez que é formada por uma camada fina de peptidoglicano, rodeada por uma camada externa de lipopolissacarídeo e proteína(64).
As bandas de absorção observadas na região IV entre aproximadamente 1800 cm-1 e 400 cm-1 surgem principalmente a partir dos modos de vibração fundamentais e podem ser atribuídas a determinados grupos funcionais. A partir da literatura podem ser obtidas descrições de espetros de vários m.o. e correlações entre estrutura de espetros e as macromoléculas biológicas mais importantes. A interpretação dos espetros IV de moléculas
biológicas baseia-se principalmente na análise de estruturas conhecidas (Tabela 2). Os ácidos nucleicos, proteínas, lípidos e hidratos de carbono estão sempre presentes em diferentes quantidades e numa grande diversidade de células microbianas(61).
Tabela 2. Atribuição do número de onda ao grupo funcional existente nas células microbianas (adaptado(61)).
Número de
onda (cm-1) Designação
3500 Movimentos de distensão O-H de grupos hidroxilo 3200 Movimentos de distensão N-H das proteínas
2955 Movimentos de distensão assimétricos C-H de -CH3 em ácidos gordos
2930 Movimentos de distensão assimétricos C-H de >CH2
2918 Movimentos de distensão assimétricos C-H de >CH2 em ácidos gordos
2898 Movimentos de distensão C-H em grupos metilo 2870 Movimentos de distensão simétricos C-H de –CH3
2850 Movimentos de distensão simétricos C-H de >CH2 em ácidos gordos
1740 Movimentos de distensão >C=O dos ésteres
1715 Movimentos de distensão >C=O dos ácidos carbónicos 1680-1715 Movimentos de distensão >C=O dos ácidos nucleicos 1695 Componentes da banda da amida I
1685 Resultante de folhas β -antiparalelas 1675 Configuração β das proteínas 1655 Estruturas α–helicoidais da amida I 1637 Estruturas das folhas -β da amida I 1550-1520 Amida II
1515 Banda da tirosina
1468 Deformação C-H de >CH2
1400 Movimentos de distensão simétricos C=O de COO 1310-1240 Banda da amida III das proteínas
1250-1220 Movimentos de distensão assimétricos P=O de >PO2 de fosfodiésteres
1200-900 Movimentos de distensão de C-O e C-C e deformação de C-O-C de hidratos de carbono
1090-1085 Movimentos de distensão simétricos P=O de >PO2
720 Vibração de rotação da ligação C-H de >CH2
900-600 Região da impressão digital (fingerprint)
Na maioria das implementações descritas na literatura, os m.o. são analisados na forma de colónias (tal como neste estudo), dando origem a espetros muito complexos, uma vez que o espetro resultante depende da composição química total das células (do material intracelular, da membrana e da parede). Devido à complexidade do espetro resultante, é indispensável uma avaliação por intermédio de análise multivariada (quimiometria)(63).
A técnica FTIR-ATR para identificação de contaminantes microbiológicos tem como principais vantagens(52,61):
• técnica aplicável a todos os m.o.;
• os requisitos de biomassa podem ser reduzidos para colónias individuais tomadas diretamente a partir das placas de cultura;
• os resultados estão disponíveis em poucos minutos após a obtenção de amostras adequadas de cultura pura;
• classificação dos m.o. em níveis muito diferentes de discriminação taxonómica sem qualquer pré-seleção de estirpes por outros critérios taxonómicos;
• a especificidade do método é de um modo geral extremamente alta;
• a deteção in situ de componentes celulares específicos é possível (p.e. materiais de armazenamento de formação de esporos e a encapsulação de m.o.).
Por outro lado, também existem desvantagens(52):
• apenas os m.o. que podem ser cultivados em cultura e estão disponíveis como culturas puras devem ser analisados;
• culturas mistas só podem ser investigadas com a ajuda de um microscópio de IV; • a classificação e identificação são baseadas na análise de impressões espectrais; • as informações específicas sobre um ou apenas alguns compostos específicos
presentes nas células geralmente não está disponível;
• a técnica proporciona uma impressão digital espectral de todos os constituintes celulares, resultados estáveis só podem ser obtidos desde que os parâmetros microbiológicos (meio de cultura, tempo de cultivo, e temperatura) sejam controlados de forma rígida;
• as bases de dados espetrais que são utilizadas em diferentes laboratórios exigem a utilização de meios de cultura adequados disponíveis no mercado;
• a aceitação da técnica de IV pela comunidade científica pressupõe que a informação espetral pode ser transformada com esquemas de classificação que sejam pelo menos semelhantes aos obtidos por quimio-taxonómica ou por técnicas de genética molecular (tarefa realizada apenas por profissionais qualificados)(61).
Figura 6. Espetro FTIR representativo de bactérias com respetiva identificação das principais bandas (Adaptado de (64)).
1.4. Análise Multivariada
Para processar os dados espetrais complexos (de isolados intactos) obtidos por espetroscopia de IV são necessários métodos estatísticos multivariados ou quimiométricos. A quimiometria pode ser definida como uma ciência relacionada a medidas realizadas num sistema ou processo químico, obtendo informações sobre o estado do sistema através da aplicação de métodos matemáticos ou estatísticos(65,66). Para garantir robustez dos modelos quimiométricos baseados em análises FTIR-ATR de m.o., é necessária uma base de dados muito completa de isolados. Apenas deste modo é possível a validação e estabelecimento do método para análise de rotina. Todos estes métodos requerem um conjunto muito completo de isolados e técnicas de pré-processamento espetrais adequadas de modo a construir modelos que permitam inequivocamente a discriminação de m.o.(54).
Na análise de espetros IV de m.o. são utilizadas duas abordagens quimiométricas(67): • análise por métodos não supervisionados - os dados são analisados independentemente
da identidade da amostra. Estes são considerados métodos qualitativos uma vez que o objetivo é a distinção de microrganismos;
• análise por métodos supervisionados - parte-se de um conjunto de amostras, bem caracterizadas, para a construção de um modelo que será posteriormente utilizado para a identificação de uma amostra desconhecida.
1.4.1. Pré-processamento espetral
O tratamento quimiométrico dos espetros deve ser feito após uma primeira etapa designada por pré-processamento espetral. Esta etapa consiste na aplicação de métodos
Número de onda (cm-1) Ab so rv â nc ia ( u .a .)
corretivos dos espetros. As interferências nos espetros devem-se ao facto dos aparelhos das medições espetrais serem afetadas por vários fenómenos tanto ao nível químico como físico. Por exemplo, variações de temperatura, quantidade de amostra e efeito causado pela dispersão da luz devem ser eliminados dos espetros de forma a que estes evidenciem apenas informação química ou bioquímica da amostra. Também podem ocorrer distorções causadas pela saturação do detetor ou relacionadas com o ruído atribuível ao detetor. Os métodos de pré-tratamento permitem eliminar algumas destas perturbações de forma a melhorar o desenvolvimento dos modelos quimiométricos(68). Existem vários tipos de pré-processamento que podem ser aplicados aos espetros, traduzindo diferentes alterações nos dados. Os mais utilizados são as derivadas (combinados com filtros de Savitzky-Golay), variação de padrão normal (SNV) e correção multiplicativa de sinal (MSC)(69).
Derivação
Os filtros de Savitzky-Golay têm como principais objetivos reduzir o ruído existente nos espetros originais e derivação do sinal para remoção de efeitos de linha de base. Esta operação envolve a derivação matemática de uma função, sendo a função o espetro de uma amostra em vários comprimentos de onda. Estes filtros são essencialmente funções locais que são aplicadas a cada espetro sendo posteriormente aplicadas as derivadas. A primeira ou segunda derivada do espetro original amplificam as diferenças entre espetros enquanto minimizam perturbações causadas pelos fenómenos descritos atrás(72,73).
Variação de padrão normal
A variação de padrão normal (SNV) é um pré-processamento que centra cada espetro em torno do zero, por subtração da média e divisão pelo desvio padrão. Este método permite reduzir interferências causadas pelo tamanho de partícula e pelas diferenças de densidade das amostras(71). Para este método o espetro de uma amostra i transformado X(i)’, é dado pela Equação 1. X(i) representa o espetro da amostra i inicial, mi a média das leituras espetrais
para a amostra i e si o desvio padrão(72).
𝑋(𝑖)! =! ! !!!
!! [Equação 1]
Correção multiplicativa de sinal
A correção multiplicativa de sinal (MSC) é um pré-processamento que reduz o efeito de luz dispersa em espetros de refletância. A MSC seleciona um espetro de referência no
conjunto total de espetros disponíveis para análise. Na maioria das vezes o espetro de referência consiste no espetro médio do conjunto usado no desenvolvimento do método (calibração). Através de regressão linear são corrigidos todos os espetros tendo o espetro de refência como alvo(72,73).
1.4.2. Técnicas quimiométricas
Análise de Componentes Principais
A análise de componentes principais (PCA) é um método utilizado para reduzir a dimensão de um conjunto de dados tornando-o mais interpretável. A PCA evidencia em que aspeto uma determinada amostra difere de outra e quais as variáveis que mais contribuem para essa diferença. Esta análise permite criar um padrão das amostras, formando grupos entre as amostras semelhantes. Esta técnica é utilizada para identificar correlações entre um conjunto de variáveis e transformar o conjunto original de variáveis num novo conjunto de variáveis não correlacionadas denominadas por componentes principais (CPs)(67). Os CPs são uma combinação linear das variáveis originais, neste caso as absorvâncias em diversos comprimentos de onda. Os CPs individuais podem ser interpretados através da sua conexão com as variáveis originais. Ou seja, mostram-se os mesmos dados num sistema de coordenadas diferente. As relações entre amostras são reveladas através das suas projeções nos CPs (scores). A distância entre amostras pode ser avaliada no espaço dos PCs constituindo uma medida mais fiável para análise de similaridade. A tabela de dados original é transformada numa nova matriz reorganizada cuja estrutura revela as relações entre linhas (amostras) e colunas (variáveis) que podem estar ocultas na matriz original, assim, a nova estrutura constitui a parte explicada dos dados originais(74). Em termos matriciais o modelo de PCA pode ser descrito através da Equação 2 e da Figura 7.
𝑋 = 𝑇×𝑃!+ 𝐸 [Equação 2]
Figura 7. Ilustração do método de PCA (adaptado de ((74)). X : Matriz original de dados (n linhas e p colunas)
T : Matriz de scores (n linhas e d colunas)
E : Matriz de resíduos ( mesmo tamanho de X) p : Número de variáveis (absorvâncias)
n : Número de amostras (isolados) d : Número de fatores
O objetivo do modelo de PCA é encontrar as direções no espaço de dados que descrevem a maior variação do conjunto de dados. Cada direção é uma combinação linear das variáveis iniciais que mais contribuem para a variação real entre amostras. Pela construção, CPs são ortogonais entre si e também são classificados de modo que cada um carrega mais variância que qualquer um dos que se seguem. A prioridade é dada à interpretação destes CPs, começando com o primeiro, que incorpora a maior variação e, portanto, constitui um sistema alternativo menos complexo que é mais adequado para a interpretação da estrutura de dados. Normalmente, apenas os primeiros CPs contêm informações pertinentes.
Os resíduos (E) mantêm a variação que não está incluída no modelo, como uma medida de quão bem as amostras ou variáveis se enquadram no modelo. Se todos os CPs fossem retidos, não haveria nenhuma aproximação e o ganho de simplicidade consistiria apenas em ordenar a variação dos próprios CPs pelo tamanho. Decidir sobre o número de componentes a reter num modelo de PCA é um compromisso entre simplicidade e robustez(67,75).
Análise hierárquica
A análise hierárquica de grupos (HCA) permite comparar e categorizar os dados de acordo com as ponderações de importância/variabilidade das variáveis. Assim, os dados serão incluídos em grupos de acordo com a sua similaridade. Um agrupamento consiste num grupo de objetos semelhantes entre si. Os pontos de um agrupamento específico partilham algumas características comuns que os diferenciam daqueles reunidos em outros agrupamentos. Estes são caracterizados tendo em conta três propriedades principais: tamanho, forma e distância para o agrupamento mais próximo. HCA é um modelo não supervisionado de análise de dados que é utilizado tanto para análise explicativa como para a confirmatória. Este método difere da análise discriminante (técnica de classificação supervisionada) em que um objeto não rotulado é atribuído a um grupo de objetos pré-classificados(74,76).
O dendrograma consiste na representação gráfica dos resultados. O agrupamento hierárquico encontra aglomerados em modo aglomerativo (de baixo para cima) ou divisor (de cima para baixo) para formar uma estrutura em forma de árvore. O modo aglomerativo começa com a definição de cada ponto como seu próprio agrupamento e a estes juntam-se
aglomerados semelhantes em pares até que o conjunto de dados seja classificado. O eixo horizontal de um dendrograma descreve a variância ou heterogeneidade crescente. A magnitude dessa heterogeneidade depende do número de espetros num agrupamento e das semelhanças entre eles. Os dendrogramas são usados para identificar as semelhanças entre espetros de bactérias que representam a mesma espécie ou género, e além disso pode identificar uma bactéria desconhecida através do cálculo da distância espectral entre ela e bactérias conhecidas(53). O modo divisivo começa considerando todo o conjunto de dados como um único agrupamento, que é então recursivamente dividido até que somente agrupamentos contendo pontos de dados exclusivos sejam obtidos. Algoritmos diferem na forma como eles calculam a similaridade entre agrupamento(73,74).
Regressão por mínimos quadrados parciais – discriminante (PLS-DA)
O PLS-DA consiste na clássica regressão PLS onde a variável resposta é categórica e representativa de classes(76). O PLS-DA é realizado para modelar a separação entre grupos de observações(77). PLS-DA é um caso especial de PLS onde as variáveis independentes (matriz X) é regredida numa matriz Y consistindo de uns e zeros identificativa das classes. Estes uns e zeros indicam a classe à qual as amostras pertencem(74). O PLS-DA permite melhorar a separação entre classes quando comparado com o método PCA, pois força a que se obtenha a máxima separação entre classes(75). Este método supervisionado requer o conhecimento da identidade de cada amostra (isolado). Com um conjunto de amostras bem caracterizadas, um modelo pode ser calibrado para que possa prever a identidade de novas amostras, relacionando um conjunto de variáveis de resposta Y (classes) com um conjunto de variáveis X (espetros)(34). Esta abordagem visa maximizar a covariância entre as variáveis independentes X (espetros) e a variável dependente Y correspondente (classes, grupos, isto é, a classe que se deseja prever). Permite a previsão da variável Y com base num número reduzido de fatores (variáveis latentes). Estes fatores descrevem o comportamento das variáveis dependentes Y e abrangem o espaço no qual as variáveis independentes X são projetadas (Figura 8).
Figura 8. Ilustração de um modelo de PLS-DA com três classes em que o i é o número de amostras e o j o número de
variáveis (Adaptado de (78)).
Por exemplo, se considerarmos a divisão de amostras em diferentes classes, então a variável Y será composta por vetores que terão uma entrada 0 para todas as amostras na primeira classe e uma entrada 1 para todas as amostras da segunda classe (ou vice-versa)(78).
Para avaliar um modelo de PCA e de PLS-DA avaliam-se normalmente duas estatísticas: a estatística de Hotelling’s T2 que incorpora a informação retida nos CPs e a estatística dos resíduos que permite avaliar a componente não modelada (ou seja a componente que não ficou retidas nos CPs). A avaliação destas duas estatísticas permite-nos concluir sobre isolados atípicos, ou seja, ‘outliers’ do modelo construído(73).
PLS-DA ! ! X (espetros) Y (classes) C las se 1 C las se 2 C las se 3 PLS-DA ! ! X (espetros) Y (classes) C las se 1 C las se 2 C las se 3 X (espetros) Y (espetros) Cl a ss e 2 Cl a ss e 1 Cl a ss e 3 PLS-DA
2. Revisão da literatura
O potencial da espetroscopia FTIR o âmbito da identificação de microrganismos está documentado desde a década de 90. A técnica tem sido aplicada em muitos campos diferentes, como a microbiologia alimentar, diagnóstico médico e ecologia microbiana. A sua aplicação pode abranger identificação de patogenos, contaminantes alimentares, culturas probióticas e microrganismos ambientais. Alguns estudos centram-se apenas na diferenciação de espécies enquanto outros tentaram mostrar bases de dados abrangentes para aplicação em microbiologia(79). Existe um elevado número de estudos sobre este tema. Alguns especialmente relevantes e recentes encontram-se listados na Tabela 3.
Tabela 3. Revisão dos estudos publicados recentemente sobre identificação e discriminação de m.o. com o uso de
espetroscopia. DA: análise discriminante; CVA: análise canónica variada; PAS: espetroscopia foto-acústica; CA: análise de clusters; SOM: (self-organized maps) mapas auto-organizados; KNN: (K-nearest neighbor); ANN: redes neuronais artificiais.
Género(s)/ Espécie(s) Técnica espetroscópica
Percentagem de identificação/discriminação
correta
Abordagem
quimiométrica Ref.
Células microbianas intactas FTIR; NIR Raman (Estudo pioneiro) CA, ANN (60) Bactérias intactas e fungos ATR (Estudo pioneiro) CA, ANN (61)
Staphylococcus Transmissão 97% dos isolados de S. aureus PCA, FDA (80)
Salmonella Enteritidis Transmissão 100% dos tipos de fagos (utilizando extratos das proteínas de membrana externa)
PLS-DA (34)
E.coli Transmissão 77,92% das estirpes de E. coli PLS-DA (59)
Lactobacillus casei, B. cereus, E.coli, S.cerevisiae, A.niger e F.verticilliodes
PAS 100% das estirpes de E. coli CVA; Distância de Mahalanobis
(55)
Pichia Pastoris ATR 100% entre células cultivadas sob condições de nitrogénio e de limitação de carbono
PCA; PLS-DA (4)
E. coli; S. aureus; B. subtilis; C.albicans
Espetroscopia
Microscópica Elevada precisão HCA; CA (62)
Acinetobacter baumannii; A.
calcoaceticus ATR Superior a 90% PLS-DA; HCA (81)
Listeria Espetroscopia Microscópica 93% CVA; PCA; PLSDA (82)
S. aureus ATR 98% pela KNN 93% pelos SOM 92% pelo PCA PCA; KNN; SOM (83) L. plantarum, L. acidophilus, L. kefirgranum, L. kefiranofaciens e L. cassei
ATR 100% ao nível da espécie HCA (84)
O. limosa, A. platensis, P. laminosum, Phormidium sp, S. javanicum, N. punctiforme e P. angustissimum
ATR 80% PCA; SIMCA (85)
A. flavus, A. fumigatus e A.
parasiticus ATR 75%-100% ao nível da toxigenicidade HCA; DA (86)
S. aureus ATR 100% ao nível das estirpes ANN (87)
Yersinia enterocolitica Transmissão 79,9% ao nível da espécie ANN (88)
B. cereus, Salmonella
enterica, E. Coli, Listeria ATR 94% PCA; SIMCA (89)
Burkholderia cepacia
3. Objetivos
O principal objetivo deste trabalho é avaliar o potencial da técnica FTIR como técnica de identificação e discriminação de microrganismos contaminantes de medicamentos (formas sólidas). Os objetivos deste trabalho são:
• a construção de uma biblioteca no FTIR-ATR para os principais microrganismos contaminantes de medicamentos sólidos não estéreis;
• a identificação de estirpes através do método FTIR-ATR;
• a comparação do desempenho do método baseado em FTIR-ATR com o método VITEK usado em rotina;
• a diminuição do tempo de identificação de microrganismos com consequente diminuição do tempo de análise microbiológica de um medicamento.
4. Materiais e métodos
A parte prática desta dissertação foi realizada no Laboratório de Microbiologia da Generis Farmacêutica SA na Venda Nova, Lisboa.
4.1. Isolados
As amostras biológicas são colónias recolhidas de crescimentos bacterianos presentes na análise de produtos e no controlo microbiológico ambiental feito nas áreas de produção e embalagem da unidade fabril da Generis Farmacêutica SA. Foram recolhidos 52 microrganismos cujas identificações pelo método VITEK correspondiam a microrganismos dos géneros Staphylococcus e Pseudomonas (Tabela 4).
Para além de bactérias isoladas da rotina do laboratório foram ainda utilizados microrganismos padrão de E. coli, S. aureus e P. aeruginosa. Estes padrões foram preparados a partir de microrganismos Bioball(90). Estes padrões contém um número exato de UFC viáveis. A Bioball é uma bola liofilizada de aproximadamente 3 mm de diâmetro que é conservada individualmente em frascos entre -33ºC a -18ºC.
As abreviaturas referentes a cada microrganismo dão-nos informação sobre a origem do mesmo, uma vez que a primeira letra ‘a’ ou ‘p’, refere-se ao fato desta colónia ter sito obtida da monitorização ambiental ou da análise de um produto. A segunda letra ao género: ‘s’ – Staphylococcus e ‘p’ - Pseudomonas. As terceira e quarta letras indicam a espécie e o número distingue isolados da mesma espécie.
Tabela 4: Codificação e origem dos isolados utilizados neste estudo.
Abreviatura m.o. Origem
Staphylococcus
ST01 S. aureus Bioball ST02 S. aureus Bioball ST03 S. aureus Bioball ST04 S. aureus Bioball psau01 S. auricularis produto psep01 S. epidermidis ambiente psep02 S. epidermidis ambiente asep03 S. epidermidis ambiente asep04 S. epidermidis ambiente asep05 S. epidermidis ambiente aslt01 S. lentus ambiente pslt02 S. lentus produto pslt03 S. lentus produto pslt04 S. lentus produto pslt05 S. lentus produto ascc01 S. cohnii ssp cohnii ambiente ascc02 S. cohnii ssp cohnii ambiente asxy01 S. xylosus ambiente asxy02 S. xylosus ambiente ashh01 S. hominis ssp hominis ambiente pshh02 S. hominis ssp hominis ambiente assp01 S. saprophyticus ambiente assp02 S. saprophyticus ambiente assp03 S. saprophyticus ambiente assp04 S. saprophyticus ambiente assp05 S. saprophyticus ambiente aswa01 S. warneri ambiente aswa02 S. warneri ambiente pswa03 S. warneri ambiente aswa04 S. warneri ambiente aswa05 S. warneri ambiente pssc01 S. sciuri produto pssc02 S. sciuri produto psvt01 S. vitullinus produto psvt02 S. vitullinus produto ascp01 S. capitis ambiente ascp02 S. capitis ambiente ascp03 S. capitis ambiente ascp04 S. capitis ambiente ascp05 S. capitis ambiente
Pseudomonas
PS01 Pseudomonas
auriginosa Bioball
PS02 Pseudomonas auriginosa Bioball PS03 Pseudomonas auriginosa Bioball PS04 Pseudomonas auriginosa Bioball apfr01 Pseudomonas fluorescens ambiente apst01 Pseudomonas stutzeri ambiente apst02 Pseudomonas stutzeri ambiente ppor01 Pseudomonas oryzihabitans produto
Escherichia
EC01 E. coli Bioball EC02 E. coli Bioball EC03 E. coli Bioball EC04 E. coli Bioball