UNIVERSIDADE FEDERAL DO CEARÁ CENTRO DE CIÊNCIAS
DEPARTAMENTO DE BIOQUÍMICA E BIOLOGIA MOLECULAR PROGRAMA DE PÓS-GRADUAÇÃO EM BIOQUÍMICA
JOSÉ EDNÉSIO DA CRUZ FREIRE
MODIFICAÇÕES PÓS-TRADUCIONAIS REVELAM QUE Mo-CBP3, UMA
ALBUMINA 2S LIGANTE DE QUITINA DE Moringa oleifera, É UMA MISTURA COMPLEXA DE ISOFORMAS
JOSÉ EDNÉSIO DA CRUZ FREIRE
MODIFICAÇÕES PÓS-TRADUCIONAIS REVELAM QUE Mo-CBP3, UMA ALBUMINA
2S LIGANTE DE QUITINA DE Moringa oleifera, É UMA MISTURA COMPLEXA DE ISOFORMAS
Tese de Doutorado apresentada ao Programa de Pós-Graduação em Bioquímica, do Departamento de Bioquímica e Biologia Molecular da Universidade Federal do Ceará, como requisito parcial para obtenção do título de Doutor em Bioquímica. Área de concentração: Bioquímica Vegetal.
Orientador: Profº. Dr. Thalles Barbosa Grangeiro
JOSÉ EDNÉSIO DA CRUZ FREIRE
MODIFICAÇÕES PÓS-TRADUCIONAIS REVELAM QUE Mo-CBP3, UMA ALBUMINA
2S LIGANTE DE QUITINA DE Moringa oleifera, É UMA MISTURA COMPLEXA DE ISOFORMAS
Tese de Doutorado apresentada ao Programa de Pós-Graduação em Bioquímica, do Departamento de Bioquímica e Biologia Molecular da Universidade Federal do Ceará, como requisito parcial para obtenção do título de Doutor em Bioquímica. Área de concentração: Bioquímica Vegetal.
APROVADA EM: _____ / _____ / _____
BANCA EXAMINADORA
Profº. Drº. Thalles Barbosa Grangeiro (Orientador) Universidade Federal do Ceará (UFC)
Prof. Dr. Geancarlo Zanatta Universidade Federal do Ceará (UFC)
Prof. Dr. Bruno Lopes de Sousa Universidade Estadual do Ceará (UECE)
Prof. Dr. Ito Liberato Barroso Neto Centro Universitário Unichristus
A Deus pela ciência necessária para desenvolver este trabalho.
A minha queria e compreensiva esposa, Cindy;
Aos saudosos, Genésio e Edite (meus pais);
A minha Madrasta, Maria por ter ajudado sempre que necessário;
Aos meus Irmãos, Benedito, Genedito, Evandito, Genedier, Efigênia e Lidiene,
AGRADECIMENTOS
Ao Profº. Dr. Thalles Barbosa Grangeiro, por me acolher em seu laboratório e dedicar uma valiosa orientação. Pelos questionamentos, explicações, soluções e exemplo, o qual tentarei seguir na minha caminhada científica.
Ao Profº. Dr. José Edvar Monteiro Júnior, por sua grande contribuição para a realização deste trabalho, por meio de sugestões e explicações e co-orientação.
Aos Profº. Dr. Geancarlo Zanatta, Dr. Bruno Lopes de Sousa, Dr. Ito Liberato Barroso Neto e Dr. Rômulo Farias Carneiro por aceitarem o convite para participar desta banca.
A todos os professores do Departamento de Bioquímica e Biologia Molecular que, de alguma forma, colaboraram para a realização deste trabalho.
A todos os integrantes dos Laboratórios de Genética de microrganismos.
A Deus, pela ciência necessária para a execução deste trabalho.
AGRADECIMENTOS INSTITUCIONAIS
Este trabalho foi realizado graças ao auxílio das seguintes Instituições:
Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES).
Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq).
Fundação Cearense de Apoio ao Desenvolvimento Científico e Tecnológico (FUNCAP).
RESUMO
O estudo de albuminas 2S expressas em sementes de Moringa oleifera tem sido um processo muito complexo, especialmente quando empregadas técnicas proteômicas convencionais devido à presença de muitas isoformas. A grande diversidade de isoformas proteicas pode advir, em parte, devido à origem multigênica, além do intenso processamento pós-traducional. Neste contexto, este trabalho teve como objetivos investigar a ocorrência de novas isoformas de Mo-CBP3 e caracterizar estas proteínas quanto à presença de modificações
pós-traducionais (MPTs). O trabalho empregou uma abordagem experimental que incluiu (1) clonagem e sequenciamento de fragmentos de DNA genômico codificando Mo-CBP3; (2)
análise de massas moleculares por LC-ESI-MS; e (3) análise computacional, com o objetivo de correlacionar massas teóricas e experimentais. Um total de 32 clones diferentes de Mo -CBP3 foram obtidos e completamente sequenciados. Após alinhamento múltiplo das
sequências de aminoácidos deduzidas foi possível agrupá-las em oito grupos distintos. Destas, quatro isoformas foram descritas anteriormente (Mo-CBP3-1, Mo-CBP3-2, Mo-CBP3-3 e Mo
-CBP3-4), enquanto que as isoformas Mo-CBP3-2A (163 resíduos de aminoácidos), Mo-CBP3
-2B (162 resíduos), Mo-CBP3-3A (160 resíduos) e Mo-CBP3-3B (160 aa) foram descritas pela
primeira vez neste trabalho. Análises das sequências de aminoácidos, deduzidas de DNA genômico, sugeriram que as isoformas de Mo-CBP3 são sintetizadas como preproproteínas,
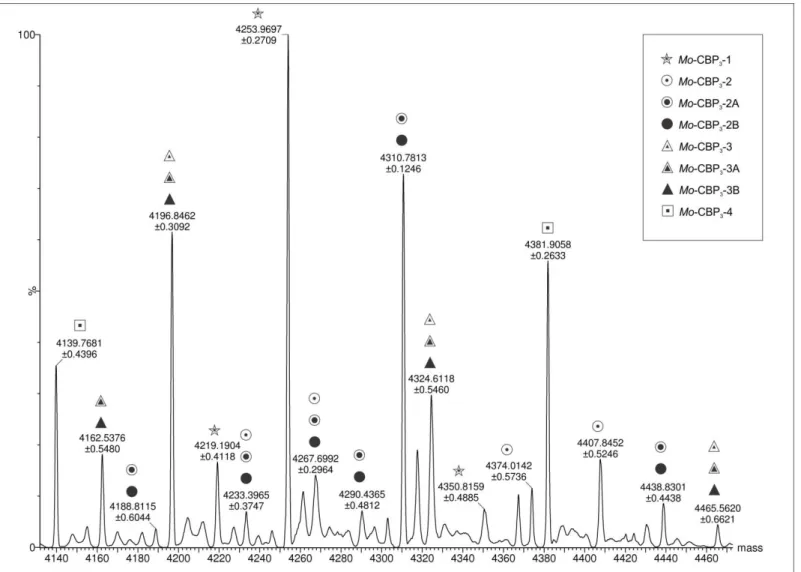
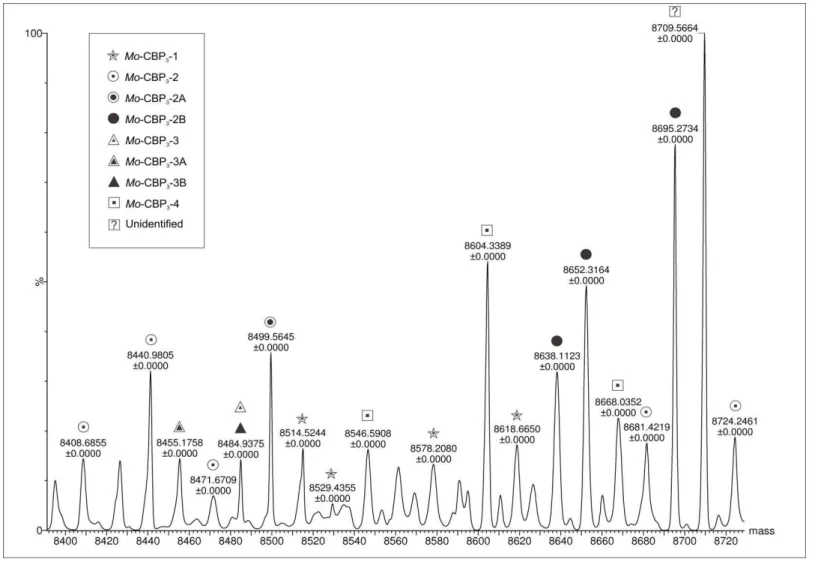
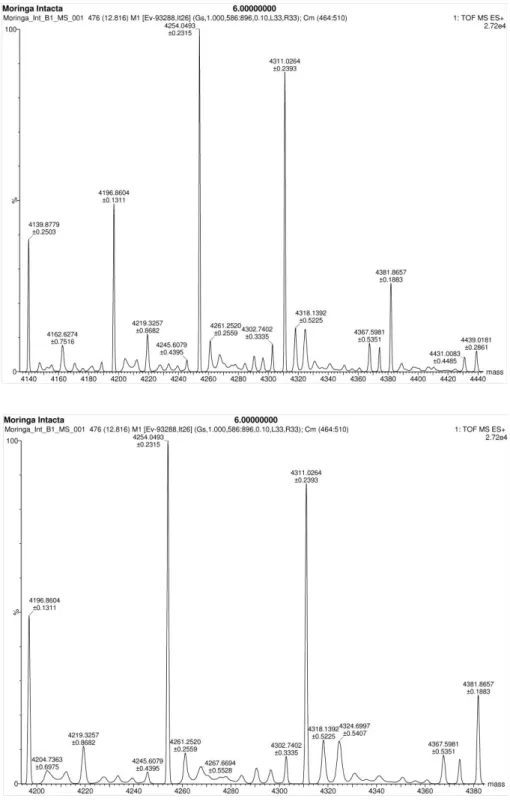
contendo um peptídeo sinal N-terminal, um propeptídeo N-terminal, uma cadeia menor com cerca de 4 kDa, um peptídeo de ligação entre as duas cadeias, uma cadeia maior com aproximadamente 8 kDa, e uma extensão C-terminal. ESI-MS revelaram 147 valores distintos de massas moleculares, dos quais 89 correspondiam às variantes da cadeia menor e 58 às variantes da cadeia maior. Valores de massa molecular foram calculados a partir das sequências de aminoácidos, assumindo diferentes graus de processamento proteolítico nas extremidades N- e C-terminais de cada cadeia. Além disso, possíveis alterações produzidas por diferentes PTMs, como hidroxilação de prolina, fosforilação de serina ou treonina, oxidação de metionina e cliclização de glutamina N-terminal em piroglutamato (pGlu), foram também levados em consideração. Usando essa estratégia, a maioria das massas experimentais da cadeia menor (72 de um total de 89) e quase todas as massas da cadeia maior (57 de um total de 58) puderam ser atribuídas a sequências específicas de aminoácidos, das diferentes isoformas de Mo-CBP3. Estes resultados sugerem que: i) as isoformas de Mo-CBP3 são
multigênica; ii) uma vez processados, os precursores codificados a partir dos diferentes mRNAs originam duas cadeias, uma menor (4 kDa) e outra maior (8 kDa), unidas por pontes dissulfeto; as extremidades N- e C-terminais de ambas as cadeias podem sofrer processamento proteolítico, resultando na remoção de um ou alguns resíduos de cada sequência, gerando cadeias com massas ligeiramente distintas; iii) modificações pós-traducionais em determinados resíduos de uma das cadeias polipeptídicas ou de ambas são uma terceira fonte de variação na massa molecular, aumentando substancialmente o número de isoformas. Em conclusão, uma mistura extremamente complexa de isoformas de Mo-CBP3,
oriundas de alguns poucos genes, é produzida mediante diferentes combinações de PTMs distintas. Estes resultados constituem importantes avanços na compreensão dos mecanismos pós-traducionais inerentes que operam durante a biossíntese das albuminas 2S nas sementes da M. oleifera.
ABSTRACT
The study of 2S albumin expressed in Moringa oleifera seeds has been a very complex process, especially when using conventional proteomic techniques due to the presence of many isoforms. The great diversity of protein isoforms may be dueto the multigenic origin, in addition to the intense post-translational processing. In this context, the aim of this work was to investigate the occurrence of new Mo-CBP3 isoforms and characterize these proteins for the
presence of post-translational modifications (PTMs). The work employed an experimental approach which included (1) cloning and sequencing of genomic DNA fragments encoding
Mo-CBP3; (2) molecular mass analysis through LC-ESI-MS; and (3) computational study,
with the objective to correlate theoretical and experimental masses. A total of 32 different
Mo-CBP3 clones were obtained and completely sequenced. After multiple alignments of the
deduced amino acid sequences it was possible to categorize them into eight distinct isoform groups. Four of these isoforms have been previously described (Mo-CBP3-1, Mo-CBP3-2, Mo
-CBP3-3 and Mo-CBP3-4), whereas Mo-CBP3-2A (163 amino acid residues), Mo-CBP3-2B
(162 residues), Mo-CBP3-3A (160 residues) and Mo-CBP3-3B (160 aa) isoforms were first
reported in this work. Analysis of the amino acid sequences, deduced from the genomic DNA, suggested that Mo-CBP3 isoforms are synthesized as preproproteins containing an N-terminal
signal peptide, an N-terminal propeptide, a small chain of about 4 kDa, a peptide linkage between the two chains, a large chain with approximately 8 kDa, and a C-terminal extension. ESI-MS experiments revealed 147 distinct values of molecular mass, of which 89 corresponded to variants of the small chain and 58 to variants of the large chain. Molecular mass values were calculated from the amino acid sequences, assuming different degrees of proteolytic processing at the N- and C-terminal ends of each chain. In addition, possible changes produced by different PTMs, such as proline hydroxylation, serine or threonine phosphorylation, methionine oxidation, and the cyclization of the N-terminal glutamine into pyroglutamate (pGlu) were also taken into account. Using this strategy, the majority of the experimental masses of the small chain (72 of a total of 89) and almost all experimental masses corresponding to the large chain (57 out of 58) could be attributed to specific amino acid sequences of the different Mo-CBP3 isoforms. These results suggest that: i) Mo-CBP3
residues from each sequence, yielding chains with slightly different masses; iii) post-translational modifications in certain residues of one of the polypeptide chains or both are a third source of variation in the molecular mass, substantially increasing the number of isoforms. In conclusion, an extremely complex mixture of Mo-CBP3 isoforms, derived from a
few genes, is produced by different combinations of distinct PTMs. These results constitute important advances in understanding the inherent post-translational mechanisms that operate during the biosynthesis of 2S albumins in seeds of M. oleifera.
SUMÁRIO
1 INTRODUÇÃO GERAL... 13
2 FUNDAMENTAÇÃO TEÓRICA... 16
3 HIPÓTESE... 30
4 OBJETIVOS... 31
5 POST-TRANSLATIONAL MODIFICATIONS REVEAL THAT Mo -CBP3, A 2S MORINGA OLEIFERA CHITIN-BINDING ALBUMIN, IS A COMPLEX MIXTURE OF ISOFORMS... 39
6 CONSIDERAÇÕES FINAIS... 69
REFERÊNCIAS... 70
1 INTRODUÇÃO GERAL
A Moringa oleifera é uma planta pertencente à família Moringaceae, oriunda da região sub-himalaia, a noroeste da Índia, sendo mundialmente conhecida por vários codinomes dentre eles: drumstick tree (RAMACHANDRAN; PETER; GOPALAKRISHNAN, 1980) e árvore do milagre, devido as suas inúmeras propriedades benéficas, incluindo seu potencial nutricional (SAHAY; YADAV; SRINIVASAMURTHY, 2017; KOU et al., 2018; SREELATHA; JEYACHITRA; PADMA, 2011).
No Brasil, notadamente na região Nordeste, a M. oleifera foi introduzida por volta da década de 1950, sendo largamente utilizada, especialmente na purificação de água para fins de uso doméstico devido a presença de moléculas floculantes naturais em suas sementes (VICENTE et al., 2017; ABIYU et al., 2018). Além disso, assementes de M. oleifera detêm grande importância industrial alimentícia, uma vez que apresentaelevada quantidade de óleo de boa qualidade, sendo o principal constituinte o ácido oléico com até 78,7% (LEONE et al., 2016). Outra razão, pela qual a M. oleifera tem chamado de muitos pesquisadores diz respeito aos efeitos deletérios causados sobre vírus (LIPIPUN et al., 2003), bactérias (ELGAMILY et al., 2016), fungos (ELGAMILY et al., 2016; NETO et al., 2017), artrópodes (FERREIRA et
al., 2009) e nematódeos(ONYEKE; AKUESHI, 2012).
Destra as moléculas antifúngicas identificadas na M. oleifera se destacam as proteínas ligantes à quitina chamada genericamente de Mo-CBP (chitin-binding protein from Moringa
oleifera), com especiais destaques: a Mo-CBP3, uma proteína termoestável capaz de flocular e
inibir atividade contra as espécies de fungos fitopatogênicos Fusarium solani, F. oxysporum,
Colletotrichum musae e C. gloesporioides (GIFONI et al., 2012) e a Mo-CBP2 capaz de inibir
Candida albicans, C. parapsilosis, C. krusei e C. tropicalis (NETO et al., 2017),
respectivamente.
No que diz respeito a Mo-CBP3, além de seu potencial antifúngico, possui ainda
resistência a uma ampla faixa de temperatura e pH. Segundo Freire e colaboradores (2015), a
Mo-CBP3 constitui produtos de origem multigênica, uma vez que são conhecidas atualmente
quatro isoformas: Mo-CBP3-1, Mo-CBP3-2, Mo-CBP3-3 e Mo-CBP3-4. Embora tenha sido
KREBBERS et al., 1988; (MYLNE; HARA-NISHIMURA; ROSENGREN, 2014), que ainda compartilham características que incluem resíduos catiônicos, ligações dissulfeto e peptídeos antimicrobianos (NAWROT et al., 2014).
Muitas dessas proteínas são sintetizadas como pré-pro-albuminas, as quais apresentam uma sequência N-terminal (peptídeo sinal), uma extensão do N-terminal que separa o peptídeo sinal da cadeia maior da cadeia menor, um linker ou pequena sequência de aminoácidos que liga ambas as cadeias polipeptídicas e, uma sequência C-terminal, regiões essas ausentes na proteína madura, a exemplo a pré-pro-Mabinlin-II de Capparis masaikai (NIRASAWA et al., 1993) e a pré-pro-Mo-CBP3 de M. oleifera (FREIRE et al., 2015).
Embora, as albuminas 2S apresentem diferentes tamanhos de precursores, a estruturas dessas proteínas têm sido muito conservadas, sendo sua estabilidade mantida por oito resíduos de cisteínas ligadas por pontes dissulfeto (..C..C../..CC..CXC..C..C..) (PANTOJA-UCEDA et al., 2002). De modo geral a estrutura das albuminas 2S são organizadas em quatro α-hélices, sendo mantido esse padrão por quatro pontes dissulfeto de maneira similar aos encontrados nos inibidores bifuncionais de α-amilase/tripsina e nas proteínas transportadoras de lipídios não-específicas – nsLTPs, de non-specific Lipid Transfer Proteins (CÂNDIDO et al., 2011; EDSTAM et al., 2011; NAWROT et al., 2014). Entretanto, há relato de algumas albuminas 2S que exibem apenas três α-hélices como a Ber e 1 (PDB: 2LFV) de Bertholletia excelsa, enquanto que outras podem apresentar até cinco α-hélices como o caso observado na proteína Napin BnIb (PDB: 1PNB) de Brassica napus (RICO, et al., 1996).
Apesar dos avanços na caracterização e do modo de ação de Mo-CBP3 esta proteína não
é compreendida completamente. Acredita-se que Mo-CBP3 possa influênciar negativamente a
atividade das bombas de prótons (H+-ATPase) presentes na membrana celular, além de causar
deformações na parede de células de fungos fitopatogênicos (BATISTA et al., 2014), tais como: F. solani, F. oxysporum, Colletotrichum musae e C. gloesporioides (GIFONI et al., 2012). Resultados obtidos por espectrometria de massas e PAGE-SDS admitiram que Mo -CBP3 é formada por duas cadeias polipeptídicas, em torno de 4,1 kDa e 8,1 kDa (BATISTA
et al., 2014; FREIRE et al., 2015).
Apesar de recentes estudos estarem buscando respostas sobre o mecanismo de ação desta proteína, é necessário grande esforço a fim de descrever todas as isoformas existentes de
Mo-CBP3, bem como entender o papel fisiológico de cada uma delas, visto a grande
importância fisiológica das albuminas durante o desenvolvimento dos vegetais, além dos mecanismos de defesa que estas estão envolvidas. Embora existam isoformas de Mo-CBP3
2 – FUNDAMENTAÇÃO TEÓRICA
Moringa oleifera Lamarck
Considerações gerais
A Moringa oleifera (Figura 1) é uma planta pertencente à família Moringaceae, originária da região sub-himalaia, a noroeste da Índia, sendo mundialmente conhecida como
drumstick tree (RAMACHANDRAN; PETER; GOPALAKRISHNAN, 1980; GANGULY;
GUHA, 2008), pode ainda ser conhecida como horseradish (SHIH et al., 2011) ou árvore do milagre, devido seus inúmeros atributos benéficos, incluindo seu potencial nutricional (SAHAY; YADAV; SRINIVASAMURTHY, 2017; KOU et al., 2018; SREELATHA; JEYACHITRA; PADMA, 2011).
FIGURA 1– Características morfológicas da Moringa oleifera.
Fonte: elaborada pelo autor.
brasileiro devido seu emprego no tratamento de água para uso doméstico. O gênero Moringa é constituído por treze espécies (RANI; HUSAIN; KUMOLOSASI, 2018) bastante estudadas, em razão, de propriedades floculantes presentes em suas sementes e, consequentemente, sua aplicação para purificar água para consumo humano e animal. Muitas espécies pertencentes a gênero Moringa, especialmente a M. oleifera, se caracteriza, também, por sua resistência a diferentes espécies de pragas, facilidade de cultivo e rapidez no crescimento (RAMACHANDRAN; PETER; GOPALAKRISHNAN, 1980).
Aproveitamento da Moringa oleifera
Devido o atributo floculante apresentado pelas sementes de M. oleifera, esta espécie tem sido de grande relevância, sobretudo, em países subdesenvolvidos, onde é comum o uso de suas sementes para purificar a água de uso doméstico (VICENTE et al., 2017; ABIYU et al., 2018). As propriedades floculantes presentes em sementes de M. oleifera foram atribuídas a polieletrólitosde 3,0 kDa, globulinas e albuminas (OKUDA et al., 2001; ZAKU et al., 2015; BAPTISTA et al., 2017). Outros estudos relacionaram essa propriedade a proteínas catiônicas com massa molecular entre 6,5 e 13,0 kDa (GASSENSCHMIDT et al., 1995; NDABIGENGESERE; NARASIAH; TALBOT, 1995; GHEBREMICHAEL et al., 2005; GOYAL et al., 2007), ou a polipeptídeos (CHEN, 2009; MANGALE; CHONDE; RAUT, 2012). Além disso, as sementes de M. oleifera detêm importância industrial, pois apresentaelevada quantidade de óleo de boa qualidade, sendo o principal constituinte o ácido oléico com até 78,7% (LEONE et al., 2016).
Além dos atributos ponderados acima, a M. oleifera tem sido emprega a séculos na medicina popular, em diversos continentes. Por essa razão, diversos estudos têm sido delineados a fim de isolar moléculas com potencial biotecnológico a partir dessa planta. Em um estudo desenvolvido por Lipipun e colaboradores (2003), foi demonstrado que extrato etanólico obtido a partir de folhas de M. oleifera possui efeito antiviral contra o HSV-1 (vírus
Herpes simplex tipo 1), inibindo o desenvolvimento viral in vitro, minimizando a mortalidade
de murinos previamente infectados com o HSV-1.
Há décadas, agentes antimicrobianos são identificados e isolados a partir de sementes de M. oleifera. Tanto extratos etanólicos quanto aquosos foram capazes de inibir o crescimento dos microrganismos: Bacillus subitilis, Escherichia coli, Pasturella multocida,
Pseudomonas aeruginosa e Staphylococcus aureus, S. mutans e Vibrio cholerae (CÁCERES
2016). Efeitos adversos contra espécies de fungos foram comprovados em: Basidiobolus
haptosporus, B. ranarum, Candida albicans, Colletotrichum gloeosporioides, C. musae,
Fusarium solani, F. oxysporum, Microsporum canis, Rhizopus solani, Trichophyton rubrum e
T. mentagrophytes (NWOSU; OKAFOR, 1995; JABEEN et al., 2008; ROCHA et al., 2011;
GIFONI et al., 2012; BATISTA et al., 2014; ELGAMILY et al., 2016; NETO et al., 2017). Efeito deletério sobre o mosquito Aedes aegypti (FERREIRA et al., 2009) e nematicida contra o Meloidogyne incognita (ONYEKE; AKUESHI, 2012), também foram observados.
Grande parte das propriedades discorridas acima tem sido atribuída a moléculas isoladas a partir das sementes da planta M. oleifera. Segundo Gallão e colaboradores (2006), para cada 100 g de farinha há o equivalente a 39,3 g de proteínas nas sementes de moringa. Portanto, é possível que muitas classes de proteínas expressas nas sementes de M. oleifera possam estar diretamente relacionadas a várias propriedades atribuídas a esta parte da planta.
Dentre as proteínas já purificadas de sementes de moringa destacam-se: uma proteína catiônica com capacidade de flocular partículas suspensas em água (AGRAWAL; SHEE; SHARMA, 2007). Makkar e Becker (1997), sugeriram que propriedades antinutricionais inerentes às sementes dessa espécie podem estar associadas a uma lectina. A hipótese foi corroborada após a identificação de duas lectinas hemaglutinantes básicas chamadas cMol (30 kDa) e WSMoL (20 kDa), termoestáveis e com atividade nas faixas de pH 4.0-9.0 e de 4.5-9.5, respectivamente (SANTOS et al., 2009; ROLIM et al. 2011).
Proteínas ligantes à quitina chamada Mo-CBP4 apresentaram propriedades
antiinflamatória e antinociceptiva (PEREIRA et al., 2011), em seguida foi descrita outra proteína denominada Mo-CBP3 termoestável capaz de flocular e inibir as espécies de fungos
fitopatogênicos Fusarium solani, F. oxysporum, Colletotrichum musae e C. gloesporioides (GIFONI et al., 2012). Mais recentemente foi caracterizada uma proteína chamada de Mo -CBP2 capaz de inibir Candida albicans, C. parapsilosis, C. krusei e C. tropicalis.
Devido ao potencial antifúngico, resistência a ampla faixa de temperatura e pH, a proteína Mo-CBP3 pode ser útil no desenvolvimento de medicamentos antifúngicos. Esta, no
entanto, constitui produtos de origem multigênica (FREIRE et al., 2015), sendo, conhecidas atualmente quatro isoformas identificadas como: Mo-CBP3-1, Mo-CBP3-2, Mo-CBP3-3 e Mo
-CBP3-4. Embora tenha sido identificada como uma proteína ligante à quitina, estudos
Albuminas 2S Vegetais
Considerações gerais
O termo “albumina” foi um codinome genérico atribuído inicialmente para qualquer proteína solúvel em água (MYLNE; HARA-NISHIMURA; ROSENGREN, 2014). Outrossim, essas proteínas já foram chamadas de arabins, devido a sua similaridade com a napin (Brassica napus), primeira albumina 2S descrita (HEATH et al., 1986).
Atualmente, as albuminas 2S constituem uma classe de pequenas (1,7S – 2,2S) proteínas de armazenamento em plantas, ricas em aminoácidos cisteínas e glutaminas em regiões conservadas, geralmente classificadas com base em seu coeficiente de sedimentação (YOULE; HUANG, 1981; KREBBERS et al., 1988), ainda compartilham características que incluem resíduos catiônicos, ligações dissulfeto e peptídeos antimicrobianos (NAWROT et al., 2014). Essas proteínas pertencem à superfamília das prolaminas e, compreendem as principais proteínas em sementes e frutos de cascas rígidas, sendo amplamente distribuídas em plantas monocotiledôneas e dicotiledôneas (BREITENEDER; RADAUER, 2004; GUPTA; GAUR; SALUNKE, 2008).
Como proteínas de armazenamento, as albuminas 2S ficam estocadas sob forma de corpos proteicos (protein storage vacuoles - PSVs), sendo empregadas como fonte de nutrientes (aminoácidos) e esqueletos de carbono durante a germinação e crescimento de plântulas (YOULE; HUANG, 1981; LI et al., 2006; CANDIDOet al., 2011). Acredita-se que o transporte das proteínas de reserva energética para os PSVs é regulado por enzimas proteolíticas presentes nas sementes (TAN-WILSON; WILSON, 2012), com base na co-localização subcelular tanto das enzimas e das proteínas reserva, bem como das condições adequadas para a atividade das enzimas proteolíticas (WILSON et al., 2016).
Para Wang e Bunkers (2000), as plantas podem usar proteínas de armazenamento para gerar proteínas antimicrobianas que as protegem contra o ataque de seus inimigos naturais, especialmente durante a germinação das sementes, quando as plântulas encontram-se vulneráveis devido à falta do sistema de defesa induzível mais organizado.
Biossíntese das albuminas 2S
sofre extenso processamento proteolítico em vacúolos, resultando em duas subunidades, sendo a maior entre 8-14 kDa e a menor entre 3-10 kDa (SHEWRY; NAPIER; TATHAM, 1995; MORENO; CLEMENTE, 2008; NAWROT et al., 2014).
Muitas dessas proteínas são sintetizadas como pré-pro-albuminas, as quais apresentam uma sequência N-terminal (peptídeo sinal), uma extensão do N-terminal que separa o peptídeo sinal da cadeia maior da cadeia menor, um linker ou pequena sequência de aminoácidos que liga ambas as cadeias polipeptídicas e, uma sequência C-terminal, regiões essas ausentes na proteína madura, a exemplo a pré-pro-Mabinlin-II de Capparis masaikai (NIRASAWA et al., 1993) e a pré-pro-Mo-CBP3 de M. oleifera (FREIRE et al., 2015). Um
esquema geral é mostrado na figura 2.
FIGURA 2- Esquema geral de uma pré-pro-albumina.
Fonte: elaborada pelo autor.
Dentre os eventos inerentes aos processos de maturação das albuminas 2S em sementes, a sequência N-terminal se destaca, sendo ela a responsável pelo direcionamento dessas proteínas ainda inativas para o vacúolo de armazenamento, consistindo, portanto, um mecanismo geral de ontogenia nesses órgãos (VITALE; GALILI, 2001). É provável que o direcionamento das albuminas 2S para os vacúolos de armazenamento seja devido à maior abundância deste quando comparados aos vacúolos líticos (MARTINOIA; MAESHIMA; NEUHAUS, 2007).
No que diz respeito aos eventos metabólicos inerentes as albuminas 2S, em muitos casos, o processamento parece ser muito dispendioso, uma vez que os precursores apresentam peso molecular elevado, a exemplo da albumina 2S de Helianthus annuus (HaG5)
Peptídeo sinal
Extensão N-terminal
Cadeia menor
Cadeia maior
Extensão C-terminal
que possui massa molecular de 38 kDa, sendo reduzido em dois peptídeos de tamanhos aproximados de 19 kDa (ALLEN et al., 1987). Em sementes da planta Ricinus communis, um precursor da albumina 2S (2S ASP-Ib) com peso molecular de 29,3 kDa, após os processos proteolíticos pode resultar em até em duas isoformas com subunidades menores 4 kDa e 4,3 kDa e as subunidades maiores em torno de 7 kDa e 8 kDa, respectivamente (SHARIEF; LI, 1982; IRWIN et al., 1990; SILVA et al., 1996).
Há, no entanto, algumas plantas que parecem ter desenvolvido mecanismos para evitar essa perda energética durante a síntese de albuminas, produzindo precursores muito menores, como as albuminas Ses i 1 e Ses i 2 de Sesamum indicum que possui um precursor conhecido com massa molecular próximo de 17,5 kDa (AF091841) e 17,4 kDa (ID: AF240005), respectivamente (TAI et al., 1999; TAI et al., 2001; MORENO et al., 2005).
Após os processos proteolíticos inerentes as proteínas Ses i 1 e Ses i 2, o resultado são dois heterodímeros, sendo o correspondente a proteína Ses i 1 madura uma cadeia menor em torno de 4 kDa e a cadeia maior em trono de 8 kDa, e para a proteína Ses i 2 uma cadeia menor em torno de 4,9 kDa e a maior em trono de 8,7 kDa (TAI et al., 1999; MORENO et al., 2005). A Malva parviflora é outra espécie de planta que compartilha mecanismos que evitam a perda de energia durante a síntese da albumina 2S CW-1, após seu processamento resulta em duas subunidades de 5 kDa e 3 kDa (WANG; BUNKERS, 2000). Há ainda albuminas 2S que apresentam apenas um único polipeptídio (KHAN et al., 2016).
É possível que em precursores menores de albuminas 2S, forme-se apenas uma subunidade como é o caso da albumina 2S (SFA-8), isoladas de sementes de H. annuus que possui massa molecular de 12,1 kDa (KORTT; CALDWELL, 1990). Há, no entanto, pesquisas mais recentemente que indicam haver albuminas 2S sintetizadas a partir de precursor de tamanho aproximadamente 13 kDa (HUMMEL; WIGGER; BROCKMEYER, 2015), resultando em proteínas maduras compostas por subunidades de cerca de 9 a 10 kDa (subunidade maior) e 3 a 4 kDa (subunidade menor), respectivamente.
FIGURA 3 - Representação esquemática do padrão de pontes de sulfuto formadas entre os oito resíduos de cisteínas conservados na família de albumina 2S.
Fonte: elaborada pelo autor.
Há relatos que algumas albuminas 2S são organizadas estruturalmente em quatro α-hélices, sendo mantido esse padrão por quatro pontes dissulfeto de maneira similar aos encontrados nos inibidores bifuncionais de α-amilase/tripsina e nas proteínas transportadoras de lipídios não-específicas – nsLTPs, de non-specific Lipid Transfer Proteins (CÂNDIDO et al., 2011; EDSTAM et al., 2011; NAWROT et al., 2014). Porém, até a presente data, exceto a albumina 2S: Ber e 1 (PDB: 2LFV) de Bertholletia excelsa que apresenta três α-hélices (RUNDQVIST et al., 2012), todas as estruturas (Figura 4) de albuminas 2S resolvidas apresentam cinco α-hélices como observadas nas proteínas: Napin BnIb (PDB: 1PNB) de
Brassica napus (RICO, et al., 1996), RicC3 (PDB: 1PSY) de Ricinus communis
(PANTOJA-UCEDA, et al., 2003), napin BnIb (PDB: 1SM7) de Brassicanapus (PANTOJA-UCEDA, et al., 2004a), SFA-8 (PDB: 1S6D) de Helianthus anuus (PANTOJA-UCEDA, et al., 2004b), Ara h 2 (PDB: 1W2Q) de Arachis hypogaea (LEHMANN, et al., 2006), Mabinlin II (PDB: 2DS2) de Capparis masaikai (LI, et al., 2008) e Mo-CBP3-4 (PDB: 5DOM) de Moringa
oleifera (ULLAH, at al., 2015).
Atividades biológicas já descritas para as albuminas 2S
de reserva), como mostrado na Arabidopsis thaliana (VAN DER KLEI et al., 1993), em
Bertholletia excelsa (MORENO et al., 2004), S. indicum (ORRUÑO; MORGAN, 2011), na
Corylus avellana (PFEIFER et al., 2015), na M. oleifera (FREIRE et al., 2015) e no A.
hypogaea (BOUALEG; BOUTEBBA, 2017), por exemplo. Em consequência, o isolamento
de albumina específica tem se mostrado um processo complexo, quando se emprega técnicas convencionais devido à presença de muitas isoformas.
É plausível que a diversidade de formas pode ser resultante, em parte, devido suas origens multigênicas e, por consequente, intenso processamento pós-traducional que essas proteínas sofrem (CÂNDIDO et al., 2011; PFEIFER et al., 2015). Além desses fatores, há ainda a influência de fenômenos ambientais incluindo temperatura, umidade e salinidade que podem induzir a ocorrência de variações alélicas em sequências de proteínas entre os genótipos de plantas, resultando em uma heterogeneidade no pool de proteínas quando a amostra não é de origem geneticamente uniforme.
Em muitos casos, é imperativo a clonagem e expressão de albuminas 2S presentes em uma dada amostra, para então caracterização dessas individualmente como ocorreu com as albuminas de B. excelsa (GANDER et al., 1991), de Glycine max (LIN et al., 2006) e de C.
avellana (GARINO et al., 2010b).
Potencial antifúngico de albuminas 2S
Há décadas, estudos científicos têm demonstrado o grande potencial que as albuminas 2S detêm contra espécies fúngicas. Dentre essas moléculas, destacam-se alguns exemplos isolados a partir de sementes das plantas:
A. hypogaea: A albumina heterodimérica 2S-1 pode existir em pelo menos três
isoformas (DUAN et al., 2013), suporta variação de 30 a 100 °C durante 10 minutos e variação de pH de 2,0 a 11,0. Possui potencial inibitório contra o fungo Aspergillus flavus (DUAN et al., 2013).
Capsicum annuum: A albumina 2S chamada Ca-Alb estruturalmente caracterizada
como um heretodímero (4 kDa e 9 kDa), e funcionalmente ativo contra as espécies de leveduras Candida tropicalis, Kluyveromyces marxiannus e Saccharomyces cerevisiae (RIBEIRO et al., 2011a).
Cucurbita maxima: A albumina 2S (sem nomenclatura) apresentou atividade
FIGURA 4 - Estrutura de albuminas 2S já resolvidas. (A) 2LVF de Bertholletia excelsa, (B) 1PNB de B. napus, (C) 1PSY de Ricinus communis, (D) 1SM7 de Brassica napus, (E) 1S6D de Helianthus annuus, (F), 1W2Q de Arachis hypogaea, (G) 2DS2 de Capparis masaikai e (H) 5DOM de Moringa oleifera. As α-hélices em vermelho (cadeia menor) e α-hélices em azul (cadeia maior).
Fonte: elaborada pelo autor.
A B C D
2LFV 1PNB 1PSY 1SM7
E F G H
1W2Q 2DS2
H. annuus: Um pool de albuminas identificadas como Ha-AF15, embora tenha
apresentado atividade minimizando o crescimento das espécies Sclerotiniasclerotiorum e F.
solani, não foi capaz de inibir completamente a germinação de esporos desses fungos
(REGENTE; DE LA CANAL, 2001).
M. parviflora: Duas albuminas 2S nomeadas como CW-1 e CW-4 até o presente
momento foram identificadas. A isoforma CW-1 caracteriza-se como uma proteína heterodimérica, possuindo massa molecular da cadeia maior em torno de 6 kDa e da cadeia menor entorno de 3 kDa (WANG; BUNKERS, 2000). Análises adicionais com a CW-1 evidenciaram que essa proteína suporta variações de concentração de sal, e ainda exerce efeitos deletérios contra o fungo F. graminearum (WANG; BUNKERS, 2000). Segundo Wang e colaboradores (2001), a isoforma CW-4 possui uma cadeia polipeptídica de 5 kDa, e mesmo depois de ser incubada a 100 ºC durante 20 minutos, ainda permanece ativa contra o fungo Phytophthora infestans.
M. oleifera: Várias albuminas 2S foram identificadas, sendo elas formas
heterodiméricas com cadeias polipeptídicas com massa moleculares próximas de 8,1 kDa e 4,1 kDa (FREIRE et al., 2015). Estas isoformas possuem termoestabilidade permanecendo ativas após serem incubadas durante 1 h a 100 ºC (GIFONI et al., 2012). Atualmente, são denominadas de Mo-CBP3-1, Mo-CBP3-2, Mo-CBP3-3, Mo-CBP3-4 (FREIRE et al., 2015) e
Mo-CBP2 (NETO et al., 2017). Estas proteínas possuem atividade inibitória contras as
espécies F. solani, F. oxysporum, C. musae and C. gloeosporioides (GIFONI et al., 2012; BATISTA et al., 2014).
Passiflora alata: Até a presente data apenas a albumina 2S chamada Pa-AFP1 foi
identificada. Essa proteína é formada por duas cadeias polipeptídicas, sendo a massa da cadeia maior 7 kDa e a massa da cadeia menor 4,5 kDa, e tem apresentado atividade inibitória contra o fungo C. gloeosporioides (RIBEIRO et al., 2011b).
P. edulis f. flavicarpa: Entre as albuminas 2S purificadas desta planta podemos
destacar três heterodiméricas, sendo elas Pf1 (3,5 kDa e 8 kDa), Pf2 (3,5 kDa e 8 kDa) e a Pf -Alb (5 kDa e 9 kDa) (AGIZZIO et al., 2003; RIBEIRO et al., 2011a). Em um ensaio in vitro, as proteínas Pf1 e Pf2, bem como a associação contendo ambas as proteínas, inibiram o crescimento dos fungos F. oxysporum e F. solani, bem como a levedura S. cerevisiae e inibiu fortemente a acidificação estimulada pela glicose do meio por F. oxysporum de forma dose dependente (AGIZZIO et al., 2003). A Pf-Alb foi capaz de causar efeitos deletérios nas espécies de leveduras C. albicans, C. guilliermondii, C. parapsilosis, C. tropicalis
Putranjiva roxburghii: A albuminas 2S identificada como putrin, possui duas
cadeias polipeptídicas, a maior em torno de 7,5 KDa e a menor em torno de 4,5 kDa. Análise realizada a fim de evidenciar potencial antifúngico, demonstrou que a putrin é ativa contra
Aspergillusflavus, F. oxysporum e Phanerochaetechrysosporium (TOMAR et al., 2014b).
Raphanus sativus: Conhecidas como Rs-2Ss (Rs-2S1 - Rs-2S5), este grupo de
isoformas são formadas por duas cadeias polipeptídicas, sendo a maior em torno de 10 kDa e a menor próximo de 4 kDa (TERRAS et al., 1992). Ensaios realizados têm demonstrado que o
pool proteico formado por essas albuminas, mesmo após tratamento a 100 ºC durante 10
minutos é capaz de inibir as espécies fúngicas: Alternariabrassicola, Ascochyta pisi, Botrytis
cinerea, C. lindemuthianum, F. culmorum, F. oxysporum f.sp. lycopersici, F. oxysporum f.sp.
pisi, Mycosphaerella fijiensis var. fijiensis, Nectria haematococca, Phoma betae,
Phytophthora infestans, Pyricularia oryzae, Trichoderma hamatum e Verticillium dahliae
(TERRAS et al., 1992; TERRAS et al., 1993).
Taraxacum officinale: Cinco isoformas de albuminas 2S foram identificadas,
porém a capacidade inibitória contra fungos foi observada somente nas isoformas: To-A1, To -A2, To-A3, todas elas apresentando características heterodiméricas, com cadeia maior entrono de 10 kDa e a menor entrono de 4 kDa (ODINTSOVA et al., 2010). Os ensaios antifúngicos mostraram que as isoformas To-A diferiram em seu efeito antifúngico: To-A1 foi capaz de inibição a germinação das espécies Helminthosporium sativum (Bipolaris sorokiniana) e
Phoma betae; To-A2 apresentou igual efeito contra as espécies H. sativum, P. betae e F.
oxysporum; enquanto que a To-A3 foi ativa contra os microrganismos H. sativum, P. betae e
Verticillum albo-atrum (ODINTSOVA et al., 2010).
Embora, muitos dos estudos científicos que abordam atividade inibitória de albuminas 2S contra fungos tenham sido, de maneira geral, direcionados para fungos fitopatogênicos, é provável que estas proteínas compartilhem essa mesma propriedade funcional quando testadas contra fungos de interesse clínico. Tal hipótese tem sido corroborada por algumas albuminas 2S, por exemplo, a Mo-CBP2, uma proteína purificada de
sementes de M. oleifera, que foi capaz de inibir crescimento de espécies de fungos de interesse hospitalar, apresentando atividade contra as espécies, C. albicans, C. parapsilosis,
Potencial antibacteriano de albuminas 2S
Nos últimos dez anos, estudos científicos têm demonstrado também que algumas albuminas 2S possuem atividade antibacteriana. Dentre essas proteínas, destaca-se a albumina 2S (SiAMP2) isolada de sementes de Sesamum indicum, que apresentou atividade inibitória contra espécies pertencentes ao gênero Klebsiella sp., (MARIA-NETO et al., 2011), a albumina 2S (Putrin) isolada de sementes de Putranjiva roxburghii, apresentou atividade contras as espécies Bacillus subtilis e Micrococcus flavus (TOMAR et al., 2014b), e a albumina (Rc-2S-Alb) isolada de R. communis, apresentou atividade contra as espécies B.
subtilis, Klebsiella pneumonia e Pseudomonas aeruginosa (SOUZA et al., 2016).
Potencial alergênico de albuminas 2S
Durante décadas, muitas proteínas presentes em sementes de plantas têm sido associadas a moléculas potencialmente alergênicas, dentre essas, muitas são albuminas. Dentre as amêndoas, as quais as albuminas constituem as principais moléculas alergênicas, merecem destaque as albuminas: Sin a I isolada de Sinapis Alba (MENÉNDEZ-ARIAS et al., 1988), SFA-8 isolada de H. annuus (PANTOJA-UCEDA et al., 2004), Ana o 3 isolada de
Anacardium occidentale (ROBOTHAM et al., 2005), Pis v 1 isolada de Pistacia vera (AHN
et al., 2009), Cor a 14 isolada de C. avellana (GARINO et al., 2010; PFEIFER et al., 2015), Car i 1 isolada de Carya illinoinensis (SHARMA et al., 2011), e as albuminas Gly m 5 e Gly m 6 isoladas de G. max (EBISAWA et al., 2013), todas elas têm induzindo sérios efeitos nocivos locais e/ou sistêmicos.
Potencial atividade nucleásica de albuminas 2S
fragmentar de DNA, sendo esta isolada de semente da planta M. oleifera (NETO et al., 2017) e, mais recentemente a albumina 2S (WTA), purificada de sementes de Wrightia tinctoria que apresentou atividade DNásica (SHARMA et al., 2017).
Outras atividades relatadas para as albuminas 2S
É ainda relatada para esse grupo de proteínas atividade anticancerígena. Dentre o grupo de albuminas, esta atividade foi relatada para a albumina 2S (não nomeada) de C.
maxima, que exibiu forte atividade anticancerígena contra células de câncer de mama
(MCF-7), teratocarcinoma de ovário (PA-1), câncer de próstata (PC-3 e DU-145) e linhagens celulares de carcinoma hepatocelular (HepG2) (TOMAR et al., 2014).
Algumas albuminas 2S são potencialmente capazes de criar orifícios permitindo a permeabilidade em membranas celulares, sendo este efeito já descrito para as albuminas Pf1 e Pf2 expressas em semente de Passiflora edulis f. flavicarpa (AGIZZIO et al., 2006), bem como para a albumina 2S (Mo-CBP2) purificada de sementes de M. oleifera (NETO et al.,
2017). Ademais, foi demonstrado que a Mo-CBP2 é capaz de aumento substancialmente a
quantidade de espécies reativas de oxigênio, bem como apresenta potencial, quando incubadas com células de leveduras (NETO et al., 2017).
Outra atividade comprovada para esse grupo de proteínas e sua competência em emulsificar, sendo essa atividade demonstrada pela proteína SFA-8 isolada de H. annuus (BURNETT et al., 2002; PANTOJA-UCEDA et al., 2004).
Tem sido também, evidenciado que algumas albuminas 2S são capazes de inibir proteases serínicas e subtilisin (protease não específica), sendo observada essa atividade em albuminas isoladas de sementes de Brassica juncea e B. nigra (GENOV et al., 1997; MANDAL et al., 2002). Recentemente, a Rc-2S-Alb purificada de semente de R. communis apresentou potencial inibitório contra tripsina (SOUZA et al., 2016). E a albumina Cb2SA, uma proteína heterodimérica (5 kDa e 9 kDa) isolada de Caryocar brasiliense, apresentou atividade inibitória de tripsina in vivo na espécie Spodoptera frugiperda (COSTA et al., 2015).
Ademais, uma das cadeias presente na albumina PA1b, albumina 2S isolada de
Pisum sativum, apresentou atividade in vivo em ensaios contra Culex pipiens e Aedes aegyptii
(GRESSENT et al., 2011). Em estudos posteriores utilizando as espécies de gorgulos do arroz
Sitophilus oryzae, S. oryzae e Tribolium castaneum foi demonstrado que ocorre uma interação
desses insetos (EYRAUD et al., 2017). De igual modo, atividade apoptótica tem sido relatada para a albumina 2S (não nomeada) de C.máxima (TOMAR et al., 2014), é possível que outras albuminas vegetais possam compartilhar essa função biológica.
Mo-CBP3, uma albumina 2S de Moringa oleifera
Apesar dos avanços na caracterização e do modo de ação de Mo-CBP3 esta
proteína não é compreendida completamente. Mo-CBP3 parece ter influência sobre as bombas
de prótons (H+-ATPase) presentes na membrana celular, além de causar deformações na
parede de células de fungos fitopatogênicos (BATISTA et al., 2014), tais como: F. solani, F.
oxysporum, Colletotrichum musae e C. gloesporioides (GIFONI et al., 2012). Espectrometria
de massas e PAGE-SDS mostram que Mo-CBP3 é formada por duas cadeias polipeptídicas,
em torno de 4,1 kDa e 8,1 kDa (BATISTA et al., 2014; FREIRE et al., 2015). Apesar de recentes estudos estarem buscando respostas sobre o mecanismo de ação desta proteína, são necessários maiores esforços a fim de descrever todas as isoformas existentes de Mo-CBP3,
3 HIPÓTESE
Mo-CBP3 constitui um conjunto de isoformas de origem multigênica presente na
4 OBJETIVOS
Objetivo geral
Entender os padrões de modificações pós-traducionais (PTMs) possíveis em isoformas de Mo-CBP3, empregando técnicas de biologia molecular e proteômicas (ESI-MS).
Objetivos específicos
- Isolar e purificar gDNA a partir de folhas jovens de Moringa oleifera; - Amplificar a região codificadora de isoformas de Mo-CBP3;
- Clonar e sequenciar a região codificadora de isoformas Mo-CBP3;
- Identificar todas as sequências que codificam isoformas de Mo-CBP3 no genoma de M.
oleifera;
- Comparar e analisar as sequências de nucleotídeos obtidas e as sequências aminoácidos deduzidas para as isoformas de Mo-CBP3 com sequências disponíveis em bancos de dados
públicos;
- Reduzir e alquilar amostras de Mo-CBP3 e submeter a análises por espectometria de massas
(ESI-MS);
- Identificar todas as massas obtidas as para ambas as cadeias de Mo-CBP3;
REFERÊNCIAS
ABIYU, A. et al. Wastewater treatment potential of Moringa stenopetala over Moringa olifera as a natural coagulant, antimicrobial agent and heavy metal removals. Cogent Environmental Science, v. 4, n. 1, p. 1–13, 2018.
AGIZZIO, A. P. et al. A 2S albumin-homologous protein from passion fruit seeds inhibits the fungal growth and acidification of the medium by Fusarium oxysporum. Archives of
Biochemistry and Biophysics, v. 416, p. 188–195, 2003.
AGIZZIO, A. P. et al. The antifungal properties of a 2S albumin-homologous protein from passion fruit seeds involve plasma membrane permeabilization and ultrastructural alterations in yeast cells. Plant Science, v. 171, p. 515–522, 2006.
AGRAWAL, H.; SHEE, C.; SHARMA, A. K. Isolation of a 66 kDa Protein with coagulation activity from seeds of Moringa oleifera. Research Journal of Agriculture and Biological Sciences, v. 3, n. 5, p. 418–421, 2007.
AHN, K. et al. Identification of two pistachio allergens, Pis v 1 and Pis v 2, belonging to the 2S albumin and 11S globulin family. Clinical and Experimental Allergy, v. 39, p. 926–934, 2009.
ALLEN, R. D. et al. Sequence and expression of a gene encoding an albumin storage protein in sunflower. Molecular & General Genetics, v. 210, n. 2, p. 211–218, 1987.
ASHRAF, M. et al. Microscopic evaluation of the antimicrobial activity of seed extracts of Moringa oleifera. Agriculture, v. 40, n. 4, p. 1349–1358, 2008.
BAPTISTA, A. T. A. et al. Protein fractionation of seeds of Moringa oleifera lam and its application in superficial water treatment. Separation and Purification Technology, v. 180, p. 114–124, 2017.
BATISTA, A. B. et al. New insights into the structure and mode of action of Mo-CBP3, an
antifungal chitin-binding protein of Moringa oleifera seeds. PLoS ONE, v. 9, n. 10, p. e111427, 2014.
BOUALEG, I.; BOUTEBBA, A. Purification of water soluble proteins (2S albumins)
extracted from peanut defatted flour and isolation of their isoforms by gel filtration and anion exchange chromatography. Scientific Study & Research, v. 18, n. 2, p. 135–143, 2017. BREITENEDER, H.; RADAUER, C. A classification of plant food allergens. Journal of Allergy and Clinical Immunology, v. 113, p. 821–830, 2004.
CÁCERES, A. et al. Pharmacological properties of Moringa oleifera. 1: Preliminary
screening for antimicrobial activity. Journal of Ethnopharmacology, v. 33, p. 213–6, 1991. CÂNDIDO, E. DE S. et al. Plant storage proteins with antimicrobial activity: novel insights into plant defense mechanisms. The FASEB Journal, v. 25, n. 1, p. 3290–3305, 2011. CHEN, M. Elucidation of bactericidal effects incurred by Moringa oleifera and chitosan. Journal of the U.S. SJWP, v. 4, n. 2009, p. 65–79, 2009.
DA SILVA, J. G. et al. Amino acid sequence of a new 2S albumin from Ricinus communis which is part of a 29-kDa precursor protein. Archives of Biochemistry and Biophysics, v. 336, n. 1, p. 10–18, 1996.
DUAN, X. H. et al. Some 2S albumin from peanut seeds exhibits inhibitory activity against Aspergillus flavus. Plant Physiology and Biochemistry, v. 66, p. 84–90, 2013.
EBISAWA, M. et al. Gly m 2S albumin is a major allergen with a high diagnostic value in soybean-allergic children. Journal of Allergy and Clinical Immunology, v. 132, n. 4, 2013. ELGAMILY, H. et al. Microbiological assessment of Moringa oleifera extracts and its
incorporation in novel dental remedies against some oral pathogens. Macedonian Journal of Medical Sciences, v. 4, n. 4, p. 585–590, 2016.
EYRAUD, V. et al. The interaction of the bioinsecticide PA1b (Pea Albumin 1 subunit b) with the insect V-ATPase triggers apoptosis. Scientific Reports, v. 7, n. 1, p. 1–10, 2017. FANG, E. F. et al. Biochemical characterization of the RNA-hydrolytic activity of a pumpkin 2S albumin. FEBS Letters, v. 584, n. 18, p. 4089–4096, 2010.
FERREIRA, P. M. P. et al. Larvicidal activity of the water extract of Moringa oleifera seeds against Aedes aegypti and its toxicity upon laboratory animals. Anais da Academia
Brasileira de Ciências, v. 81, p. 207–16, 2009.
FREIRE, J. E. C. et al. Mo-CBP3, an antifungal chitin-binding protein from Moringa oleifera
seeds , is a member of the 2S albumin family. PLoS ONE, v. 10, n. 3, p. 1–24, 2015. GALLÃO, M. I.; DAMASCENO, L. F.; BRITO, E. S. Avaliação química e estrutural da semente de moringa. Revista Ciência Agronômica, v. 37, n. 1, p. 106–9, 2006.
GANGULY, R.; GUHA, D. Alteration of brain monoamines & EEG wave pattern in rat model of Alzheimer’s disease & protection by Moringa oleifera. Indian Journal of Medical Research, v. 128, n. p. 744–751, 2008.
GARINO, C. et al. Isolation, cloning, and characterization of the 2S albumin: A new allergen from hazelnut. Molecular Nutrition and Food Research, v. 54, n. 9, p. 1257–1265, 2010. GASSENSCHMIDT, U. et al. Isolation and characterization of a flocculating protein from Moringa oleifera Lam. Biochimica et Biophysica Acta, v. 1243, n. 3, p. 477–81, abr. 1995. GENOV, N. et al. A novel thermostable inhibitor of trypsin and subtilisin from the seeds of Brassica nigra: Amino acid sequence, inhibitory and spectroscopic properties and
thermostability. Biochimica et Biophysica Acta - Protein Structure and Molecular Enzymology, v. 1341, n. 2, p. 157–164, 1997.
GHEBREMICHAEL, K. A. et al. A simple purification and activity assay of the coagulant protein from Moringa oleifera seed. Water Research, v. 39, p. 2338–2344, 2005.
GIFONI, J. M. et al. A novel chitin-binding protein from Moringa oleifera seed with potential for plant disease control. Biopolymers, v. 98, n. 4, p. 406–415, 2012.
GRESSENT, F. et al. Pea albumin 1 subunit b (PA1b), a promising bioinsecticide of plant origin. Toxins, v. 3, n. 12, p. 1502–1517, 2011.
GUPTA, P.; GAUR, V.; SALUNKE, D. M. Purification, identification and preliminary crystallographic studies of a 2S albumin seed protein from Lens culinaris. Acta
Crystallographica Section F: Structural Biology and Crystallization Communications, v. 64, p. 733–736, 2008.
HEATH, J. D. et al. Analysis of storage proteins in normal and aborted seeds from embryo-lethal mutants of Arabidopsis thaliana. Planta, v. 169, n. 3, p. 304–312, 1986.
IRWIN, S. D. et al. The Ricinus communis 2S albumin precursor: A single preproprotein may be processed into two different heterodimeric storage proteins. Molecular and General Genetics, v. 222, n. 2–3, p. 400–408, 1990.
KHAN, S. et al. Purification and characterization of 2S albumin from Nelumbo nucifera. Bioscience, Biotechnology and Biochemistry, v. 80, n. 11, p. 2109–2114, 2016. KOU, X. et al. Nutraceutical or Pharmacological Potential of Moringa oleifera Lam. Nutrients, v. 10, n. 3, p. 343, 2018.
KREBBERS, E. et al. Determination of the processing sites of an Arabidopsis 2S albumin and characterization of the complete gene family. Plant physiology, v. 87, p. 859–866, 1988. LEONE, A. et al. Moringa oleifera seeds and oil: Characteristics and uses for human health. International Journal of Molecular Sciences, v. 17, n. 12, p. 1–14, 2016.
LEHMANN, K. et al. Structure and stability of 2S albumin-type peanut allergens:
implications for the severity of peanut allergic reactions. Biochemical Journal, v. 395, p. 463-472, 2006.
LI, D.-F. et al. Crystal structure of Mabinlin II: A novel structural type of sweet proteins and the main structural basis for its sweetness. Journal of Structural Biology, v. 162, p. 50-62, 2008.
LI, L. et al. MAIGO2 is involved in exit of seed storage proteins from the endoplasmic reticulum in Arabidopsis thaliana. The Plant cell, v. 18, n. 12, p. 3535–3547, 2006. LIPIPUN, V. et al. Efficacy of Thai medicinal plant extracts against Herpes simplex virus type 1 infection in vitro and in vivo. Antiviral Research, v. 60, p. 175–180, 2003. MAKKAR, H. P. S.; BECKER, K. Nutrients and antiquality factors in different
morphological parts of the Moringa oleifera tree. The Journal of Agricultural Science, v. 128, p. 311–322, 1997.
MANDAL, S. et al. Precursor of the inactive 2S seed storage protein from the Indian mustard Brassica juncea is a novel trypsin inhibitor. Characterization, post-translational processing studies, and transgenic expression to develop insect-resistant plants. Journal of Biological Chemistry, v. 277, n. 40, p. 37161–37168, 2002.
MARTINOIA, E.; MAESHIMA, M.; NEUHAUS, H. E. Vacuolar transporters and their essential role in plant metabolism. Journal of Experimental Botany, v. 58, n. 1, p. 83–102, 2007.
MENÉNDEZ-ARIAS, L. et al. Primary structure of the major allergen of yellow mustard (Sinapis alba L.) seed, Sin a I. European journal of Bochemistry/FEBS, v. 177, n. 1, p. 159–166, 1988.
MORENO, F. J. et al. Mass spectrometry and structural characterization of 2S albumin isoforms from Brazil nuts (Bertholletia excelsa). Biochimica et Biophysica Acta - Proteins and Proteomics, v. 1698, p. 175–186, 2004.
MORENO, F. J. et al. Thermostability and in vitro digestibility of a purified major allergen 2S albumin (Ses i 1) from white sesame seeds (Sesamum indicum L.). Biochimica et Biophysica Acta - Proteins and Proteomics, v. 1752, p. 142–153, 2005.
MORENO, F. J.; CLEMENTE, A. 2S albumin storage proteins: What makes them food allergens? The Open Biochemistry Journal, v.2, p. 16-28, 2008.
MYLNE, J. S.; HARA-NISHIMURA, I.; ROSENGREN, K. J. Seed storage albumins: biosynthesis, trafficking and structures. Functional Plant Biology, v. 41, p. 671–7, 2014. NAWROT, R. et al. Plant antimicrobial peptides. Host Defense Peptides and Their Potential as Therapeutic Agents, v. 59, p. 181–196, 2014.
NDABIGENGESERE, A.; SUBBA NARASIAH, K.; TALBOT, B. G. Active agents and mechanism of coagulation of turbid waters using Moringa oleifera. Water Research, v. 29, p. 703–710, 1995.
NETO, J. X. S. et al. A chitin-binding protein purified from Moringa oleifera seeds presents anticandidal activity by increasing cell membrane permeability and reactive oxygen species production. Frontiers in Microbiology, v. 8, p. 1–12, 2017.
NIRASAWA, S. et al. Cloning and sequencing of a cDNA encoding a heat-stable sweet protein, mabinlin II. Gene, v. 181, p. 225–7, 1993.
NWOSU, M.; OKAFOR, J. I. Preliminary studies of the antifungal activities of some medicial plants against Basidiobolus and some other pathogenic fungi. Mycoses, v. 38, p. 191–5, 1995. ODINTSOVA, T. I. et al. Antifungal activity of storage 2S albumins from seeds of the
invasive weed dandelion Taraxacum officinale Wigg. Protein and Peptide Letters, v. 17, n. 4, p. 522–529, 2010.
OKUDA, T. et al. Isolation and characterization of coagulant extracted from Moringa oleifera seed by salt solution. Water Research, v. 35, n. 2, p. 405–410, 2001.
ONYEKE, C. C.; AKUESHI, C. O. Infectivity and reproduction of Meloidogyne incognita (Kofoid and White) Chitwood on African yam bean, Sphenostylis stenocarpa (Hochst Ex. A. Rich) Harms accessions as influenced by botanical soil amendments. African Journal of Biotechnology, v. 11, n. 67, p. 13095–103, ago. 2012.
PANTOJA-UCEDA, D. et al. Solution structure and stability against digestion of rproBnib, a recombinant 2S albumin from rapeseed: relationship to its allergenic properties.
Biochemistry, v. 43, p. 16036-16045, 2004a.
PANTOJA-UCEDA, D. et al. Solution structure of a methionine-rich 2S albumin from sunflower seeds: Relationship to its allergenic and emulsifying properties. Biochemistry, v. 43, p. 6976-6986, 2004b.
PANTOJA-UCEDA, D. et al. Solution structure of RicC3, a 2S albumin storage protein from Ricinus communis. Biochemistry, v. 42, p. 13839-13847, 2003.
PEREIRA, M. L. et al. Purification of a chitin-binding protein from Moringa oleifera seeds with potential to relieve pain and inflammation. Protein and Peptide Letters, v. 18, p. 1078– 1085, 2011.
PFEIFER, S. et al. Cor a 14, the allergenic 2S albumin from hazelnut, is highly thermostable and resistant to gastrointestinal digestion. Molecular Nutrition & Food Research, v. 59, n. 10, p. 2077–2086, 2015.
RAMACHANDRAN, C.; PETER, K. V.; GOPALAKRISHNAN, P. K. Drumstick (Moringa oleifera): A Multipurpose Indian Vegetable. Economic Botany, v. 34, n. 3, p. 276–283, 1980. RANI, N. Z. A.; HUSAIN, K.; KUMOLOSASI, E. Moringa Genus: A review of
phytochemistry and pharmacology. Frontiers in Pharmacology, v. 9, n. p. 1–26, 2018. REGENTE, M.; DE LA CANAL, L. Do sunflower 2S albumins play a role in resistance to fungi? Plant Physiology and Biochemistry, v. 39, p. 407–413, 2001.
RIBEIRO, S. F. F. et al. Antifungal and other biological activities of two 2S albumin-homologous proteins against pathogenic fungi. Protein Journal, v. 31, p. 59–67, 2011a. RIBEIRO, S. M. et al. Identification of a Passiflora alata Curtis dimeric peptide showing identity with 2S albumins. Peptides, v. 32, n. 5, p. 868–874, 2011b.
RICO, M. et al. H NMR assignment and global fold of napin BnIb, a representative 2S albumin seed protein. Biochemistry, v. 35, p. 15672-15682, 1996.
ROBOTHAM, J. M. et al. Ana o 3, an important cashew nut (Anacardium occidentale L.) allergen of the 2S albumin family. Journal of Allergy and Clinical Immunology, v. 115, p. 1284–1290, 2005.
ROCHA, M. F. G. et al. Extratos de Moringa oleifera e Vernonia sp. sobre Candidaalbicans e Microsporum canis isolados de cães e gatos e análise da toxicidade em Artemia sp. Ciência Rural, v. 41, p. 1807–1812, 2011.
ROLIM, L. A. D. M. M. et al. Genotoxicity evaluation of Moringa oleifera seed extract and lectin. Journal of Food Science, v. 76, p. T53-T58, 2011.
RUNDQVIST, L. et al. Solution structure, copper binding and backbone dynamics of
SAHAY, S.; YADAV, U.; SRINIVASAMURTHY, S. Potential of Moringa oleifera as a functional food ingredient: A review. International Journal of Food Science and Nutrition, v. 2, n. 5, p. 31–37, 2017.
SANTOS, A. F. S. et al. Isolation of a seed coagulant Moringa oleifera lectin. Process Biochemistry, v. 44, p. 504–508, 2009.
SAPANA, M. M.; SONAL, G. C.; RAUT, P. Use of Moringa oleifera (Drumstick) seed as natural absorbent and an antimicrobial agent for ground water treatment. Research Journal of Recent Sciences, v. 1, n. 3, p. 31–40, 2012.
SHARIEF, F. S.; LI, S. S. Amino acid sequence of small and large subunits of seed storage protein from Ricinus communis. The Journal of biological chemistry, v. 257, n. 24, p. 14753–14759, 1982.
SHARMA, A. et al. Purification and characterization of 2S albumin from seeds of Wrightia tinctoria exhibiting antibacterial and DNase activity. Protein and Peptide Letters, v. 24, n. 4, p. 368–378, 2017.
SHARMA, G. M. et al. Cloning and characterization of 2s albumin, Car i 1, a major allergen in pecan. Journal of Agricultural and Food Chemistry, v. 59, p. 4130–4139, 2011. SHEWRY, P. R.; NAPIER, J. A; TATHAM, A S. Seed storage proteins: structures and biosynthesis. The Plant cell, v. 7, n. July, p. 945–956, 1995.
SHIH, M. C. et al. Effect of different parts (leaf, stem and stalk) and seasons (summer and winter) on the chemical compositions and antioxidant activity of Moringa oleifera.
International Journal of Molecular Sciences, v. 12, p. 6077–6088, 2011.
SOUZA, P. F. N. et al. A 2S albumin from the seed cake of Ricinus communis inhibits trypsin and has strong antibacterial activity against human pathogenic bacteria. Journal of Natural Products, v. 79, p. 2423–31, 2016.
TAI, S. S. K. et al. Molecular cloning of 11S globulin and 2S albumin, the two major seed storage proteins in Sesame. Journal of Agricultural and Food Chemistry, v. 47, p. 4932–8, 1999.
TAI, S. S. K. et al. Expression pattern and deposition of three storage proteins, 11S globulin, 2S albumin and 7S globulin in maturing sesame seeds. Plant Physiology and Biochemistry, v. 39, n. 11, p. 981–992, 2001.
TAN-WILSON, A. L.; WILSON, K. A. Mobilization of seed protein reserves. Physiologia Plantarum, v. 145, n. 1, p. 140–153, 2012.
TERRAS, F. R. et al. A new family of basic cysteine-rich plant antifungal proteins from Brassicaceae species. FEBS letters, v. 316, n. 3, p. 233–240, 1993.
TERRAS, F. R. G. et al. Analysis of two novel classes of plant antifungal proteins from radish (Raphanus sativus L.) seeds. Journal of Biological Chemistry, v. 267, p. 15301– 15309, 1992.
TOMAR, P. P. S. et al. Purification, characterization and cloning of a 2S albumin with DNase, RNase and antifungal activities from Putranjiva Roxburghii. Applied Biochemistry and Biotechnology, v. July, p. 1–12, 2014b.
ULLAH, A. et al. Crystal structure of mature 2S albumin from Moringa oleifera seeds. Biochemical and Biophysical Research Communications, v. 468, p. 365-371, 2015. VAN DER KLEI, H. et al. A fifth 2S albumin isoform is present in Arabidopsis thaliana. Plant Physiology, v. 101, n. 4, p. 1415–1416, 1993.
VICENTE, T. et al. Tratabilidade de água superficial utilizando coagulantes naturais à base de tanino e extratos de sementes de Moringaoleifera. Ensaios e Ciência: C. Biológicas,
Agrárias e da Saúde, v. 1, n. 3, p. 152–155, 2017.
VIEIRA, H.; CHAVES, L. H. G.; VIÉGAS, R. A. Acumulação de nutrientes em mudas de moringa (Moringa oleifera Lam) sob omissão de macronutrients. Revista Ciência
Agronômica, v. 39, n. 1, p. 130–136, 2008.
VIERA, G. H. F. et al. Antibacterial effect (in vitro) of Moringa oleifera and Annona muricata against Gram positive and Gram negative bacteria. Revista do Instituto de Medicina Tropical de São Paulo, v. 52, n. 3, p. 129–132, 2010.
VITALE, A; GALILI, G. The endomembrane system and the problem of protein sorting. Plant Physiology, v. 125, n. 1, p. 115–118, 2001.
WANG, X. et al. Purification and characterization of three antifungal proteins from
cheeseweed (Malva parviflora). Biochemical and Biophysical Research Communications, v. 282, n. 5, p. 1224–1228, 2001.
WANG, X.; BUNKERS, G. J. Potent heterologous antifungal proteins from cheeseweed (Malva parviflora). Biochemical and Biophysical Research Communications, v. 279, n. 2, p. 669–673, 2000.
WILSON, K. A. et al. Role of vacuolar membrane proton pumps in the acidification of
protein storage vacuoles following germination. Plant Physiology and Biochemistry, v. 104, p. 242–249, 2016.
YOULE, R. J.; HUANG, A. H. C. Occurrence of low molecular weight and high cysteine containing albumin storage proteins in oilseeds of diverse species. AmericanJournal of Botany, v. 68, n. 1, p. 44, 1981.
5 POST-TRANSLATIONAL MODIFICATIONS REVEAL THAT
MO-CBP3, A 2S MORINGA OLEIFERA CHITIN-BINDING ALBUMIN, IS A
COMPLEX MIXTURE OF ISOFORMS
Abstract
Mo-CBP3 is a mixture of proteins (Mo-CBP3-1, Mo-CBP3-2, Mo-CBP3-3 and Mo-CBP3-4)
from Moringa oleifera seeds, as described previously, and it is classified as member of 2S albumin seed storage proteins. The four isoforms share the same molecular weight, but slightly diverge in terms of primary sequence. X-ray diffraction studies revealed that Mo -CBP3-4 is a heterodimeric protein consisting of a small and a large polypeptide chain
stabilized by disulfide bonds. This work aimed to investigate the occurrence of other Mo -CBP3 isoforms and characterize these proteins regarding to physicochemical properties and
post-translational modifications (PTM). DNA isolation, molecular cloning and DNA sequencing along with the analysis of deduced amino acid sequences demonstrated the existence of four undescribed Mo-CBP3 isoforms, which are most closely related to Mo-CBP3
-2 and Mo-CBP3-3. In addition, the native Mo-CBP3 protein complex extracted from M.
oleifera seeds was purified and further separeted and characterized on a ultra performance
liquid chromatography system coupled to an electrospray ionization tandem mass spectrometer (MS). Both the intact and the reduced and alkylated proteins were used. These analysis allowed to separate and identified peaks corresponding to the four known and the four new Mo-CBP3 isoforms. Furthermore, MS/MS experiments revealed the existence of an
impressive number of clusters of peptides (89 and 57, small and large chain respectively) presenting fixed and variable PTM, which may account for an extensive diversity of Mo -CBP3 isoforms. The biological meaning and implications for this findings are discussed.
Keywords: Isoform. Post-translational modification. Processing. 2S albumins. PTMs.
Introduction
Mo-CBP3 is a complex mixture of seed storage proteins from the tree Moringa
oleifera belonging to the 2S albumin group [1], a class of water-soluble proteins that