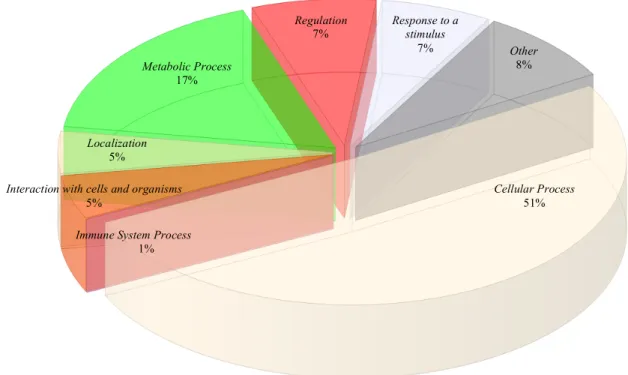
OralOme : o contributo dos microrganismos revelado por estudos de proteómica
Texto
Imagem
Documentos relacionados
Note on the occurrence of the crebeater seal, Lobodon carcinophagus (Hombron & Jacquinot, 1842) (Mammalia: Pinnipedia), in Rio de Janeiro State, Brazil.. On May 12, 2003,
occurring in more than just enterprise and technology companies (2002: US – 83% / Europe – 47%).. • In the UK, France and Germany
- Se o estagiário, ou alguém com contacto direto, tiver sintomas sugestivos de infeção respiratória (febre, tosse, expetoração e/ou falta de ar) NÃO DEVE frequentar
O software PcModel pode ser uma ferramenta bastante útil, quando aplicado na previsão e estimativa do cálculo de energia envolvendo as conformações mais e menos estáveis do
Nessa situação temos claramente a relação de tecnovívio apresentado por Dubatti (2012) operando, visto que nessa experiência ambos os atores tra- çam um diálogo que não se dá
O score de Framingham que estima o risco absoluto de um indivíduo desenvolver em dez anos DAC primária, clinicamente manifesta, utiliza variáveis clínicas e laboratoriais
4.5 Conclusões: Este trabalho mostrou um modelo criado para representar uma linha de transmissão monofásica através de uma cascata de circuitos π, cujos parâmetros longitudinais
O objetivo principal deste estudo de caso era verificar o grau de valorização da medicina popular de plantas entre o próprio povo, tendo por base uma comunidade com diferentes