Pró-reitoria de Pós-Graduação e Pesquisa
–
PRPGP
Programa de Pós-Graduação em Ciências
Genômicas e Biotecnologia
MicroRNAs ASSOCIADOS A
COPY NUMBER
VARIATION
EM CÂNCER COLORRETAL
Autora: Nina Jardim Gasparini
Orientadora: Dra. Rosângela Vieira de Andrade
NINA JARDIM GASPARINI
MicroRNAs ASSOCIADOS A COPY NUMBER VARIATION EM CÂNCER COLORRETAL
Dissertação apresentada ao Curso de Mestrado em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial à obtenção do título de Mestre em Ciências Genômicas e Biotecnologia.
Orientadora: Dra. Rosângela Vieira de Andrade
Co-orientadores: Dr. Robert Pogue Ms. Túlio Lins
Ficha elaborada pela Biblioteca Pós-Graduação da UCB 12/08/2013
G249m Gasparini, Nina Jardim
MicroRNAS associados a copy number variation em câncer colorretal / Nina Jardim Gasparini – 2013.
114f. : il ; 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília, 2013. Orientação: Profa. Dra. Rosângela Vieira de Andrade.
Coorientação: Prof. Dr. Robert Pogue Coorientação: Prof. Ms. Túlio Lins
1. Câncer colorretal. 2. Cólon (Anatomia) Doenças. 3. Biotecnologia. I. Andrade, Rosângela Vieira de, orient. II. Pogue, Robert, coorient. III. Lins, Túlio, coorient. IV.Título.
Dissertação de autoria de Nina Jardim Gasparini, intitulada “MiRNAs associados a
Copy Number Variation em câncer colorretal.”, apresentada como requisito parcial para obtenção de grau de Mestre em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, em 28 de junho de 2013, defendida e aprovada pela banca examinadora abaixo assinada:
____________________________________________________________________ Profa. Dra. Rosângela Vieira de Andrade
Orientadora
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia- UCB)
____________________________________________________________________ Prof. Dr. Robert Edward Pogue
Coorientador
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia- UCB)
_____________________________________________________________________ Profa. Dra. Fábia Aparecida Carvalho Lassance
Membro Interno
(Universidade Católica de Brasília - UCB)
_____________________________________________________________________ Profa. Dra. Gisele Pereira de Carvalho
Membro Externo
(Pontifícia Universidade Católica do Rio Grande do Sul)
_____________________________________________________________________ _Profa. Dra. Juliana Forte Mazzeu de Araújo
Membro Externo
(Universidade de Brasília - UnB)
AGRADECIMENTO
Primeiramente, gostaria de agradecer à minha orientadora, Rosângela Vieira de Andrade, por ter me dado a oportunidade de iniciar meus estudos nessa instituição, e pelo carinho com que me acompanhou no desenvolvimento da minha pesquisa. Ao meu co-orientador Robert Pogue, por ter me amparado sempre que precisei. Ao meu co-co-orientador e amigo, Túlio Lins, por todas as horas que passamos conversando e resolvendo nossos problemas. Ao pessoal do Hospital São Lucas, da Pontifícia Universidade Católica do Rio Grande do Sul, em especial a Dra. Gisele Pereira e Bernardo Garicochea, por terem gentilmente cedido as amostras usadas nesse estudo, e por terem sido tão solícitos sempre que foi preciso. Ao Dr. Luis Sakamoto, por sua disponibilidade e paciência em me ajudar na estatística e todas as questões a serem abordadas. À colega de mestrado Silmara Andreoli, por ter compartilhado comigo a pesquisa. Aos meus alunos de iniciação científica e amigos, Marcos Dumont, Gabriela Lopes, e especialmente o aluno Jonatas Cunha, por ter colaborado em grande peso para o desenvolvimento do nosso trabalho. À minha colega Larissa Lemos, por ter colaborado muito com a pesquisa, e por ter sido uma grande amiga.
Ao longo de dois anos muitas pessoas passaram pela minha vida. Cada uma à sua maneira, elas me ajudaram a seguir com a pesquisa em frente. Gostaria de agradecer à minha família por ter me proporcionado o ambiente e as condições perfeitas para que eu me tornasse o que hoje sou. Meu amado pai, que não está mais entre nós, gostaria muito poder ver o seu rosto neste dia, e saber que tem orgulho de mim. Sei que sente isso, onde você estiver. Minha mãe e padrastro, Goretti e Marciano, sem o amor e a dedicação que têm por mim, eu não seria nada. Meu irmão, cunhada e sobrinho, Pedro, Karina e Arthur, vocês me proporcionaram muita alegria e amparo. Obrigada! Meus avós Mário (in memorian) e Zayda, que me proporcionaram o estudo necessário para chegar aqui. Sem o amor da minha família eu não chegaria tão longe.
afinco. Meus queridos amigos Jacqueline Tirotti, Rafael Burtet, Nézio Neri, Getúlio Júnior, Gustavo Eustáquio, Rafael Faria, Wander Martins e Pedro Renato. Gostaria também de lembrar a importância da dança, e dos amigos que fazem parte dela, que me ajudaram na manutenção do meu bem-estar mental e físico. Especialmente, gostaria de agradecer à minha eterna irmã Paula Sallas, por ter sempre me amparado nos momentos difíceis, e Laureane Pavanelli, por ter sido a parceira inseparável. Ao meu querido e amado namorado Luis Renato Leite de Sá, por ser um exemplo e por ter me incentivado a continuar em frente por toda a etapa de finalização do trabalho.
“Em todo ser vivo, aquilo que designamos
como partes constituintes forma um todo inseparável, que só pode ser estudado em conjunto, pois a parte não permite reconhecer o todo, nem o conjunto deve
ser reconhecido nas partes.”...
RESUMO
Referência: GASPARINI, Nina. Título: MicroRNAs associados a Copy Number Variation em câncer colorretal. 114 f. Mestrado em Ciências Genômicas e Biotecnologia. Universidade Católica de Brasília. Brasília.
CNV relacionada ao miR150. Por fim, uma correlação não-paramétrica para avaliar a ligação entre a variação de número de cópias e níveis de expressão de miRNAs foi feita. Apenas para um miRNA (miR16 na amostra tumoral) essa correlação foi significativa, o que indica que para essas amostras o CNV provavelmente não influencia a expressão gênica de miRNAs relacionados ao desenvolvimento de CCR. Esse estudo é essencial para a elucidação dos mecanismos que levam ao desenvolvimento do CCR, a fim de descobrir novos marcadores moleculares e mecanismos que ajudem na compreensão da tumorigênese, o que colabora para melhores diagnósticos e prognósticos.
ABSTRACT
which indicates that in these samples CNV probably does not influence gene dosage of miRNAs related to CRC development.
LISTA DE FIGURAS
Figura 1: Etapas-chave para mutações no desenvolvimento do CCR ... 23
Figura 2: Via de transdução do sinal Wnt ... 25
Figura 3: Via de transdução do sinal MAPK/Erk ... 27
Figura 4: Diferentes tipos de câncer colorretal. ... 31
Figura 5: Vias para a deficiência em reparo de mal-pareamentos em CCR ... 33
Figura 6: Mecanismos de maturação do miRNA. ... 35
Figura 7: Mudanças na expressão de miRNAs em adenomas e carcinomas colorretais correlacionados com estágio e sobrevivência. ... 41
Figura 8: Representação esquemática do transcrito primário de um miRNA ... 46
Figura 9: Amostras de RNA (A) e DNA (B) de indivíduos com CCR selecionados. .. 65
Figura 10: Média dos miRNAs diferencialmente expressos. ... 67
Figura 11: Expressão diferencial do miR-338... 68
Figura 12: Expressão do miRNA Let-7g ... 69
Figura 13: Expressão diferencial do miR-1... 69
Figura 14: Expressão diferencial no miR-150... 70
Figura 15: Expressão diferencial do miR-183... 71
Figura 16: Expressão diferencial do miR-650... 72
Figura 17: Expressão diferencial do miR-31... 72
LISTA DE QUADROS
LISTA DE TABELAS
Tabela 1: Características Clínicas e Histopatológicas dos indivíduos Selecionados 61
Tabela 2: Características dos Indivíduos ... 62
Tabela 3: Estadiamento ... 63
Tabela 4: Número e percentagem de individuos que apresentaram remissão e status clínico ... 64
Tabela 5: Significância da expressão diferencial dos miRNAs ... 66
Tabela 6: Sobrevida global x expressão diferencial de miRNA ... 74
Tabela 7: Frequência de CNVs em amostras normais, tumorais e linfonodais. ... 75
Tabela 8: Significância entre CNVs comparados com CNV normal ... 76
Tabela 9: Correlação entre CNVs e sobrevida global ... 78
LISTA DE SIGLAS
AJCC – American Joint Committee on Cancer APC – Adenomatous Poliposis Coli
CCR – Câncer colorretal
CCS – Síndrome Conkhite-canada CNV – Copy Number Variation CS – Síndrome de Cowden
DGV – Database of Genomic Variants EGRF – Epidermal Growth Factor Receptor ERK – Extracellular Receptor Kinase
FAP – Polipose Adenomatosa Familiar
GRB2 – Growth-factor-receptor Bound Protein-2 HMPS – Síndrome Poliosa Hereditária Mista HNPCC – Síndrome de Linch
JPS – Síndrome Poliposa Juvenil
LCV – Large-segment Copy-number Variations MAP – Polipose Associada a Mih
MAPK – Mitogen-activated Protein Kinase MLH1 – MutL Homolog 1
MMR – Mismatch Repair
MSI – Instabilidade de Microssatélite PJS – Síndrome de Peutz-Jeghers
RISC – RNA-induced Silencing Complexes ROS – Espécie Reativa de Oxigênio
SUMÁRIO
1. INTRODUÇÃO ... 19
1.1CÂNCER COLORRETAL ... 19
1.2EPIDEMIOLOGIA ... 21
1.3 MECANISMOS RELACIONADOS COM O DESENVOLVIMENTO DO CCR ... 22
1.3.2 A via canônica de transdução do sinal Wnt/β-CATENINA e a proteína APC 24 1.3.3 A proteína RAS na via de transdução de sinal MAPK/ERK ... 26
1.3.4 P53 ... 28
1.4 TIPOS DE CCR ... 29
1.4.1 CCR esporádico ... 29
1.4.2 CCR hereditário ... 30
1.5 MICRORNAS (MiRNAS) ... 34
1.5.1 MiRNAs relacionados com CCR ... 37
1.6 VARIAÇÃO DE NÚMERO DE CÓPIAS (CNV) ... 42
1.6.1 miRNA x CNV ... 45
2 OBJETIVOS ... 49
2.1 OBJETIVO GERAL ... 49
2.2 OBJETIVOS ESPECÍFICOS ... 49
3 DELINEAMENTO EXPERIMENTAL ... 50
4 MATERIAL E MÉTODOS ... 51
4.1 COLETA DAS AMOSTRAS ... 51
4.2 EXTRAÇÃO DO RNA E DNA ... 51
4.3 VERIFICAÇÃO DA QUALIDADE E QUANTIFICAÇÃO DAS AMOSTRAS ... 53
4.4 SELEÇÃO DOS MIRNAS E CNVS ... 53
4.5 SÍNTESE DE cDNA PARA MiRNAS ... 56
4.7 ANÁLISE DOS DADOS DE MIRNA ... 57
4.8 ANÁLISE DOS DADOS DE CNV ... 58
4.9 ANÁLISES ESTATÍSTICAS ... 58
5 RESULTADOS E DISCUSSÃO ... 60
5.1 ANÁLISE CLÍNICA ... 60
5.2 ANÁLISE MOLECULAR ... 64
5.2.1 Qualidade dos ácidos nucleicos ... 64
5.2.2 Análise da Expressão dos miRNAs ... 65
5.2.3 Análise de CNVs ... 74
5.2.4 Correlação de miRNA x CNVs ... 80
6 CONCLUSÃO ... 83
REFERÊNCIAS ... 85
APÊNDICE A ... 109
1.INTRODUÇÃO
1.1 CÂNCER COLORRETAL
O câncer colorretal (CCR) é um tipo de neoplasia maligna que afeta a região do cólon e do reto. Sua importância é global por ser o terceiro tipo de câncer mais comum na população mundial. Ele surge a partir de lesões pré-malignas, os polipos, que se desenvolvem ao longo da vida nos indivíduos. Essas lesões, após cerca de 10 a 15 anos, podem evoluir para adenocarcinomas, dependendo da propensão genética e de hábitos cotidianos que aumentam o seu risco de desenvolvimento (American Cancer Society, 2012) (SUEHIRO; HINODA, 2008).
Indivíduos sedentários, acima do peso, com dieta rica em carnes vermelhas e processadas, dieta pobre em vegetais e cálcio, baixos níveis séricos de vitamina D, fumantes e indivíduos que consomem moderadamente álcool, possuem maior risco para o desenvolvimento de um CCR. Alguns fatores de risco também importantes, porém não mutáveis, são idade, histórico de presença de pólipos no intestino, histórico familiar de CCR, presença de mutações hereditárias, etnia (afro-americanos e judeus da Europa Oriental) e presença de diabetes tipo 2. Entretanto, os fatores de risco relacionados a hábitos podem ser facilmente diminuídos pela promoção de campanhas informativas, gerando mudanças de postura no cotidiano das populações, e a remoção dos polipos nos indivíduos em idade de risco (American Cancer Society, 2013).
O estadiamento do câncer estima o seu nível de penetração, e permite que se escolham as melhores formas de tratamento para cada indivíduo. As estimativas feitas pelos sistemas de classificação se baseiam na extensão da invasão do tumor primário, no comprometimento dos linfonodos anexos e na presença de metástases distantes.
O sistema TNM, criado pela AJCC (American Committee on Cancer) e pela UICC (Internacional Union for Cancer Control) classifica clinicamente a doença por meio de três
categoria “N”, que avalia o grau de comprometimento de linfonodos regionais, e a
categoria “M”, que avalia a presença de metástase (quadro 1).
Quadro 1: Classificação TNM
Tumor Primário (T)
Tx Tumor primário não pode ser avaliado
T0 Sem evidência de tumor primário
Tis Carcinoma in situ
T1, T2, T3, T4 Tamanho e/ou extensão do tumor primário
Linfonodos Regionais (N)
NX Linfonodos regionais não podem ser
avaliados
N0 Sem comprometimento de linfonodos
N1, N2, N3 Envolvimento de linfonodos (número de linfonodos e/ou extensão do tumor) Metástase (M)
MX Metástases distantes não podem ser
avaliadas
M0 Sem metástase
M1 Presença de metástase
Fonte: American Cancer Society, 2012
agrupou essa classificação de forma que os estádios são classificados em categorias de I a IV: quanto mais alta a classificação, mais avançado é o estádio da doença e pior o prognóstico (quadro 2).
Quadro 2: Classificação AJCC
Estádio
AJCC Estádio TNM Critérios
0 Tis No Mo Tumor em estádio primário. Carcinoma in situ.
I T1-2 No Mo
Tumor invade submucosa (T1) ou muscularis propria (T2)
II-A T3 No Mo Tumor invade submucosa ou além
II-B T4a No Mo
Tumor invade a parede do cólon/reto mas não atinge tecidos anexos
II-C T4b No Mo Tumor cresce anexo a outros tecidos III-A T1-2 N1-2a Mo Acomete 1-3 linfonodos (N1) ou 4-6 (N2) III-B T1-4a N1-2b Mo Acomete de 1 a mais de 7 linfonodos (N2b)
III-C T3-4b N1-2b Mo
Acomete de 1 a mais linfonodos com tumor em estádios diferentes
IV-A Tqualquer Nqualquer M1a Metástase em um órgão distante
IV-B Tqualquer Nqualquer M1b Metástase em mais de um órgão distante
Fonte: American Cancer Society (2013)
1.2 EPIDEMIOLOGIA
O CCR é o terceiro tipo de câncer mais comum em homens e o segundo tipo mais comum em mulheres (FERLAY et al., 2010; MACEDO; LIMA; SASSE, 2012). Todo ano, um milhão de pessoas são diagnosticadas com CCR, representando 9,7% de todos os casos registrados de câncer, sendo que aproximadamente metade dos indivíduos diagnosticados morrem dessa doença (PEACOCK et al., 2012).
Unidos, é estimado que existam mais de 50 mil mortes em 2013 (American Cancer Society, 2013), e no Brasil, segundo dados do Instituto Nacional do Câncer, a estimativa de incidência é de aproximadamente 30 mil casos por ano (14.180 homens e 15.960 mulheres) (INCA, 2012).
A taxa de incidência e mortalidade do CCR pode ser facilmente diminuída por meio de exames de rastreamento, como o de sangue oculto em fezes e exames diagnósticos (colonoscopia e sigmoidoscopia). Uma varredura para CCR começa primeiramente pela pesquisa de sangue oculto nas fezes de indivíduos assintomáticos em idade de risco (50 a 74 anos), que deve ser feito periodicamente. Se positivo para sangue oculto, o paciente é encaminhado para realizar outros exames mais eficazes, como a colonoscopia e sigmoidoscopia (BUECHER; DE PAUW, 2012).
A prática do exame de sigmoidoscopia feito apenas uma vez em pacientes entre 55 e 64 anos reduz a taxa de mortalidade em 40% e a taxa de incidência em 33% (ATKIN et al., 2010), e a prática de exames de endoscopia do intestino grosso demonstra uma redução na mortalidade por CCR de 60-76% e uma redução na incidência de 76-90% (COURTNEY et al., 2012).
Quando o tumor é detectado em um estádio inicial, pode apresentar taxas de sobrevida de até 90% (CRUK, 2009). Assim, a prática de exames preventivos na população de risco e o acompanhamento multidisciplinar evita que muitos casos cheguem a estádios mais avançados (COURTNEY et al., 2012; LIPTON; TOMLINSON, 2006; PEACOCK et al., 2012).
Recentemente, estudos da American Cancer Society relataram que a taxa de mortalidade em CCR vem diminuindo nos últimos 20 anos devido ao avanço de tratamentos e maior uso dos exames de rastreamento (American Cancer Society, 2013).
1.3 MECANISMOS RELACIONADOS COM O DESENVOLVIMENTO DO CCR
evolução acompanha uma série de etapas estabelecidas: algumas mutações são consideradas clássicas no seu desenvolvimento, e se iniciam em consequência de instabilidades genômicas. Os principais genes envolvidos são: APC (Adenomatous Poliposis Coli), K-RAS e p53 (MALHOTRA et al., 2013; PARSONS; MYERS, 2013), como pode ser visto na figura 1, pela qual é demonstrado de forma simplificada o desenvolvimento de um carcinoma colorretal e os momentos em que os genes-chave se tornam mutados.
Figura 1: Etapas-chave para mutações no desenvolvimento do CCR
Fonte: SOBHANI et al. (2013)
1.3.2 A via canônica de transdução do sinal Wnt/β-CATENINA e a proteína APC
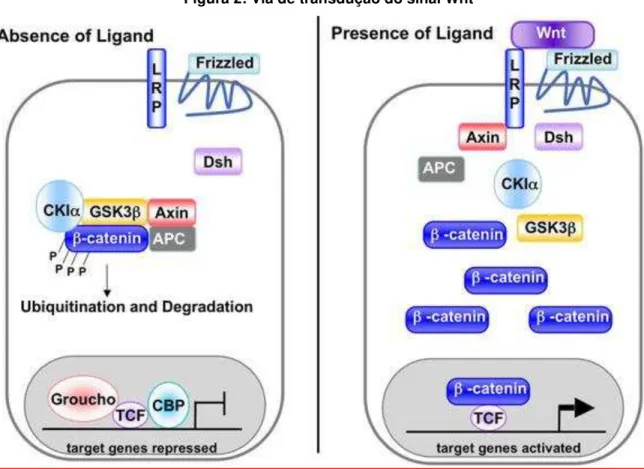
A via Wnt (Wingless-Int) é a principal via de regulação da proliferação e renovação do epitélio no lúmen colorretal, na qual sua principal proteína é a β-CATENINA (RAMAN et al., 2012). A β-CATENINA faz parte de um complexo de proteínas com função de aderência celular. Essas proteínas são essenciais para a manutenção das junções de adesão em tecidos epiteliais, ou seja, mantêm em equilíbrio o crescimento e adesão entre essas células. Quando a β-CATENINA não se encontra associada às proteínas do complexo de adesão, ela permanece livre no citoplasma, onde interage com outras proteínas e realiza funções distintas, como por exemplo, a transcrição de genes (EISENMANN, 2005; REINTSCH et al., 2005).
Um complexo de proteínas, na ausência da ativação da via Wnt, é responsável pela degradação da β-CATENINA (figura 2). Esse complexo é composto pelas proteínas AXIN, GSK3, CK1 e APC, e ele desestabiliza a molécula de β-CATENINA pela fosforilação, levando à sua ubiquitinação e degradação por proteassomas (SCHOLER-DAHIREL et al., 2011; YANG, Y., 2012). Em 80% dos casos de CCR esporádico, a proteína APC é encontrada mutada. Quando essa proteína está mutada, o complexo de proteínas não se mantém coeso (MARKOWITZ; BERTAGNOLLI, 2009).
Figura 2: Via de transdução do sinal Wnt
Fonte: EISENMANN (2005)
1.3.3 A proteína RAS na via de transdução de sinal MAPK/ERK
As Proteínas ERK (Extracellular Receptor Kinase), também conhecidas como MAPK (Mitogen-Activated Protein Kinase), fazem parte de uma família de proteínas que são ativadas por meio da fosforilação dos seus resíduos de tirosina em resposta a fatores de crescimento. A via de transdução do sinal MAPK/ERK tem importante papel na mediação de propagação de sinais oncogênicos e inflamatórios, como controle de mitose, produção de enzimas que degradam matriz celular, angiogênese e degeneração óssea (CHANDHANAYINGYONG et al., 2012).
Figura 3: Via de transdução do sinal MAPK/Erk
Fonte: KIM; BAR-SAGI (2004)
A proteína RAS faz parte da superfamília de GTPases monoméricas, e se localiza na periferia da célula, ligada à face interna da membrana celular por possuir domínios lipídicos que se ligam a ela (XU, X. M. et al., 2011).
que por sua vez fosforila a proteína ERK (KIM; BAR-SAGI, 2004). A proteína fosforilada MAPK (ou ERK) passa então para o núcleo celular onde fosforila e ativa fatores de transcrição que controlam a expressão de genes relacionados a crescimento celular, diferenciação e apoptose (KIM; BAR-SAGI, 2004).
Mutações nas proteínas da via MAPK/ERK estão relacionadas com a proliferação celular descontrolada relativa à progressão dos tumores. Cerca de 30% dos tumores humanos possuem formas mutantes hiperativas de RAS, que contribuem para a proliferação descontrolada das células cancerosas (mais comuns em fenótipos que apresentam instabilidade cromossomal, em pólipos hiperplásicos, adenomas serreados e localizados no cólon proximal). Quando uma proteína RAS é mutada, ela se mantém no estado ativo de forma permanente, não permitindo que outras proteínas GTPases a inativem. Essa situação ocorre em 37% dos casos de CCR, e é importante se estabelecer quando um indivíduo possui essa mutação porque pacientes com mutações em K-RAS não respondem a tratamento de anticorpos monoclonais anti-EGRF (Epidermal Growth Fator Receptor), sendo necessário que se façam tratamentos alternativos que permitam a melhora no prognóstico da doença (OUERHANI et al., 2013; PARSONS; MYERS, 2013).
1.3.4 p53
A inativação da p53 pode acontecer por meio de mutações pontuais, pela deleção do cromossomo 17p, ou por mutações em genes que atuam na biogênese da proteína. Tal falha na funcionalidade da proteína está relacionada com a transição de grandes adenomas para carcinomas invasivos, presente em 51% a 74% dos casos de CCR. (FABER; KIRCHNER; HLUBEK, 2009; MARKOWITZ; BERTAGNOLLI, 2009).
A presença de mutações nos genes de todas as proteínas citadas anteriormente de forma simultânea é muito rara. Especialmente é muito raro a mutação das proteínas K-RAS e APC ao mesmo tempo e no mesmo tumor. Isso reforça que essas mutações acontecem por vias de forma alternada no desenvolvimento do CCR (XU, X. M. et al., 2011).
Desta forma, o CCR evolui a partir de vários mecanismos, que acontecem em momentos diferentes do desenvolvimento do tumor e se acumulam em diferentes proporções, dependendo do tipo de tumor.
1.4 TIPOS DE CCR
1.4.1 CCR esporádico
O CCR esporádico é o tipo mais comum, representando cerca de 80% de todos os CCR. Não acontecem a partir de genes vindos de linhagem germinativa, mas a partir de células somáticas, que sofrem instabilidades genômicas, como por exemplo, a metilação de regiões promotoras de genes de reparo por pareamento errado (MMR). O Risco para desenvolvimento do CCR na população em geral é de 5% a 6% (RUSTGI, 2007; SOBHANI et al., 2013).
RAF-MAP kinase) são por sua vez responsáveis por 10% dos casos. O estudo das síndromes hereditárias em CCR tem grande importância no entendimento da patogênese molecular dos CCR esporádicos, uma vez que as vias pelas quais o câncer se desenvolve são as mesmas (ARMAGHANY et al., 2012).
Pelo fato de o CCR esporádico se iniciar a partir de mutações não-herdadas, fica claro a influência das mudanças epigenéticas que o meio externo promove sobre os genes dos indivíduos. Vários estudos verificaram o crescente aumento de casos de CCR em populações e etnias orientais a medida que estas absorvem cada vez mais hábitos alimentares e cotidianos do ocidente (RAMAN et al., 2012; SINHA et al., 2013).
A maioria dos CCR esporádicos são decorrentes de polipos adenomatosos, e as alterações associadas a ele começam principalmente com mutações no gene APC.
1.4.2 CCR hereditário
Figura 4: Diferentes tipos de câncer colorretal.
Referências: Al-tassan et al. (2002); Calva; Howe (2008); Chen, H. M.; Fang (2009); Colas et al. (2012); Jass (2008) e Lynch; De La Chapelle (2003)
As formas poliposas são mais raras, representando 1% de todos os tipos de câncer colorretal (JASS, 2008). Dentre elas a FAP é uma síndrome autossômica dominante, associada a mutações truncadas da linhagem germinativa no gene APC. O portador possui apenas um alelo funcional do gene, e este alelo pode também sofrer mutações (LYNCH; DE LA CHAPELLE, 2003).
A síndrome poliposa harmatomatosa é um grupo heterogêneo de desordens que representa menos de 1% das síndromes hereditárias com predisposição a CCR (CHEN, H. M.; FANG, 2009). Incluem as síndromes: Síndrome Poliposa Juvenil (JPS), Síndrome Peutz-Jeghers (PJS), Síndrome Bannayan-Riley-Ruvalcaba (BRRS), Síndrome de Cowden (CS), Síndrome Cronkhite-Canada (CCS), Síndrome Poliposa Hereditária Mista (HMPS) (CALVA; HOWE, 2008) e também Síndrome de Neoplasia Múltipla Endócrina 2B, Síndrome Nervosa de Célula Basal e Neurofibromatose 1 (CHEN, H. M.; FANG, 2009).
Essas síndromes têm em comum o aparecimento de polipos no trato gastrointestinal com risco aumentado para progressão ao câncer, mas com mecanismos diferentes aos encontrados nas síndromes adenomatosas (CALVA; HOWE, 2008).
O HNPCC, síndrome na forma não poliposa, é uma doença autossômica dominante e também o CCR hereditário mais frequente: representa 3% a 4% de todos os CCR, sendo uma doença de difícil diagnóstico, tanto clínica como molecularmente (COLAS et al., 2012; ZLOBEC et al., 2010).
Fu et al. (2013) evidenciam que portar mutações nos genes MMR confere fator de risco para o desenvolvimento do câncer colorretal. Pacientes portadores dessa mutação possuem risco para CCR de 74% em populações de países ocidentais e 90% na população da China.
Todos os pacientes com HNPCC possuem defeito na linhagem germinativa em um dos genes MMR (figura 5), e quando existe uma mutação somática, isso causa uma instabilidade em sequências repetitivas, como os microssatélites. O tumor afetado passa então a apresentar fenótipo de instabilidade de microssatélite (MSI) (FU et al., 2013; SINICROPE; SARGENT, 2012).
Os genes-alvo de MSI são conhecidos como TGFΒIIR, E2F4, e BAX, sendo que as mutações nas linhagens germinativas de MLH1, MSH2 e MSH6 (genes MMR) estão presentes e mutadas em 60-80% desses genes (VASEN; BOLAND, 2005).
A instabilidade de microssatélite pode ser mensurada como alta, baixa ou estável, usando marcadores moleculares recomendados do painel do National Cancer Institute. A determinação da presença de MSI é importante para o tratamento da doença, pois indica o prognóstico e resposta a medicamentos (SINICROPE; SARGENT, 2012).
Figura 5: Vias para a deficiência em reparo de mal-pareamentos em CCR
1.5 MICRORNAS (MiRNAS)
Primeiramente descritos por Lee; Feinbaum e Ambros (1993), os miRNAs são pequenas moléculas com 21 a 24 nucleotídeos de RNA não-codificante, cuja função é regular a expressão gênica por meio da clivagem ou inibição da tradução de mRNAs alvo (CARTHEW; SONTHEIMER, 2009), com grande importância na regulação de vários processos celulares como o crescimento celular, diferenciação, proliferação e apoptose (SU et al., 2011).
Existem atualmente uma série de bancos de dados de miRNAs, nos quais se armazenam as informações básicas sobre cada miRNA descoberto, como sequência gênica e sequência do miRNA, e se fazem ligações por bioinformática sobre as possíveis conexões de cada miRNA com doenças e processos celulares. De acordo com o principal banco de dados, o MIRbase, foram descritos até o presente momento 2237 miRNAs na espécie humana (MIRBASE, 2013).
A sequência de DNA que codifica para miRNAs pode ser encontrado em regiões de íntrons, bem como em regiões intergênicas (INUI; MARTELLO; PICCOLO, 2010). Eles são responsáveis por regular a expressão de 5300 genes, ou seja, cerca de 30% de todos os genes humanos (LEWIS; BURGE; BARTEL, 2005). Os miRNAs possuem caráter pleiotrópico: um só miRNA pode regular vários mRNAs, e em contrapartida, vários miRNAs podem regular um só mRNA (VOLINIA et al., 2010).
Como pode ser visto na figura 6, o miRNA é transcrito por uma RNApolimerase II a um miRNA primário (pri-miRNA). Essa molécula forma então uma estrutura em grampo
com o tamanho de 70 nucleotídeos, na qual a extremidade 5’ se complementa com a
própria molécula, e então é adicionada uma cauda poli A em uma extremidade da
Figura 6: Mecanismos de maturação do miRNA.
Fonte: Adaptada de Faber; Kirchner; Hlubek (2009)
O pri-mRNA sofre processamento com a ajuda das proteínas DROSHA e DGCR8, levando à formação de um pré-miRNA (CARTHEW; SONTHEIMER, 2009; INUI; MARTELLO; PICCOLO, 2010). As DROSHA são proteínas nucleares de aproximadamente 130-160KDa, e possuem 2 sítios catalíticos de RNAse III (nucleases específicas de RNA fita-dupla) e um domínio para ligação de RNA fita-dupla (dsRBD). A RNAse III atua em conjunto com a dsRBD em um complexo conhecido em mamíferos como DGCR8, que atua processando o pri-miRNA a pré-miRNA (FILIPOWICZ et al., 2005).
200 Kda e são classificadas pelos seus domínios: o domínio ATPase/RNA helicase, domínio DUF 283, domínio PAZ (compartilhado com proteínas ARGONAUTAS), dois domínios de RNAse III seguidos e domínios de ligação de RNA fita-dupla.
A molécula de miRNA, então processada pela DICER e DROSHA a dois fragmentos de fita-dupla, é incorporada ao complexo RISC (RNA-Induced Silencing Complexes), um conjunto de proteínas que variam dependendo do complexo e da espécie, onde a ARGONAUTA é a mais importante e presente em todos os conjuntos.
Os produtos de miRNA sofrem separação e ocorre uma associação estável da proteína ARGONAUTA com apenas uma das fitas que foram separadas. Dessa forma, a fita não associada é descartada, enquanto que a outra direciona o complexo por pareamento de bases ao mRNA alvo (CARTHEW; SONTHEIMER, 2009). Existem indicações a respeito do critério para se escolher qual das fitas será incorporada ao complexo: a fita com a extremidade 5’ mais termodinamicamente instável fará parte do
complexo miRISC (FILIPOWICZ et al., 2005).
O miRNA atua então como guia para o miRISC (SU et al., 2011), reconhecendo e dessa forma regulando RNAs. Geralmente, os mRNAs alvo são reconhecidos pelos miRNAs por meio das regiões 3’UTR (região não-traduzida) (MASAFUMIINUI, 2010). Na maioria dos casos esse reconhecimento de bases acontece com um grau de complementaridade imperfeito (especificamente, a região chave está nos nucleotídeos 2-8, e a falta de complementaridade está principalmente na parte central do miRNA) (CARTHEW; SONTHEIMER, 2009; FILIPOWICZ et al., 2005).
Uma complementaridade perfeita leva à clivagem do mRNA catalizada pela proteína ARGONAUTA, já uma complementaridade imperfeita não leva à clivagem, mas à repressão da tradução do mRNA: nos dois casos há a diminuição da transcrição do mRNA alvo. Existem teorias a respeito do momento em que essa repressão de tradução acontece (CARTHEW; SONTHEIMER, 2009; SU et al., 2011).
elas as mais importantes a inibição da circularização do mRNA (por deadenilação), competição por sítios com a cauda Cap do mRNA, impedir que os ribossomos permaneçam ligados ao mRNA (inibição pós-iniciação), promover a degradação do mRNA (deadenilation e decapping). Disso se entende que a repressão da tradução acontece tanto na iniciação quanto durante a elongação ou outro estágio posterior. Carthew; Sontheimer (2009) sugerem que a repressão acontece durante a elongação e o miRNA promove a dissociação de ribossomos do mRNA.
A maturação do miRNA pode ser o elemento chave para a regulação da sua expressão: estudos com câncer indicam que alguns precursores de miRNA se encontram em grande quantidade durante a embriogênese, porém não são processados a miRNAs maduros (INUI; MARTELLO; PICCOLO, 2010).
Os miRNAs possuem uma maquinaria complexa que modela as suas moléculas precursoras até se tornarem miRNAs maduros. Modificações em qualquer uma das proteínas que o processam podem modificar o padrão de expressão dos miRNAs que atuam em genes-chave para uma série de atividades celulares, como os genes que participam de processos carcinogênicos em vias de transdução do sinal (INUI; MARTELLO; PICCOLO, 2010).
1.5.1 MiRNAs relacionados com CCR
A desregulação da expressão dos miRNAs em um organismo pode provocar uma série de modificações nos padrões de migração celular, mitose e controle de apoptose. Esses padrões alterados são típicos em tumores e podem levar à evolução de um câncer (PEACOCK et al., 2012; SHIMONO et al., 2009).
mudanças epigenéticas (como metilações). Sendo assim, as regiões codificantes para muitos miRNAs estão localizadas em sítios de frequente deleção ou amplificação em câncer, bem como regiões de fragilidade genômica (CALIN et al., 2004), onde pode ocorrer a perda de heterozigosidade (de um gene supressor de tumor), uma amplificação (que pode conter um ongogene) ou translocações e inserções de vírus associados ao câncer (CALIN et al., 2004; VISONE; CROCE, 2009).
Uma das principais causas epigenéticas para a mudança do padrão de expressão de miRNAs está na metilação de ilhas CpG, pois cerca de 50% dos miRNAs estão associados a essas regiões. Quaisquer mutações no miRNA maduro, no pré-miRNA ou regiões genômicas próximas a proteínas da maquinaria de processamento do miRNA podem afetar seu processamento, levando a uma redução na expressão das proteínas da maquinaria processadora e gerando miRNAs que não pareiam com o mRNA como previsto. Isso gera um padrão de expressão diferenciado, que apresenta níveis de expressão de miRNA maduro baixos em relação aos tipos imaturos. Esse fato pode ser explicado pela perda da função de proteínas processadoras como a RNAse III DROSHA. Estudos com murinos indicam a perda da enzima DICER tornando impossível que animais com essa deficiência ultrapassarem a fase de gastrulação pela falta de células-tronco multipotentes (VISONE; CROCE, 2009).
Estudos em humanos indicam que o nível de expressão de DROSHA, DICER e DGCR8 se encontram notavelmente alterados em linhagens celulares de câncer gastrointestinal, quando comparado com linhagens normais (JAFARI et al., 2013).
A quantidade de miRNAs expressos em um tipo celular varia, então, entre os tecidos, e entre tecidos normais e patológicos. Logo, cada tipo celular apresenta um padrão de expressão de miRNAs que podem estar mais ou menos expressos em determinado momento. Isso cria uma assinatura celular que aponta para a identificação da mesma, implicando utilidade diagnóstica e prognóstica desses padrões de expressão (LU, J. et al., 2005).
amostras de câncer na região do cólon, 30 amostras de região do reto e 30 amostras de tecido normal (10 da região do cólon distal, 10 do cólon proximal e 10 do reto) para comparação dos perfis entre amostras normais e cancerosas relativas à mesma região em que o câncer estava situado. Apenas entre as amostras de tecido normal foram encontrados 287 miRNAs diferencialmente expressos entre as regiões do cólon. Isso evidencia a importância em se comparar perfis de miRNA de amostras que estejam em regiões similares. Foram selecionadas também amostras que possuíam particularidades, como instabilidade de microssatélite (MSI), metilação de ilhas CpG (CIMP+), amostras mutadas para o gene KRAS e mutações para o gene tp53. Para cada grupo uma comparação foi feita com suas respectivas amostras de tecido normal, e 143 miRNAs diferencialmente expressos em MSI, 129 miRNAs diferencialmente expressos em CIMP+,143 miRNAs em mutações em KRAS e 136 miRNAs em mutações em tp53 foram relatados (SLATTERY et al., 2011).
Estudos têm identificado miRNAs em câncer colorretal, a fim de usá-los como possível ferramenta para diagnóstico. Luo et al. (2011) identificaram 164 miRNAs com variações significantes em CCR em relação a amostras saudáveis, sendo que as variações mais significativas foram a superexpressão do miR-31 e a subexpressão do miR-145, sendo isso confirmado em mais oito estudos. Manne et al. (2010) identificou a diminuição dos miRNAs miR-133b, miR-143 e miR-145 e o aumento dos níveis de miR17-92, miR-18a, miR-20a, miR-31, miR-miR17-92, miR-96, miR135b e miR-183 como variações relacionadas ao CCR. O aumentos nos níveis de miR-17-92 é associado com aumento na expressão de C-MYC para carcinoma colorretal, e o aumento da expressão desse miRNA está diretamente associado com a progressão do câncer. Também, os miRNAs miR-135a e miR-135b foram correlacionados com níveis mais baixos de mRNA do gene APC (DIOSDADO et al., 2009; MANNE et al., 2010). A expressão do miR-21 aumentada também foi correlacionada com a progressão de tumores, uma vez que sua expressão em pólipos não-neoplásicos não é aumentada, mas em adenomas pré-cancerosos e tumores sua expressão aumenta progressivamente (YAMAMICHI et al., 2009).
tumorais. O quadro 3 mostra os principais miRNAs diferencialmente expressos em CCR em distintos momentos.
Quadro 3: MiRNAs diferencialmente expressos em CCR relacionados a distintas fases tumorais
Fases MiRNA
Screening miR‐17‐92, miR‐20a, miR‐21, miR‐92, miR‐96, miR‐106a, miR‐135, miR‐144, miR‐203, miR‐326
miRNAs mais frequentemente
alterados Subexpressos: miR‐133b, miR‐143 e mir‐145. Superexpressos: miR‐17‐92, miR‐18a, miR‐20a, miR-21, miR‐31, miR‐92, miR‐96,miR‐135b e miR‐183
Associados com pior prognose miR‐18a, miR‐21, miR‐20a, miR‐31, miR‐143, miR‐145, miR‐155, miR‐181b, miR‐200c, miR‐203 e miR‐106a Associados com metástase miR‐17‐92, miR‐21, miR‐31, miR‐135a, miR‐335, miR‐206, miR‐10b, miR‐146a/b e let7a/b
Fonte: MENENDEZ et al. (2013)
Figura 7: Mudanças na expressão de miRNAs em adenomas e carcinomas colorretais correlacionados com estágio e sobrevivência.
Fonte: FABER; KIRCHNER; HLUBEK (2009)
1.6 VARIAÇÃO DE NÚMERO DE CÓPIAS (CNV)
As CNVs, também conhecidas como Large-segment Copy-Number Variations (LCVs) ou Copy-Number Polymorphisms (CNPs) (IAFRATE et al., 2004; WEISE et al., 2008), são variações estruturais do genoma que apresentam tamanho a partir de 1Kb (TALSETH-PALMER et al., 2013). Essas sequências de DNA resultam de deleções, e duplicações que ocorrem em um determinado segmento de DNA, resultando em um número de cópias diferente. Tais mudanças do DNA são estáveis, e podem ser herdadas ou adquiridas (eventos de novo) (ALMAL; PADH, 2012; BREHENY et al., 2012).
Em 2006, Redon et al. (2006) construíram o primeiro mapa de CNVs no genoma humano. O estudo, que agrupou 270 indivíduos de variadas ancestralidades, observou um total de 1.447 regiões de variação do número de cópias, abrangendo cerca de 12% do genoma. Dentro dessas regiões foram reconhecidos centenas de genes, incluindo loci de elementos essenciais para manutenção celular e relacionados a doenças, que possuíam variações entre as populações.
Muitas vezes, as CNVs podem ser grandes o suficiente para afetar vários genes e regiões de regulação dos mesmos, e dessa forma influenciar na expressão de um gene. Assim, seu papel no fenótipo dos organismos é importante: ela influencia desde características fundamentais para a manutenção da vida, até diversas patologias (DHAWAN; PADH, 2009; MORENO-DE-LUCA; CUBELLS, 2011; VAISHNAVI et al., 2013).
Para se determinar se uma CNV é oriunda de linhagem germinativa (herdada) não é simples, pois para estabelecer se a CNV foi herdada ou se é um evento de novo é necessário que se analise o padrão de CNVs dos pais do indivíduo, e isso abre espaço para interpretações errôneas. Por exemplo, quando dois pais são heterozigotos para uma determinada CNV (possuem uma ou mais cópias em um alelo e deleção no outro alelo) e o filho herda o padrão homozigoto (uma ou mais cópias nos dois alelos): não se pode determinar com certeza se em um dos alelos o padrão de CNVs foi herdado ou se houve uma mutação de novo (FREEMAN et al., 2006). Essa é uma das razões pelas quais muita informação é perdida em relação a essas variações estruturais (FEUK; CARSON; SCHERER, 2006).
É importante ressaltar que em uma patologia poligênica, raramente apenas uma CNV determina suscetibilidade para a doença. Geralmente isso depende de outros fatores, como outras CNVs, outros tipos de variantes genômicas e o ambiente (ALMAL; PADH, 2012). Uma prova de que a CNV sofre alterações somáticas é o mosaicismo, no qual o ambiente altera o padrão de CNVs germinativas para linhagens celulares diferentes, e um mesmo indivíduo não mais possui o seu genoma idêntico para todas as células (FANCIULLI; PETRETTO; AITMAN, 2010).
Angstadt et al. (2013) identificou que duas enzimas ligadas a CNVs (UGT2B17 e UGT2B28), responsáveis pela desintoxicação do organismo e por várias patologias como obesidade e câncer de pulmão, são comumente deletadas no genoma como uma forma de polimorfismo (57% dos caucasianos possuem pelo menos uma deleção). Sabendo que os dois genes alteravam a expressão gênica de células neoplásicas em CCR, eles avaliaram 665 pacientes caucasianos de uma região onde o consumo de carne é alto para avaliar uma ligação entre estes genes e o ambiente como fatores de risco para CCR. Eles identificaram o UGT2B17 como sendo um gene que confere fator de proteção ao CCR nos indivíduos testados, principalmente homens, e que a exposição aumentada a hidrocarbonetos aromáticos policíclicos e aminas heterocíclicas encontrados em carnes vermelhas e processadas aumenta o risco do desenvolvimento de CCR, sugerindo que pode haver uma interação desses componentes com o gene associado à CNV UGT2B17.
Talseth-Palmer et al. (2013) avaliaram 100 indivíduos portadores de HNPCC que foram genotipados para associação com CNVs. Cerca de 28% dos pacientes sofreram ganhos de número de cópias no cromossomo 7q11.21 (contra 0% dos controles) e 46% em 16p11.2 (contra perda em 23% dos controles). A validação do resultado mostrou significâncias na associação dessas regiões com o HNPCC, ressaltando também o aumento da instabilidade genômica nesse tipo de síndrome. Reforçando o aumento de instabilidade genômica, Chen, W. et al. (2013) evidenciaram a instabilidade de microssatélite em HNPCC relacionada ao perda de heterozigosidade e alteração em número de cópias. Eles identificaram 10 amplificações de ganho (7p14.1, 7p15.3, 7q31.2, 8q22.11, 3q31.3, 13q32.4 e 20p11.23) e três de perda tumor-específicas (17p13.11, 18q21.33 e 15q26.1) provavelmente envolvidas com o desenvolvimento da síndrome. Nessas regiões foram encontrados genes relacionados ao desenvolvimento celular e de câncer, e particularmente dois genes foram encontrados superexpressos significantemente em relação a amostras normais, podendo ser candidatos genes responsáveis pelo desenvolvimento da doença.
desenvolvimento induzido do CCR. Com mapeamento gênico estudando os efeitos desse locus, encontrou variações de número de cópias diferentes em relação a tecidos normais no íntron 15 do gene NFKB1. Essa proteína faz parte da via Nfkb, uma via relacionada à regulação da homeostase e resposta inflamatória no tecido intestinal. A ativação dessa via de forma anormal é associada com CCR (MEUNIER et al., 2013).
1.6.1 miRNA x CNV
A variação do número de cópias pode ser uma forma comum de variabilidade para moléculas de miRNA (MARCINKOWSKA et al., 2011). Wong et al. (2007) reportaram a presença de miRNAs detectados em regiões de CNV e reconheceu as potenciais consequências deste achado. Uma outra busca genômica de CNVs identificou algumas regiões duplicadas abrigando genes de miRNA, alguns deles relacionados com doenças, como esquizofrenia e autismo (ROGAEV; ISLAMGULOV; GRIGORENKO, 2008). Na
figura 8 está descrito um esquema de como o miRNA pode ser influenciado na presença de CNVs.
Figura 8: Representação esquemática do transcrito primário de um miRNA. A posição da sequencia do pré-miRNA está indicada como uma estrutura em forma de grampo. Linhas pontilhadas representam transcritos que não devem ser produzidos pela falta de regiões promotoras ou sequencias iniciadoras. Os quadros laranja representam as regiões de CNV em deleções, duplicações ou duplicações dispersas. Cada grupo (A, B, C e D) representa a CNV localizada em
diferentes partes do gene do miRNA. Os sinais “+” , “-“ e “0” indicam potencial para aumento, diminuição ou nenhuma mudança para a expressão do miRNA
Fonte: Marcinkowska et al. (2011)
Pelo fato de as CNVs participarem de 18% das variações na expressão de genes, existe uma correlação plausível em que a expressão dos miRNAs pode ser modificada pelo número de cópias em CNVs, bem como sabe-se que os genes de miRNAs estão geralmente amplificados ou perdidos em tumores. Marcinkowska et al. (2011) analisaram a co-localização de loci de miRNAs em regiões de CNV, e identificaram e validaram 209 miRNAs nessa condição usando banco de dados DGV.
classificações de tumores. As regiões de CNV identificadas em comum para todos os tipos de tumor não eram compostas de DNA codificante, e foram encontradas, em outras regiões de CNVs, sequências codificantes para três miRNAs, 12 mRNAs, deleções de genes envolvidos com modificação de histonas e remodelamento de cromatina. Isso reforça a idéia que a variação do número de cópias de alguma forma influencia a expressão de moléculas não codificadas, como os miRNAs.
Garcia-Orti et al. (2012) avaliaram a influência das CNVs na expressão de miRNAs em Leucemia Mielóide Aguda. Dezenove miRNAs possuiam associação significante entre a sua expressão e a CNV na qual estavam relacionados. Dentre eles, o miR-370 estava em uma região de alta amplificação e superexpresso. Foi visto que o alvo desse miRNA é um supressor tumoral (NF1) comumente subexpresso na Leucemia Mielóide Aguda. Uma análise funcional mostrou que a superexpressão do miR-370 levou à inativação do NF1, evidenciando que a influência da CNV sobre o miR-370 é um mecanismo envolvido na subexpressão ou inativação do NF1 (AN et al., 2013; DI FIORE et al., 2013). Nesse estudo não foi feita a correlação estatisticamente válida da influência da CNV sobre a expressão do miR-370, porém é demonstrado que isso, de fato, pode acontecer.
Estudos mostram que as CNVs podem influenciar na dosagem de genes associados a elas, incluindo miRNAs e unidades codificadoras de proteínas processadoras de miRNA, como a DICER1 e ARGONAUTE2. Wu; Zhang; Li (2012) descreveram uma alta frequência em CNVs que contêm genes processadores de miRNA. Um total de 209 miRNAs foram relatados em regiões de CNV. Foram encontrados 1.134 genes regulados exclusivamente por CNV-miRNAs. Concluiu-se que as CNV-miRNAs tendem a regular em média um número maior de genes-alvo, e que esses alvos tendem a ter uma variabilidade maior de expressão em populações. Esses genes-alvo também tendem a ter maior expressão diferencial entre tecidos e estados de desenvolvimento celular.
inibição de mRNAs-alvo. Porém, se esse gene-alvo também se encontra em uma região de CNV, e esta região se duplica, pode não haver mudança alguma no seu produto gênico. Mesmo dessa forma, há alterações que levam a diferenças fenotípicas, devido ao efeito pleiotrópico dos miRNAs (VAISHNAVI et al., 2013).
O câncer colorretal é uma doença de importância global, segundo dados epidemiológicos. A elucidação de mecanismos isolados que influenciam a tumorigênese tem importante papel na compreensão das desordens causadoras do câncer. Dentro do contexto dos miRNAs e CNVs exercendo influência sobre as doenças, não se pode ignorar que não apenas um, mas vários caminhos moleculares, em nível de mutações e alterações epigenéticas, regulam paralela e conjuntamente as diferentes vias de sinalização que levam ao desenvolvimento do câncer colorretal.
A análise da expressão diferencial dos miRNAs tem sido elucidada, bem como a variação do número de cópias. Alguns estudos já relacionam esses eventos com algumas características de desfecho clínico, como remissão e óbito, porém, até o momento, não existem relatos na literatura relacionando a influência dos CNVs no padrão de expressão de miRNAs em câncer colorretal. Uma análise profunda nesse assunto se torna essencial, em consequência da crescente disponibilidade de informação em bancos de dados de miRNAs e CNVs, uma vez que já possuímos o conhecimento de vários miRNAs diferencialmente expressos, bem como de CNVs no câncer colorretal.
2 OBJETIVOS
2.1 OBJETIVO GERAL
Correlacionar a expressão diferencial de microRNAs com CNVs em espécimes de câncer colorretal visando compreender se a variação do número de cópias influencia na diferença de níveis expressão de miRNAs em CCR.
2.2 OBJETIVOS ESPECÍFICOS
Analisar a expressão diferencial de miRNAs entre tecidos normais, tumorais e linfonodais por qPCR;
Analisar a Variação do Número de Cópias entre tecidos normais, tumorais e linfonodais por qPCR;
Comparar os dados obtidos nos experimentos, verificando se as alterações de número de cópias de CNV no genoma podem modificar os níveis de expressão dos miRNAs em CCR;
4 MATERIAL E MÉTODOS
4.1 COLETA DAS AMOSTRAS
Fragmentos de amostras de 31 indivíduos foram coletadas no Hospital Universitário da Pontifícia Universidade Católica do Rio Grande do Sul (Hospital São Lucas) e cedidas durante procedimento cirúrgico realizado em portadores de câncer colorretal. De cada um, foi retirada amostra de tecido tumoral, amostra de tecido saudável do intestino e linfonodos. Após a retirada dos tecidos, as amostras foram armazenadas em RNAlatter (Invitrogen) e estocadas a -80oC.
Os pacientes ainda não haviam sido submetidos a tratamento com quimioterapia ou radioterapia no momento da cirurgia, e todas as amostras foram avaliadas histopatologicamente. Foi assinado um Termo de Consentimento Livre e Esclarecido, encontrado no Anexo A. As informações clínicas de todos os pacientes, incluindo idade, sexo, presença de histórico familiar, localização do tumor, tipo histológico, classificação TNM e localização de metástase, remissão e sobrevida global estavam disponíveis.
Esse projeto foi aprovado pelo comitê de ética da Universidade Católica de Brasília com o número de aprovação 160/2009.
4.2 EXTRAÇÃO DO RNA E DNA
RNAses, por três tempos de 2 minutos a 28Hz. Assim, esta segunda metodologia foi adotada posteriormente para o rompimento das células.
Para a extração de RNA total, a metodologia do Trizol reagent® (invitrogen) foi utilizada com algumas modificações: a amostra previamente triturada foi colocada em 750 µL de Trizol e incubada 10 minutos a temperatura ambiente, agitada vigorosamente em intervalo três vezes durante 30 segundos. Após essa etapa, a amostra foi centrifugada a 12.000 r.p.m. durante 10 minutos a 4oC e o sobrenadante foi transferido para um novo tubo. Em seguida, 200 µL de clorofórmio foram adicionados. A amostra foi misturada vigorosamente por 15 segundos e incubada por 10 minutos, depois centrifugada por 15 minutos a 12.000 r.p.m. a 4oC. Nessa etapa, se formam duas fases: uma com Trizol (onde se encontra o DNA) e outra transparente (onde se encontra o RNA). A fase aquosa transparente foi então transferida para um novo tubo para dar prosseguimento ao protocolo (e a fase que contém o DNA foi armazenada a -20oC para posterior extração do seu DNA). Em seguida, 500µL de isopropanol foram adicionados à amostra, e esta foi incubada durante 10 minutos a temperatura ambiente ou durante a noite a -20oC. Posteriormente, foi centrifugada a 12.000 r.p.m. por 10 minutos a 4oC. O Pellet foi lavado com 900 µL de etanol 75% gelado, centrifugado a 12.000 r.p.m. durante 5 minutos a 4oC e seco a temperatura ambiente por 10 minutos. O RNA foi ressuspenso em 30µL de água Milli-Q e armazenado a -80oC.
As amostras de DNA foram extraídas a partir do protocolo de extração de RNA pela fase de Trizol separada. Foi adicionado 500 µL de tampão de extração e a amostra foi incubada por 30 minutos a 45oC, invertendo o tubo a cada 5 minutos. Após essa etapa, foi
centrifugada a 12.000 r.p.m. a 4oC por 15 minutos. O sobrenadante foi transferido para um
4.3 VERIFICAÇÃO DA QUALIDADE E QUANTIFICAÇÃO DAS AMOSTRAS
As amostras de RNA e DNA foram quantificadas utilizando Qubit® 1.0 fluorometer (Qiagen) conforme recomendação do fabricante. A qualidade foi verificada por meio do método de eletroforese em gel de agarose (1%), utilizando como agente intercalante fluorescente o brometo de etídio (10µg/mL). A visualização das bandas foi realizada no transiluminador de ultra-violeta DyDNA Light (Labnet) conjuntamente com o foto-documentador Lab Trade.
4.4 SELEÇÃO DOS MIRNAS E CNVS
Quadro 4: Lista de primers relativos aos miRNAs e CNVs relacionados com CCR
miRNA Cromossomo
Ensaio TaqMan® de
CNV
hsa-miR-650 22 Hs03322330_cn
hsa-miR-570 3 Hs03487806_cn
hsa-let-7g 3 Hs03459881_cn
hsa-mir-183 7 Hs04967643_cn
hsa-miR-31 9 Hs03281785_cn
hsa-miR-338–
3p 17 Hs03984183_cn
hsa-mir-150 19 Hs04012189_cn
hsa-mir-1-1 20 Hs00892069_cn hsa-miR-16-1 13 Hs03846573_cn RNU-6B
(controle) 10 Hs05225739_cn
Para melhor visualização, cada CNV foi nomeada de acordo com o miRNA a ela relacionada. Dessa forma, a CNV relacionada ao hsa-miR-650 foi chamada de CNV-miR-650, e assim por diante.
Quadro 5: Principais pontos que evidenciam a relevância dos miRNAs escolhidos neste estudo.
miRNA Características Referência
miR-570 Inibe a tradução da proteína b7-h1, associada com progressão de câncer gástrico quando superexpressa. Um dos 4 miRNAs superexpressos fortemente associados com pacientes sem recorrência de CCR.
WANG, W. et al., 2013 ; CHRISTENSEN et al., 2013
miR-16 Supressor de tumor. Associado com grau histológico de diferenciação. Subexpresso em 67% dos CCR. Ligado à via de si alização p53. Se liga à região 3’UTR do RNA codifica te para COX-2 (fator encontrado superexpresso no epitélio intestinal tumoral)
MA, Q. et al., 2013 ; YOUNG et al, 2012
miR-338 Supressor tumoral. Subexpresso em CCR. Regula a expressao da proteína Smoothened (SMO), associada à via Hedgehog e com grau de agressividade do CCR.
GAEDCKE et al, 2012 ; SUN, K. et al., 2013
Let-7g Função de supressão tumoral. Altos níveis associados com melhor taxa de sobrevida. Tem como alvo o mRNA do gene KRAS (que é um fator negativo na resposta a tratamentos com EGFRs - anticorpos monoclonais)
RUZZO et al., 2012 ; KJERSEM et al., 2012 ; SMITS et al., 2011
miR-1 Um dos miRNAs mais subexpressos em cânceres e em CCR. Associado à inibição de TAGLN2 (Transgelin 2 gene)
KANAANRAIEICHENBERGERROBERTS et al., 2012 ; HAMPFJORD et al., 2012
miR-150 Subexpresso em CCR e outros cânceres. Função de supressão tumoral. Baixa expressão indica pior prognose. Potencial biomarcador para CCR. Predileção por bioinformática indicam que ele se liga ao mRNA da proteína p53 (possível função protooncogênica)
MA, Y. et al., 2012 ; BOUSQUET et al., 2013 ; LIN et al., 2011 ; WANG, D. T. et al., 2013
miR-183 Superexpresso em CCR, associado a CCR com MSI. Pode ter como alvo membros da familia FOX
EARLE et al., 2010 ; BRANDES et al., 2006 ; MOTOYAMA et al., 2009
miR-650 Envolvido com metástase linfocítica e distante em CCR.
Associado ao gene NDRG2 (N-Myc Downstream –regulated gene
2), um supressor de tumor associado com crescimento celular, diferenciação e apoptose, subexpresso em vários tipos de câncer devido à metilação de ilhas CpG. Ele foi identificado como co patível à ligação de sua região 3’UTR co o iR-650
FENG et al., 2011
miR-31 Superexpresso em CCR. Tem como alvo RASA1 (uma proteína que quando inibida leva à ativação da via de sinalização RAS em CCR.
BARNDRES et al., 2006 ;
MOTOYAMA et al., 2009 ; XU, R. S. ET AL., 2013 ; SUN, D. et al., 2013
como controle constitutivo. No entanto, para amostras de CCR aqui utilizadas, o RNU6B demonstrou diferença na expressão entre os indivíduos normais e tumorais, não sendo considerado como constitutivo para o estudo.
4.5 SÍNTESE DE cDNA PARA MiRNAS
As reações de síntese de cDNA a partir de transcriptase reversa foram feitas utilizando o kit miRNA reverse transcription (Applied Biosystems®). A reação consiste em: 5,0 µL de amostra (contendo 10 ng de RNA), 3,0 µL de primer, 1,5 µL de tampão a 10X, 0,15 µL de ddNTPs, 1,0 µL de multiscribe RT, 0,19 µL de inibidor de RNAse e 4,16 µL de água milli-Q.
As reações foram ajustadas para um volume final de 15 microlitros e incubadas no termociclador Veriti Cycler (Applied Biosystems) por 30 minutos a 16oC, 30 minutos a 42oC, 5 minutos a 85oC e armazenadas a 4oC.
4.6 PCR EM TEMPO REAL (qPCR)
As reações de qPCR dos miRNAs ocorreram utilizando o kit TaqMan MicroRNA Assay (Applied Biosystems) e os ensaios Taqman (Applied Biosystems). As proporções dos reagentes foram: 0,665 µL de amostra, 0,5 µL de primer Taqman, 5 µL de Universal PCR Mastermix e água para o volume final de 10 microlitros. O ensaio de referência (controle constitutivo) utilizado foi o RNU48.
As reações foram feitas utilizando a máquina Step One Plus® (Applied Biosystems), incubadas a 95oC por 10 minutos, seguido por 40 ciclos de 95oC por 15 segundos e 60oC
Para os testes de CNVs, as proporções dos reagentes foram: 4 µL de Universal MasterMix, 3,2 µL de água milli-Q, 0,4 µL do primer relativo a cada CNV, 0,4 µL de ensaio de referência e 2 µL de cada amostra. O ensaio de referência utilizado foi o RNase P, relativo a uma ribonucleoproteína com regiões altamente conservadas, com função de processamento de pré-tRNAs (Nossent, Rischlueller & S, 2012).
4.7 ANÁLISE DOS DADOS DE MIRNA
Os ensaios TaqMan são constituídos de um oligonoucleotídeo de fita-única. Esses ensaios amplificam de forma específica sobre a sequência-alvo do cDNA, e a cada ciclo, quando novas moléculas são amplificadas, há a geração de sinal luminescente, a ser lido e interpretado em forma de gráfico.
A quantidade mínima de luminescência emitida pela amplificação das moléculas de cDNA a ser detectada é denominada Cq. Logo, se um produto de qPCR leva poucos ciclos para apresentar um dado de Cq, ele possui boa quantidade de produto gênico. Todavia, para esse estudo, a quantidade de produto gerado na qPCR foi quantificado de forma relativa, ou seja, sua quantidade foi mensurada em relação a uma amostra padrão.
No caso dos miRNAs, a quantificação relativa (RQ) das amostras foi determinada utilizando o método comparativo de ΔΔCq. Este é um método no qual a expressão do gene da amostra é medido com base em uma amostra de referência, ou controle endógeno (calibrador). Para os miRNAs, o controle endógeno utilizado foi o RNU48. O
método de ΔΔCq calcula as diferenças nas expressões gênicas primeiramente calculando
o ΔCq (média da triplicata do Cq do gene alvo menos a média da triplicata do Cq do gene
constitutivo). A partir desses dados, se calcula o ΔΔCq (subtração de dois ΔCq que se quer analizar, como o ΔCq do tumor ou linfonodo menos o ΔCq do normal). A partir daí, é
4.8 ANÁLISE DOS DADOS DE CNV
Após a amplificação das CNVs, seguindo as mesmas técnicas para miRNA até a determinação do Cq (usando como calibrador o RNAse P), os arquivos de dados que contém os valores da amostra foram exportados para uma ferramenta de software de análise recomendada pela Applied Biosystems, o software CopyCaller TM. Esse software quantifica o número de cópia dos dados experimentais, e assume-se que o calibrador tem 2 números de cópia.
4.9 ANÁLISES ESTATÍSTICAS
Para verificar a significância estatística dos dados gerados, o programa IBM SPSS statistics foi utilizado como ferramenta para a análise.
5 RESULTADOS E DISCUSSÃO
5.1 ANÁLISE CLÍNICA
Tabela 1: Características Clínicas e Histopatológicas dos indivíduos Selecionados
Indivíduos Sexo
Idade ao diagn. Inf. a
50 anos Loc. do tumor TNM AJCC Remissão Status 30 M Sim Ileocecal T3N0M0 II-A Sim
Vivo s/doença 39 M Não Retossigmóide T2N0Mx I Sim
Vivo s/doença 48 F Não Ileocecal T3N0Mx II-A Sim
Vivo s/doença 53 M Não Retossigmóide T3N2Mx IV Sim Óbito 55 M Não Retossigmóide T3N1Mx III-B Sim
Vivo s/doença 58 M Não Retossigmóide - - Não Óbito 60 M Não Retossigmóide T3N1Mx IV Não Óbito 70 M Não Retossigmóide T3N2M1 IV Não Óbito 73 M Não Ileocecal T3N1Mx III-B Sim
Perda de contato 75 M Não Retossigmóide T3N1Mx IV Sim Óbito 77 M Não Retossigmóide T3N2Mx IV Sim Óbito 85 M Sim Retossigmóide T3N0M0 II-A Sim
Vivo s/doença 92 M Não Retossigmóide T3N1M1 IV Não Óbito 94 M Não Retossigmóide T1N0M0 I Sim
Vivo s/doença
95 F Não Retossigmóide T3N0M0 II-A Sim
Vivo c/ doença 96 M Não Ileocecal T3N0M1 IV Não
Vivo c/ doença 97 M Não Retossigmóide T3N1M1 IV Sim Óbito 98 F Sim Retossigmóide T3N0M0 II-A Sim
Vivo s/doença 99 F Não Ileocecal T3N1M1 IV Não Óbito 101 M Não Retossigmóide T3N0M0 II-A Sim
Vivo s/doença 104 F Não Ileocecal T3N0M0 II-A Sim Óbito 105 M Não Ileocecal T3N0M0 II-A Sim
Vivo s/doença 107 M Não Retossigmóide T3N1Mx IV Não Óbito 143 F Não Retossigmóide T3N1M1 IV Não Óbito 144 M Não Retossigmóide T3N2M1 IV Não Óbito 146 F Sim Retossigmóide T3N2M0 III-C Sim
Vivo c/ doença 147 F Não Ileocecal T3N0M1 IV Não
Vivo c/ doença 152 F Sim Retossigmóide T3N2M2 IV Sim
Vivo s/doença 153 M Não Retossigmóide T3N0M0 II-A Sim
Vivo s/doença 154 F Não Retossigmóide T3N2Mx III-C Sim
Vivo s/doença 155 M Não Ileocecal T3N0M0 II-A Sim
Para melhor visualização, as características clínicas foram organizadas de acordo com idade ao diagnóstico, sexo, localização do tumor, estadiamento (TNM e AJCC), remissão e estado atual dos indivíduos em relação à doença.
A idade dos indivíduos ao diagnóstico variou entre 26 (indivíduo 146) e 86 (indivíduo 95), com a média de idade de 64 anos. Quanto à faixa etária, observou-se predomínio de indivíduos com idade ao diagnóstico igual ou superior a 50 anos (83,8%) em relação aos indivíduos com idade inferior a 50 (16,12%). Quando os indivíduos foram analisados quanto ao sexo, a maioria selecionada é do sexo masculino (67,7%) e a minoria do sexo feminino (32,25%). De acordo com os dados histopatológicos, 28 indivíduos possuíam tumores do tipo adenocarcinoma (90,3%), enquanto que apenas um apresentou carcinoma coloide (3,2%), e dois indivíduos apresentaram polipos (6,4%). Quanto à localização do tumor, vinte e dois (70,9%) se encontravam na região retossigmóide, enquanto que 9 (29%) se encontravam na região ileocecal (Tabela 2). Para os indivíduos selecionados neste trabalho, a história familiar não foi considerada como um critério de análise, uma vez que somente dois indivíduos (85 e 97) possuíam estes dados.
Tabela 2: Características dos Indivíduos
Características dos indivíduos
Variável Categorias N. de indivíduos %
Idade ao diagnóstico < 50 anos 5 16,1
> 50 anos 26 83,8
Sexo Masculino 21 67,7
Feminino 10 32,2
Tipo de tumor
Adenocarcinoma 28 90,3
Carcinoma coloide 1 3,2
Polipo 2 6,4
Localização do tumor Retossigmóide 22 70,9
Em relação ao estadiamento (tabela 3), os indivíduos foram classificados pelo sistema TNM: T3 (93,5%), T2 (3,2%) e T1 (3,2%) e AJCC, no qual foram classificados em: I (6,6%), II-A (33,3%), III-B (6,6%) III.C (6,6%) e IV (46,6%). A presença de metástase linfonodal e distante também foi considerada. A primeira foi diagnosticada em 16 indivíduos e a outra em 14. A metástase distante, quando avaliada, foi observada em 7 pacientes, sendo 3 hepáticas; 3 pulmonares e 1 hepática e esplênica no mesmo indivíduo. Todavia, em muitos casos não foi possível avaliar a presença do tumor distante representados como Mx na tabela 1.
Tabela 3: Estadiamento
Estadiamento e metástase
Classificação Categoria N. de indivíduos %
TNM
T1 1 3,2
T2 1 3,2
T3 29 93,5
AJCC
I 2 6,6
IIA 10 33,3
IIIB 2 6,6
IIIC 2 6,6
IV 14 46,6
Metástase Linfonodal 16 53,3
Distante 14 46,6
Tabela 4: Número e percentagem de indivíduos que apresentaram remissão e status clínico
Remissão e óbito
Variável Categorias N. de indivíduos %
Remissão Sim 21 67,7
Não 10 32,2
Status Vivo com doença Vivo sem doença 13 4 13,3 43,3
Óbito 13 43,3
5.2 ANÁLISE MOLECULAR
5.2.1 Qualidade dos ácidos nucleicos