PRÓ-REITORIA DE PÓS-GRADUAÇÃO E PESQUISA
STRICTO SENSU EM CIÊNCIAS GENÔMICAS E
BIOTECNOLOGIA
CENTRO DE ANÁLISES PROTEÔMICAS E BIOQUÍMICAS
Tese de Doutorado
DESENVOLVIMENTO DE NOVAS TERAPIAS NO COMBATE A LINHAGENS DE E n t e r o b a c t e r c l o a c a e RESISTENTES À COLISTINA.
Autora: Thais Bergamin Lima
Orientador: Dr. Octávio Luiz Franco
THAIS BERGAMIN LIMA
DESENVOLVIMENTO DE NOVAS TERAPIAS NO COMBATE A
LINHAGENS DE E n t e r o b a c t e r c l o a c a e RESISTENTES À COLISTINA.
Tese apresentada ao Programa de Pós-Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia da Universidade Católica de Brasília, como requisito parcial para obtenção do Título de Doutor em Ciências Genômicas e Biotecnologia.
Orientador: Dr. Octávio Luiz Franco Coorientadora: Beatriz Simas Magalhães
.
AGRADECIMENTOS
Ao meu orientador Octávio Luiz Franco, por me direcionar a atingir meus objetivos e ainda por lapidar-me durante esses quatro anos, para que eu me tornasse a profissional que sou hoje. À minha co-orientadora professora Beatriz, que deu asas a minha imaginação e base para que compreendesse melhor o mundo obscuro da espectrometria de massa e suas aplicações.
À professora Simoni, que sempre foi solícita para a discussão e desenvolvimento dos experimentos.
À professora Nádia, que transmitiu-me seus conhecimentos a cerca do cultivo de micro-organismos em biorreatores.
Ao professor Robert e a professora Rosângela, ao Aulus, ao Renato e a Lauriane pela extração de DNA e RNA.
À professora Cristine Barreto e sua aluna Cristiane pelo sequenciamento das bactérias por 16S.
A Betty e ao Nelson por me auxiliarem na parte na elaboração dos géis bidimensionais e por discutir os problemas associados a esta técnica.
Aos meus antigos alunos de iniciação científica Carlos, Alícia, Michele e Kleber que me auxiliaram nos experimentos e se disponibilizaram a vir até mesmo nos finais de semana e feriados.
Ao Osmar que sempre esteve disposto a me ajudar e ainda pela sua amizade.
As minhas amigas Keyla, Suzana, Dielle, Michelle, Amanda e Mariana que congelaram comigo na sala do Maldi, discutiram experimentos, corrigiram a minha tese e até mesmo cuidaram da minha filha para que eu pudesse trabalhar, a vocês minha imensa gratidão e carinho.
As minhas irmãs de consideração Simone, Lauriane e Tatiane, que por mais de 10 anos, mesmo que separadas geograficamente, estão sempre presentes na minha vida e estarão para sempre no meu coração.
Ao meu maridão Juceli, pelo companherismo, paciência, apoio e compreensão que fez esta jornada passível de ser cumprida. Sem seu auxílio nunca teria conseguido. Te amo meu amor...
Aos meus pais e meu irmão pelo apoio e incentivo, que durante estes quatros anos foram fundamentais para que eu prosperasse.
"Toda a nossa ciência, comparada com a realidade, é primitiva e infantil - e, no entanto, é a coisa mais preciosa que temos."
RESUMO
Referência: LIMA, Thais Bergamin. Desenvolvimento de novas terapias no combate a linhagens de E n t e r o b a c t e r c l o a c a e resistentes à colistina.2013.
170f. Tese de Doutorado em Ciências Genômicas e Biotecnologia - Universidade Católica de Brasília, Brasília, 2013.
As infecções relacionadas a assistência a saúde (IrAS), juntamente com o aparecimento de isolados multirresistentes, representam um grave problema de saúde pública. Deste modo, novas terapias de controle de tais isolados bem como a avaliação de proteínas envolvidas no mecanismo de resistência à colistina em Enterobacter cloacae foram estudados neste
trabalho. Para isto foram estabelecidos os padrões de crescimento bacteriano de E. cloacae
(ATCC 49141), bem como a indução de resistência à colistina. O padrão fenotípico de resistência foi determinado por quatro métodos usualmente empregados incluindo Vitek, MicroScan, disco difusão e microdiluição. Estes métodos demonstraram que as isolados são potenciais produtoras de β-lactamases de espectro estendido (ESBL), além de apresentarem
resistência à eritromicina, vancomicina e colistina. Objetivando identificar as proteínas ou peptídeos envolvidos no mecanismo de resistência, as isolados foram cultivadas em biorreatores e seus sub-proteomas avaliados por 2DE e espectrometria de massa. Através destas análises foi possível averiguar que, aparentemente não existe nenhuma molécula proteíca secretada em reposta ao estresse causado pela colistina nas isolados resistentes a este antibiótico. Nas análises de proteínas de membrana pôde-se identificar as proteínas de membrana externa, OmpX e OmpA, que estão correlacionadas com a virulência deste patógeno bem como outras moléculas. Para a implementação de novas terapias no combate a estas isolados, avaliou-se o efeito combinado de colistina com diferentes antibióticos convencionais bem como a colistina com o peptídeo antimicrobiano, HHC-10 e, este combinado com cloranfenicol. Como resultado verificou-se efeito sinérgico de cloranfenicol com HHC-10 assim como o do cloranfenicol com colistina. Entretanto, ao avaliarmos os efeitos citotóxicos destes antimicrobianos em combinação, em células da linhagem RAW 264.7 de monócitos, verificou-se que HHC-10 apresenta citoxicidade mesmo quando empregado juntamente com cloranfenicol, inviabilizando a sua possível utilização clínica. Em contrapartida, não foi evidenciado efeitos citotóxicos de colistina com cloranfenicol em concentração duas vezes maiores do que as concentrações onde se observou o efeito sinérgico. Desta forma os dados deste trabalho sugerem uma nova estratégia para o controle de linhagens de E. cloacae multirresistentes, demonstrando a possível empregabilidade da
colistina e cloranfenicol no tratamento clínico.
ABSTRACT
The infections related to health care (IRAs), along with the emergence of multiresistant isolates represent a serious public health problem. Thus, new control therapies as well as, proteins involved in the Enterobacter cloacae colistin
resistance mechanism were studied in this work. Therefore were established standards of bacterial growth of E. cloacae (ATCC 49141) and colistin resistance induction.
The phenotypic resistance pattern was determined by four methods usually employed including Vitek, MicroScan, disk diffusion and microdilution. These methods showed that the isolated are potential extended spectrum β -lactamase (ESBL), and were resistant to erythromycin, vancomycin and colistin.In order to identify the proteins or peptides involved in the mechanism of resistance, the strains were grown in bioreactors and its sub-proteome analyzed by 2DE and mass spectrometry. Apparently there is no protein molecule secreted in response to stress caused by colistin in strains resistant to this antibiotic.In the analysis of membrane proteins could identify the outer membrane protein, OmpA and OmpX, which are correlated with the virulence of this pathogen as well as other molecules. For the implementation of new therapies to fight these isolates, we evaluated the combined effect of colistin with different conventional antibiotics and colistin plus antimicrobial peptide, HHC-10, and this combined with chloramphenicol (CHL). By broth microdillution checkerboard method it was observed the synergistic effects in combined use of CST and CHL, well as HHC-10 with CHL. However, to evaluate the cytotoxic effects of these antibiotics in combination, cells of RAW 264.7 monocyte lineage, it was found that HHC-10 shows cytotoxicity alone and when used together with chloramphenicol, preventing its possible clinical use. In contrast, there were no evidence cytotoxic effects of colistin with chloramphenicol at a concentration twice higher than the concentrations where there was a synergistic effect. Thus, this study suggests a new strategy for the control of E. cloacae multiresistant, demonstrating the potential use of
colistin and chloramphenicol in clinical treatment.
LISTA DE FIGURAS
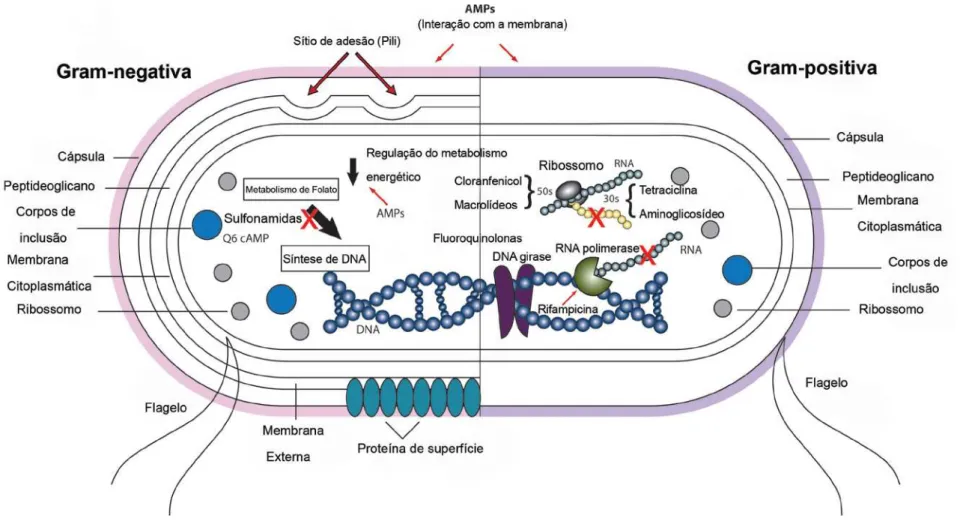
Figur a 1 : Repr esentação do s pr incip ais alvo s intr acelulares d e antibió ticos. Os antib iótico s q ue são capazes de inibir o metabolismo do folato, q ue é um co mpo nente necessário par a a síntese de ADN (sulfo na midas). Além disso, o s antibiótico s q ue inib em a biossíntese d e proteínas podem agir sobre o s ribosso mo s utilizando a sub nuid ade 30 S (tetr aciclina e a mino glicosídeo s) o u 50S (cloranfenico l e macro líd eo s) . Anti micro biano s envo lvido s na r eplicação de DNA e RNA pod em causar uma inibição da tr anscrição po r ligação a RNA po lime rase, prevenindo assim a sua ligação no DNA (r ifamp icina). Finalmente antibió tico s que se ligam na DNA girase (q uino lo nas). Fonte: ( Lima e t a l .,
2013). ... 26
Figur a 2: E str utur a tr idimensio nal de magainina II em so lução aquo sa. Estr utur a deter minad a por RM N, depositad a no PDB so b o código 2 mag ( Gesell e t a l ., 1997) e visualizad a pelo so ft war e UCSF Chimer a. ... 28
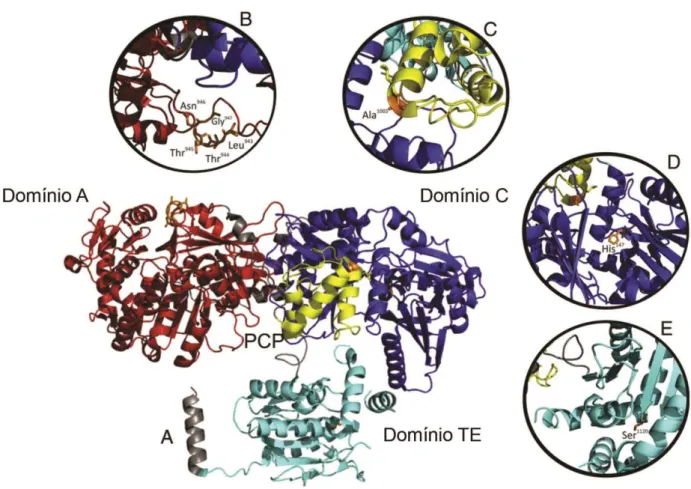
Figur a 3: Estr utura 3 D dos NRP S ( A) SRFA-C, um NRPS de B a c i l l u s s u b t i l i s , e o s seus
do mínio s catalítico s relacio nado s repr esentado s na cor laranja. (B) o sítio catalítico Leu943 -T hr944 -T hr945 - Asm946 -Gly947. ( C) d o mínio catalítico PCP e Ala1003 (D) o do mínio C e His147 (E) o do mínio T E e Ser1120 . Fo nte: (M andal e t a l ., 2013) ... 30
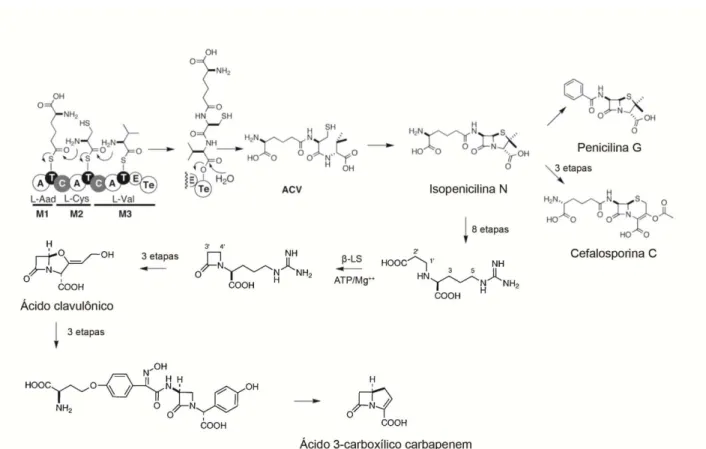
Figur a 4 : U ma visão geral da bio ssíntese de ácido clavulânico e do s membro s
representativos dos antibióticos β-lactâmico s. Na bio ssíntese de penicilina, d -(
L-aminoad ipoilo) -L-cisteinil-D-valina ( ACV) (1) é ciclizado para isop enicilina N através d a isop enicilina N sintetase. Bio ssíntese de ácido clavulânico, d ifere na ciclização de N2 (2 -carbo xietil) -arginina (CEA) pelo ação da enzima β-lactâmico sintetase (β-LS) . Várias transfor mações sub seq uentes desta molécula são necessárias p ara for mar o ácido clavulânico, mo nobactêmico s e carbapenens. Fonte: (Bach mann e t a l ., 1998; Felnagle e t a l ., 2008) ... 32
Figur a 5: E str utura q uímica do s fár maco s glico peptíd eos ( A) vanco micina hidrocloridr ico, (B) telavancina e ( C) teicoplanina. Fo nte: P ub Chem. ... 34
Figur a 6: Uma visão geral da bio ssíntese do s lipopepttídeo s. O mód ulo de iniciação (IM ) consiste Dp tE ( AL, acil-Co A-ligase ho mólo go) e DptF (t, do mínio T ACP). Os demais são co mpo nentes d a NRP S e sub str ato s não p roteino gênico s ( LOr n, Lor nitina; 3 mGlu, 3 -metil-L-Glu; Kyn, kynur enine). Fo nte: ( Felnagle e t a l ., 2008). ... 38
Figur a 7: E str utura q uímica do s fár maco s polipeptíd eos ( A) bacitr acina, (B) polimixina B e ( C) co listina. Fo nte: Pub Chem ... 40
Figur a 8: E vo lução da resistência microbiana aos antibió tico s co nvencio nais em u m período d e 1950 a 201 0. Asterisco s indicam que algu mas linhagens destes p ató geno s atualmente não podem ser tratadas co m o s fár maco s antimicrob iano s. Legenda: ( Verd e) Fungos. ( Ver melho) Gr am-negativo. ( Ro xo) Gr am-positivo. ( Azul) Gram -po sitivo/ ácid o resistente. ... 44
Figur a 9: T écnicas utilizadas par a ob tenção da cur va de crescimento ( a) S p r e a d - p l a t e (b) e
micro go ta. ... 69
Figur a 11: Cur va de crescimento de E . c l o a c a e utilizando a técnica s p r e a d - p l a t e. Leitura
realizad a e m espectro fotô metro a 595 nm. E nsaio feito em tr iplicata e barr as ver ticai s correpo sndem ao desvio padrão. ... 92
Figur a 12 : Deter minação da CI M de colistina co ntra a iso lado de E . c l o a c a e AT CC
49141.Cultivo em meio LB sup lementado co m diferentes co ncentrações de co listina. Co ntrole negativo ( C-) água. Análise r ealizada no leito r de microplaca co m temp eratura (37 ° C) e agitação co nstante (40 rp m) , a densidade óptica ( DO) a 595 nm foi mensurad a em inter valo s de 30 min. Desvio padrão representado pela b arra vertical. ... 94
Figur a 13: Deter minação do CI M de colistina nas sub -cultur as resistentes a co listina: A) R1; B) R2 ; C) R3. Cultivo em meio LB suplementado co m diferentes co ncentrações de colistina. Análise realizada no leitor de microp laca co m te mperatura ( 37 ° C) e agitação constante (40 rp m), a d ensidade óp tica (DO) a 595 nm fo i mensurad a em inter valos d e 30 min. Desvio p adrão r epresentado pela barra vertical. ... 96
Figur e 14: Esp ectro de massa M ALDI -T OF das iso lado s de E . c l o a c a e [ (E . c l o a c a e AT CC
49141; isolado s não r esistentes à colistina (NR 1, 2 e 3); isolado s resistentes à co listina (R1, 2 e 3)]. As proteínas foram analisadas no espectro metro de massa Micro Flex LT em modo linear po sitivo , usando o método MBT . ... 101
Figur a 16: E spectro de massa do tipo M ALDI -T OF. Detecção de r esistência a p enicilina G por meio da esp ectro metria de massa. ( A) matriz CHCA; (B) meio LB + p enicilina (256µ g. mL- 1); ( C) M eio LB apó s o cultivo da iso lado d e
E . c l o a c a e AT CC 49141 co m
penicilina G (256µ g. mL- 1). ... Erro ! Indicado r não definido .
Figur a 17: Cur va de cr escimento das isolado s de E. cloacae NR1 e R1, cultivadas em biorreator de de 2 L (Bioflo 110, Ne w Br unswick Scientific Co.), a 37 º C, p H 6.8, aer ação de 100 % e agitação de 300 rp m. I nó culo inicial de 5 x105 CFU. mL- 1. I solado R1 cultivada
em presença de colistina a uma co ncentração de 8 µ g. mL- 1. Erro ! Indicado r não definido .
Figur a 18: Análise do p erfil de massa molecular obtido por SDS -P AGE 12% d e proteínas presentes no secreto ma de isolados não resistentes ( A) NR1; (B) NR2 ; ( C) NR3 e resistentes a co listina ( D) R1 ; (E) R2; ( F) R3. MM ind ica o marcador de massa molecular, valores ap resentado s em kDa. ... Erro ! Indicado r não definido .
Figur a 19: Análise co mparativa do s géis 2 -DE de proteínas citosó licas dos isolado s d e E . c l o a c a e não r esistentes à colistina ( NR1 -3) e não resistentes à colistina (R1 -3). ... Erro ! Indica dor não def inido .
Figur e 20: Análise do s frag mento s tríp tico s das proteinas de me mbrana de E . c l o a c a e não
resistente ( NR1 -3) e resistentes à colistina ( R1 -3), após o cultivo em LB, no s b iorreatores e extr ação d ifer encial. Nas isolados R1 -3 o meio LB fo i suple mentado co m co listina. Espectro s de massa M ALDI -T OF ob tido s no AutoFlex, e m modo RP com faixa d e detecção de 700 -3500Da. ... Erro ! Indicado r não definido .
Figur e 21: Análise dos frag mento s tríp tico s das proteinas d e membrana de E. cloacae não resistente ( NR1 -3) e resistentes à colistina ( R1 -3), após o cultivo em LB, no s b iorreatores e extr ação d ifer encial. Nas isolados R1 -3 o meio LB fo i suple mentado co m co listina. Espectro s de massa M ALDI -T OF ob tido s no AutoFlex, e m modo RP com faixa d e detecção de 700 -3500Da. ... Erro ! Indicado r não definido .
das células tr atadas fo i co mp arad a co m co m o controle. As barras de erro representam o desvio padrão d a méd ia da viabilid ade celular de três experimentos ind ependentes realizado s em trip licata. ... 140
LISTA DE TABELAS
T abela 1 – Princip ais mecanismo s d e resistência anti microbiana. ... 48
T abela 2 – Número de Ir AS em unid ades de terapia intensiva no s Estudo s Unido s por local de ocorrência e or ganismo. ( ND) : Não detectad o; ( *) Número to tal de Ir AS fo i de 92.45 4 detectad as em unidades de ter apia intensiva em um p eríodo d e oi to ano s. ... 55
T abela 3 – Distrib uição dos diferentes soro tipo s de E . c l o a c a e e seus p adrões d e
resistência a agentes antimicrob iano s. Abr eviações: NT (não tipad o); multiresistante (resistente a cefoperozo na ( CP Z, 75 µ g), cefotaxima ( CT X, 30 µ g), imipenen (IP M, 10 µ g) , cipro flo xacino ( CI P, 5 µ g), tetr aciclina ( TT , 30 µ g), cloranfenicol ( CLO, 30 µ g), sulfameto xazo l-tr imetro pim (SUT , 1,25/23,75 µ g), a mpicilina ( AP , 10 µ g), cefalotina ( CF, 30 µ g), gentamicina ( GN, 10 µ g) , e cefalexina ( CX, 30 µ g) . GONÇALVEZ et al, 2000. .. 59
T abela 4: I nterpretação dos testes bioq uímico s realizado s co m o kit d e identificação de enterob actérias ( Ne wpr ov), Análises co nd uzid as co m as iso lado s de E . c l o a c a e AT CC
49141 e suas sub -cultur as não r esistentes (NR1 -3) e ind uzidas a resistência à colistina (R1 -3). Fer mentador o u produtor (+) , Não fer me ntador o u não prod utor ( -). ... 98
T abela 5. Co mparação d os halo s de inibição d e diferentes antibió tico s obtido s através do teste d e disco d ifusão para as iso lado s de E . c l o a c a e AT CC 49141 e suas sub -cultura s
suscep tíveis à co listina (NR1 -NR3) e r esistentes à co listina (R1 -R3 ) cultivadas e m meio Muller -Hinto n. ... 108
T abela 6: Co mpar ação entre as co ncentrações inibitó rias mínimas ( CI M s) para E . cloacae deter minad as p elo s sistemas VIT EK 2 e M icro Scan. ... 110
T abela 7. Interpr etação dos testes de suscep tibilidad e antimicrobiana das iso lado s de
E n t e r o b a c t e r c l o a c a e p or método s auto matizad os, disco difusão e teste de microdiluição.
Resultado s obtido s co nfor me os cr itério s de interpretação estabelecido s no CLSI M100 -S21.(V), VIT EK® 2 ( bioMérieux); (M) , M icro Scan® W alkAwa y® ( Siemens); (D), disco difusão ; (T ), teste de microd iluição ; ( NT ), não testado; AT CC, E . c l o a c a e AT CC 4 9141;
(NR1, NR2 e NR3) E . c l o a c a e susceptíveis à colistina; (R1, R2 e R3), E . c l o a c a e
resistentes à colistina; ( S), sensível; (I), inter mediár io; ( R) , resistente. ... 113
T abela 8 : Identificação das proteínas apó s a extr ação diferencial para ob tenção das proteínas d e membrana em isolado s de E . c l o a c a e não r esistentes e resistentes a co listina.
A id entificação proteica fo i obtida atr avés do sequencia mento dos espectros M S/MS. ... 131
T abela 9: Avaliação do efeito co mbinado de colistina (COL) e cloranfenicol ( CLO) co ntr a iso lado s de E . c l o a c a e . ... 135
LISTA DE ABREVIATURAS
AmpC - β-lactamase classe C
ANVISA - Agência Nacional de Vigilância Sanitária
ATCC - American Type Culture Collection
Blastp - Basic local alignment search tool protein
BLEEs - β-lactamases de espectro estendido (extended spectrum β-lactamase - ESBL)
CHCA -
CIM - Concentração inibitóriamínima
CLSI -Clinical and Laboratory Standards Institute
Da - Daltons
2-DE - eletroforese bidimensional em gel (two-dimensional gel electrophoresis)
2D DIGE - gel bidimensional de eletroforese diferencial com fluorescência (2-D Fluorescence Difference Gel Electrophoresis)
D.P - Desvio padrão
Domínio T - domínio de tiolação
Domínio A - domínio de adenilação
Domínio CV - domínio de clivagem
Domínio E - domínio de epimerização
Domínio MT - domínio de metilação
Domínio TE - domínio de tioesterificação
ESI - El ectrospray i onization
FDA – Food and Drug Administration
kDa – KiloDalton
KPC - K lebsi ella pneumoni ae carbapenemase
LACEN-DF -Laboratório Central de Saúde Pública do Distrito Federal
LB - Meio Luria Bertani
LPS – lipopolissacarídeo
LTD - Desaminação do L-Triptofano
MALDI-TOF-TOF -Matrix-Assisted Laser Desorption Ionization Time-of-Flight.
MBL - metalo β-lactamase
MM - Marcador molecular
2-MPA - ácido 2-mercaptopropiônico
MRR - regiões de multirresistência
MS - Espectrometria de massa (Mass spectrometry)
MSP - dendrograma da projeção do espectro médio (mai n spectrum proj ection dendrogram)
m/z - Massa/carga
MW – Massa molecular (Molecular weight)
NCBI – Centro Nacional de Informações Biotecnológicas (National Center for Biotechnology Information)
NRPSs - Peptídeos produzidos por sintetases não-ribossomais
OMS - Organização Mundial da Saúde (World Health Organization)
OD – Densidade óptica
OMP- Proteína de membrana externa
PCP - proteína transportadora peptidil
PBP - Peni cillin Bi nding Protei ns
pI - Ponto isoelétrico
pH - Potencial hidrogenionico
PIC – Coquetel inibidor de proteinase (p roteina se inhibit or cocktail)
RPM - Rotações por minuto
SDS - Dodecil sulfato de sódio (Sodium dodecyl sulfate)
SDS-PAGE - Eletroforese em gel de poliacrilamida com dodecil sulfato de sódio (sodium dodecyl sulfate polyacrylamide gel electrophoresis)
TCA - Ácido tricloroacético
TFA - Ácido trifluoroacético
UFC - Unidade formadora de colônia
SUMÁRIO
LISTA DE FIGURAS ... 10
LISTA DE TABELAS ... 13
LISTA DE ABREVIATURAS ... 14
1. INTRODUÇÃO ... 21
2. REVISÃO DE LITERATURA ... 23
2.1. ANTIBIÓTICOS ... 23
2.1.1. Antibióticos peptídicos ... 25
2.1.1.1. Glicopeptídeos ... 33
2.1.1.2. Lipopeptideos ... 35
2.1.1.3. Polipeptídeos ... 39
2.2. RESISTÊNCIA BACTERIANA ... 43
2.3. RESISTÊNCIA À β-LACTÂMICOS ... 49
2.4. RESISTÊNCIA AOS GLICOPEPTÍDEOS ... 51
2.5. RESISTÊNCIA AOS POLIPEPTIDEOS ... 52
2.6. INFECÇÕES RELACIONADAS À ASSISTÊNCIA A SAÚDE (IRAS) 53 2.7. ENTEROBACTER ... 58
3. JUSTIFICATIVA ... 64
4. HIPÓTESE ... 65
5. OBJETIVO ... 65
6. METAS ... 65
7. MATERIAL E MÉTODOS ... 67
7.1. Cultivo e produção da curva de crescimento microbiano dos isolados 67 7.2. Preparação de E . c l o a c a e resistente a colistina ... 70
7.3. Identificação de E . c l o a c a e ... 70
7.3.1. Identificação bacteriana utilizando-se provas bioquímicas 71 7.3.1.1. Kit comercial ... 71
7.3.1.2. Identificação da espécie por métodos automatizados (Vitek 2 e MicroScan) ... 72
7.3.2. Identificação das espécies bacterianas via MALDI BioTyper 73 7.4. Dendrograma da Projeção do Espectro Médio (MSP) ... 74
7.5. Avaliação da susceptibilidade de E . c l o a c a e ATCC 49141 e suas sub-culturas a antibióticos de classes distintas ... 74
7.5.2. Determinação da Concentração Mínima Inibitória (CIM)
pelo método de microdiluição ... 76
7.5.3. Antibiograma por disco difusão ... 76
7.6. Análise proteômica comparativa de E . c l o a c a e susceptível e resistente a colistina ... 77
7.6.1. Produção de E . c l o a c a e em bioreatores ... 77
7.6.2. Preparo das amostras ... 79
7.6.3. Secretoma ... 81
7.6.3.1. Avaliação do crescimento bacteriano de E . c l o a c a e e secreção proteica dos isolados resistentes e suscetíveis a colistina ... 81
7.6.3.2. Análise do perfil proteico presente no secretoma dos isolados de E . c l o a c a e resistentes e não- resistentes a colistina ... 81
7.6.3.3. Análises eletroforéticas do perfil de massas moleculares . 82 7.6.4. Citosol ... 82
7.6.4.1. Eletroforese bidimensional (2-DE) ... 82
7.6.5. Membranas ... 83
7.7. Análise do perfil proteico de E . c l o a c a e em resposta ao estresse a diferentes antibióticos. ... 84
7.7.1. Análise do lisado celular ... 84
7.9. Ensaio de citotoxicidade ... 88
8. RESULTADOS E DISCUSSÃO ... 90
8.1. Determinação dos padrões de cultivo bacteriano de E . c l o a c a e ATCC 49141 ... 90
8.2. Preparação da isolado bacteriana antibiótico-resistente ... 93
8.3. Identificação da isolado ATCC e suas sub-culturas ... 97
8.4. Avaliação da susceptibilidade da ATCC 49141 e suas sub-culturas 104 8.5. Análise proteômica comparativa de E . c l o a c a e susceptível e resistente a colistina ... 117
8.5.1. Secretoma ... 119
8.5.2. Citosol ... 122
8.5.3. Membranas ... 125
8.5.4. Avaliação do sinergismo ... 132
8.5.5. Ensaio de citotoxicidade ... 138
1. INTRODUÇÃO
Os antibióticos têm mecanismos de ação distintos capazes de modificar a tradução, a replicação de DNA e síntese da parede celular (Morar e Wright, 2010). Apesar disso, nas últimas duas décadas, têm sido relatados inúmeros casos de resistência à maioria de classes de antibióticos utilizados na prática clínica. Tal fato tornou-se um dos maiores problemas de saúde pública principalmente em países em desenvolvimento, porque estas infecções causam alta mortalidade e morbidade, que culminam na perda de recursos humanos e econômicos (Askarian et al ., 2013).
De acordo com Aminov e colaboradores (2010) o custo do tratamento de infecções relacionadas a ambientes de saúde foi de no mínimo $1,8 bilhões de dólares em 2006, sendo este valor superior a despesa anual com infecções causadas pelo vírus influenza. Além do custo de tratamento e métodos de prevenção para redução de estirpes resistentes em ambientes de saúde, ressalva-se que em muitos casos as terapias empregadas não são eficazes, desperdiçando-se um grande volume financeiro e principalmente, resultando na morte de milhões de pessoas em todo o mundo (Grundmann et al., 2011).
Algumas estratégias são considerados imprescindíeis para uma melhora deste panorama tais como: implementação de medidas preventivas para limitar a disseminação dos micro-organismos resistentes; desenvolvimento de uma política de uso racional de antimicrobianos; rapidez no diagnóstico; elaboração de novas drogas com mecanismos de ação distintos aos antibióticos usuais (Côrrea, 2008).
Uma série de fatores contribuiu para o declínio da produção de antibióticos. Primeiramente, há uma dificuldade técnica para descoberta de novos antibióticos, especialmente aqueles capazes de atuarem sobre Gram-negativas. Além disso, a consolidação da indústria farmacêutica através de fusões e aquisições, bem como a extinção de muitas empresas farmacêuticas que contribuíram durante o chamada “era do ouro”, promoveram uma redução do número de empresas com capacidade e experiência para desenvolver novos agentes antibacterianos (Livermore et al., 2011). Ressalva-se ainda que, o
modelo econômico atual não favorece o desenvolvimento de antibacterianos, uma vez que, o retorno financeiro com a comercialização dos antibióticos não é atrativo para as indústrias farmacêuticas, pois, há um elevado investimento para a produção dos mesmos, estes são empregados em tratamento por um pequeno período e ainda, com o surgimento da resistência a antimicrobianos, estes são utilizados durante um curto prazo (Livermore et al., 2011; White et al., 2011). Sendo assim, a visão mercadológica destas empresas tem sido
voltada para a produção de outros fármacos empregados para o tratamento de doenças pulmonares crônicas obstrutivas bem como, no tratamento da exacerbação aguda da bronquite crônica, além de fármacos para doenças cardiovasculares e câncer (White et al., 2011).
Diante deste panorama na prática clínica, fármacos anteriormente abandonados, por apresentarem espectro de ação similar aos usuais, porém com uma maior toxicidade, têm sido reintroduzidos (Maalej et al ., 2011).
Além disso, no tratamento clínico adotou-se como estratégia uma melhor utilização dos antibióticos licenciados, sendo empregado o uso combinado desses agentes terapêuticos a fim de obter uma melhora no tratamento (Elouennass et al ., 2012). Entretanto, em alguns casos a combinação pode
resultar em alterações dos parâmetros farmacocinéticos e consequentemente, em uma maior toxicidade (Petrosillo et al ., 2008).
ou envolve outros mecanismos? A colistina poderia ser utilizada em associação com outro antibiótico no combate a isolados multirresistentes e até mesmo contra as isolados resistentes à colistina?
A fim de sanar estas questões foram levantadas algumas hipóteses. A primeira hipótese é que a resistência à colistina é ocasionada por alterações nas proteínas de membrana uma vez que, este antibiótico atua causando uma ruptura da membrana da bactéria. A segunda hipótese é que ao ser resistente a colistina, a bactéria pode ser resistente a outro peptídeo antimicrobiano que tenha o mesmo mecanismo de ação. A terceira hipótese é que a colistina ao ser empregado em combinação com outros antibióticos com mecanismos de ação diferentes proporciona uma maior eficiência terapêutica e diminui os efeitos toxicológicos.
Deste modo, o objetivo do nosso trabalho foi fornecer subsídios para o desenvolvimento de uma nova terapia de controle de isolados multirresistentes bem como, avaliar proteinas envolvidas no mecanismo de resistência de Ent erobacter cloacae à colistina.
A relevância deste trabalho dar-se-á, sobretudo pela possibilidade de utilização da colistina em associação com cloranfenicol no combate a isolados multirresistentes. A combinação de ambos os antibióticos aumenta a potencialidade da ação antimicrobiana bem como, reduz os efeitos citotóxicos destes antimicrobianos, quando empregados sozinho e em altas doses.
2. REVISÃO DE LITERATURA
Os antibióticos são, indiscutivelmente, as drogas mais bem sucedidas implantadas ao longo dos últimos 100 anos, pois contribuíram para o controle de doenças infecciosas que foram as principais causas de morbidade e mortalidade da população (Aminov, 2010). Os primeiros antibióticos foram preparados sinteticamente, como por exemplo, a arsfenamina. Este composto apresenta em sua estrutura o arsênico e foi previamente nomeado como Salvarsan, sendo descoberto por Ehrlich na virada do século 20 e, as sulfonamidas, que são inibidores do metabolismo de folato desenvolvidos pela Domagz em 1930 (Jesman et al ., 2011). Posteriormente em 1940, iniciou-se a
produção em escala industrial da penicilina, um composto isolado por Alexander Fleming em 1928 ao estudar o fungo Peni cilli um chrysogenum
(Jesman et al., 2011). Este período entre 1940 e 1970 foi considerado a “era
do ouro” devido ao surgimento de novos antimicrobianos logo após a descoberta e disseminação do uso das sulfonamidas e da penicilina que propiciaram o tratamento de infecções consideradas graves e potencialmente fatais (Chopra et al., 2002).
Desde então, muitos outros antibióticos têm sido desenvolvidos a partir de fontes diferentes, com diversos alvos e modos de ação sendo mais eficazes e apresentando menos efeitos colaterais (Kong et al., 2010). Atualmente são
empregados na clínica diversos antibióticos com distintos mecanismos de ação causando a morte celular ou simplesmente cessando o crescimento microbiano.
Os diferentes mecanismos de ação destas moléculas podem ser visualizados na Figura 1.
2.1.1. Antibióticos peptídicos
2.4.1.1.Peptídeos oriundos de proteínas sintetizadas por ribossomos
As magaininas, (do hebraico “magain” que significa escudo, proteção) constituem um grupo de potentes peptídeos antimicrobianos obtidos da pele de sapos da África do Sul, da espécie Xenopus laevis, conhecido também
como sapo africano com garras (Zasloff, 1987; Soravia et al ., 1988).
Em 1987 foram isolados os dois primeiros membros dessa família, as magaininas I e II, as quais apresentam estruturas helicoidais (Figura 2) similares formadas por 23 resíduos de aminoácidos e potencialmente anfipáticas, diferindo apenas nas substituições nas posições 10 de glicina (Gly) por lisina (Lys) e 22 de lisina (Lys) por asparagina (Asn) (Zasloff, 1987; Maria-Neto et al., 2012).
As magaininas apresentam um amplo espectro de ação, sendo capazes de inibir, em concentrações reduzidas, o crescimento de fungos, bactérias Gram-positivas e -negativas. O mecanismo de ação proposto menciona que esse peptídeo ao entrar em contato com a membrana de micro-organismos assume uma estrutura secundária helicoidal anfipática capaz de interagir com a bicamada de fosfolipídeos, interferindo na integridade da membrana, e promovendo a sua desestabilização e ruptura (Duclohier, 1994; Bechinger, 2011).
A catelicidina LL-37 (hCAP18), é um peptídeo catiônico encontrado em humanos no epitélio bucal e na saliva, são derivados dos neutrófilos e das glândulas salivares (Murakami et al., 2002). Além da atividade microbicida, a
LL-37 apresenta importantes funções adicionais na defesa do hospedeiro, incluindo propriedades quimiostáticas e modulação da resposta inflamatória, atraindo neutrófilos, monócitos, células T e mastócitos (Bowdish et al., 2004;
Figura 2: Estrutura tridimensional de magainina II em solução aquosa. Estrutura determinada por RMN, depositada no PDB sob o código 2mag (Gesell et al., 1997) e visualizada pelo software UCSF Chimera.
2.4.1.2.Peptídeos não ribossomais
2.4.1.2.1 Peptídeos produzidos por sintetases não-ribossomais (NRPSs)
Estas sintetases são compostas de proteínas organizadas em módulos, sendo cada um deles responsável pela incorporação de um específico aminoácido no peptídeo final (Abdo et al., 2012) De um modo geral, a
montagem de um peptídeo por NRPS envolve uma série de passos que são catalisadas pelas ações coordenadas dos três domínios catalíticos principais: o domínio (A) adenilação, o domínio de tiolação (T ou proteína transportadora peptidil, PCP), e os domínios (C) de condensação (Felnagle et al., 2008). O domínio A é responsável pelo reconhecimento do substrato e
adenilato (Felnagle et al ., 2008; Roongsawang et al ., 2010) O domínio T faz
uma ligação covalente com o intermediário acil-ciclase para formar o domínio aminoaciltioéster e o domínio C, que é responsável pela formação da ligação peptídica entre o aminoacil adenilato e aminoaciltioéster, promovendo o enlogamento da cadeia peptídica (Felnagle et al., 2008; Roongsawang et al .,
2010).
Para que o peptídeo seja liberado da enzima, um domínio adicional está presente na porção C-terminal da NRPS, o domínio de tioesterificação (domínio TE) ou clivagem (domínio CV). Além dos domínios A, T, C e TE básicos para todas as NRPS, há outros domínios adicionais como o domínio de epimerização (Domínio E) que faz a conversão de L-aminoácidos a D-isomeros, ou ainda, o domínio de metilação (MT) que promove metilações ao longo do peptídeo (Felnagle et al., 2008; Roongsawang et al., 2010),
Figura 3: Estrutura 3D dos NRPS (A) SRFA-C, um NRPS de Bacillus subtilis, e os seus domínios catalíticos
O peptídeo pode ser liberado por hidrólise na forma de um peptídeo linear ou ainda sofrer uma reação intramolecular originando um peptídeo cíclico (Roongsawang et al., 2010). Há diversos antibióticos comerciais que
não são sintetizados pela via ribossomal tais como, os β-lactâmicos, a lipopeptídeos, polipeptídeos e glicopeptídeos.
2.4.1.2.1. β-lactâmicos
Os β-lactâmicos são uma grande classe de antibióticos, que inclui as
penicilinas, cefalosporinas, carbapenens, monobactâmicos e inibidores de β -lactamases (Diaz-Mejia et al ., 2009). A penicilina e cefalosporina são
produzidos por fungos dos gêneros Peni cil lium, Cephalospori um, e Aspergi llus e por bactérias incluindo Strept omyces, Nocardia, Flavobact erium
e espécies L ysobact er. Independentemente do micro-organismo produtor, a
biossíntese dos produtos naturais derivados de penicilina e cefalosporina envolve a formação do intermediário comum isopenicilina N (Bachmann et al., 1998). Este intermediário é formado pela modificação do tripeptídeo L-α
-aminoadipato-L-Cys-D-Val (ACV) por isopenicilina N sintetase, que catalisa
a formação dos β-lactâmicos e anéis da tiazolidina da isopenicilina N. A
montagem deste tripeptidio é catalisada por uma NRPS, conhecida como a ACV sintetase (Weber et al., 2012). Enquanto, para os demais antibióticos
dessa classe como, carbapenens, monobactâmicos e os inibidores de β
-lactamases são produzidos por St reptomyces cl avuligerus e sua biossíntese
O mecanismo de ação dos β-lactâmicos é por meio da reação de inativação da transpeptidação da biossíntese da parede celular, que conduz à lise da célula (Diaz-Mejia et al., 2009).
2.1.1.1. Glicopeptídeos
Dentre os glicopeptídeos mais empregados destacam-se a vancomicina, isolada de Amycolat opsis orientali s, ativa contra bactérias Gram-positivas,
incluindo estafilacocos resistentes à meticilina. Além disto, este antibiótico tem sido utilizado contra enterococos resistentes (Scherl et al., 2006). Este
fármaco tem afinidade pelo resíduo N-acyl D-ala-D-ala do peptídeoglicano da bactéria estabelecendo 5 pontes de hidrogênio que por sua vez, acarretam em uma inibição da síntese da parede celular (Drummelsmith et al., 2007). A
vancomicina atua nas transpeptidases ou PBP (Penicillin Binding Proteins), proteínas envolvidas na síntese do peptidoglicano, que se encontram na face exterior da membrana citoplasmática e utilizam um grupo amino como nucleófilo para formar uma acyl-enzima intermediária entre o sítio hidroxila da serina e a carbonila da D-alanina (Navarre e Schneewind, 1999; Scherl et al., 2006).
2.1.1.2. Lipopeptideos
Os lipopeptídeos são uma classe de antibióticos, também conhecidos como acilpeptídeos, que apresentam uma molécula lipídica conjugada com um peptídeo que apresenta uma porção peptídica catiônica ou aniônica (Lima et al., 2013). De um modo geral, os lipopeptídeos apresentam 11 a 13 resíduos
de aminoácidos, dentre os quais alguns são sintetizados por via não ribossomal e ainda, apresentam diferentes ácidos graxos na porção N-terminal facilitando assim, a sua inserção na bicamada lipídica das membranas bacterianas e, por conseguinte a atividade antibacteriana devido a sua diversidade estrutural (Strieker e Marahiel, 2009).
Alguns deles até chegaram a um estado de antibiótico comercial, como daptomicina, a caspofungina, micafungina e anidulafungina (Mandal et al.,
Estes fármacos são em geral oriundos do metabolismo secundáio de micro-organismos do gênero Bacill us, Pseudomonas e Strept omyces
(Raaijmakers et al ., 2010). Nesta classe estão inclusos a anfomicinas, a
aspartomicina, a A5145 e a daptomicina, sendo este último o único empregado clinicamente (Strieker e Marahiel, 2009). A5415, produzido por Streptomyces fradi ae, é um composto cíclico contendo 10 resíduos de aminoácidos e uma
cadeia exocíclica de 3 resíduos de aminoácidos, cuja estrutura assemelha-se à daptomicina, conforme observado na Figura 6 (Alexander et al., 2011). Este
lipopeptídeo contém diversos aminoácidos não-proteinogênicos tais como, hidroxiasparagina (hAsn3), sarcosina (Sar5), ácido metoxiaspártico (moAsp9) e ácido 3-metil glutâmico (3mGlu12) (Gu et al., 2011). De acordo com este
autor, a localização dos aminoácidos coincidem com a localização dos D-aminoácidos da daptomicina, enfatizando assim, a possibilidade de ambos terem estrutura tridimensional semelhante e consequentemente o mesmo modo de ação.
A daptomicina consiste em um lipopeptídeo cíclico natural de
Streptomyces roseosporus e enquadrada na família A21978C, que em geral
contém um núcleo peptídico com 6 aminoácidos não proteigênicos: D- isômeros de Asn, Ala e Ser, bem como, ornitina, L-Cinurenina and 3- ácido metilglutamico (3mGlu) (Nguyen et al ., 2011). A daptomicina contém 13
aminoácidos hidrofílicos e uma cadeia lateral lipofílica. Dentre estes 13 aminoácidos, 10 formam uma estrutura cíclica devido as ligações éster entre o amino terminal de L-cinurenina e o grupo hidroxila da treonina (Cottagnoud, 2008; Beiras-Fernandez et al ., 2010). Além disso a daptomicina também
consiste no primeiro antibiótico lipopeptídico cíclico aprovado na Europa para infecções graves de pele e tecidos moles bem como, endocardites e bacteremias causadas por S. aureus (Beiras-Fernandez et al ., 2010).
A mesma dose 3x ao dia pode ser usada para o tratamento das infecções graves de pele associadas a Est reptococos (S. pyogenes, S. agalactiae e S. dysgalacti ae) e E. faecali s (Alder, 2005). Este lipopeptídeo contém uma
cadeia carbônica C10 que é produzida pela adição de ácido decanóico no meio de cultura durante a fermentação (Cottagnoud, 2008).
2.1.1.3. Polipeptídeos
Esta classe de antibióticos abrange a bacitracina, polimixina B e polimixina E, conhecida como colistina, que se diferenciam pelas cadeias laterais, como mostrado na Figura 7.
A bacitracina consiste em um peptídeo produzido por via não ribossomal, que contém anéis heterocíclicos, incorporados pelo domínio de ciclização (Cy) durante a sua biossíntese. Sintetizado por Baci llus licheni formis e algumas isolados de Bacillus subtili s como parte do
metabolismo secundário de proteção contra outras bactérias (Duerfahrt et al .,
2004). Apresenta atividade semelhante à da penicilina, sendo mais ativa contra micro-organismos Gram-positivos, incluindo estafilococos produtores
de β-lactamase, porém é comumente utilizada como uma pomada antibiótica
O mecanismo de ação envolve a ligação undecaprenyl pirofosfato e, o lipídeo carreador responsável pela translocação dos precursores da parede celular oriundos do citosol em direção à superfície, inibindo assim, a formação da parede celular (Hiron et al., 2011). Cabe ressaltar que, o
desenvolvimento de resistência em Bacil lus subitilis é decorente do sistema
de transporte ABC e uma membrana histidina quinase (HK), que atua como um sinalizador (Hiron et al., 2011). A histidina quinase pertence à subfamília
intramembrana quinase (IMSK), que em geral estão envolvidas na resposta ao estresse celular e são caracterizadas por um domínio amino-terminal composto por duas hélices transmembrana separadas por alguns aminoácidos (Hiron et al., 2011).
Segundo Li et al (2009) o transportador ABC, Spr0812/0813, foi necessário para desenvolvimento de resistência intrínseca à bacitracina, mas uma superexpressão de uma permease Spr0813 mutante resultou surpreendentemente numa susceptibilidade reduzida a vancomicina, um antibiótico de ácido tetramérico (2,4-pirrolidina). A bacitracina possui graves efeitos tóxicos sobre o rim e, por conseguinte, é adotada somente para uso tópico em infecções da boca, nariz, olhos e pele (Majchrzykiewicz et al .,
2010).
As polimixinas constituem uma família de decapeptídeos descobertos em 1947 e originalmente produzidos por Bacill us pol ymyxa e Bacillus coli stinus (Saar-Dover et al ., 2012). De um modo geral, esta classe de
antibióticos apresenta em sua estrutura uma cadeia de ácidos graxos na porção N-terminal, além de resíduos de ácido 2,4-diaminobutírico conferindo assim, o caráter anfipático e catiônico (Fernandez-Reyes et al., 2009). As
polimixinas apresentam amplo espectro contra bactérias Gram negativas, E. coli , Klebsiella sp., Acinetobact er sp,, Enterobact er sp., e P. aeruginosa
(Lin, X. M. et al ., 2010b). De um modo geral, atuam causando uma ruptura da
por sua vez, acarreta em morte celular (Fernandez-Reyes et al ., 2009; Sun et al., 2009).
A família das polimixinas compreendem 5 compostos químicos diferentes (polimixinas A, B, C, D e E), contudo, são empregadas em uso clínico somente a polimixina B e E, também conhecida como colistina (Sun et al., 2009). A colistina (polimixina E) surgiu em 1949 como um produto da
fermentação de Baci llus colisti nus, porém em virtude da alta incidência de
neurotoxicidade e nefrotoxicidade a sua utilização foi suspensa na década de 70 e 80 (Sun et al ., 2009; Lin, K. H. et al., 2010). Entretanto, com o
surgimento de bactérias Gram-negativas multiresistentes, em especial A. baumannii o agente etiológico da meningite, bem como, a necessidade do
desenvolvimento de novos antibióticos para o tratamento de infecções causadas por P. aeruginosa em pacientes com fibrose cística houve uma maior
utilização desta droga (Sun et al., 2009). Cabe ressaltar que, apesar da
colistina ter sido empregada com maior frequência nesses últimos 20 anos, a sua administração deve ser cautelosa, uma vez que, a nefrotoxicidade ocorre em 14.3 a 24% dos pacientes tratados com colistina nas Unidades de Terapia Intensiva (UTI). Além disso, ressalva-se que, sepses, hipotensão e o uso de outros agentes nefrotóxicos podem contribuir para um agravamento da função renal (Fulnecky et al ., 2005; Gounden et al., 2009). Apesar da toxicidade das
polimixinas, vários estudos respaldam o uso clínico da colistina quando empregada em associação com outros antibióticos. Estudos “in vit ro” têm
demonstrado sinergismo entre polimixina B associada à azitromicina, rifampicina ou imipenem contra A. baumannii e colistina associada à
rifampicina contra P. aerugi nosa (Saugar et al ., 2006). Isto por sua vez,
ocorre por que as polimixinas podem atuar aumentando a permeabilidade da membrana externa da parede celular bacteriana e permitir a passagem da outra droga para o interior da célula, executando, assim, efeito antimicrobiano acumulativo (Mitsugui et al ., 2011).
plasmático afim de, averiguar as concentrações plasmáticas que podem acarretar em uma nefrotoxicidade (Gounden et al ., 2009). Atualmente, são
empregadas comercialmente duas formas de colistina, que incluem o sulfato de colistina para uso oral e tópico e metanosulfato de colistina para uso parenteral (Peleg et al., 2008). O metanosulfato de colistina é um pro-fármaco
convertido em colistina através de uma reação de hidrólise. Entretanto, ressalva-se que a inalação de metanosulfato de colistina não é aprovado pelo FDA, uma vez que, sua forma bioativa, pode ser tóxica em alguns órgãos (Peleg et al., 2008). Além disso, tem se mostrado a eficácia do uso
intravenoso de metanosulfato de colistina em pacientes com infecções por diferentes tipos de bactéria Gram-negativa, principalmente, as infecções causadas por isolados multiresistentes de P. aerugi nosa e A. baumannii, bem
como Kl ebsi ell a pneumoniae e E. cloacae (Sun et al ., 2009).
2.2. RESISTÊNCIA BACTERIANA
A “era de ouro” dos antibióticos não durou muito tempo, e um novo obstáculo surgiu, sendo este a resistência bacteriana. Esse fenômeno, causado pelo uso maciço e inadequado dos antibióticos culminou na seleção de isolados resistentes a múltiplas drogas, deixando os pacientes sem opções de tratamento e vulneráveis a doenças perigosas (Nisnevitch, 2013).
O primeiro relato de isolado resistente a antibióticos foi descrito em 1930, onde se verificou a existência de Staphyl ococcus aureus resistentes à
penicilina (Sykes, 2010). Posteriormente, em 1940, relatou-se a N eisseri a gonorrhoeae resistente à penicilina, em 1970, um isolado de Haemophi lus influenzae produtor de β-lactamases, e, por conseguinte, S. aureus resistentes
a meticilina. Ao final da década de 70 e início da década de 80, inúmeras espécies foram descritas como resistentes aos antibióticos incluindo
Gram-negativas resistentes tais como Shigella sp., Salmonella sp., Vibri o cholerae, E. coli, K. pneumoniae, A. baumanii, P. aeruginosa, entre outras
(Allen et al ., 2010; Davies e Davies, 2010; Sykes, 2010). Através da pesquisa
realizada por Mandigan em 2010, foi constatado que em 60 anos inúmeros fungos e bactérias desenvolveram resistência aos antibióticos, como observado na Figura 8.
Figura 8: Evolução da resistência microbiana aos antibióticos convencionais em um período de 1950 a 2010. Asteriscos indicam que alguns isolados destes patógenos atualmente não podem ser tratadas com os fármacos antimicrobianos. Legenda: (Verde) Fungos. (Vermelho) Gramnegativo. (Roxo) Grampositivo. (Azul) Gram -positivo/ ácido resistente.
Candida albicans*
Acinetobacer sp
Enterococcus faecium* Streptococcus pneumoniae Mycobacterium tuberculosis*
Haemophilus ducreyi
Salmonella typhi
Haemophilus influenzae
Neisseria gonorrhoeae
Pseudomonas aeruginosa* Salmonella sp
Shigella dysenteriae
Shigella sp
Staphylococcus aureus*
Fonte: (Mandigan et al., 2010)
Atualmente, tem sido descrito que várias bactérias como as
Pneumococcus sp. Staphyl ococcus sp. e Streptococcus sp., desenvolveram
resistência aos antibióticos e ainda, mais de 70 % delas causam infecção. Vale ressaltar que, apesar dos graves quadros de infecção proeminentes dessas bactérias Gram-positivas, o maior alerta de saúde pública está em relação aos membros da família Enterobacteriaceae, pois apresentam
resistência a ampicilina (β-lactâmicos), trimetropin, sulfametoxazol,
tetraciclina, cloranfenicol, vancomicina e ácido nalidíxico (Mosquito et al.,
2011).
Desta forma, observa-se que a frequência de infecções causadas principalmente por bactérias Gram-negativas tem se elevado rapidamente e as opções terapêuticas estão se esgotando pois, medicamentos não vêm sendo desenvolvidos em tempo suficiente para combatê-las (Chopra et al., 2008).
Entre 1930 e 1962 mais de 20 classes de antibióticos foram comercializadas, porém na última década, somente 2 novas classes chegaram ao mercado (Lewis, 2013). Dentre as razões que culminam para a diminuição da produção de novos antibióticos encontram-se o alto custo de produção juntamente com a rapidez com que os microrganismos adquirem resistência (Akhtar et al.,
2012) De um modo geral, em cerca de dois anos um antibiótico comercializado torna-se ineficaz devido ao surgimento de resistência microbiana tornando inviável a sua produção pela indústria farmacêutica, uma vez que outros grupos de medicamentos, como anti-inflamatórios e cardiovasculares permanecem por tempo maior no mercado (Coates et al.,
2011).
com tamanho grande e/ou hidrofóbicos, como alguns antibióticos (Vidaillac et al., 2012). Tal fato é extremamente preocupante, pois embora análogos das
classes de antibióticos existentes estejam sendo comercializados, eles não têm sido suficientes para conter a resistência aos antibióticos, principalmente entre bactérias Gram-negativas (Marr et al., 2006).
A resistência aos antibióticos em enterobactérias está frequentemente relacionada com as regiões de multirresistência (MRR), constituída de elementos móveis tais como, IS, cassete de genes/integrons, transposons de Tn3 e familia Tn5053 que ao se recombinarem promovem os mecanismos diferenciais de resistência (Partridge, 2011). De modo geral, a resistência bacteriana pode ser inata ou adquirida em relação a um ou mais antibióticos. A resistência inata corresponde a uma característica da espécie bacteriana, que “naturalmente” cria uma barreira de permeabilidade contra o antibiótico, como por exemplo, a presença de membrana externa nas bactérias Gram-negativas. A resistência adquirida pode ocorrer por mutações espontâneas, recombinação ou por transferência horizontal de genes de um micro-organismo para o outro através de plasmídeos (conjugação ou transformação), transposons (conjugação), integrons ou bacteriófagos (tradução) (Giedraitiene et al., 2011). È importante salientar que a
resistência pode ser adquirida com concentrações subletais promovendo alterações genéticas em diferentes vias, que resultam no aumento de espécies reativas de oxigênio, e como consequência, danos em componentes celulares como, proteínas, lipídios e DNA. Esses danos podem levar a erros no DNA que se não forem reparados podem levar a mutações permanentes (Kohanski et al., 2010; Rodriguez-Rojas et al., 2013).
As bactérias Gram-positivas podem desenvolver resistência através do efluxo de antibióticos utilizando uma coleção de proteínas associadas à membrana que funcionam como bomba de efluxo ou ainda, através da síntese
de enzimas como, por exemplo, as β-lactamases, que rompem o anel lactâmico
e causam mutações em PBPs (proteínas de ligação a penicilinas) conferindo
Além disso, em Gram-positivas também ocorrem alterações no sítio de ligação que conferem resistência aos macrolídeos ou então, na reprogramação da via de biossíntese da parede celular, como na resistência aos antibióticos glicopeptídicos (Aktas et al., 2007; Cetin et al., 2010).
Em bactérias Gram-negativas, são descritos as β-lactamases e PBPs que
conferem resistência aos β-lactâmicos e apresentam ainda uma ação seletiva
de porinas, que dificultam a entrada do fármaco, e a presença de bombas de efluxo, que bombeiam a antibiótico para fora da célula bacteriana (Llarrull et al., 2010). O fenótipo de resistência pode ser perceptível em função da
Tabela 1 –Mecanismos de resistência bacteriana aos principais antibióticos utilizados na prática clínica.
Família de
antibióticos Mecanismo de ação Mecanismos de resistência Genes relacionados
β- Lactâmicos Interfere na síntese de peptideoglicano, componente
necessário para formação da parede bacteriana.
β- lactamases: enzimas que se caracterizam por hidrolisar a ligação amida
do núcleo β- lactâmico, inativando dessa maneira
o antibiótico.
Genes que codificam β-
lactamases: blaTEM, blaSHV, blaCARB, blaOXA, blaCTX-M e
blaGES.
Quinolonas Inibe a ação das Topoisomerases e da DNA
girase bacterianas
Mutações pontuais que resultam na troca de aminoácidos na região
alvo do antibiótico.
Sistemas de expulsão do antibiótico.
Presença de genes plasmidiais de resistência
antibiótica.
Mutações em gyrA (gene que codifica uma subunidade
da DNA girase) e parC
(gene que codifica uma subunidade da topoisomerase IV). AcrAB-like (sistemas presentes en diferentes
enterobacterias) Família de genes qnr (A, B,
C, D S) que codificam proteínas Qnr que impedem
estericamente a união do antibiótico ao sítio ativo.
Gene que codifica a variante cr da acetiltransferase 6’ (AAC (6’)-Ib-cr), capaz de
acetilar fluoroquinolonas.
Tetraciclinas Unem-se ao ribossomo bacteriano, inibindo a síntese
de proteínas.
Presença de bombas de efluxo específicas para
tetraciclinas.
Genes tetA y tetB que
codificam sistemas de efluxo.
Cloranfenicol Inhibidor da biossíntese de proteínas.
Inativação enzimática por acetilação. Exportadores específicos
de Cloranfenicol.
Gene cat que codifica
aenzima cloranfenicol acetiltranferase. Genes foR e cmlA.
Trimetoprima/
Sulfametoxazol Inibe a síntese da enzima diidropteroato sintase (sulfametoxazol) e da enzima
diidrofolato redutase (trimetoprima), as quais são enzimas necessárias na rota
do ácido fólico.
Presença de genes que codificam formas mutantes da enzima.
Genes sul1 e sul2
(sulfametoxazol) e genes dfr
(trimetoprima).
2.3. RESISTÊNCIA À Β-LACTÂMICOS
No contexto hospitalar, as enzimas mais prevalentes pertencem ao diversificado grupo das beta-lactamases. Usualmente, as β-lactamases são classificações de duas maneiras distintas: de acordo com a classificação molecular, proposta por Ambler (1980) e pela classificação funcional proposta por Bush (2010). A classificação molecular é baseada na sequência
de aminoácido e divide as β-lactamases por homologia. Assim, as classes A,
C e D compreendem as enzimas que utilizam serina para a hidrólise do anel β
-lactâmico (serino-β-lactamases); enquanto a classe B correspondem as metaloenzimas (metalo-β-lactamases) que requerem íons zinco divalentes para a hidrólise do substrato.
A classificação funcional se baseia nas propriedades funcionais das enzimas, levando em consideração o perfil do substrato e do inibidor, buscando agrupar as enzimas de modo que elas possam ser correlacionadas com seu fenótipo em isolados clínicos (Harada et al ., 2008; Bush, 2010) O
sistema de classificação funcional inclui: grupo 1 (classe C) cefalosporinases; grupo 2 (classes A e D) largo-espectro (broad-spect rum), inibidor-resistente, e β-lactamases de espectro estendido (BLEEs ou ESBL, do inglês e xtended-spectrum β-lactamases) e serino-carbapenemases; enquanto no grupo 3 estão
as metalo-β-lactamases.
Até o momento, as β-lactamases de interesse clínico e que apresentam
espectro de ação distinto sobre os β-lactâmicos são quatro: β-lactamase de
espectro estendido (ESBL), K. pneumoni ae carbapenemase (KPC), metalo β
-lactamase (MBL) e β-lactamase classe C (AmpC). De um modo geral, os genes
que codificam essas enzimas podem ser encontrados no cromossomo bacteriano ou no plasmídeo (Mendes et al., 2007).
As β-lactamases do tipo AmpC são enzimas codificadas pelo DNA
carbapenêmicos, não são inibidas por inibidores de β-lactamases tais como (sulbactam, tazobactam e ácido clavulânico) e são encontradas em uma grande variedade de bactérias Gram-negativas, sendo mais prevalentes no grupo AMPCES (Aeromonas spp., Morganell a morganii, Prot eus spp., Cit robact er freundii, Ent erobact er spp. e Serratia spp.) (Philippon et al ., 2002).
BLEEs (β-lactamases de espectro estendido) são enzimas capazes de
hidrolisar todos os agentes β-lactâmicos, excluindo-se os carbapenêmicos, são
inibidas por inibidores de β-lactamases, o que as deferenciam das β
-lactamases do tipo AmpC, são codificadas por genes plasmidiais, conferindo
uma maior facilidade de disseminação entre diferentes espécies bacterianas (Livermore et al., 2011).
Carbapenemases são enzimas capazes de hidrolisar todos os agentes β
-lactâmicos incluindo os carbapenems, pode ser codificado por elementos móveis ou por genes presentes no cromossomo bacteriano. São divididas em dois grupos: as serina carbapenemases, enzimas capazes de hidrolisar a penicilina, cefalosporinas e as carbapenemases de primeira geração, não hidrolisam as cefalosporinas de terceira geração e o aztreonam (Poirel et al .,
2011).
As metalo-β-lactamases são enzimas que degradam todos os β -lactâmicos, exceto, o monobactâmico, aztreonam. Essas enzimas caracterizam-se por necessitarem de dois íons divalentes, usualmente zinco, como co-fator para atividade catalítica, por terem a mesma estrutura tridimensional e por apresentarem resíduos conservados, os quais são responsáveis pela interação da enzima com cátions divalentes (Mendes et al .,
Além das β-lactamases descritas, pode-se citar a AmpC, que é capaz de
hidrolisar penicilinas, monobactâmicos e cefalosporinas até terceira geração. Nas últimas duas décadas, a transferência de plasmídeos tem sido reportada com frequência (Walsh et al., 2005).
2.4. RESISTÊNCIA AOS GLICOPEPTÍDEOS
Dentre as alterações presentes nas isolados resistentes à vancomicina encontram-se mudanças no metabolismo energético glicolítico, bem como, distúrbios nos níveis de mRNA que codificam enzimas da via biossintética de purina, composto presente na parede celular, acarretando em alterações morfológicas na parede celular, que incluem o espessamento da parede celular, substituição de gD-Gln com D-Glu na muropeptídeo, esterificação de ácidos teicóicos com D-Ala alterando a carga negativa do ácido teicóico presente no peptideoglicano bem como, um crescimento mais lento destas isolados (Pieper et al., 2006). De acordo com Cui et al (2009) o
espessamento da parede celular deve-se ao aumento da concentração de glutamina sintetase que, por sua vez, catalisa a síntese de glutamina, que serve como doador de NH4 + para a reação de amidação de iso-D-glutamato a partir do muropeptídeo (MurP-NA).
Segundo Drummelsmith et al (2007) a isolado de S. aureus resistente a
vancomicina (Vn) em geral, apresentam MIC variantes de 4 a 16 mg.L- 1 . Porém, estes níveis de resistência não estão correlacionados à presença do gene van, como visto em Enterococcos, sugerindo que a resistência pode ter ocorrido por inúmeras mutações, dada a complexidade estrutural e regulatória associada com à parede celular e a variedade de alterações fenotípicas associadas à resistência. Espécies de Enterococcus (Enterococcus f aecal is e Ent erococcus faecium) também podem causar infecções graves e fatais,
precursores de parede celular que não se ligam a glicopeptídeos, devido a aquisição de um grupo de genes associados à plasmídeos denominados genes vanA ou vanB (Leclercq e Courvalin, 2002).
2.5. RESISTÊNCIA AOS POLIPEPTIDEOS
A resistência a colistina pode ocorrer em decorrência da alteração do lipídeo A ou por bomba de efluxo. De acordo com Burtnick et al (1999) as bactérias Gram-negativas resistentes a polimixina modificam a estrutura do lipídio A ao utilizar o açúcar L-Ara4N na constituição deste lipopolissacrídeo. Além disso, a modificação do lipopolissacarídeo está correlacionada a indução de dois componentes de sistemas de regulação, PhoP-PhoQ e pmrA-PmrB (Mcphee et al., 2003)como mostrado em P. aeruginosa. Cabe ressaltar que, estudos recentes demonstram que, o operon
pmrCAB atua como mediador de resistências à colistina em A. baumannii
através da modificação da fosfoetalonamina presente no lipídios A (Arroyo et al., 2011; Beceiro et al., 2011).
De um modo geral, o mecanismo de resistência a colistina ocorre em virtude da adição de unidades de ácido palmítico causando assim, um aumento da hidrofobicidade dos LPS, ou por esterificação dos grupos fosfato no lipídeo A, causando uma diminuição da carga negativa acarretando em alteração da membrana celular e consequentemente distúrbio osmótico (Fernandez-Reyes et al., 2009). Além disso, as bactérias podem empregar
sistema de efluxo como mostrado por Padilla et al (2010) em K. pneumonia
resistente à polimixina B ocorre uma super expressão do sistema AcrAB, uma bomba de efluxo comumente associado à resistência a múltiplas drogas.
mais aprofundado sobre a base molecular dos mecanismos de resistência para que, a partir da determinação do modo de infecção usado por essas bactérias, possam-se desenvolver novas estratégias para o controle bacteriano.
2.6. INFECÇÕES RELACIONADAS À ASSISTÊNCIA A SAÚDE (IRAS)
As infecções relacionadas à assistência a saúde (IrAS) são caracterizadas por infecções adquiridas em unidades hospitalares ou em qualquer outra instituição de assistência a saúde, como por exemplo enfermarias, ambulatórios e consultórios, podendo ser diagnosticada a partir de 72 h após a internação. O termo IrAs também está relacionado às infecções que são manifestadas durante o período de internação ou após a alta do paciente (Côrrea, 2008). Cabe ressaltar que, as infecções em âmbito hospitalar podem ser adquiridas não apenas por pacientes, que apresentam maior susceptibilidade, mas também, embora menos frequentemente, por visitantes e funcionários do próprio hospital (Lipsitch et al ., 2000).
As IrAS derivam da interação de vários fatores como a presença de micro-organismos no ambiente hospitalar, a presença de hospedeiros imunodeprimidos e a cadeia de transmissão existente no hospital. A presença de um dos fatores isolados aparentemente não é suficiente para que a infecção ocorra, porém a interação dos três fatores representa riscos para a aquisição do quadro infeccioso (Tortora et al., 2012).
Ainda que muitos esforços sejam realizados para eliminar ou impedir o crescimento de micro-organismos em hospitais, vale destacar que o ambiente hospitalar é um reservatório importante de uma variedade de patógenos (Tortora et al ., 2012). Em Unidades de Terapia Intensiva (UTI), por exemplo,
Essas infecções ocorrem mundialmente e acometem países desenvolvidos, em desenvolvimento e subdesenvolvidos (Padoveze et al .,
2010). De acordo com o Cent ers for Disease Cont rol and Preventi on dos
Estados Unidos, estima-se que as IrAS representam cerca de 1,7 milhões dos casos de infecções, sendo que 99.000 mortes ocorrem em decorrência dessas infecções todos os anos somente nos Estados Unidos (CDC, 2010). Destas infecções, 32% referem-se às infecções do trato urinário, 22% são infecções de sítio cirúrgico, 15% são relativas à pneumonia (infecção pulmonar) e 14% são infecções da corrente sanguínea (CDC, 2010). Algumas espécies patogênicas causadoras destas infecções estão descritas simplificadamente na Tabela 2 onde é possível observar os patógenos e sua ocorrência na corrente sanguínea, pulmões e/ou trato urinário isolados em UTI’s de hospitais americanos (Mandigan et al., 2010).
De forma similar em hospitais irlandeses averigou-se que, as IrAS mais comuns foram infecções do trato respiratório (2,65%) e infecções de sítio cirúrgico (1,7%) (O'neill et al ., 2010). Em países em desenvolvimento
inúmeros casos de óbito surgem em decorrência deste cenário, o que resulta em um aumento dos custos de saúde pública e ressalva a necessidade de uma maior atenção aos procedimentos laboratoriais (Lipsitch et al ., 2000; Matlow