Energie, force et temps
Quelques dispositifs
Dans le cas de la chromatine, son analité avec les surfaces (interactions non spécifiques) limite l'utilisation de l'AFM comme détecteur de force [177]. Le dispositif Bre décrit précédemment (Fig. 1.21 (b)) permet la rotation de la micropipette autour de son axe.
Comportement élastique de l'ADN : modèles
Le modèle qui décrit la molécule d'ADN de cette manière n'est pas le modèle FJC, mais le modèle Worm Like Chain, qui est expliqué ci-dessous. Modèle Kratky-Porod. La molécule d'ADN n'est pas un polymère parfaitement flexible décrit par le modèle FJC.
Comportement en torsion
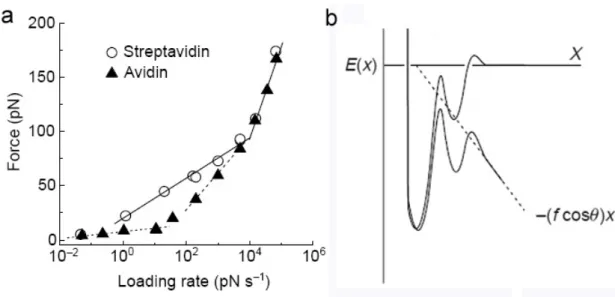
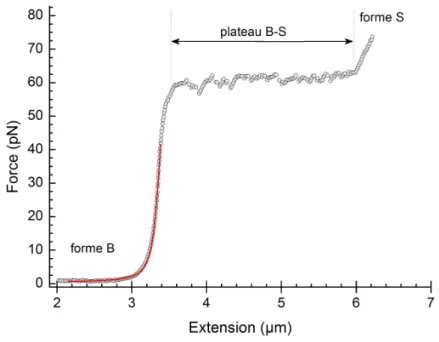
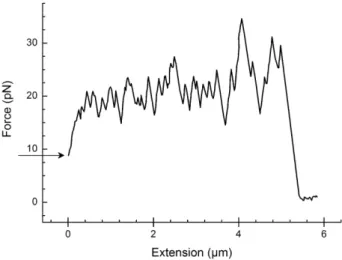
L'allongement d'une molécule d'ADN nécessite d'autant plus d'énergie que l'allongement est proche de la courbe. 132] ont noté un comportement frappant : lorsque l'extension dépasse cette longueur courbe, d'un coup, pour une force d'environ 60 pN, la molécule s'étire de 70 % par rapport à sa forme classique d'ADN-B.
Etirement d'une bre de chromatine
Divers types de chromatine étudiés
Etirements sur les substrats chromatinien
Piège optique
On mesure les déplacements de la bille piégée dans le plan perpendiculaire à l'axe optique (OXY). Comme décrit par la figure (2.8), nous projetons l'ombre de la balle piégée sur la surface du PSD. Les deux extrémités de la molécule d’ADN sont désormais marquées avec le même antigène.
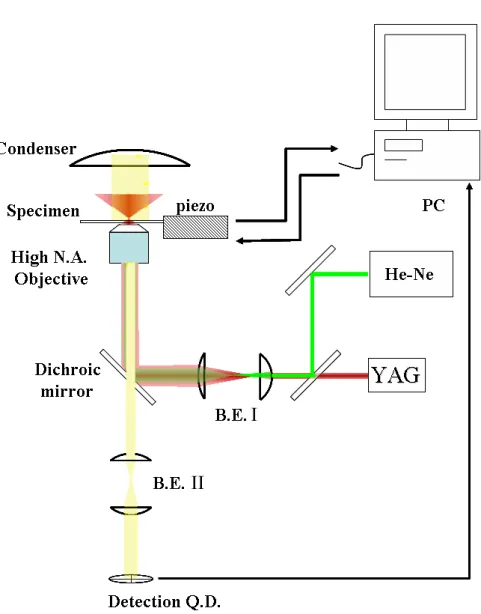
Cellule d'expérimentation
Détection et sensibilité
Calibration
Force de Stokes
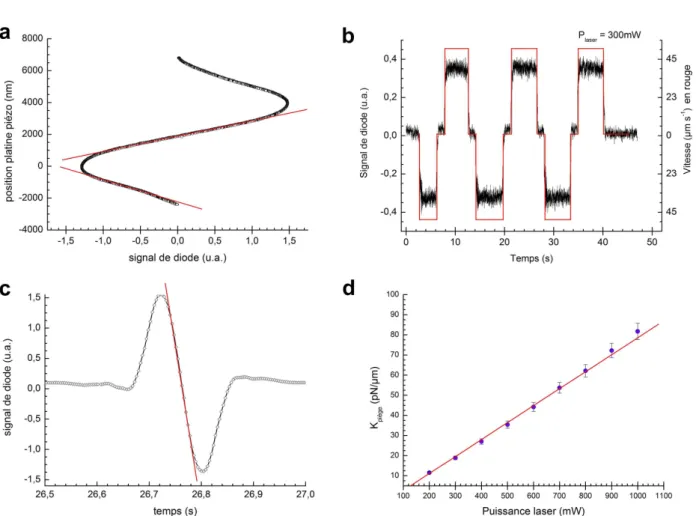
La force la plus simple à générer expérimentalement est celle d'un ux hydrodynamique laminaire sur la balle : la force de Stokes, FStokes = 6πηrv, où r est le rayon de la balle, η la viscosité dynamique du milieu et v la vitesse d'écoulement. La sphère est de taille micrométrique, la vitesse du courant généré est d'environ 100 µm s−1, le tout en milieu aqueux. On utilise donc un écoulement hydrodynamique contrôlé à la vitesse v, autour de la sphère piégée, de diamètre d, au moyen de l'étage motorisé et contrôlé via un générateur de fonctions.
Analyse des uctuations
Deux zones où la relation signal diode-déplacement bille est linéaire peuvent être exploitées (en rouge). Après conversion signal-déplacement du signal de diode (voir a), la pente nous renseigne sur la vitesse de l'étage motorisé et donc sur la vitesse de l'écoulement hydrodynamique ressenti pendant b. Ces dérives lentes (de l'ordre de 100 nm/min) tendent à élargir les distributions et conduisent à une sous-estimation de la constante de rigidité.
Vers la pince magnéto-optique
L'intensité du champ magnétique est modifiée en modifiant le courant circulant dans les bobines. Champ magnétique généré Nous avons mesuré l'amplitude du champ magnétique généré par les bobines sur l'échantillon avec une sonde Hall. Si nous faisons tourner le champ magnétique dans un plan perpendiculaire à la surface du verre, alors ces types de billes suivent le champ et tournent avec lui (Figure 2.14).

Choix et préparation des billes
Ensuite il faut prendre en compte la composition de la surface des billes et le tampon utilisé. Pour éviter la liaison des deux extrémités de l’ADN à la même bille, nous avons modifié les deux extrémités de la molécule avec deux antigènes différents. Tout au long des préparations et lors du stockage, nous utilisons des tubes en verre afin de minimiser les pertes de billes dues au collage aux parois.
Préparations biochimiques
Préparation de l'ADN
Le choix de l'enzyme de restriction (ici EcoR1) dépend de la séquence d'ADN ainsi que de l'endroit choisi pour couper et marquer une molécule d'ADN, ici initialement supposée circulaire (plasmide). Nous complétons ensuite les extrémités simple brin à l'aide de l'ADN polymérase II (Fermentas) (5' → 3') avec des nucléotides marqués avec des antigènes correspondant aux anticorps à la surface des billes, typiquement dATP-biotine. (Gibco) ou dUTP-digoxygénine (Roche). Répétition Recommencez le protocole précédent en modifiant les dNTP marqués ; utilisez la dUTP digoxigénine (Roche).
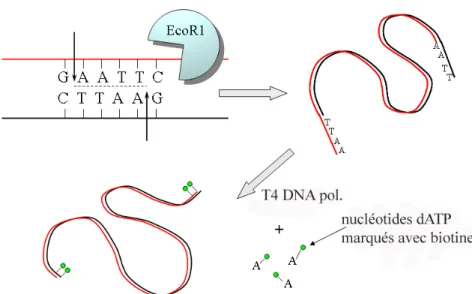
Chromatine reconstituée
100 ng d'ADN libre radiomarqué et une quantité égale d'ADN impliqué dans un cerveau à chromatine reconstituée sont digérés pendant 2 minutes dans une solution de Ca2+ 1 mM et 0,03 et 0,1 unité de Mnase, respectivement. Cependant, s'il est impossible de reconstituer un cerveau chromatinien en mélangeant l'ADN et les histones directement dans un tampon physiologique, une telle reconstitution est possible en utilisant des protéines chaperonnes des histones telles que NAP1 ou la nucléoplasmine et N. Avant que les histones et l'ADN ne soient mélangés pour former une hypothétique chromatine. , nous incubons les histones seules avec de la nucléoplasmine pendant 30 min dans du tampon nal 1x TE, 150 mM NaCl.
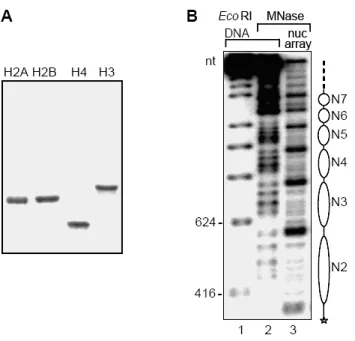
Chromatine native
Nous avons vérifié que les profils d'élongation de l'ADN ou de la chromatine ne différaient pas selon le mode d'élongation. Face à une telle reproductibilité, nous nous sommes intéressés à l'effet de la séquence d'ADN sur ces proles [119]. Comme pour l’extrait de Xenopus Laevis, chaque spicule est caractérisé par la libération d’ADN nucléosomal.
Déroulement d'une expérience type
Incubation
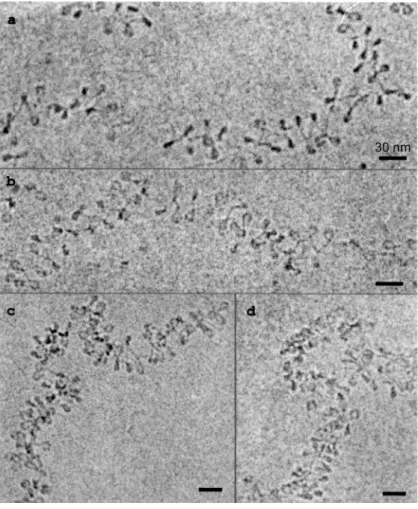
Insérer un histogramme de la répartition des forces de rupture lors de l'étirement de cette chromatinbre et ajuster avec une courbe de Gauss (courbe en bleu). Nous avons essayé de confirmer nos résultats par une expérience de biochimie. Nous avons montré qu'à de faibles concentrations de chromatine (~ 1 nM, conditions diluées), les dimères H2A-H2B sont libérés par le NCP.
Accrochage proprement dit
Etirement
Si la réponse à l’instruction du piézoboard est rapide ; de l'ordre de la ms, les contraintes subies par la molécule étirée à un tel taux de charge sont négligeables tant que l'amplitude du pas ne dépasse pas l'amplitude des oscillations browniennes de la sphère. Mais l'amplitude des pas doit rester nettement inférieure à une longueur de l'ADN nucléosomal pour pouvoir détecter le signal qui nous intéresse : le signal de cassure de nucléosomes individuels. Les résultats que nous mentionnons sur la transition B-S de l'ADN ont été établis en extrayant les molécules aux extrémités 3' dans des tampons physiologiques : PBS 1× ou TE 1×, 100 mM NaCl.
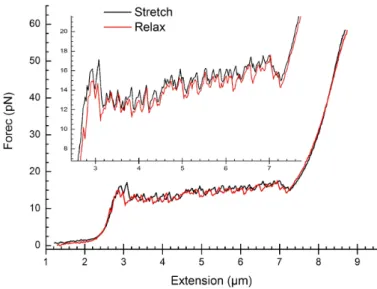
D'autre part, parce que c'est in fine sur ce substrat, en association avec les histones, que l'on sera amené à dessiner : une bonne connaissance de la réaction du substrat seul est donc également indispensable et permet de détecter des signaux confus, propres au substrat. Molécule d'ADN (généralement l'hystérésis sur le plateau B-S) avec d'autres spécifiques à l'association ADN-histone. Avec la pratique, on peut également distinguer les cas où une ou plusieurs molécules d'ADN pontent les billes : la forme du profil d'élongation est très différente, et la longueur curviligne apparente de la molécule peut varier (voir aussi [38]). On peut estimer cette longueur curviligne en mesurant la distance séparant les centres des deux boules et en soustrayant la somme des rayons des boules.
Plateau B-S
Le profil irrégulier du plateau peut s'expliquer par des changements dans la répartition des paires A-T le long de la molécule. Ces pics sont la signature de la libération d'ADN essentiellement au contact du tétramère H3-H4. Pour la chromatine reconstituée à partir d’extrait d’œuf de Xenopus Laevis, nous avons étudié l’influence du rapport quantité d’ADN/quantité de protéines sur la cinétique de formation de bre.

Prols particuliers des plateaux de transitions
Discussion
Modèle de Cluzel & coll
Modèle de Williams & coll
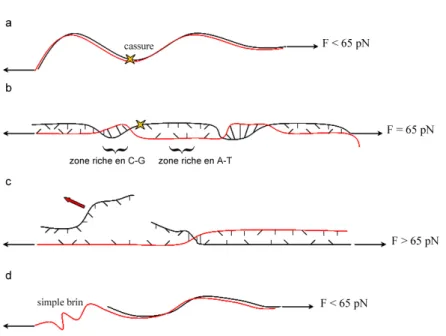
Dans ce modèle, c’est la force qui détermine la transition entre l’ADN double brin de forme B et deux brins d’ADN très proches l’un de l’autre qui sont encore localement appariés, en particulier dans les régions riches en CG. Les auteurs distinguent deux étapes dans leur modèle ; La première consiste en une transition d’équilibre au cours de laquelle les domaines d’ADN-S se nucléent puis s’étendent jusqu’à une force de 65 pN. Au-delà du plateau B-S, nous sommes confrontés à un processus hors équilibre au cours duquel les régions d’ADN encore appariées sont détruites et les deux brins se séparent, de 68 pN à plus de 150 pN, selon les taxes.
Interprétations
L'hystérésis observée lors de la relaxation est cependant plus difficile à interpréter dans le contexte d'un modèle où l'étirement n'affecte que l'arrangement interne de la molécule tandis que les bases restent appariées [163]. Après la transition, la plupart des paires de bases, y compris C-G, deviennent désappariées, conduisant à la configuration de deux polyélectrolytes chargés dans une interaction complexe [39] : répulsion des squelettes phosphate chargés négativement (1 e-par pb) et attraction entre les bases sur différentes chaînes. . ou sur le même brin. Ainsi, lorsque le squelette sucre-phosphate de la molécule d’ADN est brisé, il est possible, par un simple étirement à travers le plateau B-S, de séparer complètement la partie simple brin du reste de la molécule (Figure 3.12).
Commentaire sur la méthode : eet de la protéine MENT
Expériences
Discussion
Mais nous commencerons par discuter des problèmes liés au taux de remodelage de la chromatine avec l’extrait d’œuf de Xenopus Laevis. Une explication possible pourrait provenir de la longueur totale de l’ADN qui interagit avec une protéine de liaison. Selon les données de microscopie électronique [20] et la modélisation moléculaire [23], l'histone de liaison est localisée de manière transitoire à la sortie du NCP.
Extraits d'÷ufs de Xenopus Laevis
Contournement des problèmes dus aux impuretés de l'extrait
La double longueur, qui correspondrait à la rupture simultanée de deux nucléosomes, est intégrée dans la queue de la distribution. On remarque cependant une très nette réduction de la distance entre les deux billes au fur et à mesure de la formation de la fibre chromatinienne. La présence à la sortie du NCP du remplacement de l'histone linker dans l'extrait, la protéine B4, est une des explications envisagées.
En biologie, il est généralement admis que le temps de formation de la chromatine à partir de l'extrait d'œuf de Xenopus Laevis est long, de l'ordre d'une heure [136][43]. Chromatine native En analysant l'histogramme de la figure (4.20), nous pouvons distinguer jusqu'à quatre populations (figure 5.2).
Distributions
Cinétique de formation
Eet du tampon
Une incubation de quelques minutes dans l'extrait a ainsi pu former des nucléosomes sur notre molécule d'ADN. Dans un premier temps, nous ajustons l'extension de la molécule d'ADN entre nos deux billes pour que seuls les LDN A−Lbead−bead=L0= 300 nm soient disponibles pour former une bre à l'arrivée de l'extrait. Le premier cycle d'élongation-relaxation est réalisé dans l'extrait et est suivi d'une deuxième phase d'incubation de 3 minutes, où nous avons réduit la distance entre les deux billes par rapport au premier étirement, L0 = 1200nm.
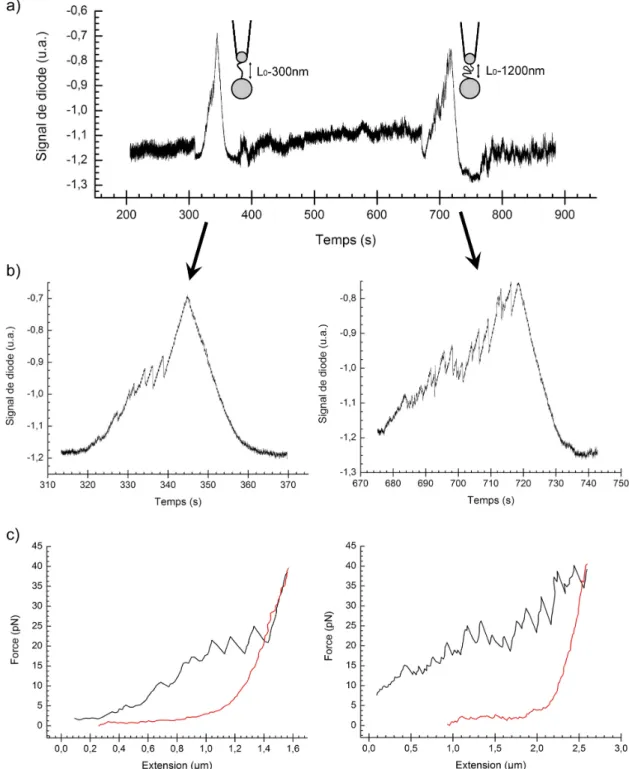
Déplétion de la protéine B4
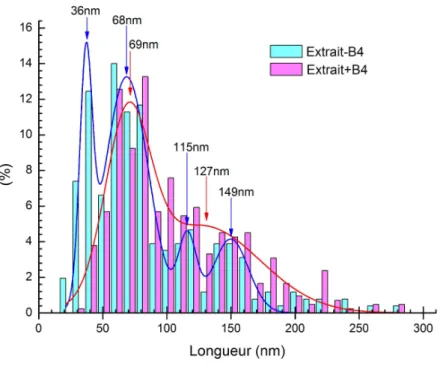
Une longueur d'environ 23,5 nm est clairement perceptible et la queue de distribution n'est pas négligeable. En raison de la symétrie du problème, le vecteur normal n¯ appartient au plan (OY Z), tandis que l'axe de la dyade coïncide toujours avec l'axe (OX). Si la force moyenne appliquée ne varie pas trop (on passe de 17 pN pour une chromatine reconstituée sur une séquence de type 5S, à une moyenne de 20 pN pour ce type de chromatine, au même taux de chargement), c'est aussi le cas pour la distribution de longueur de l'ADN libéré.
En revanche, on enregistre une diminution de la longueur de l'ADN libéré, le pic principal de la distribution passant de 68 nm (avec B4) à 60 nm (sans B4). Toujours dans ce contexte de régulation et de rôles multiples, nous pouvons discuter de la manière dont l’histone de liaison contribue à la répression de la transcription.
Chromatine reconstituée sur une séquence répétée de 5S ADN-r
Chromatine native
Conditions stabilisantes
Chromatine reconstituée sur une séquence 5S d'ADN-r
Chromatine native
Expériences de dilutions successives et analyse sur gel
Dilution de NCP
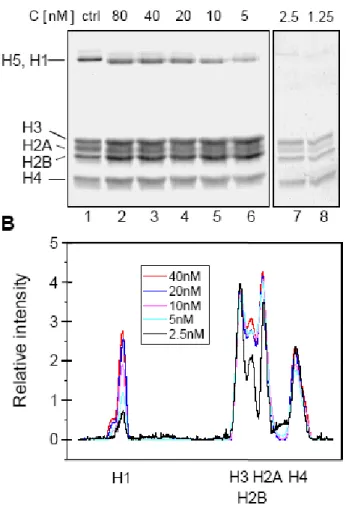
Nous avons ensuite étudié la stabilité de ces particules avec leur dilution dans des solutions de 1× TE, 10 mM NaCl et 1× PBS (≈ 160 mM Na + ). Conformément à ce qui a été établi pour la stabilité des NCP, l'effet de la dilution sur la stabilité de ces NCP est beaucoup plus prononcé pour les expériences réalisées à des concentrations d'ions modérées (PBS1x) qu'à de faibles concentrations de sels monovalents (NaCl 10 mM). A une concentration en sel (sel monovalent) de 130 mM (PBS1×), les NCP sont plus sensibles à la dilution qu'à une concentration en sel plus faible, 10 mM : les dimères H2A-H2B se dissocient du tétramère H3-H4.
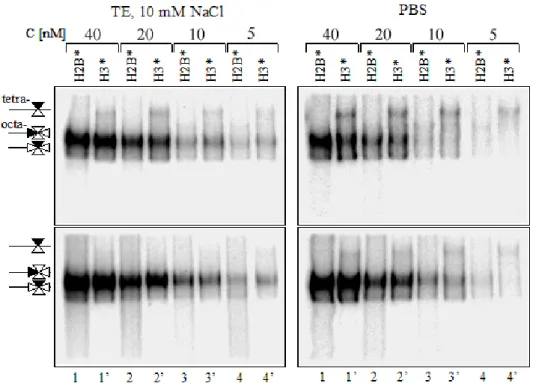
Dilution de nucléosomes
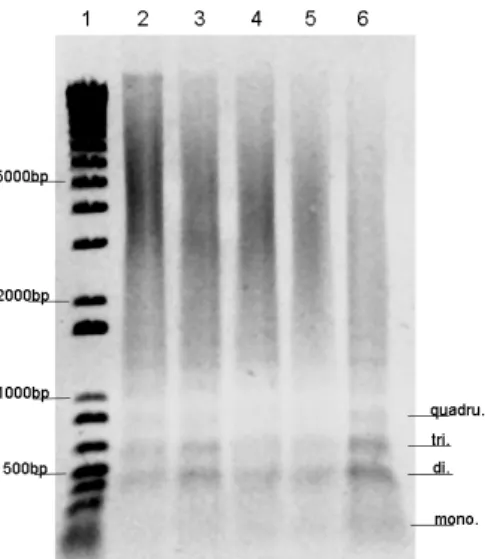
Les positions de migration des histones centrales ainsi que des histones de liaison sont indiquées à gauche.
Modèle de déroulement du nucléosome
Brève description du modèle
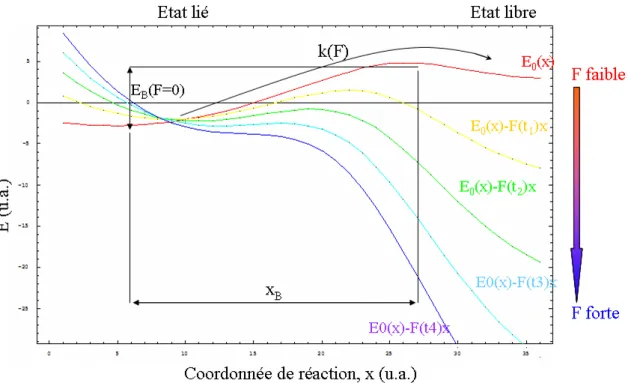
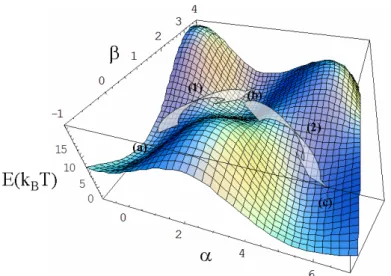
On peut alors étudier l'influence de la force et/ou de l'énergie d'adhésion de l'ADN sur les barrières énergétiques, toujours dans un but d'interprétation. Pour une plage de force d'environ 10 pN, l'ADN est décollé symétriquement de la surface de l'octamère jusqu'à ce qu'il ne reste qu'un seul tour d'ADN enroulé. Barrière énergétique Une hypothèse retenue est la présence d'une barrière énergétique à des positions ±40 pb par rapport à l'axe de la dyade NCP [27].
En particulier, l'enchevêtrement des queues N-terminales des histones entre les deux spires de l'ADN nucléosomal (H4, H2A) ou de part et d'autre (H3, H2B) joue certainement un rôle en plus de la seule énergie d'adsorption. Lorsque la répression transcriptionnelle doit être forte, la chromatine est organisée en une membrane de chromatine dense, avec la participation de l'histone de liaison au repliement (69).
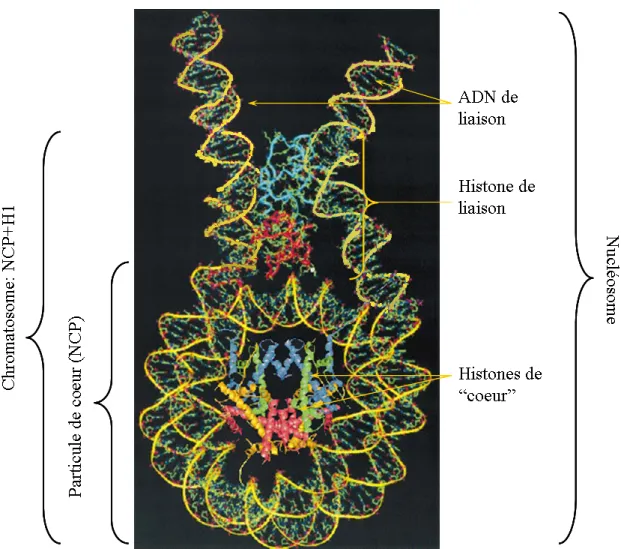
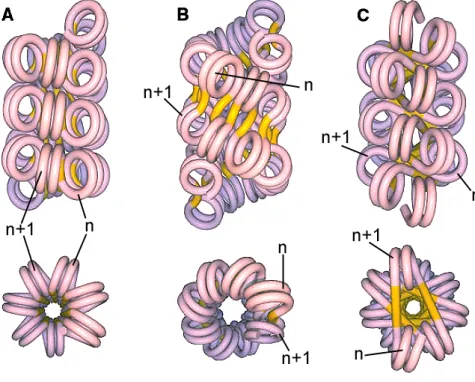
![Fig. 1.11 Structure cristallographique de la NCP. Résolution à 2.8 Åd'après [96]. Vues de dessus (A) et de prol (B)](https://thumb-eu.123doks.com/thumbv2/1bibliocom/465189.70513/26.892.230.699.606.934/fig-structure-cristallographique-ncp-résolution-åd-vues-prol.webp)