Je souhaite à l'équipe Optique et Biologie beaucoup de succès dans l'activité "ADN". En plus d'étirer les molécules d'ADN, j'ai également enseigné les bases de la physique à des jeunes étudiants de Paris 6 et j'ai beaucoup apprécié cette activité.
Localisation par les prot´eines de s´equences-cibles sur l’ADN
Phase initiale : diffusion tridimensionnelle en solution
- Diffusion des prot´eines en solution
- Estimation du taux d’association maximal par dif-
Nous allons estimer le coefficient de diffusion des protéines en solution et en déduire le taux d'association d'une protéine avec sa cible, qui résulterait de la seule diffusion tridimensionnelle, sans prendre en compte ici les interactions avec les parties non spécifiques des molécules d'ADN. Ainsi, la liaison d’une protéine à l’ADN n’est possible que si sa conformation interne et son orientation par rapport à la molécule d’ADN le permettent.
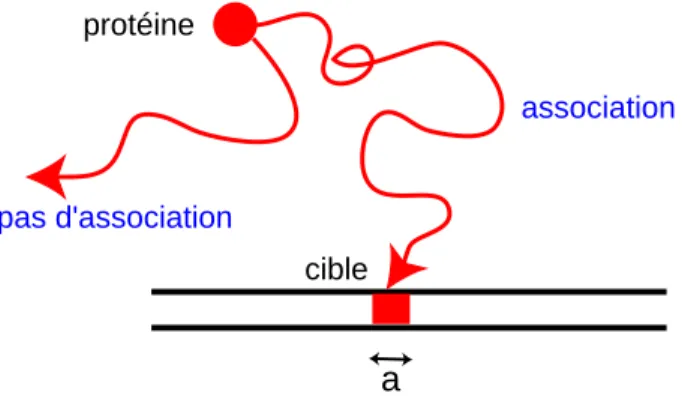
Diffusion dans le domaine d´elimit´e par une mol´ecule d’ADN 12
- Mod´elisation
Un modèle simple permet d'estimer le temps nécessaire pour explorer une molécule d'ADN de longueur L grande par rapport à la longueur de persistance de l'ADN. Dans ce modèle, une protéine alterne entre des phases de diffusion le long de l'ADN (glissement) et des phases de diffusion 3D (saut) au sein du domaine composé de molécules d'ADN.
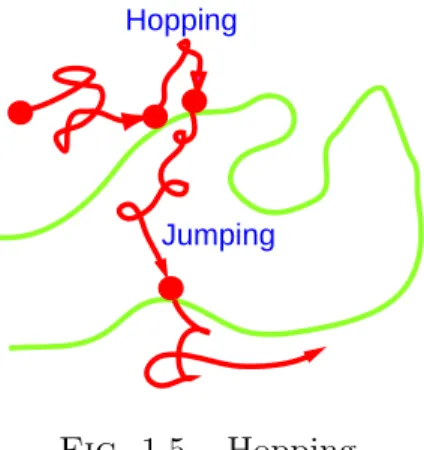
Enzymes de restriction de type II
- Syst`emes de restriction-modification chez les bact´eries
- Int´erˆet des enzymes de restriction de type II
- Mise en ´evidence exp´erimentale de l’interaction des en-
- Mise en ´evidence exp´erimentale du sliding
- Action sur des mol´ecules d’ADN contenant deux
- Influence de la pr´esence d’obstacles le long de l’ADN 24
- Pertinence de ces ´etudes pour l’interaction ADN-
- Mise en ´evidence exp´erimentale du hopping
- Action sur diff´erents substrats d’ADN
- Effet du surenroulement de l’ADN
- Quelle est l’importance respective des m´ecanismes 1D et 3D ? 27
- exp´eriences de processivit´e
1.17 – Action d'une enzyme sur une molécule d'ADN contenant deux séquences cibles proches d'une extrémité. Les expériences de processivité utilisent des molécules d'ADN qui contiennent deux séquences cibles pour l'enzyme étudiée.
![Fig. 1.15 – Vitesse de clivage de mol´ecules d’ADN par EcoRV en fonction de leur longueur [39]](https://thumb-eu.123doks.com/thumbv2/1bibliocom/465083.70417/31.892.238.655.704.873/fig-vitesse-clivage-ecules-adn-ecorv-fonction-longueur.webp)
M´ethodes non optiques
- Microscopie de force atomique
- Techniques de micromanipulation (pinces optiques
Il est donc possible d'étirer et même de tordre des molécules d'ADN (dans le cas des pinces magnétiques). Ils ne conviennent pas pour étudier le mouvement des enzymes qui diffusent le long de l’ADN sans appliquer des forces de cette amplitude ni modifier la longueur de l’ADN.
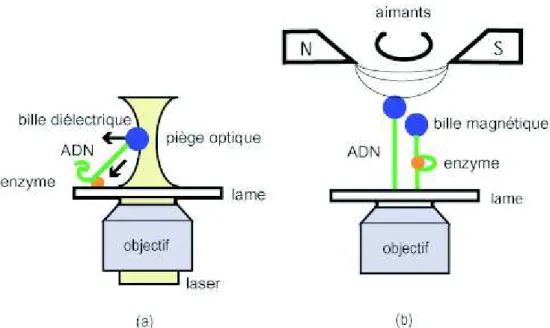
M´ethodes optiques
- Utilisation de fluorophores pour la visualisation
- Utilisation des m´ethodes optiques en vue de l’´etude
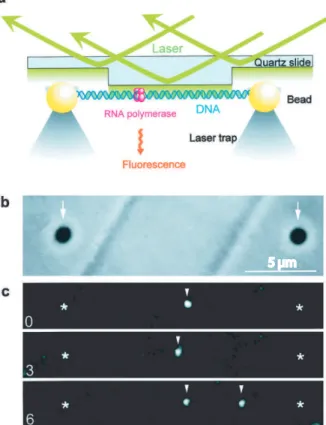
Cette expérience a permis d'observer le glissement d'ARN polymérases individuelles le long de l'ADN. Comment placer les molécules d'ADN dans une configuration optimale pour observer leur interaction.
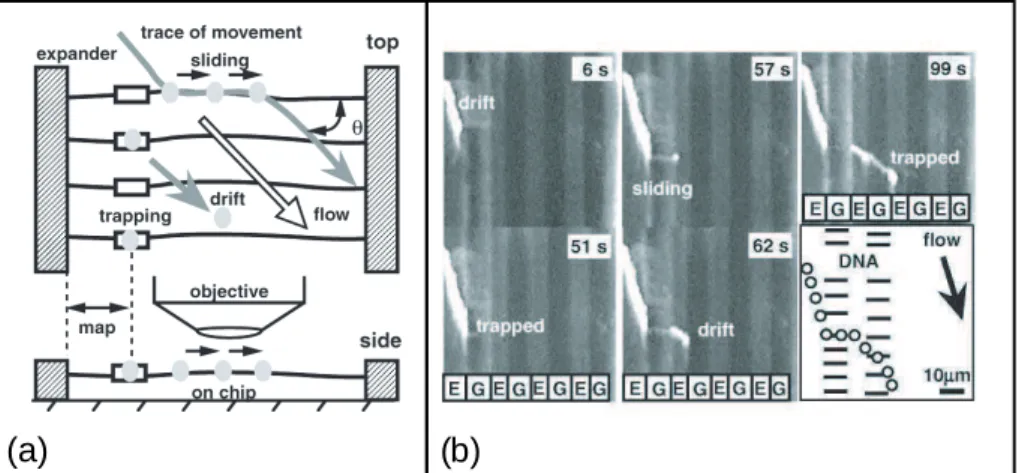
Observation de mol´ecules d’ADN individuelles en microsco-
- Marquage fluorescent de l’ADN
- Microscopie de fluorescence
Cette technique permet d'étirer un grand nombre de molécules d'ADN en parallèle sur une surface. 2.3 – Schéma du dispositif optique utilisé pour étirer les molécules d'ADN et les observer.
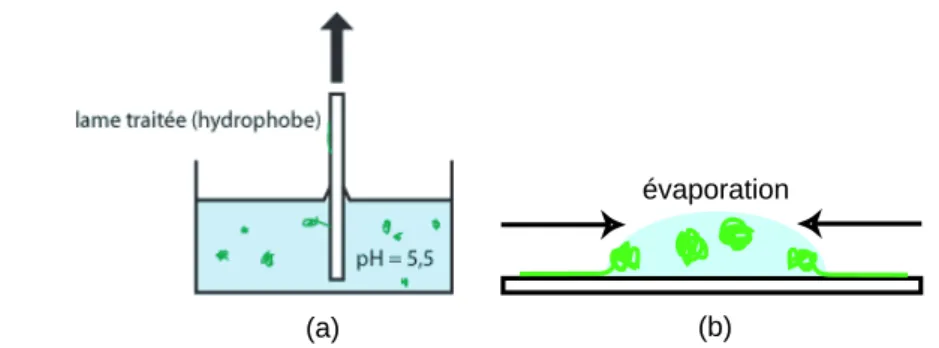
Peignage
- R´ealisation exp´erimentale
- Ancrage sp´ecifique des extr´emit´es de l’ADN
- Etat des mol´ecules peign´ees
- Mat´eriel et m´ethodes pour le peignage
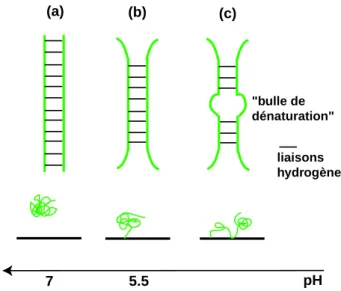
Ainsi, sans que la raison ne nous soit claire, il est possible de peigner des molécules d'ADN à pH 7,5. Le comportement des molécules d’ADN en solution sur de telles surfaces dépend fortement du pH de la solution. À un pH inférieur à 5,5, les molécules d’ADN s’adsorbent rapidement et de manière irréversible à la surface, sous forme de pastilles.
Le peignage est un procédé qui peut être utilisé pour une très large gamme de tailles de molécules d'ADN (jusqu'aux mégabases).
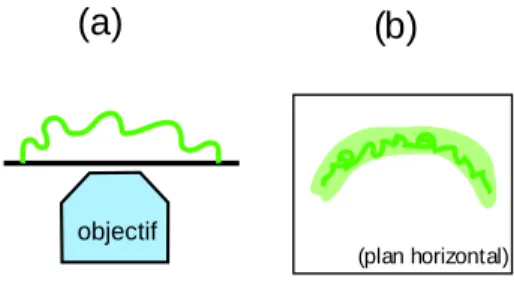
Etirement
- Principe
- R´esultats exp´erimentaux
- Mat´eriel et m´ethodes pour l’´etirement
Le photoblanchiment des fluorophores utilisés pour marquer l’ADN (YOYO-1, SYBR Gold) peut conduire à la photodestruction des molécules d’ADN. Les molécules d'ADN étirées préalablement marquées avec YOYO-1 sont facilement détectables en excitant ce fluorophore avec une lampe à mercure et en collectant la fluorescence. La durée des films produits, limitée par la photodestruction des molécules d'ADN marquées, est de l'ordre de la minute.
Après quelques minutes, cette solution est introduite dans la cavité d'écoulement contenant l'ADN étiré.
Conclusion
Analyse des fluctuations transverses
- Elasticit´e de l’ADN
- Mod`ele de la chaˆıne librement jointe
- Mod`ele du ver
- Mod`ele utilis´e pour l’analyse des fluctuations transverses . 54
- Choix du nombre de billes
- Dynamique
- Discussion : pertinence de la description hydrody-
- R´esultats du mod`ele pour une ´elasticit´e lin´eaire
- Analyse dans le cas de l’´elasticit´e r´eelle (non lin´eaire)
- Simulations num´eriques
- R´esultats exp´erimentaux
- Analyse des films exp´erimentaux
- Etirement de l’ADN dans le flux hydrodynamique 58
- Conclusion
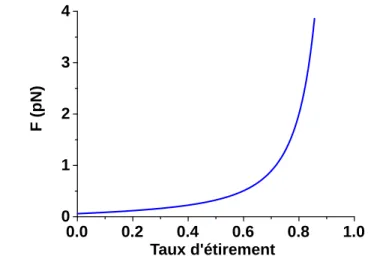
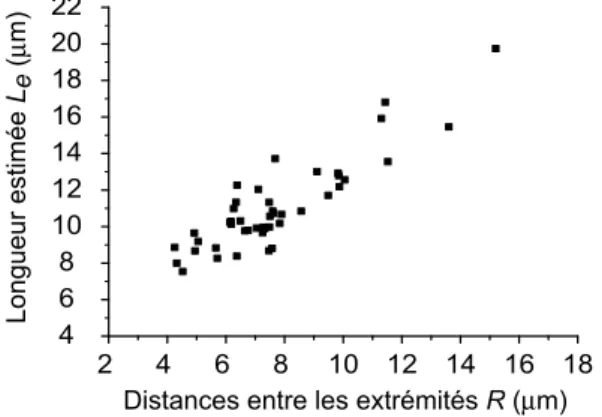
De plus, dans nos expériences, le degré d’alignement des molécules d’ADN est élevé et varie peu. On peut cependant y revenir, à condition que le degré d'extension des molécules d'ADN considérées soit suffisamment élevé. La vitesse d’étirement des molécules d’ADN est liée à la force d’écoulement appliquée pendant le processus d’étirement.
L'étude de l'amplitude des fluctuations transversales permet une mesure indirecte de la taille des molécules d'ADN.
D´etection de mol´ecules d’ADN peign´e `a l’aide de nanocristaux
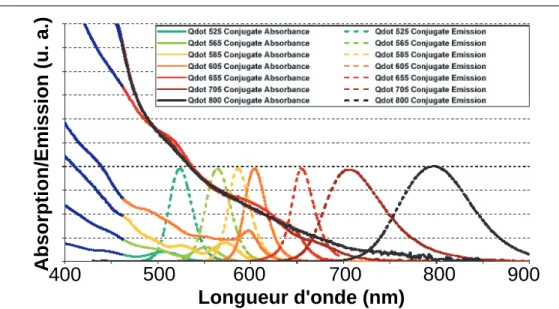
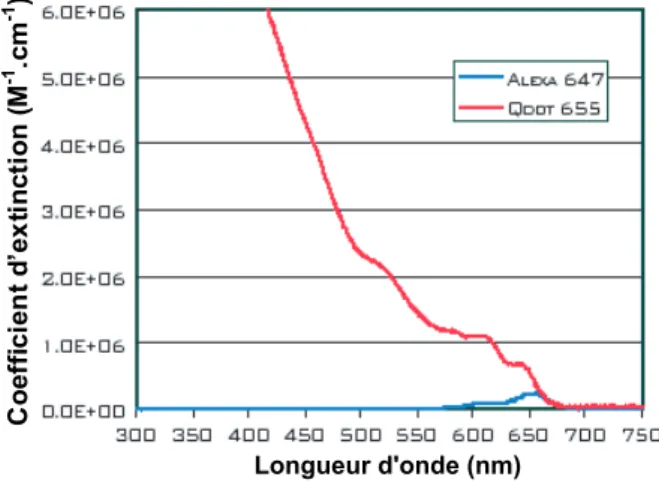
- Fluorophores organiques vs nanocristaux
- Inconv´enients des fluorophores organiques
- Les nanocristaux : des fluorophores aux propri´et´es
- Mise en oeuvre exp´erimentale
- Pr´eparation d’ADN avec extr´emit´es biotine et/ou
- Attachement des NCs sur les mol´ecules d’ADN
- Mat´eriels et m´ethodes
- R´esultats
- Efficacit´e de d´etection des extr´emit´es modifi´ees . 72
- Estimation de la probabilit´e de localisation cor-
- D´etection d’ADN ´etir´e
- Conclusions
Leur photoblanchiment limite la durée d'observation des molécules d'ADN et peut conduire à leur photodestruction. Les molécules d'ADN ont été ligaturées aux deux extrémités de la biotine, marquées avec YOYO-1 et peignées. Expérimentalement, il nous est possible de détecter certains NC aux extrémités des molécules d’ADN.
En revanche, davantage de molécules d'ADN (7 %) comportant des NC aux deux extrémités sont détectées.
Conclusion g´en´erale du chapitre
L’enzyme de restriction EcoRV
Cette enzyme reconnaît une séquence de six paires de bases sur l'ADN double brin, en la coupant au milieu. La capacité d'EcoRV à reconnaître et à cliver spécifiquement sa séquence cible dépend de la présence d'ions divalents. En l'absence de tels ions, EcoRV montre peu de spécificité pour sa séquence cible, qu'il est incapable de cliver (88).
En présence de ces ions, EcoRV, en revanche, se lie beaucoup plus fortement à sa séquence cible qu'à des fragments d'ADN non spécifiques.
Couplage des prot´eines `a des fluorophores : imp´eratifs et
- Choix du fluorophore
- Int´erˆet d’un couplage site-sp´ecifique de la prot´eine 88
- Synth`ese d’enzymes EcoRV biotinyl´ees (Wolfgang
- Couplage aux nanocristaux
- Mat´eriel et m´ethodes
- Tests biochimiques de l’activit´e des NC-EcoRV . 92
Cependant, une résolution spatiale de 1,5 nm a été obtenue dans cette expérience avec un temps d'exposition de 500 ms. La structure tridimensionnelle d'EcoRV suggère que cette cystéine, située « à l'intérieur » de l'enzyme, n'est pas accessible. Clivage de l'ADN : comme le montre la figure 3.5, la présence de l'ADN λ, qui contient 21 séquences cibles pour EcoRV, avec NC-EcoRV conduit au clivage de cet ADN.
Au cours de ces tests, nous avons également constaté que les concentrations de NC-EcoRV nécessaires au clivage total de l’ADN étaient supérieures à celles des enzymes libres.
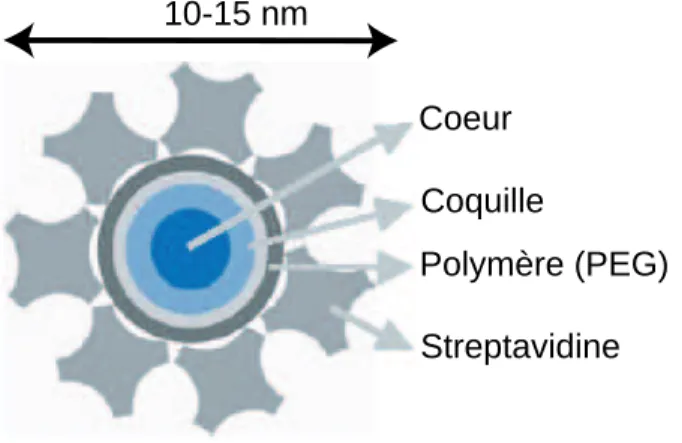
Choix de l’ADN utilis´e et de son mode de d´etection
- Choix de l’ADN du bact´eriophage T7
- D´etection de l’ADN par le SYBR Gold
Les protéines capables de se lier à l’ADN peuvent également se lier à l’héparine ; cette fixation est stable à faible salinité. Pour cette raison, l’héparine est largement utilisée pour la purification de protéines capables de se lier à l’ADN. Pour cette raison, nous avons choisi SYBR Gold (Molecular Probes) pour détecter l’ADN, un fluorophore qui s’insère dans le petit sillon des molécules d’ADN [96].
Un protocole permettant de fixer des nanocristaux aux extrémités des molécules d’ADN représenterait une excellente méthode de détection.
Choix des conditions d’interaction
- Traitement des surfaces
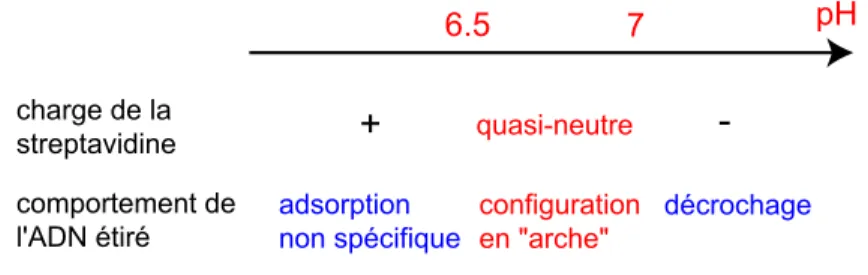
- pH d’interaction
- Concentration des NC-EcoRV
La plage de pH de 6,5 à 7,0 est également optimale pour la stabilité des molécules d'ADN étirées sur les surfaces recouvertes de caséine, y compris le point iso. Ce comportement est probablement dû aux interactions électrostatiques répulsives entre l'ADN et la caséine, dont la force augmente avec le pH car les charges négatives de l'ADN et de la caséine augmentent. La fréquence des événements d'association de NC-EcoRV avec une molécule d'ADN étirée est d'autant plus grande que leur concentration est élevée.
Il peut s'agir d'agrégats de NC-EcoRV qui interagissent avec l'ADN via diverses enzymes.
Films exp´erimentaux
Aucune interaction des nanocristaux avec l'ADN n'a pu être observée, même en augmentant la concentration des nanocristaux à 1 nM. Nous avons étudié trois conditions salines différentes pour tester l'influence de ce paramètre sur les interactions de NC-EcoRV avec l'ADN.
Mat´eriel et m´ethodes
- Interaction des NC-EcoRV avec l’ADN
- R´ealisation des films exp´erimentaux
- Programme d’analyse
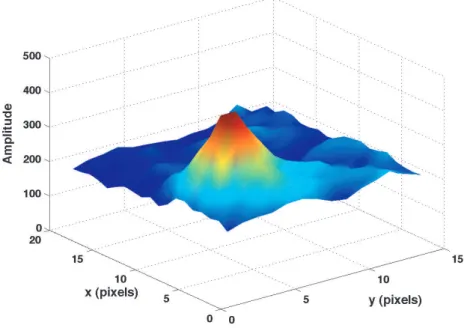
- Estimation de la r´esolution spatiale
A la fin de l'analyse par le programme Matlab nous aurons la trajectoire des spots lors de leur interaction avec l'ADN. La valeur obtenue a ensuite été comparée au résultat d'un calcul prenant en compte les différentes sources d'incertitude sur la localisation des spots. a) Mesure directe de la résolution spatiale. La résolution spatiale obtenue est finalement de l'ordre d'une dizaine de nanomètres avec un temps de pose de 20 ms. c) Nanocristaux en mouvement.
Le mouvement d'un NC-EcoRV lié à l'ADN pendant texp=20 ms est donc de l'ordre d'une dizaine de nanomètres.
R´esultats
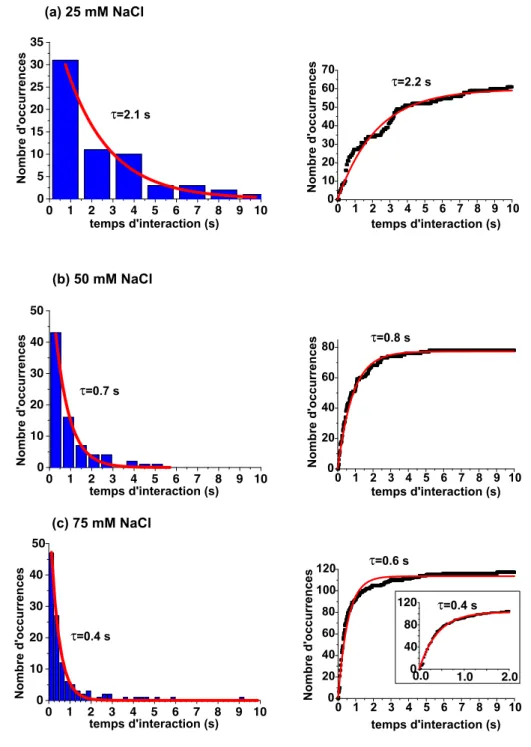
Dur´ees d’interaction des NC-EcoRV avec l’ADN
- M´ethode d’analyse
- R´esultats
- Discussion
En effet, le clignotement des nanocristaux conduit à sous-estimer le temps d’interaction des taches qui s’associent ou se dissocient de l’ADN à l’état sombre. Nos résultats expérimentaux (Figure 3.12) montrent que la durée de l'interaction du NC-EcoRV avec l'ADN diminue à mesure que la concentration de NaCl augmente. Nos expériences ont montré qu'un NC-EcoRV restait lié à l'ADN pendant environ 1 seconde avant de se dissocier.
La longue durée mesurée de l'interaction NC-EcoRV/ADN fournit donc une première indication, pointant vers l'existence d'un mouvement d'EcoRV sur l'ADN pendant la durée de l'interaction.
Mise en ´evidence du mouvement des NC-EcoRV le long de
- Mod´elisation du mouvement thermique d’un point
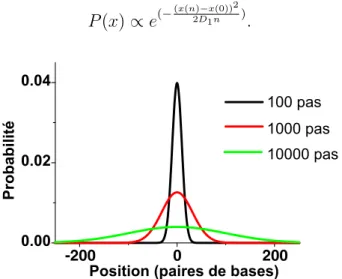
- Une grandeur appropri´ee pour l’analyse : le d´eplacement
- R´esultats
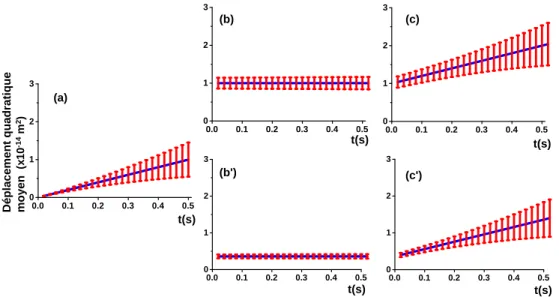
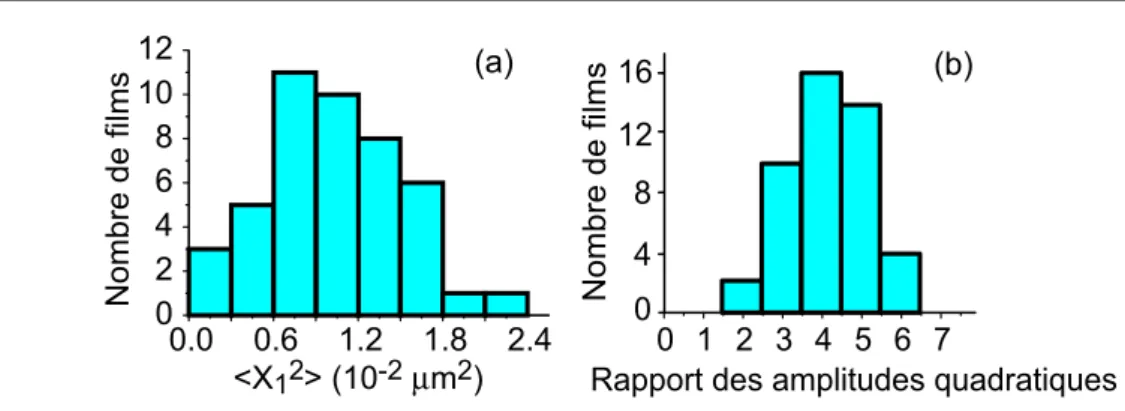
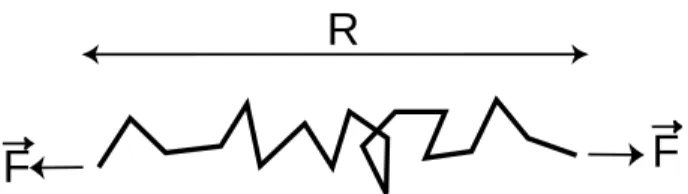
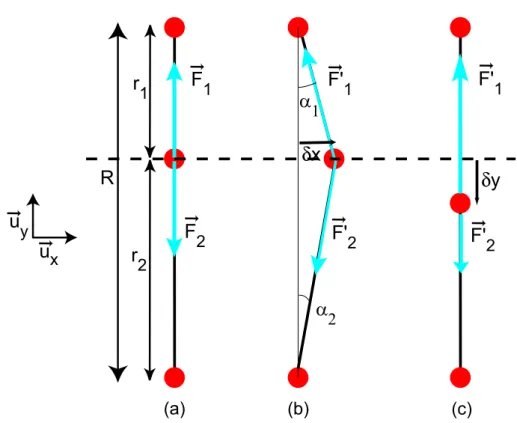
3.13 – Boule dans une molécule d'ADN étirée : (a) équilibre, (b) mouvement transversal, et (c) mouvement longitudinal. a) Rigidité transversale et longitudinale. Le mouvement de diffusion du NC-EcoRV le long de l’ADN n’a pas nécessairement une amplitude plus grande que le mouvement dû au mouvement thermique des molécules d’ADN. 3.15 – (a) Position le long de l’axe des x et (b) déplacement carré moyen d’un point fixe ; le film analysé contient 200 images. b) Calcul du déplacement quadratique moyen dans le cas d'un point fixe sur une molécule d'ADN étirée.
Dans ce cas, le mouvement transversal (longitudinal) d'un point sur une molécule d'ADN a une fonction d'autocorrélation de la forme < x(t)x(t+τ)>=X2e−τ /τx (< y(t)(y ) ( t + τ) >= Y2e−τ /τy), où X2 (Y2) indique l'amplitude quadratique moyenne des fluctuations transversales (longitudinales) et τx (τy) leur temps de corrélation.
Discussion
- Effet du couplage `a un nanocristal sur les ca-
- Discussion de la distribution large des coefficients
- Implications biologiques de nos exp´eriences
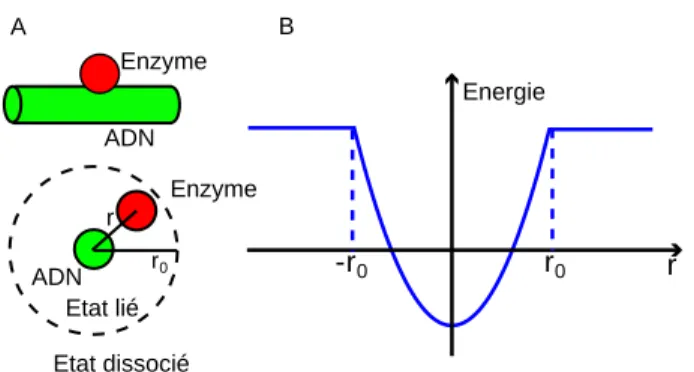
Nous avons ensuite utilisé le modèle développé par Schurr [21], qui relie le coefficient de diffusion d'une protéine sur l'ADN à sa taille. Le glissement des protéines le long du sillon majeur de l’ADN est une hypothèse souvent utilisée mais récemment contestée [35]. La rotation des protéines autour de l’ADN est également une hypothèse fondamentale du modèle Schurr.
A partir de cette observation, nous avons mesuré le coefficient de diffusion le long de l'ADN.
Am´eliorations exp´erimentales (en cours)
Etirement d’ADN biotinyl´e sur des surfaces streptavidin´ees 123
- R´esultats exp´erimentaux
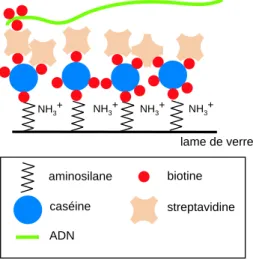
Les surfaces utilisées sont composées d'une couche d'aminosilanes, d'une couche de caséine biotinylée et d'une couche de streptavidine, qui assure la fixation des molécules d'ADN biotinylée. Pour cette raison, les molécules biotinylées telles que le PEG (polyéthylène glycol), la BSA (albumine sérique de bœuf) ou la caséine sont généralement adsorbées en surface avant le dépôt de streptavidine. Grâce à la grande stabilité de la liaison biotine-streptavidine, aucune séparation des molécules étirées n'est observée après élévation du pH, ajout de sel ou de caséine.
De plus, nous avons constaté que la distribution en longueur des molécules étirées est nettement plus étroite que sur les surfaces en polystyrène.
Application aux exp´eriences d’interaction ADN/ prot´eines 125
L’utilisation de nanocristaux pour marquer les extrémités de l’ADN nous libère de l’utilisation de molécules SYBR Gold le long de l’ADN. De cette manière, il sera possible d'éviter la photodestruction des molécules d'ADN due au colorant et de vérifier que la présence de ce fluorophore n'affecte pas les interactions ADN/EcoRV. De telles expériences ne peuvent être réalisées que si la séquence d’ADN étudiée peut être liée à des positions sur l’ADN étiré.
1 ng d'ADN biotinylé marqué au SYBR Gold dilué dans 20 mM PIPES pH 6,8 est introduit dans la cavité et interagit avec la surface pendant quelques minutes.
Nos études en microscopie à fluorescence représentent l'une des premières observations de la diffusion de protéines sur l'ADN. Plusieurs expérimentations sont prévues dans un futur proche dans le but d'améliorer notre compréhension de l'action de l'EcoRV sur l'ADN. coli selon sa localisation sur un domaine de la molécule d'ADN riche en purines (bases A et T) ou en pyrimidines (bases G et C) [11].
Ces informations seront utiles pour prédire le comportement in vivo de l'enzyme sur des molécules d'ADN recouvertes de nombreuses protéines.