J'ai également pu montrer une expression variable entre les populations de gènes de la voie piARN. 115 Figure 26 : Quantification des transcrits des gènes de la voie piARN dans les populations naturelles noires : D .
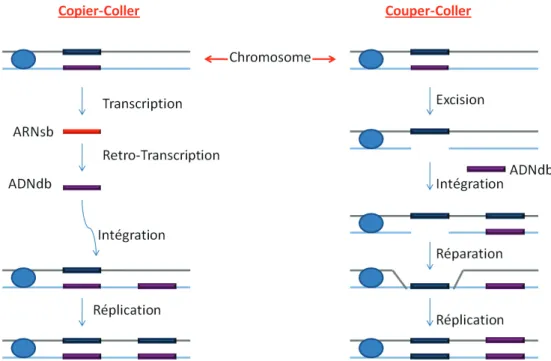
Les éléments transposables et les rétrovirus
Cependant, leur dynamique et leur impact sur les génomes sont encore mal compris. Ces expériences ont été principalement réalisées sur des organismes modèles tels que la levure Saccharomyces cerevisea (Boeke et al., 1985) et la mouche D.

Classification des éléments transposables
Les éléments à ARN (Classe I)
- Les éléments à LTR et les rétrovirus endogènes (ou ERV pour
- Les éléments sans LTR
La caractérisation chez Saccharomyces cerevisiae de la rétrotransposition de l'élément Ty fournit la première confirmation expérimentale d'un tel mécanisme (Boeke et al, 1985). Deux rétroviridae : l'un simple (Mo-MLV : Moloney Leukemia Virus), l'autre complexe (VIH : Human Immunodeficiency Virus).

Les éléments à ADN (classe II)
Autres
Cycle de rétrotransposition des éléments à LTR
Répartition des éléments transposables dans les génomes
Un exemple de RDE chez Drosophila melanogaster : le locus flam/COM . 10
La théorie égoïste de l'ADN (Doolittle & Sapienza, 1980 ; Orgel & Crick, 1980) suggère que les génomes sont constitués de deux types de séquences : l'ADN. Britten et Davidson ont été les premiers à émettre l'hypothèse que l'exaptation d'éléments transposables par le génome hôte représente un mécanisme important dans la structuration des génomes (BRITTEN & Davidson, 1969).
Implication des Éléments transposables dans l’Évolution des génomes
L'activation du rétrotransposon LTR Wis2-1A induit ainsi la régulation de l'expression d'un certain nombre de gènes chez le blé (Kashkush et al, 2003). Bailey et al ont montré que les éléments Alu seraient à l'origine de duplications dans le génome humain (Bailey et al, 2003), et seraient responsables de la translocation entre les chromosomes humains 11 et 22 (Hill et al, 2000).
Régulation épigénétique des éléments transposables
Méthylation de l’ADN
Bien que la méthylation de l’ADN soit un mécanisme de régulation épigénétique répandu, les génomes sont méthylés différemment selon les organismes. La méthylation de l'ADN dans les plantes est symétrique au niveau des cytosines de CG ou CHG (H est A, T ou C), ou asymétrique dans un contexte CHH.
Modifications d’histones
Cela nous amène à croire que chez la drosophile, la méthylation de l’ADN n’est pas impliquée dans la régulation des gènes et des ET, comme cela a été démontré chez d’autres espèces. Deux nucléosomes sont séparés l'un de l'autre par une cinquantaine de paires de bases d'ADN nu et par l'histone internucléosomale H1.
Modifications post-traductionnelles des histones
- Acétylation des histones
- La méthylation des histones
L'acétylation des histones consiste en un transfert d'un groupe acétyle vers le résidu lysine des queues amino-terminales des histones. La méthylation des histones affecte les résidus lysine et arginine des queues amino-terminales des histones.
Territoires chromatiniens
- L’euchromatine
- L’hétérochromatine
Un gène situé à proximité d’une zone d’hétérochromatine constitutive verra son expression diminuer en fonction de la distance. En effet, SU (VAR) 3-9 joue un rôle central dans l'établissement de l'hétérochromatine dans les chromocentres de la drosophile (Schotta et al, 2002).
Historique de l’ARN interférence
- Les microARN (miARN)
- Les small interfering ARN (siARN)
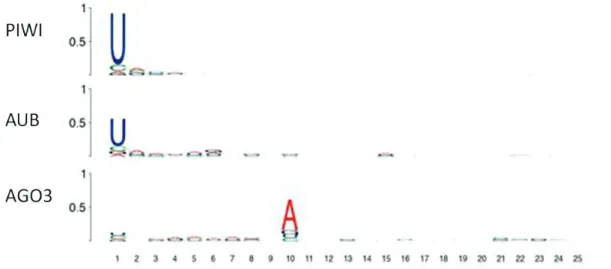
- Les Piwi-interacting RNA (piARN)
En revanche, la biogenèse des piARN secondaires par la voie d’amplification « ping-pong » est spécifique à la lignée germinale. Ces observations suggèrent donc une compartimentation de la production de piARN et de leur fonction (Lim & Kai, 2007).

La drosophile comme modèle génétique
Quelques modèles d’instabilité
- Le facteur I
- Les rétrovirus endogènes Gypsy, Idefix et ZAM
De même, les transcrits Idefix sont détectés dans les premiers stades de l'ovogenèse (germarium) uniquement dans la souche instable (Figure 20A). En revanche, la mutation de la protéine Piwi entraîne une dérépression de Gypsy, ZAM et Idefix dans les tissus reproducteurs (Desset et al, 2008) (Sarot et al, 2004) (Figure 20C).

Les populations naturelles de drosophiles pour comprendre la dynamique
- Variabilité en ET dans les populations naturelles de drosophiles
Cependant, jusqu'à présent, les données concernant les tailles efficaces respectives de D. melanogaster et de D. simulans sont contradictoires. Une autre façon d’essayer de comprendre ces différences est de considérer que les deux espèces ont des histoires différentes en matière de régulation des ET. Une telle différence peut s'expliquer a) par la capacité du génome de D. simulans à réguler ses ET plus strictement que D. melanogaster, b) par une perte de copies d'ET qui seraient devenues rapidement inactives chez D. simulans, c) à partir de le fait que le génome de D.
Ces deux espèces envahissantes originaires des régions d'Afrique de l'Est ont des histoires écologiques et de colonisation du monde différentes par D. Notre laboratoire travaille depuis plusieurs années sur le contrôle de l'expression du rétrotransposon qui attire D. Marsano et al. 2000) ont analysé tyran dans quatre souches de D. Ces quatre souches ont au moins une copie complète de tyran ainsi que des copies supprimées.
Dans un premier temps, j'ai déterminé les types d'histones associés aux copies de pullers en utilisant la technique d'immunoprécipitation de la chromatine (CHIP), et j'ai démontré un effet de l'insertion de pullers sur les modifications des histones et sur l'expression du gène tkv (Article 2).

Discussion
- L’élément tirant : un modèle d’étude de prolifération de rétrovirus
- L’expression de tirant dépend de la population
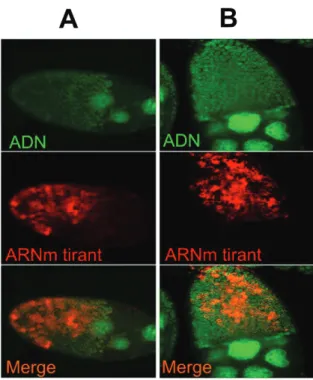
La présence d'une copie cravate codant pour une protéine d'enveloppe potentiellement fonctionnelle dans la population de Mayotte conforte l'idée que des particules virales infectieuses peuvent être assemblées dans les ovaires de cette population. Dans la population de Mayotte, les résultats de l'immunocoloration anti-ENV et de l'hybridation in situ d'ARN pull-down montrent que la protéine et l'ARN sont localisés au niveau de la membrane apicale. On peut donc supposer que le site d’assemblage des particules de bielle est situé sur la membrane apicale.
Dans la plupart des populations naturelles, l'élément attracteur est inactif comme dans la population Chicharo, malgré le fait que nos résultats montrent que ces populations contiennent les gènes complets de l'enveloppe attractive. L'analyse de la distribution des liaisons dans le génome de l'hôte a montré que le nombre de copies est variable entre les populations naturelles de D. Des transcrits de liaison sont détectés dans le cytoplasme des cellules folliculaires des ovaires de la population de Mayotte, par contre cette localisation folliculaire est nucléaire chez la population Makindu.
L'accumulation de transcrits de tir dans le noyau des ovaires de la population Makindu ainsi que l'absence de transposition indiquent la mise en place d'un système de régulation contrôlant la mobilisation du tir dans la lignée germinale.
Analyse des modifications d’histones associées à des insertions de tirant
Discussion
- Modification d’histones associées au rétrovirus endogène tirant
- Influence de tirant sur la chromatine des gènes voisins
Nous avons analysé la structure de la chromatine au niveau des sites d'insertion de liaisons dans trois populations différentes, en étudiant notamment une marque caractéristique de l'euchromatine, H3K4me2, et deux marques hétérochromatiques, H3K27me3 et H3K9me2. Nos résultats suggèrent une bivalence des 2 marques H3K27me3 et H3K4me2 au niveau des insertions de copies complètes de traits dans les ovaires de Drosophile. La bivalence des marques euchromatiques et hétérochromatiques au même site d'insertion du lieur avait déjà été démontrée dans le tissu somatique de D.
Nos résultats montrent que le trait est capable de modifier la structure de la chromatine au niveau de son site d'insertion, mais également en amont. En effet, l'insertion d'une copie complète d'un intron du gène tkv dans la population Makindu est responsable de la présence de la marque hétérochromatique H3K27me3 au niveau d'un promoteur tkv. La conséquence de l'hétérochromatinisation du promoteur du gène tkv par l'insertion d'un tirant est une réduction du taux d'expression de ce gène dans la population Makindu, mais pas dans les deux autres populations où cette région est dépourvue de tirant.
Pour aller un peu plus loin dans l'analyse de l'interaction entre l'insertion de l'élément et le gène tkv, nous tentons de quantifier la production de la protéine tkv dans les trois populations par Western blot.
Les piARNs germinaux déposés maternellement régulent le rétrovirus endogène
Dicussion
- Les piARN sont associés avec l’accumulation nucléaire des
- Les piARN déposé maternellement sont nécessaires à la régulation
- Interaction entre les deux voies de piARN somatique et germinale108
Nos résultats ne montrent pas d’effet des piARN secondaires dans la régulation de l’attraction des cellules nourricières. Des transcriptions de traits ont été détectées dans les cellules nourricières dans les deux sens de croisement (NRT et RT) (article Figure 1B). Par conséquent, les piARN semblent être responsables de l’accumulation nucléaire de transcrits sensoriels dans les cellules somatiques synthétisées par des copies euchromatiques de tir dans la population Makindu.
Nos résultats montrent que la surexpression de tir dans les cellules de la jonction somatique (NRT) est en corrélation avec la diminution des piARN déposés par la mère. Notre résultat montre une dérégulation des caractères dans les deux sens de croisement A (Mâle Mayotte x Femelle Chicharo) et B (Mâle Chicharo x Femelle Mayotte) (Figure 22), cohérent avec notre hypothèse. L'absence de contrôle de traction dans les cellules folliculaires dans les deux sens de croisement valide notre modèle.
Les transcriptions de tractus sont présentes dans les GSC (Figure 23B), donc le passage des piARN peut être possible entre les cellules germinales et les cellules folliculaires (Figure 23).
Polymorphisme des gènes de la voie piARN
Taux de transcrits des ET dans les populations naturelles de D. simulans et
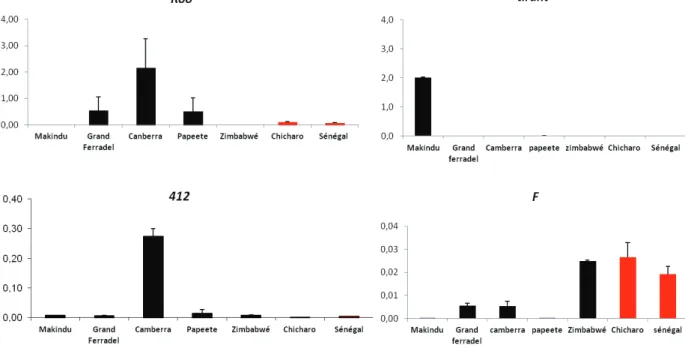
J'ai déterminé le taux de transcriptions de quatre éléments transposables (roo, 412, F et tir) dans 5 populations de D. L'élément transposable tir est exprimé dans la population Makindu, mais ce n'est pas le cas dans les autres populations. Les éléments Roo et F présentent une variabilité d'expression entre les différentes populations, et l'élément F est fortement supprimé dans les populations Makindu et Papette.
L'élément 412 est fortement exprimé dans la population de Canberra, conformément aux données de la littérature (Borie et al, 2002), et est moins exprimé dans d'autres populations. La quantité de transcrits dans l’ovaire est en corrélation avec la quantité de transcrits du gène rp49. En effet, roo et tir ont des niveaux de transcription bien plus élevés que les éléments 412 et F.
Nous avons observé 412 transcrits dans les ovaires de la population de Canberra, mais aucun marquage pour l'élément F dans la population zimbabwéenne (D. simulans) (Figure 25).
Polymorphisme de séquence et taux de transcrits des gènes de la voie
Blumenstiel JP, Hartl DL (2005) Evidence for a maternally transmitted small interfering RNA in the repression of transposition in Drosophila virilis. Bucheton A, Lavige JM, Picard G, L'Heritier P (1976) Non-Mendelian female sterility in Drosophila melanogaster: quantitative differences in the efficiency of inducers and reactive strains. Bucheton A, Paro R, Sang HM, Pelisson A, Finnegan DJ (1984) Molecular basis of I-R hybrid dysgenesis in Drosophila melanogaster: identification, cloning and properties of factor I.
Chaboissier MC, Busseau I, Prosser J, Finnegan DJ, Bucheton A (1990) Identification of a potential RNA intermediate for the transfer of a LINE-like element of factor I in Drosophila melanogaster. Crozatier M, Vaury C, Busseau I, Pelisson A, Bucheton A (1988) Structure and genomic organization of I elements involved in I-R hybrid dysgenesis in Drosophila melanogaster. Gordon A, Hannon GJ (2010) Probing the initiation and effector phases of the somatic piRNA pathway in Drosophila.
Kim AI, Belyaeva ES, Aslanian MM (1990) Autonomous transposition of gypsy mobile elements and genetic instability in Drosophila melanogaster. Lim AK, Kai T (2007) Unique germline organelle, nuage, functions to suppress selfish genetic elements in Drosophila melanogaster. Picard G, Bregliano JC, Bucheton A, Lavige JM, Pelisson A, Kidwell MG (1978) Non-Mendelian female sterility and hybrid dysgenesis in Drosophila melanogaster.