Geometric algorithms for cavity detection on protein surfaces
Texto
Imagem
Documentos relacionados
3D structures of proteins showing the binding sites (left), and main residues involved in the interaction ligand-protein (right) of R -ALA to leukotriene A4 hydrolase (A,B),
Mg-incorporated titanium oxide surfaces showed major differences of surface chemistry and crystal structure, albeit similar surface roughness values compared to the control TiO 2
Here we introduce ConCavity , a small molecule binding site prediction algorithm that integrates evolutionary sequence conservation estimates with structure- based methods
Using the 3D structure of a protein, molecular recognition studies between protein-protein or protein- ligand can be achieved and used to explain biological
A: Anteroposterior radiograph demonstrating irregularities on articular surfaces with calcifica- tions adjacent to the distal femoral (large arrow) and proximal tibial (small
Therefore the aim of this study was to validate histologically the use of DIAGNOdent (LF) for the detection and quantification of caries on intact oc- clusal surfaces;
shows the location of the binding sites on the surface of the protein structure and Table 3 shows the volume, depth and drug score for cruzain and Llacys1.. Three pockets were
Protein structure and function studies require large quantities of pure protein. Many proteins are normally expressed at very low amounts, precluding their isolation and
![Figure 8: Detecting cavities through POCKET (see [LB92]): (a) in the x -direction; (b) in the y -direction](https://thumb-eu.123doks.com/thumbv2/123dok_br/18663185.913314/52.892.89.817.125.329/figure-detecting-cavities-pocket-lb-x-direction-direction.webp)
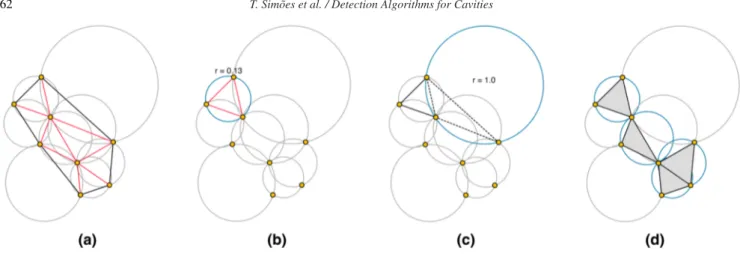
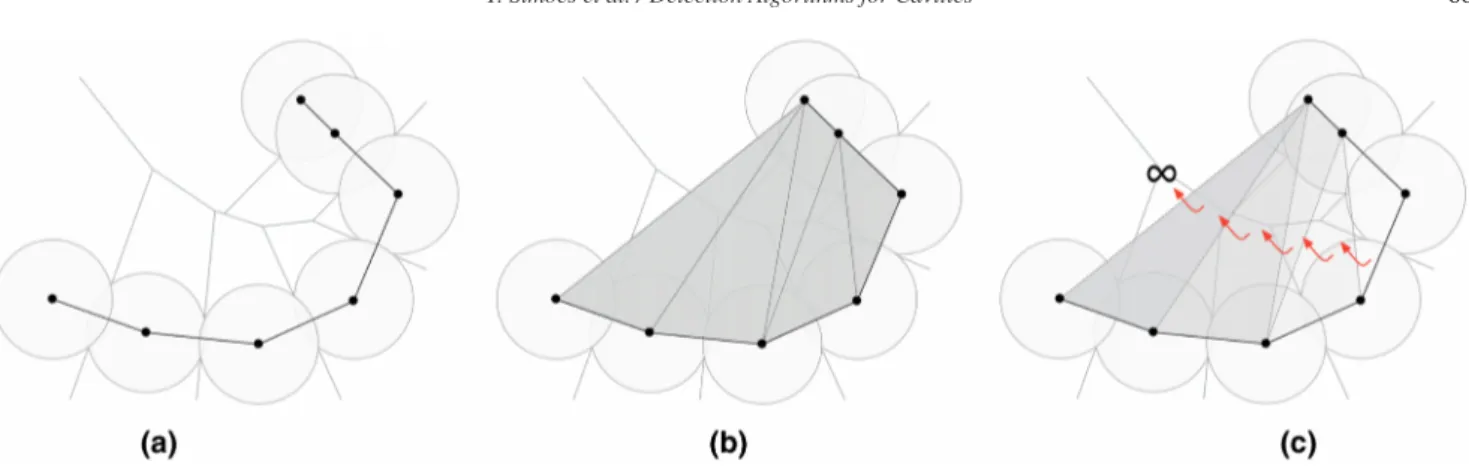
![Figure 17: GP method [XB07]: (a) C α atom-based structure (grey points); (b) convex hull (in orange) and Delaunay triangulation (in dark grey); (c) first carving procedure that removes simplexes whose edges are longer than 30.0 ˚ A (black dashed line segme](https://thumb-eu.123doks.com/thumbv2/123dok_br/18663185.913314/66.892.89.820.115.282/figure-structure-delaunay-triangulation-carving-procedure-removes-simplexes.webp)