Introduction bibliographique
Les défenses chimiques des plantes face aux insectes
La non-préférence ou antixénose définit le type de résistance induite par le rejet de la plante comme hôte de l'insecte. La ponte et l'alimentation des larves de Chalcodermus aenus sont en effet limitées par l'épaisseur de la paroi des gousses chez certains cultivars de Vigna unguiculata (Fery et Cuthbert, 1979).
Les allélochimiques non protéiques à activité entomotoxique
- Les composés azotés
- Les terpénoïdes
- Les composés phénoliques
Les produits de dégradation de ces composés ont également une activité antifongique et antibactérienne (Duncan, 1991), ce qui indique un rôle possible dans la défense des plantes contre les agents pathogènes. d) Acides aminés non protéiques. Parmi ces derniers, la tamatine, la solanine et la demissine de Solanum demissum protègent la plante contre la gourmandise du doryphore de la pomme de terre (Richter, 1993). f) Glycosides cardiotoniques.
Les protéines végétales de défense
- Les protéines entomotoxiques
- Les peptides de défense (antimicrobiens et insecticides)
BASI est impliqué dans la régulation de l'AMY2 endogène lors de la dégradation des sucres des graines (Kadziola et al., 1998). Les produits de dégradation de la chitine induisent la production de composés de défense (phytoalexines) et de SAR (résistance systémique acquise) (Brunner et al., 1998).

Conclusion
- Contexte de l'étude et présentation des travaux
Les amorces de la première PCR (flèches pleines) amplifient un premier fragment, sur lequel s'hybrident les amorces de la deuxième PCR (traits pointillés), qui amplifient alors a. La première ligne indique une maturation semblable à celle d'un pois de la préproprotéine A1 (Higgins et al., 1986). Les bibliothèques de digestion ont été réalisées de la même manière que pour la remigration.
Cependant, l'absence de la troisième cystéine (après le doublet proline) est cohérente avec la mutation de la guanine (en position deux du codon) en cytosine.
Définition de l’approche : à la découverte de la famille des A1b
Matériels et méthodes
Biologie : recherche de toxicité analogue
- Les charançons
- Le matériel végétal
- Préparation et fractionnement des farines
- Tests biologiques
Les graines destinées à la sélection (blé non traité et pois cassés) ont été placées à −80°C pendant une semaine pour tuer les insectes éventuellement présents, puis conservées à 4°C. Plusieurs plants de pois ont été conservés pour obtenir la floraison et la formation de graines, prélevés au cours de leur développement pour en extraire l'ARN. Des essais biologiques ont été réalisés sur différentes fractions pour vérifier la présence éventuelle d'une toxicité similaire (mortalité différentielle entre champignons sensibles et résistants).
Les animaux ont ensuite été placés dans le four et les morts ont été comptées quotidiennement à partir du deuxième jour pendant deux semaines.
Biochimie : recherche d’homologues protéiques
- Dosage protéique
- Electrophorèse protéique sur gel d’acrylamide (SDS-PAGE)
- Immunodétection
- HPLC (High Performance Liquid Chromatography)
- Spectrométrie de masse
- Tests d’inhibition de liaison
Les échantillons ont été mélangés à la matrice (acide sinapic ou acide 3,5-diméthoxy-4-hydroxycinnamique, LaserBio Labs) à Les filtres ont été transférés dans des tubes de comptage et la radioactivité y a été mesurée (Riastar, Packard Instrument, USA). Les données ont été analysées à l'aide du logiciel RADLIG 4 (BIOSOFT, Cambridge, Royaume-Uni) et les graphiques ont été dessinés à l'aide du logiciel ORIGIN 5 (Microcal, USA).
Des tests de déplacement de liaison7 ont été réalisés avec les fractions MeOH60 pour chaque plante.
Biologie moléculaire : recherche de gènes homologues
- Extraction d’ADN
- Amplification de fragments de gènes par PCR
- Electrophorèse sur gel d'agarose
- Clonage
- Minipréparation d’ADN plasmidique par lyse alcaline
- Séquençage
- Recherche des séquences amont des gènes
- Résultats
Une fois déposés sur la cible et séchés, ces mélanges ont été analysés au MALDI-TOF. Les réactions PCR ont été réalisées dans un thermocycleur (Perkin Elmer Gene Ampli PCR 2400) dans un volume réactionnel de 50 µl avec des quantités de matrice allant de 10 à 500 ng, avec 1,25 unités de Taq polymérase et son tampon 10X (Amersham). ) selon les schémas de la Figure 38. Pour disposer de la partie complète des gènes codant pour les homologues de PA1b, les séquences en amont de celles obtenues par PCR ont été recherchées, à l'aide du kit Universal GenomeWalkerTM (Clontech, USA), selon les instructions du fournisseur ( principe en annexe 1).
Pour chacune, une amorce du kit, s'hybridant à l'adaptateur, et une amorce spécifique, déterminée à partir de séquences incomplètes obtenues par PCR classique, ont été utilisées.

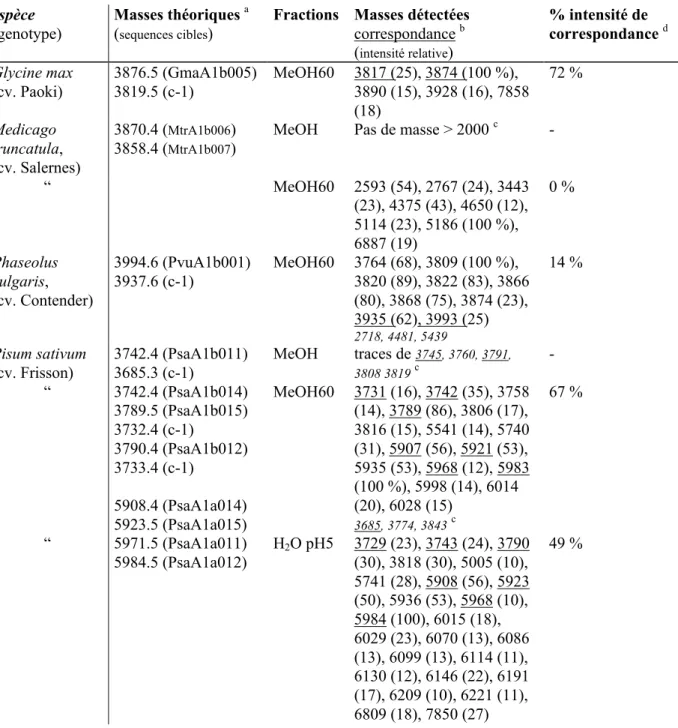
Recherche des homologues de PA1b dans les graines des plantes étudiées
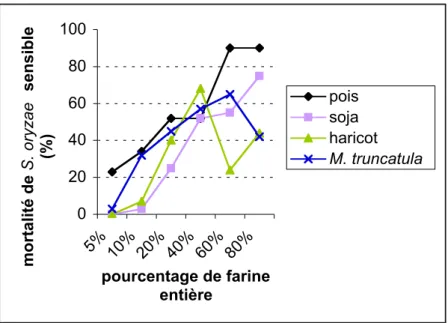
- Toxicité des farines entières
- Fractionnement et toxicité des différentes fractions
- Détection électrophorétique et immunologique d'homologues de PA1b
- Résultats des tests d’inhibition de liaison
Ceci suggère que les mortalités observées ne sont probablement pas uniquement dues à la toxicité des homologues de PA1b. Les TL50 montrent que la fraction MeOH60 de la luzerne méditerranéenne est la plus toxique, devant celle du pois et enfin du soja et des fèves (Tableau 6). b) Toxicité des autres fractions sur S. La fraction restante des 4 plantes tue les sensibles, et les résistantes dans le cas de la luzerne.
L'étude de la toxicité des différentes fractions a donc permis de localiser plus précisément la présence d'homologues de PA1b solubles (ie pour lesquels on observe une différence de mortalité entre souche témoin sensible et souche résistante).
Recherche des gènes homologues au gène codant pour PA1b dans le génome
- Extraction d'ADN des différentes plantes
- Amplification de fragments de gènes homologues à PA1b
- Recherche des séquences amont des gènes
- Analyse des séquences
Définition de l'approche : découverte de la famille A1b. http://www.toulouse.inra.fr/multalin.html). La comparaison avec les séquences publiées de pois et de soja montre que nos séquences sont de nouveaux allèles avec peu de mutations, qui sont toutes synonymes dans le cas de la séquence du soja par rapport à certaines EST. De nouveaux gènes de M. truncatula et de bean apportent de la variabilité à la famille des peptides, conduisant à la distinction de certains acides aminés particulièrement bien conservés dans A1b.
Il s'agit des six cystéines (à noter que la dernière est remarquablement absente de la séquence MtrA1b007), des cinq prolines, des glycines G5 et G30 en plus de l'arginine R21 et de la leucine L27.
Détection de peptides par spectrométrie de masse
- Discussion
- Toxicité analogue et gènes homologues, cas du pois, du soja et du haricot. 110
- Conclusion
Plusieurs problèmes de protéolyse ont été mis en évidence lors de la surexpression de protéines hétérologues. Ces systèmes ont permis la production de protéines recombinantes solubles dans le cytoplasme bactérien et correctement repliées. Cet inhibiteur de l'ARN polymérase T7 (fuite d'expression) permet un contrôle plus strict de l'expression des gènes recombinants (Moffatt et Studier, 1987).
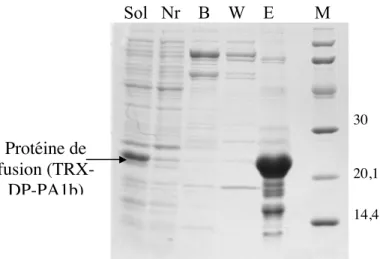
L'apparition d'une bande protéique de masse moléculaire apparente d'environ 25 kDa après induction témoigne de l'expression de la protéine de fusion (masse réelle 20 kDa).
La famille des A1b au sein de la famille des Légumineuses
Les Légumineuses
Systématique
- Présentation de la famille des Légumineuses
- Phylogénie
Les Génistoïdes comprennent une partie des Sophoreae, dont le groupe Sophora (Bolusanthus), qui apparaît alors comme sœur des Genisteae (lupin). Le clade des Galégoides, dont les tribus les plus récemment divergées seraient, d'une part, les Vicieae (pois), regroupées avec le genre Cicer et, d'autre part, les Trifolieae (Medicago) et les Loteae (arbre). Le clade Aeschynomenoid, qui comprend une partie des Dalbergieae, les genres Adesmia et Abrus, et les Aeschynomeneae (AE ; arachide).
Le clade Phaseoleae qui comprend les Phaseoleae (PH ; haricot, soja), les Robinieae, la majorité des Desmodieae (DM) et le noyau des Milletieae qui dériverait de la sous-tribu Diocleineae des Phaseoleae.

Choix des espèces testées
- Diversité structurale primaire
Matériels et Méthodes
- Matériel végétal
- Extraction d’ADN
- Recherche de fragments de gènes homologues par PCR
- Recherche de séquences amont
- Clonage et séquençage
- Northern Blot
- Analyses des séquences
L'amorce Rev6 a également été utilisée en PCR classique combinée à For1 (mêmes conditions que Rev1, Ta = 50°C) pour l'espèce testée après sa création, car elle permet d'obtenir des séquences plus longues au sein de la partie A1a. 4) Rechercher des séquences en amont. Cette bibliothèque et cet ensemble d'amorces ont été choisis pour l'intensité et le caractère unique de la bande obtenue, quelles que soient les conditions de PCR imbriquées. Après avoir vérifié la qualité et déterminé au spectrophotomètre les ARN extraits, elle a effectué une migration des ARN en vue de les transférer sur une membrane en nylon, réalisée pendant le week-end en SSC 20X (étalon de sodium citraté : NaCl 3M ; citrate de sodium 0,3M ) et eau DEPC (pyrocarbonate de diéthyle).
La sonde marquée purifiée a ensuite été placée en contact avec la membrane pendant une nuit à 42°C.
Résultats
- Extractions d’ADN
- Recherche de gènes homologues
- Marche sur le gène
Pour contourner ce problème, deux amorces imbriquées spécifiques à une séquence publiées par Ilgoutz et al. ont été définies et la technique de marche génétique a été appliquée aux différentes bibliothèques de digestion générées pour les deux espèces de lupin testées. De plus, les régions variables préférentiellement choisies pour ancrer les amorces marcheuses au gène (spécificité) ont parfois une structure qui ne correspond pas au design de ces amorces (formation de structures en épingle à cheveux, parfois stables) ou sont incompatibles avec les amorces adaptatrices universelles (partiellement séquence complémentaire). Mentionnons enfin la position de la séquence du lupin (LanU74383), très proche de celle du soja.
Le rapprochement des séquences de même espèce observé ici est également observé lors de la même analyse, en excluant la troisième base du codon.
Discussion
- Les A1b au sein des Légumineuses : une évolution récente?
- Les A1b limités à deux clades de Papilionoideae ?
- Variabilité d'activité entomotoxique
- Extractions
- Matériel animal et tests biologiques
- Tests d'inhibition de liaison
- Electrophorèses
- Variabilité de l'activité biologique
- Compléments biochimiques
- Activité biologique des extraits de graines
- Analyses complémentaires
- Indications sur le mode d'action?
L'animal doit être sélectionné en fonction de la quantité de protéines à produire (de quelques centaines de milligrammes par an pour une souris à une tonne par an pour une vache). L'induction de la synthèse des protéines recombinantes est déclenchée par l'ajout d'IPTG (isopropyl β-D-thiogalactopyranoside) 0,4 mM. Après surexpression des protéines de fusion TRX-PA1b dans Origami, une partie de la protéine recombinante produite reste sous forme soluble.
Pour augmenter la pureté de la protéine de fusion avant le clivage, une IEC a été réalisée.
Expression hétérologue de PA1b
Systèmes d'expression hétérologue
Systèmes eucaryotes
- Les Levures
- cellules d'insectes
- cellules de mammifères
- Transgénèse
Depuis son développement dans les années 1980, l'utilisation du système d'expression du baculovirus, virus infectant les insectes, pour la synthèse de protéines recombinantes dans les cellules d'insectes s'est généralisée (Luckow et Summers, 1988 ; Murhammer, 1991). Les cellules de mammifères sont généralement considérées comme le meilleur hôte pour l’expression de protéines eucaryotes qui nécessitent des modifications post-traductionnelles pour être actives. Bien que les systèmes transitoires produisent suffisamment de protéines pour les études physico-chimiques, une expression stable est préférée.
Ce travail représentera cependant un système d’activités confirmatives le plus proche de l’expression originale17.
Systèmes procaryotes
- Stratégie générale pour l’expression d’un gène chez E. coli
- Expression dans les différents compartiments cellulaires
- Matériel et méthodes
Le choix de l'espace d'expression se fera en fonction des attentes (nécessité de modifications post-traductionnelles, repliement correct de la protéine d'intérêt, etc.). La translocation à travers la membrane interne nécessite une séquence signal située à l’extrémité N-terminale de la protéine hétérologue. Les séquences signal sont ensuite séparées de la protéine d'intérêt par des peptidases liées à la membrane interne.
Un autre inconvénient potentiel de la sécrétion périplasmique est le mauvais repliement des protéines d'intérêt et donc la formation de corps d'inclusion ou leur dégradation dans le périplasme.
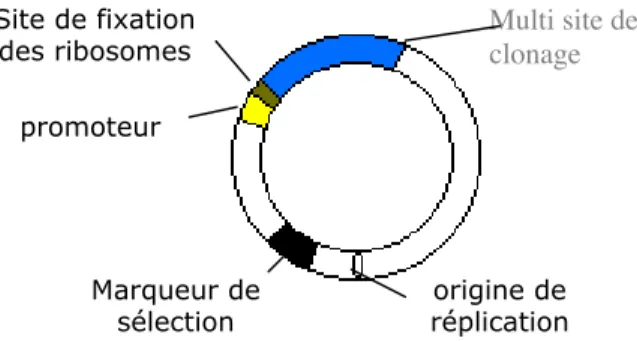
Construction génétique et expression
- Souches et plasmides
- Milieux de culture
- Extraction d’ADN plasmidique
- digestions et ligations d’ADN double brin
- Transformation d’E. coli par un plasmide
- Induction de la surexpression de protéines recombinantes en erlen
Des antibiotiques ont été ajoutés à différents milieux pour sélectionner les clones résistants : ampicilline (50 µg/mL), kanamycine (15 µg/mL) et chloramphénicol (30 µg/mL) (Sigma). 3) Extraction de l'ADN plasmidique. Les digestions d'ADN ont été réalisées par des endonucléases selon les recommandations des fournisseurs (Boeringher Manheim, Biolabs, Amersham). La ligature de l'ADN a été réalisée avec l'enzyme T4 ADN ligase (Biolabs), en utilisant le tampon recommandé par le fournisseur, pendant une nuit à 16°C.
Les culots cellulaires ont été remis en suspension, dans un dixième du volume de départ, ou dans un tampon de fixation non dénaturant (Tris-HCl 20 mM, pH 7,9 ; imidazole 10 mM ; NaCl 0,5 M), ce qui a permis d'extraire les protéines solubles. , ou dans le même liquide. tampon dénaturant (Tris-HCl 20 mM, pH 7,9 ; imidazole 10 mM ; NaCl 0,5 M ; urée 7 M) pour obtenir les protéines totales (extraction pour contrôle d'expression et non pour purification).
Purification et caractérisation des protéines
- Expression des protéines recombinantes en fermenteur
- Purification des protéines de fusion TRX-PA1b par chromatographie
- Purification des protéines fusion par chromatographie d’échange d’ion (IEC)
- Purification des protéines recombinantes par chromatographie d’affinité
- Purification des protéines recombinantes par HPLC
- Suivi des étapes de purification
- Western-Blotting et révélation immunologique
- Caractérisation des protéines par spectrométrie de masse
- Caractérisation de l'activité des protéines
- Résultats
Les culots cellulaires ont été remis en suspension, dans un dixième du volume initial, dans le tampon de fixation (Tris-HCl 20 mM, imidazole 10 mM, NaCl 0,5 M, β-mercaptoéthanol 5 mM pH 7,9) permettant l'extraction des protéines solubles. Les protéines lyophilisées ont été remises en suspension dans du méthanol à 60% (MeOH60), filtrées puis injectées sur une colonne C18 préalablement équilibrée avec 20% de tampon B (Acétonitrile-TFA du tampon A (TFA 0,1%). Les échantillons prélevés en sortie des différents les étapes de purification ont été analysées par électrophorèse.
Les protéines ont été séparées par électrophorèse sur gel dénaturant et transférées électriquement sur une membrane de nitrocellulose comme décrit précédemment.

Constructions génétiques
- Stratégie de construction
- Conditions de la surexpression de la protéine fusion
Aux fins de construction, les deux fragments double brin contiennent, à leur extrémité 3', un site unique pour l'endonucléase de restriction HinDIII, qui permet leur insertion dans le vecteur de construction pPR1 (plasmide blueprint portant une séquence supplémentaire de 39 paires de bases ( bp) ) entre les sites HindIII et EcoRI, en phase avec le gène lacZ (Elmorjani et al., 1997)). La sélection des clones positifs a été réalisée par résistance à l'antibiotique ampicilline, par vérification de la taille de l'insert et par l'acquisition de sites de restriction uniques. Le choix de la thiorédoxine comme partenaire de fusion a également été déterminé par le fait que cette protéine contient un nombre soluble de protéines dont le dépôt dans le cytoplasme d'E.
La vérification de l'expression de la protéine recombinante dans la souche Origami transformée a été réalisée dans un Erlenmeyer sous agitation normale, en milieu LB à 37°C.
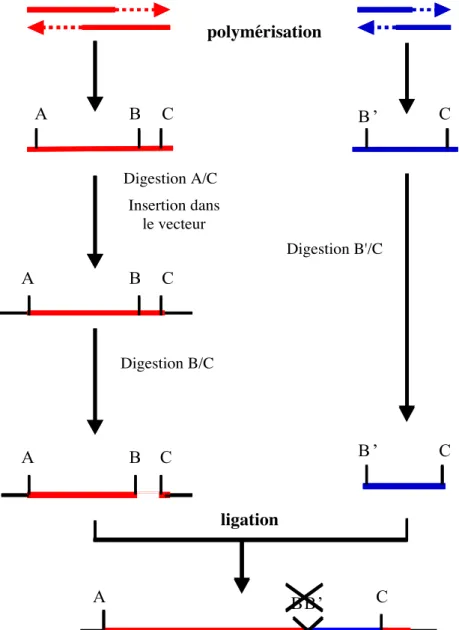
Purification et caractérisation protéiques
- Purification et caractérisation de la protéine fusion
- Clivages
- Purification de rPA1b après clivage acide
- Purification de rPA1b après clivage enzymatique
- Caractérisation de rPA1b
- Discussion
- Evolution de la famille des A1b au sein de la famille des Légumineuses
- Origine des A1b ?
- Relations structure-fonction
- Evolution of the A1b family among the Legume family
- The origins of A1b?
- Structure – function relationships
- Annexe 3 : Liste des espèces sélectionnées, origine géographique et fournisseur254
- Annexe 5 : Temps létaux 50
- Annexe 6 : Plasmide et séquences
La différence de 8,09 indique que les quatre ponts disulfure de la protéine de fusion seront bien formés (six cystéines de PA1b et deux de TRX oxydées). La mise au point de la purification du rPA1b en fin de clivage acide a nécessité de nombreux tests. Ce problème de structure du rPA1b explique également les problèmes rencontrés lors de la purification du peptide.
PA1b est le peptide responsable de la toxicité des graines de pois envers les foreurs des céréales (Delobel et al., 1998).