Une équipe du laboratoire ProBioGEM (Laboratoire des Procédés Biologiques, Ingénierie Enzymatique et Microbienne) travaille sur les peptides non ribosomiques. Nous avons ensuite procédé à l'analyse de peptides non ribosomiques putatifs issus de la séquence du génome (chapitre 6).
Synth`ese peptidique
Acides amin´es
Ainsi, les acides aminés sont classés en groupes ayant des propriétés similaires. Les acides aminés peuvent également être classés selon les propriétés physico-chimiques données par le radical [Voet et al., 2007a].

Peptides et prot´eines
Synth`ese peptidique : le dogme central de la biologie mol´eculaire
Synth`ese peptidique non-ribosomiale
- G´en´eralit´es
- Synth´etases
- Motifs caract´eristiques et sp´ecificit´e des domaines
- Biosynth`ese peptidique non-ribosomiale
Les synthétases ou NRPS (non-ribosomal peptide synthetases) sont les enzymes responsables de la synthèse des peptides non ribosomiques. Les domaines secondaires sont des domaines facultatifs qui modifient les acides aminés incorporés lors de la biosynthèse.

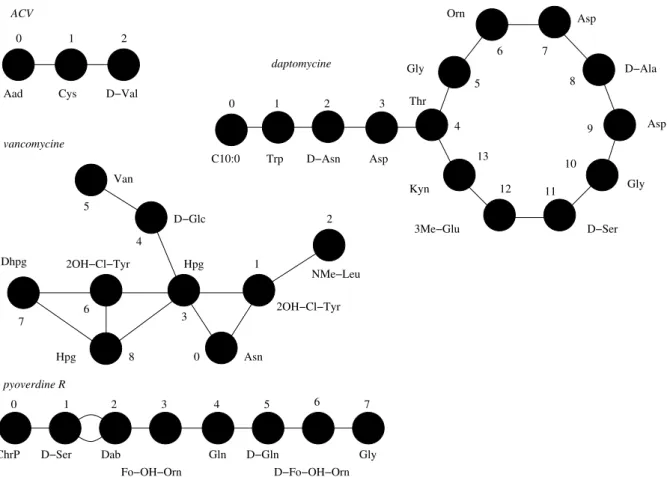
Peptides non-ribosomiaux
Diversit´e de composition
Par exemple, l'acide 3-méthylglutamique et la kynurénine sont deux acides aminés non protéogènes sélectionnés parmi les domaines A et incorporés dans la daptomycine (figure 1.24) [Miao et al., 2005]. La ciclosporine A (figure 1.24) contient des acides aminés N-méthylés dérivés de domaines de méthylation [Lawen et Zocher, 1990].
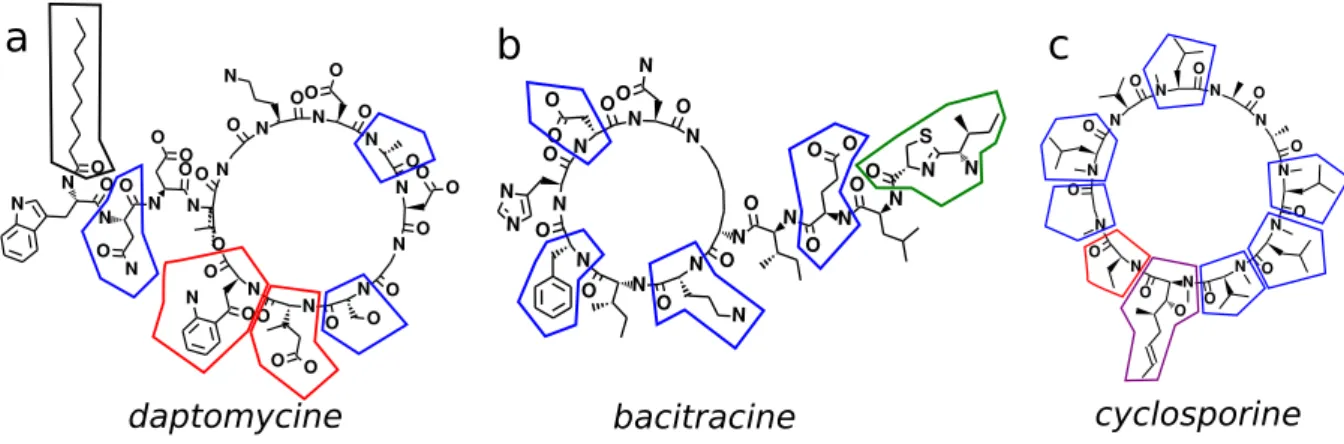
Diversit´e des structures primaires
La voie non ribosomique est souvent associée à d'autres voies de biosynthèse, telles que la voie PKS, ainsi qu'à des enzymes supplémentaires. Elle résulte de la combinaison de plusieurs voies de biosynthèse, dont la voie NRPS et la voie PKS, auxquelles s'ajoutent des réactions de glycosylation, d'oxydation et d'halogénation [Geib et al., 2008].
Diversit´e des activit´es biologiques
La bléomycine A2 et la bléomycine B2, produites par Streptomyces verticillus, sont les principaux composants du Blenoxane, utilisé pour traiter le cancer. Il existe d'autres sidérophores non ribosomiques comme la yersiniabactine, produite par Yersinia pestis [Miller et al., 2002] ou l'entérobactine produite par diverses bactéries [Raymond et al., 2003].
Approches pour l’obtention de nouveaux peptides
- Approches g´en´etiques
- Approches chemo-enzymatiques
- Banques et bases de donn´ees g´en´eralistes
- Banques et bases de donn´ees sp´ecialis´ees
La base de données de référence pour les séquences protéiques est Uniprot. Il contient un outil d'analyse NRPS/PKS (section 2.2.3) et quatre bases de données.
Outils d’analyse des synth´etases et pr´ediction du peptide produit
- Quelques d´efinitions
- PKS/NRPS Analysis Web-site
- NRPS-PKS
- NRPSpredictor
- ClustScan
- Clusean
- Codage des monom`eres
- Repr´esentation lin´eaire
- Mod´elisation par les graphes
Cette étude prouve donc que les 8 acides aminés sont suffisants pour prédire la spécificité des domaines A. Certains acides aminés non protéogènes sont également représentés par un code à trois lettres.
Recherche d’un peptide selon sa composition en monom`eres
Recherche de motifs structuraux
- Mod´elisation des motifs structuraux
- M´ethode classique
- Notre m´ethode
- Tests d’efficacit´e
- Comparaison stricte
Ainsi, la condition (2) de la méthode classique est étendue, augmentant le nombre d’arêtes dans le GC. On remarque qu’il n’y a pas d’arête entre les nœuds a et l dans le GC construit par la méthode classique. Enfin, cet exemple montre également une réduction du nombre d’arêtes dans le GC construit avec la nouvelle méthode.
Par exemple, dans le GC construit avec la méthode classique, une arête existe entre les nœuds b(0,6) et l(4,3).

Extension de la m´ethode ` a la recherche de similarit´es
- Recherche de similarit´es
- Distance entre deux peptides
- Clustering des monom`eres
- Contenu
- Alimentation de la base de donn´ees
La figure 4.3 montre une vue partielle de l'écran focalisée sur la partie « générale » du feuillet d'actinomycine D. La figure 4.4 montre une vue partielle de l'écran centrée sur la partie « structure » du feuillet d'actinomycine D. La figure 4.9 montre une vue partielle de l'écran focalisée sur la partie « structure » du feuillet d'actinomycine D. affichage de la fiche d'actinomycine D. centrée sur la rubrique "connexions".
Pour chaque peptide, la première étape est la recherche de données dans la littérature scientifique.

Interface web
- Technologies utilis´ees
- Recherche basique
- Recherche en fonction des donn´ees structurales
- Recherche de monom`eres
- Autres fonctionnalit´es
Cette section permet à l'utilisateur de rechercher dans la base de données en fonction des différents champs disponibles. Fig.4.15 – Télécharger les données peptidiques. a) liste des peptides correspondant aux critères de recherche b) formulaire de téléchargement c) format XML des données demandées par l'utilisateur. Par exemple, l'utilisateur peut obtenir la liste des peptides cycliques qui sont des tensioactifs produits par le genre Bacillus.
Cette fonctionnalité permet d’obtenir rapidement la conception d’un peptide, qui peut ensuite être modifiée par l’utilisateur.

Statistiques d’utilisation de Norine
Nous avons créé un script qui prend en entrée une liste de peptides et une liste de monomères. Comme vu précédemment, Norine contient toutes les variantes identifiées de la famille des peptides (Section 1.2.4.0). En effet, le nombre d’occurrences de monomères ubiquitaires dans une famille de peptides sera surestimé.
Tab.5.1 – Familles peptidiques présentant des variantes distinctes statut familial nombre moyen de variantes distance.

Statistiques g´en´erales
5.3 – Répartition en taille des peptides « curés » sans variants (175 peptides). 5.4 - La répartition des peptides selon les groupes d'organismes producteurs prenant en compte a) l'ensemble des peptides (1071 peptides) b) les peptides « curés » (790 peptides) issus de champignons est de 27% et celle des peptides identifiés au sein d'autres espèces eucaryotes est de 22 % . La dominance des bactéries reste majoritaire (plus de 60 %) et la proportion des « autres » organismes eucaryotes diminue (moins de 5 %). A l’inverse, la majorité des peptides identifiés dans la catégorie « autres » sont possiblement produits par la voie NRPS, mais peu d’études concernant la biosynthèse de ces peptides ont été réalisées. , plaçant ces peptides dans la catégorie « putatifs ».
Nous avons ensuite étudié la répartition des types de structures rencontrées dans les peptides présents dans la base de données.

Prot´eines ribosomiales versus peptides non-ribosomiaux
5.6 – Répartition des acides aminés protéogènes au sein des protéines et peptides ribosomiques inclus dans UniProtKB/TrEMBL. 5.7 – Répartition des acides aminés protéogènes au sein de peptides « guéris » sans variants Norine. Cela peut s'expliquer par le fait que ces acides aminés possèdent un groupe hydroxyle qui permet la formation d'une liaison covalente supplémentaire dans la chaîne latérale et ainsi l'obtention de structures primaires non linéaires.
Une autre caractéristique intéressante est que pour les protéines classiques, les acides aminés acides (Glu et Asp) sont plus courants que les acides aminés qui ont une fonction amine sur leur chaîne latérale (Gln et Asn).

En fonction des organismes producteurs
Nous avons étudié la distribution des acides aminés protéogènes dans les peptides non ribosomiques synthétisés par des bactéries et dans ceux synthétisés par des champignons. La figure 5.11 montre la répartition des acides aminés protéogènes au sein des peptides « composites » et hors variants synthétisés par les champignons (34 peptides). Nous avons calculé la répartition des acides aminés protéogènes au sein des peptides synthétisés par les métazoaires (Figure 5.12).
Nous avons calculé la distribution de la taille des peptides synthétisés par des bactéries et ceux synthétisés par des champignons.

En fonction des cat´egories chimiques
5.17 – Répartition des trente monomères les plus courants au sein des peptides (non-lipopeptides, glycopeptides et peptaibols) « guéris » et sans variants (127 peptides). 5.20 – Répartition des trente monomères les plus courants au sein d'un lipopeptide « cu-rated » sans variants (26 peptides). La figure 5.20 montre la répartition des trente monomères les plus courants au sein du lipopeptide.
5.21 – Répartition des 27 monomères identifiés au sein des peptaibols « curated » sans variants (20 peptides).

En fonction des activit´es biologiques
La figure 5.26 montre la répartition des trente monomères les plus courants. 5.27 – Répartition des trente monomères les plus courants au sein des sidérophores « cu-graded » (88 peptides). 5.28 – Répartition des trente monomères les plus courants au sein des tensioactifs « ku-rated » (130 peptides).
Fig.5.29 – Répartition des trente monomères les plus abondants au sein des toxines « composites » (142 peptides).

Aide ` a la pr´ediction de l’activit´e biologique
M´ethode
Pour chaque monomère, nous avons calculé le nombre d'occurrences de ce dernier monomère au sein des six grandes classes d'activité biologique : antibiotique, immunomodulateur, antitumoral, surfactant, toxine et sidérophore. Après avoir normalisé le nombre d’occurrences de chaque monomère, nous calculons sa fréquence au sein des différentes classes d’activité. On obtient ainsi un fichier contenant pour chaque monomère la fréquence au sein des six classes d'activité principales.
Pour prédire l'activité biologique d'un peptide, nous faisons la moyenne des fréquences de chaque monomère qui le compose au sein des six classes d'activité.
Tests
Ce premier ensemble de données provient de données confirmées de Norine. Nous souhaitons donc que la méthode de prédiction soit capable de classer correctement toutes ces données. Deuxièmement, nous avons réalisé les mêmes expériences avec un ensemble de données composé de 69 peptides pouvant représenter plusieurs activités.
Les résultats obtenus avec l'ensemble de données contenant 69 composés peptidiques pouvant présenter différentes activités sont présentés dans le tableau 5.6.

Exemples de pr´edictions
Ces tests permettent de valider notre méthode de prédiction basée sur la fréquence des monomères au sein des différentes classes d'activités. La prédiction faite pour ce peptide est fausse car elle le prédit comme un tensioactif. Dans cette section, nous avons recherché des peptides non ribosomiques « sélectionnés » non encore intégrés dans Norine pour valider notre méthode de prédiction de l’activité biologique.
Enfin, nous utilisons notre outil de prédiction d’activité biologique pour identifier l’activité potentielle du peptide d’intérêt.
Analyse du g´enome de Lactococcus lactis
Nous recherchons d’abord la structure de Norine pour voir si le peptide a déjà été identifié. On recherche alors des peptides ayant une composition en monomères proche de celle du peptide putatif, augmentant progressivement le nombre d'erreurs possibles. Ensuite, à l’aide de la recherche de motifs, nous recherchons la structure ou une sous-structure commune entre le peptide étudié et les peptides de la base de données.
La recherche de peptides similaires permet alors d’identifier des peptides dont les caractéristiques (composition et structure des monomères) sont proches du peptide étudié.
Analyse du g´enome de Pseudomonas entomophila
- Cluster 1
- Cluster 2
- Cluster 3
- Cluster 4
Dans un premier temps, nous avons recherché les gènes codant pour les synthétases impliquées dans la synthèse du chromophore. On peut donc conclure que le gène du locus PSEEN1815 ne code pas pour la synthétase responsable de la synthèse du chromophore pyoverdine. A partir des résultats de cet article, nous avons recherché les différents gènes impliqués dans la synthèse de pyoverdine potentielle dans le génome de P.
Nous avons donc validé expérimentalement la synthèse d'une nouvelle pyoverdine par cette souche.

Validations exp´erimentales
Mat´eriels et m´ethodes
Toute la vaisselle est débarrassée des traces de fer par traitement à l'acide nitrique à 10 % pendant au moins 2 heures et soigneusement rincée à l'eau ultra pure. La production de pyoverdines est suivie par mesure de l'absorbance à 400 nm (instrument Spectrophotomètre Uvikon-Kontron) après dilution dans du tampon phosphate de sodium 50 mM pH 7 (1/1, V/V). Quelques colonies isolées sur King B sont mises en suspension dans 1 ml de CAA pour ensemencer 200 ml du même milieu dans un flacon d'un litre exempt de toute trace de fer.
R´esultats - Discussion
Exploiting the selectivity-conferring code of non-ribosomal peptide synthetases for the rational design of novel peptide antibiotics. Daptomycin biosynthesis in Streptomyces roseosporus: cloning and analysis of the gene cluster and revision of peptide stereochemistry. Identification of novel, conserved, non-ribosomal peptide synthetases from fluorescent pseudomonads involved in the biosynthesis of the siderophore pyoverdine.
Identification of genes involved in the biosynthesis of the cytotoxic compound glidobactin from a soil bacterium.