Phénotype de réparation de l'ADN dans les lignées de Xeroderma pigmentosum, à l'aide d'un test multiparamétrique in vitro. Un résultat possible est la réparation de l’ADN, un processus qui restaure la séquence d’ADN intacte.

L’ADN, une molécule fragile et réparée
La molécule d’ADN
Le polymère d'ADN peut être endommagé par plusieurs mécanismes, conduisant à l'apparition de lésions dans le génome. Nous nous concentrerons dans un premier temps sur l’analyse de différents types de dommages à l’ADN puis étudierons les conséquences de ces lésions sur l’organisme vivant.

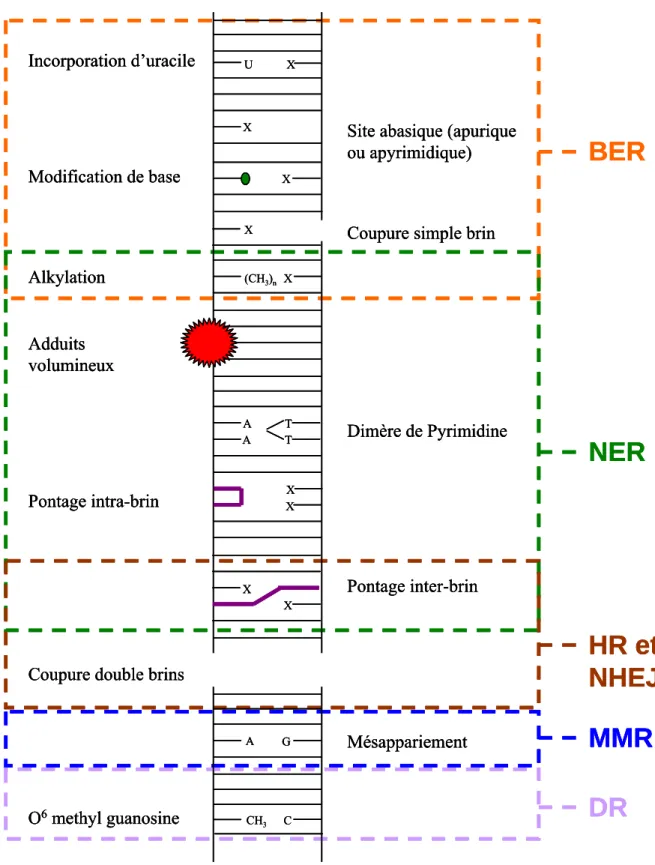
Les lésions de l’ADN
- Les « petites » lésions de l’ADN
- Les lésions volumineuses
- Les cassures de chaîne
- Les mésappariements de base
Ces lésions importantes impactent l'organisation tridimensionnelle de l'ADN (dénaturation locale, courbure de la double hélice). a) Dimères de pyrimidine. L'adduit benzo[A]pyrène (B[a]P) résulte de la réaction des HAP (hydrocarbures aromatiques polycycliques) avec l'ADN.
Lésions de l’ADNcellule
Lésions de l’ADN
Les conséquences cellulaires des lésions de l’ADN
Les différents dommages à l’ADN sont capables d’activer des points de contrôle tout au long du cycle cellulaire. Premièrement, l’ADN peut être réparé en supprimant les dommages pour retrouver une molécule d’ADN intacte.
HR et NHEJ
La réparation de l’ADN chez l’Homme
Chez l'homme, les bases d'ADN alkylées telles que l'O6-méthylguanine peuvent être réparées par réversion directe, les mésappariements de bases sont gérés par le système MMR (MisMatch Repair) et les cassures double brin sont réparées par recombinaison homologue (HR) ou par des bases d'ADN mésappariées. recombinaison terminale homologue (NHEJ). Les sites abasiques, les bases oxydées, les bases alkylées, l'uracile dans l'ADN, les cassures simple brin sont des lésions de l'ADN supportées par le système d'excision des bases (BER).

La mutagénèse et la cancérisation
Enfin, la mutation est dite non-sens lorsqu'elle introduit un codon terminateur (STOP) qui conduit à la synthèse d'une protéine tronquée et souvent inactive. Un autre modèle pour expliquer le phénomène de formation du cancer est la survenue d'une mutation primaire dans un gène associé à la réparation de l'ADN ou dans celui d'une polymérase (Loeb et al., 1974).
L’apoptose
Les dommages à l'ADN conduisent préférentiellement à la mort par apoptose, plutôt qu'à la nécrose ou à l'autophagie (Roos et al., 2006). En effet, comme nous l'avons vu précédemment, les dommages à l'ADN peuvent bloquer la réplication (Figure 9) (Roos et al., 2006).

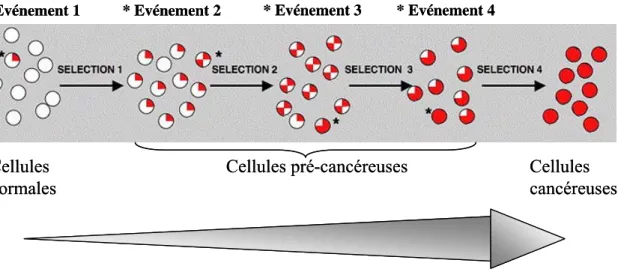
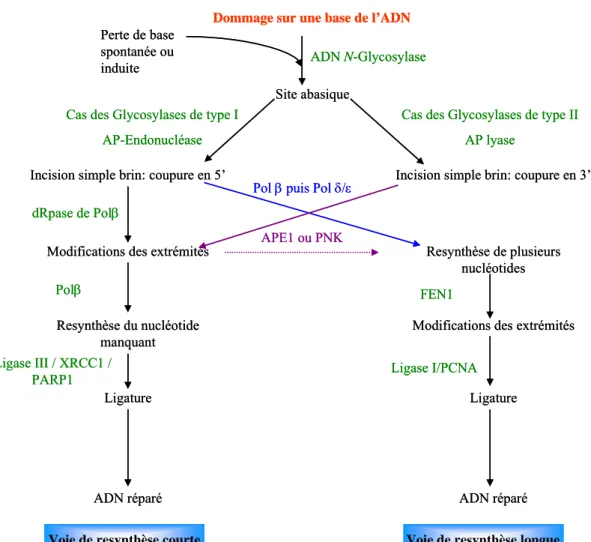
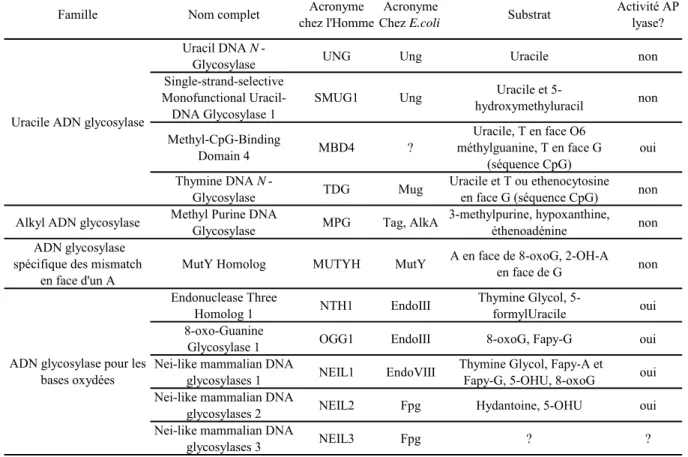
La réparation de l’ADN : deux mécanismes d’excision- resynthèse (BER et NER)
Les cellules déficientes en NER sont hypersensibles aux UV ; ceci est en grande partie dû à l'induction de l'apoptose dans ces cellules (Dunkern et al., 2001). Ces dommages modifient la structure chimique de l'ADN, mais ne déforment pratiquement pas la double hélice.
La vision classique des mécanismes de réparation par excision- resynthèse
- Réparation par excision de base : BER
- Réparation par excision de nucléotides : NER
Cependant, le rôle de l'APE2 dans le système BER semble très négligeable (Hegde et al., 2008). Le magnésium affecte à la fois l'interaction avec l'ADN et le clivage de la liaison phosphodiester (Masuda et al., 1998).
TC-NER
Voies alternatives
- AP endonucléase et la voie NIR (Nucleotide Incision Repair)
- La glycosylase NEIL, enzyme alternative pour la réparation des adduits et pontages inter-brins et pontages inter-brins
- Endonucléase V
L'endonucléase V (Endo V) purifiée dans E. coli coupe 3' de la lésion (Demple et al., 1982). L'enzyme coupe la liaison phosphodiester en 3' du nucléotide après le nucléotide endommagé. Cette coupe 3' est ensuite suivie d'une coupe 5' de la lésion avec APE1 ou le complexe MUS81/MMS4.
Interaction entre les systèmes de réparation
- Rôle des protéines XP dans la réparation des dommages oxydatifs
- Rôle des protéines CS dans la réparation des dommages oxydatifs : couplage de la BER et de la transcription couplage de la BER et de la transcription
- Le Xeroderma pigmentosum
Il a également été démontré que les facteurs hHR23 (A et B) qui jouent un rôle stimulateur de la reconnaissance des dommages par XPC lors de la NER peuvent également jouer ce rôle avec l'ADN méthyl purine glycosylase (MPG), une enzyme impliquée dans la réparation des bases alkylées. (Miao et al., 2000). Cependant, le rôle du XPA dans la réparation des dommages oxydatifs est beaucoup moins élucidé que celui du XPC (Dusinska et al., 2006). Le RPA semble pouvoir interagir avec l'UNG, une enzyme impliquée dans la réparation de l'uracile dans l'ADN (Nagelhus et al., 1997 ; Mer et al., 2000).
Signes cliniques et origines de la maladie
- Description de la maladie
- Les patients XP sont déficients pour la NER
- Spécificités cliniques des différents groupes de complémentation
Bien que les domaines hélicase soient essentiels pour le NER, ils ne le sont pas pour l'initiation de la transcription (Stary et al., 2002). Les patients atteints de XPE présentent une mutation dans la partie codante de DDB2 (Rapic-Otrin et al., 2003). f) XPF. Comme TFIIH, XPG pourrait jouer un rôle dans le couplage de la réparation et de la transcription de l'ADN (de Laat et al., 1999).

Diagnostic et traitement
Pol ζ est encore moins fidèle que Pol η, provoquant une forte probabilité de mésappariement lors de la polymérisation. Lorsque la lésion est pontée par Pol η ou ζ, les polymérases réplicatives classiques reprennent leur activité de polymérisation. Les polymérases translésionnelles semblent moins processives que les polymérases classiques, ce qui faciliterait leur clivage de l'ADN quelques nucléotides après la lésion (Washington et al., 1999).
Revue sur XPA
- Le gène XPA
- La protéine XPA
- Les différentes mutations dans le gène XPA
- Le ou les rôle(s) de XPA
- Méthodes pour étudier la réparation de l’ADN
Le renouvellement de la protéine XPA sera plus lent que prévu par les calculs théoriques (Koberle et al., 2006). Les patients XPA les plus touchés présentent des mutations homozygotes (sur les deux allèles) dans la région du gène qui permet l'attachement de la protéine XPA à l'ADN (domaine central : exons III, IV et V) (Bartels et al., 2007). Le complexe vérifiera également l'assemblage correct des facteurs de la voie NER (Missura et al., 2001).

Techniques d’imagerie : regarder la distribution des protéines de réparation
L'analyse de la cinétique de récupération de fluorescence (FRAP) permet d'en déduire le coefficient de diffusion latérale « D », caractéristique de la vitesse de déplacement des molécules dans le milieu (plus la vitesse de diffusion est élevée, plus la fluorescence augmente rapidement). L’une des limites de ces techniques réside dans l’effet de marquage fluorescent de la protéine d’intérêt. En effet, il est fort possible que la fusion avec la GFP puisse altérer la mobilité de la protéine d'intérêt (Koster et al., 2005).
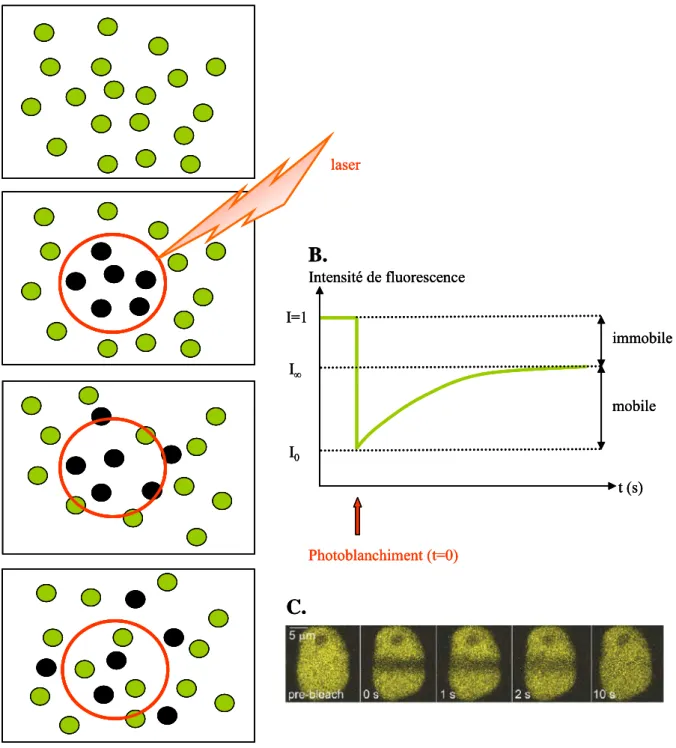
Tests in vivo : mesure d’activités de réparation au sein de la cellule Contrairement aux techniques d’imagerie, qui nous apportent une information qualitative sur
- Le test UDS (Unscheduled DNA Synthesis)
- Le test HCR (Host Cell Reactivation)
Cependant, il est important de noter que ce faible niveau de dNTP n'empêche pas la synthèse de l'ADN lors de la réparation (Friedberg et al., 2006). Le principal avantage de la synthèse non programmée de l’ADN est qu’elle permet de mesurer la réparation in cellulo. Ce test n'est pas un bon indicateur quantitatif car il dépend de la quantité cellulaire de thymidine (Friedberg et al., 2006).
Tests in vitro : mesure d’activité de réparation en dehors de la cellule
- Du lysat cellulaire au système reconstitué de protéines purifiées
- Utilisation de lysats cellulaires
- Systèmes reconstitués avec des protéines purifiées
- Comparaison de notre test avec le test de référence
- Les contraintes du test sur la puce plasmide
- Définition et illustration des paramètres mesurés sur la puce plasmide
Enfin, l'autoradiographie permet de quantifier la radioactivité incorporée lors de la réparation plasmidique. Les conditions ioniques lors de la réaction d'excision-resynthèse à partir des lysats cellulaires sont très importantes. Et si on réparait toutes ces lésions sur la puce plasmidique.

Quantification des signaux de fluorescence
La réparation du 8-oxoG est associée à une intensité de fluorescence plus élevée lorsque l'on utilise le Cy5-dGTP qu'avec le Cy5-dCTP, ce qui favorise l'incorporation d'un G à la place de la base endommagée. . Pour chaque type de lésion, les intensités de fluorescence obtenues pour les trois rapports quantité de lésion/quantité d'ADN (A, B et C) ont été additionnées. Le niveau de fluorescence obtenu pour le lysat de référence est alors fixé à 100%, et les intensités de fluorescence des autres lysats sont exprimées par rapport au lysat témoin.

Définition du phénotype réparation
Les résultats présentés dans cette thèse sont exprimés soit en intensité de fluorescence totale obtenue pour chaque type de lésion et chaque rapport lésion/ADN, soit en pourcentage de l'intensité de fluorescence totale du lysat de référence. Le lysat de référence est préparé à partir de cellules témoins, c'est à dire celles qui ne présentent pas de défauts de réparation de l'ADN.
Le niveau de fluorescence du plasmide contrôle
- Variabilité des intensités de fluorescence entre les expériences
- Caractérisation d’activités de réparation de l’ADN au niveau basal
- Caractérisation d’activités de réparation de l’ADN en réponse à un traitement génotoxique
Bien que l’intensité totale de fluorescence varie d’une expérience à l’autre, nous veillons systématiquement à ce que la fraction relative de réparation des 6 lésions reste constante. Pour chaque lysat XP, l'intensité de fluorescence totale est exprimée en pourcentage de l'intensité de fluorescence du lysat témoin. Pour chaque expérience, nous avons exprimé l'intensité de fluorescence obtenue pour un lysat XP en pourcentage par rapport au lysat de référence, à savoir le lysat de cellules normales.
Survie cellulaire après une irradiation UVB
L’irradiation UV permet-elle de distinguer les phénotypes de réparation du lysat XPA par rapport au contrôle ? Par exemple, il a été montré qu'après irradiation UVB des fibroblastes primaires à 500 J/m² (cytotoxicité de 30%), la cinétique de réparation des différents photoproduits n'est pas la même que si les cellules étaient irradiées à 50 J/m², très faible cytotoxicité. dose (Courdavault et al., 2004b). Activité de réparation de l'ADN en réponse à l'irradiation UV. Nous avons constaté que les lysats de cellules Hela siRNA XPA et XPC étaient irradiés avec.
Activité de réparation de l’ADN en réponse à une irradiation UV Nous avons constaté que les lysats de cellules Hela siRNA XPA et XPC, irradiées à
Ainsi, la stimulation des activités de réparation en réponse aux rayonnements UVB concerne paradoxalement principalement les dommages pris en charge par le BER. En effet, le niveau de réparation de toutes les lésions présentes sur la puce chute lorsque les cellules sont irradiées à cette dose. Ainsi, les activités de réparation que l'on voit stimulées 24 heures après l'irradiation des cellules avec 5 J/m² ne sont pas stimulées si les cellules étaient irradiées avec une dose plus élevée.

Caractérisation des lysats cellulaires
Dans nos conditions de lyse, la protéine XPA est bien présente dans le noyau. Cette observation contredit la localisation cellulaire du XPA décrite au chapitre I (IV. C. 2. b.), où il a été montré que le XPA est essentiellement localisé dans le cytoplasme. Cependant, la localisation de cette protéine ne semble pas clairement définie, la localisation majoritairement nucléaire de XPA ayant également été évoquée dans la littérature (Miura et al., 1991).

Mesure d’activités d’excision-resynthèse
- Excision-resynthèse d’un lysat nucléaire témoin
Nous avons ensuite cherché à connaître l'effet de la concentration en protéines lors de la réaction ER réalisée sur le support. La vitesse initiale de la réaction ER augmente d'un facteur 2,5 lorsque la concentration est multipliée par 3. Nous allons maintenant nous concentrer sur l'effet de la concentration en protéines sur la répartition de la réparation de diverses lésions.

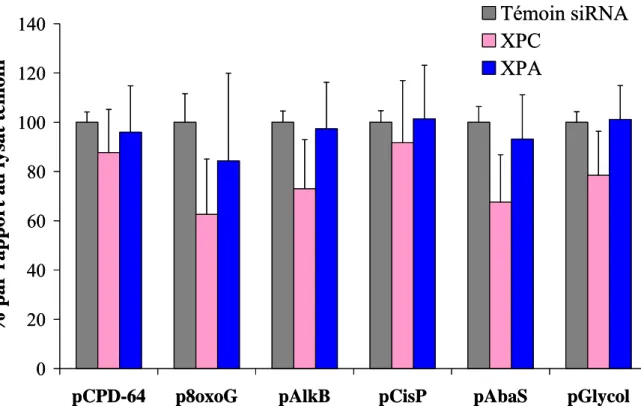
Caractérisation de phénotypes de réparation XPA et XPC
En revanche, comme pour le lysat XPC, le taux relatif de ER obtenu pour les pyrimidinediols était indépendant de la concentration protéique. En effet, le taux relatif obtenu à 1 mg/ml de protéines avec le lysat XPA est d'environ 65. Cependant, le taux de récupération des photoproduits est inférieur à celui obtenu pour le lysat témoin.

Excision seule
- Activités d’excision d’un lysat nucléaire témoin : détermination des conditions de concentrations protéiques conditions de concentrations protéiques
- Caractérisation de phénotypes d’excision XPA et XPC
Activités d'excision d'un lysat nucléaire témoin : détermination des conditions de concentration en protéines. Les conditions de concentration en protéines. Pour ce faire, nous avons réalisé une expérience sur l'oligopuce en présence d'une gamme de concentration de 0 à 20 µg/ml pour un lysat nucléaire de fibroblastes témoins (Figure 48). Le profil d'excision obtenu sur la Figure 48 A/ montre ainsi l'intensité totale de fluorescence obtenue pour un lysat nucléaire témoin par rapport à cette fluorescence 100%.