Pró-Reitoria Acadêmica
Escola de Saúde
Programa de Pós-Graduação Stricto Sensu em
Ciências Genômicas e Biotecnologia
CLONAGEM E CARACTERIZAÇÃO MOLECULAR DE GENES
DE CICLOTÍDEOS EM PLANTAS DO CERRADO
Autor: Renato Goulart de Almeida
Orientador: Prof
a. Dr
a. Simoni Campos Dias
RENATO GOULART DE ALMEIDA
CLONAGEM E CARACTERIZAÇÃO MOLECULAR DE GENES
DE CICLOTÍDEOS EM PLANTAS DO CERRADO
Dissertação apresentada ao Programa
de Pós-Graduação Stricto Sensu em
Ciências Genômicas e Biotecnologia da
Universidade Católica de Brasília, como
requisito para obtenção do Título de
Mestre em Ciências Genômicas e
Biotecnologia.
Orientador: Prof
a. Dr
a. Simoni Campos
Dias
Coorientador: Prof. Dr. Aulus Estevão
Anjos de Deus Barbosa
7,5cm
Ficha elaborada pela Biblioteca Pós-Graduação da UCB
A447c Almeida, Renato Goulart de.
Clonagem e caracterização molecular de genes de Ciclotídeos em plantas do cerrado. / Renato Goulart de Almeida – 2013.
97 f.; il: 30 cm
Dissertação (mestrado) – Universidade Católica de Brasília, 2013. Orientação: Profa. Dra. Simoni Campos Dias
Coorientação: Prof. Dr. Aulus Estevão Anjos de Deus Barbosa
1. Biotecnologia. 2. Organismos transgênicos. 3. Ciclotídeos. 4. Parigidina-br2. 5. Palicourea rigida. 6. Hyla-br1. 7. Hybanthus lanatus. I. Melo, Gislane Ferreira de, orient. II. Barbosa, Aulus Estevão Anjos de Deus, coorient. III. Título.
'\
Católica de Brasília
;
Dissertação de autoria de Renato Goulart de Almeida, intitulada "CLONAGEM E CARACTERIZAÇÃO MOLECULAR DE GENES DE CICLOTIDEOS EM PLANTAS DO CERRADO.", apresentada como requisito parcial para obtenção de grau de Mestre em Psicologia da Universidade Católica de Brasília, em 25 de junho de 2013, defendida e aprovada pela banca examinadora abaixo assinada:
Profa. Dra. Simoni Campos Dias Orientadora
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia- UCB)
Prof. Dr. Aulus Estevão Anjos de deus Barbosa Coorientador
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia- UCB)
Prof. Dr. Leonardo Liam PepinÓ de Macedo Membro Extemo
(Embrapa)
Prof.
dイセ|ャZI]gッョ。ャカ・セ@
de BruTos
Membro Intemo \
(Programa de Pós Graduação Stricto Sensu em Ciências Genômicas e Biotecnologia- UCB)
AGRADECIMENTO
Agradeço à Professora Doutora Simoni Campos Dias pela orientação dessa
Dissertação de Mestrado, por ser uma profissional presente, preocupada e
rigorosa, me ajudando de forma imensurável durante o desenvolvimento do
projeto, hora como chefe, hora como mãe.
Agradeço ao Professor Doutor Aulus Estevão Anjos de Deus Barbosa, pelos
seus conhecimentos práticos em biologia molecular, os quais foram
extremamente importantes durante todas as etapas do projeto, pela amizade e
por ser uma pessoa ímpar dentro do mundo científico, com a qual tive a honra de
dividir a bancada.
Agradeço ao Professor Doutor Antônio Américo Barbosa Viana, pela ajuda
durante finalização do projeto, pelas correções em minha escrita do idioma inglês,
pela amizade e por ser uma pessoa maravilhosa com a qual tive o prazer de
conviver ao fim do projeto.
Agradeço à Professora Doutora Cássia Beatriz Rodrigues Munhoz, pelo
auxílio na escolha das espécies deste projeto com sua experiência botânica, e
pela ajuda nas coletas realizadas no campo.
Agradeço à Professora Doutora Maria Sueli Soares Felipe, por disponibilizar
seu laboratório, alunos e reagentes para realização dos experimentos de
Southern Blot.
“Deixai toda a esperança,
ó vós que entrais!”
RESUMO
Referência: ALMEIDA, R. G.
CLONAGEM E CARACTERIZAÇÃO MOLECULAR DE
GENES DE CICLOTÍDEOS EM PLANTAS DO CERRADO.
2013. 103 folhas.
Dissertação (Mestrado em Ciências Genômicas e Biotecnologia)-Universidade
Católica de Brasília, Brasília, 2013.
Ciclotídeos representam uma classe específica de peptídeos cíclicos de plantas que possuem entre 28-37 resíduos de aminoácidos e um claro padrão de seis cisteínas ligadas por três pontes de dissulfeto, formando um motivo conhecido como CCK. Inicialmente descobertos nas familias Rubiaceae, Violaceae e Curcubitaceae, também são encontrados nas familias Fabaceae, Solanaceae e Poaceae, os ciclotídeos têm uma estrutura altamente estável e apresentam várias atividades biológicas, incluindo anti-HIV, antitumoral, antimicrobiana e inseticida. Devido ao alto potencial biotecnológico dos ciclotídeos, Palicourea rigida (Rubiaceae), Hybanthus lanatus (Violaceae) e Clitoria sp. (Fabaceae) foram usadas como alvos para estudos gênicos, com o objetivo de clonar e caracterizar genes de ciclotídeos destas espécies, que ocorrem no Cerrado. Folhas de cada planta foram submetidas à extração de RNA total e síntese de cDNA, o qual foi utilizado como molde em PCR contendo iniciadores desenhados em sequências específicas de DNA de ciclotídeos. Em P. rigida, um gene completo de ciclotideo foi isolado, denominado Pri1, possuindo uma ORF com 249 pb que codifica uma proteína precursora de 82 aminoácidos, contendo um novo ciclotideo denominado parigidina-br2. O novo gene encontrado apresenta uma configuração semelhante aos genes de Chassalia chartacea (Rubiaceae), que codificam os menores precursores conhecidos, apresentando um peptídeo sinal (ER-Signal), um pequeno pró-domínio N-terminal (NTPD), domínio de repetição do N-terminal (NTR), domínio do ciclotideo maduro (CMD) e região C-terminal (CTR). Para efeito de comparação de sequência, o DNA genômico (gDNA) de P. rigida foi extraído e utilizado como molde em PCR, gerando duas amplificações: um fragmento menor de ~400 pb e um maior com ~550 pb. O menor fragmento exibiu a mesma sequência do mRNA, enquanto o maior revelou um intron com 131 pb, encontrado dentro da região codificadora do ER-Signal, como é encontrado em todos os precursores de ciclotídeos de Rubiaceae. Para a caracterização molecular completa, o gDNA de P. rigida foi digerido com as enzimas EcoRI e AluI e posteriormente marcado com digoxigenina para análises de Southern Blot, revelando várias cópias do gene parigidina-br2 no genoma. Em H. lanatus, um novo gene de ciclotideo foi detectado, nomeado Hyla1, possuindo uma ORF com 330 pb que codifica uma proteina precursora de 109 aminoácidos. Este precursor compartilha a mesma estrutura característica dos genes de Violaceae, apresentando todos os domínios: ER-Signal, NTPD, NTR, MCD e CTR. Dentro do MCD existe uma sequência de ciclotideo totalmente nova, com 31 aminoácidos e muito semelhante a outros ciclotídeos de Violaceae, denominado hyla-br1. Nenhum gene codificador de ciclotídeos foi encontrado em Clitoria sp. Em suma, foram isolados dois novos genes que codificam precursores de ciclotídeos a partir de plantas do Cerrado, oferecendo novos conhecimentos sobre a arquitetura gênica de ciclotídeos encontrados no Brasil, podendo ser utilizados em um futuro próximo como ferramenta biotecnológica para o desenvolvimento de organismos geneticamente modificados, visando à produção de ciclotídeos em larga escala ou ao desenvolvimento de organismos transgênicos possuindo resistencia microbiana e/ou contra insetos.
ABSTRACT
Cyclotides are a specific class of plant cyclic peptides containing 28-37 amino acid
residues and a consistent pattern of six cysteines linked by three disulfide bonds,
forming a motif known as Cyclic Cysteine Knot. Cyclotides were initially isolated from
Rubiaceae, Violaceae and Curcubitaceae plant families, and further isolated from
Fabaceae, Solanaceae and Poaceae families. Cyclotides have a highly stable
structure that presents several biological activities, including anti-HIV, antitumor,
antimicrobial and insecticidal. Due to the high biotechnological potential of cyclotides,
Palicourea rigida
(Rubiaceae),
Hybanthus lanatus
(Violaceae) and
Clitoria
sp.
(Fabaceae) were used as targets for genic studies, aiming to clone and characterize
cyclotide genes from these Cerrado species. Leaves from each plant were subjected
to total RNA extraction, cDNA synthesis and used as template in PCR, with primers
designed against specific cyclotides DNA sequences. In
P. rigida,
a full length
cyclotide gene was discovered, named Pri1, with an ORF of 249 bp and a
translational product of 82 amino acids residues, encoding a precursor protein of a
novel cyclotide, named parigidin-br2. The gene isolated in this work present a very
similar configuration when compared to
Chassalia chartacea
(Rubiaceae) genes,
which encode the smallest precursors ever found, presenting an ER-Signal, a short
N-terminal Prodomain (NTPD), an N-terminal Repeat Domain (NTR), a Mature
Cyclotide Domain (MCD) and a C-terminal Region (CTR). For sequence comparison,
P. rigida
genomic DNA (gDNA) was extracted and used as template in PCR,
generating two amplicons: the smallest ~400 bp fragment and a larger ~550 bp
fragment. The smallest fragment exhibited the same sequence as the mRNA, while
the largest one revealed an 131 bp intron, comprised into the ER-Signal region, as is
found in all Rubiaceae cyclotides producers. For complete molecular
characterization, P. rigida gDNA was and digested with EcoRI and AluI enzymes and
further labeled with digoxigenin for Southern Blot analysis, revealing several copies
of parigidin-br2 gene in the plant genome. In
H. lanatus, a novel cyclotide gene was
detected, named Hyla1, with an ORF of 330 bp and a 109-amino acids residues
translational product. This precursor shares the same characteristic structural feature
of the Violaceae genes, presenting all domains previously described. Moreover, there
is a totally unknown cyclotide sequence in the MCD, with 31 amino acid residues and
very similar to other Violaceae cyclotides, named hyla-br1. No cyclotide encoding
gene was found in
Clitoria
sp. in this work. In summary, two novel genes encoding
cyclotide precursors were isolated from Cerrado plants, providing new insights into
Brazilian cyclotides genic architecture, which can be further used in a near future as
biotechnological tool for genetically modified organism development, aiming
large-scale cyclotides production or development of transgenic organisms harboring
microbial and/or insect resistance.
LISTA DE ILUSTRAÇÕES
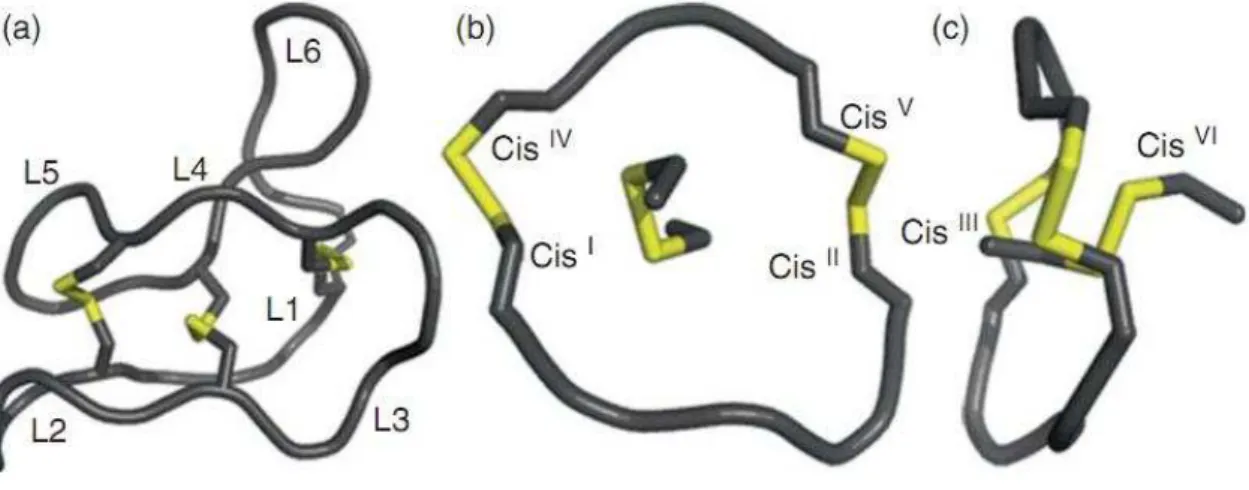
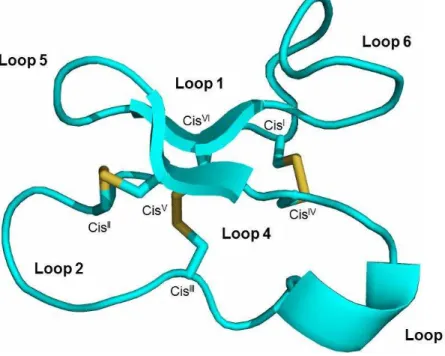
Figura 1 - Estrutura tridimensional do ciclotídeo kalata B1, exemplificando elementos gerais dos ciclotídeos, onde a cadeia principal é colorida em cinza e as pontes dissulfeto são coloridas em amarelo. (a) Os resíduos de cisteína atuam como fatores limitantes para os loops, onde os seis presentes na estrutura estão nomeados de L1-L6. (b) Além da ciclização da cadeia principal, o motivo CCK é definido por seis resíduos de cisteína que formam duas pontes dissulfeto paralelas (CisI-CisIV e CisII-CisV) (c) e por uma terceira ponte dissulfeto perpendicular
à estrutura (CisIII-CisVI), que penetra o anel a 90º de angulação. (Adaptado de WESTERMANN e
CRAIK, 2010) ... 20
Figura 2 - Alinhamento de resíduos de aminoácidos de ciclotídeos das subfamílias Braceletes, Möbius e Inibidores de Tripsina. As sequências foram alinhadas de acordo com a ordem dos loops (1-6). A cor amarela marca os resíduos de cisteína altamente conservados (I-VI); a cor vermelha realça a cis-prolina presente no loop 5 dos integrantes da subfamília Möbius. Os pontos
finais indicam o C-terminal dos ciclotídeos, onde ocorre a ciclização da molécula ... 22
Figura 3 - Organização geral de um típico precursor proteico linear de ciclotídeo. Os precursores podem ser divididos em duas regiões distintas: uma inicial que não se repete, envolvendo o ER-Signal (cinza) e o NTPD (azul); e uma posterior repetida, agrupando o NTR (verde), o MCD (roxo) e o CTR (vermelho), que pode estar presente de uma a três vezes, possuindo ciclotídeos maduros idênticos ou diferentes entre si. O domínio MCD representa o ciclotídeo maduro, no qual existe um padrão altamente conservado de seis resíduos de cisteína e é encontrado em todos os ciclotídeos conhecidos até agora, sendo responsável pela formação das pontes dissulfeto, as quais delimitam os loops do peptídeo (Loop 1-Loop 6), e pela estruturação do motivo CCK. Os números abaixo dos domínios indicam as variações no tamanho dessas regiões em quantidade de aminoácidos, que são diferentes para cada
precursor ... 27
Figura 4 - Alinhamento de quatro precursores proteicos de O. affinis (OaK1-5), com a região do ciclotídeo maduro (MCD) no centro, flanqueada pela região repetida do N-terminal (NTR) e ela cauda C-terminal (CTR). Os resíduos em vermelho indicam posições de aminoácidos conservados, como as cisteínas, a glicina inicial, os aminoácidos terminais (RN/KD) e o início da
da cauda (XLP), importantes no processamento do ciclotídeo (Adaptado de QIN et al., 2010) ... 29
Figura 5 - Etapas do processamento pós-traducional da proteína precursora linear, incluindo o enovelamento do domínio do ciclotídeo maduro (MCD) (verde), através da formação das pontes dissulfeto, sua excisão do precursor e ciclização, intermediada por endopeptidases. A região azul representa o NTR e a região amarela representa a CTR. Representação esquemática do modelo proposto de produção dos ciclotídeos, ocorrendo primeiro o enovelamento oxidativo e posteriormente a excisão e ciclização do MCD (Adaptado de QIN et
al., 2010) ... 30
Figura 6 - Diferentes precursores proteicos existentes em O. affinis, onde OaK1 representa um precursor simples, OaK2 representa um precursor duplo e OaK3 representa um precursor triplo. Em cinza: região do peptídeo sinal (ER-Signal), em azul: pró-domínio N-terminal (NTPD), em verde: região repetida N-terminal (NTR), em roxo: domínio do ciclotídeo maduro (MCD) e em vermelho: região C-terminal (CTR). Acima de cada MCD está identificado o nome do
Figura 7 - Representação esquemática da diversidade estrutural de precursores proteicos completos elucidados de diferentes espécies das famílias Rubiaceae (OaK1, OaK2 e OaK4 de O. affinis; ChaC8 de C. chartaceae), Violaceae (Mra13 de M. ramiflorus; Triciclon e Vtt1 de V. tricolor), Fabaceae (CterM de C. ternatea) e Poaceae (Umi11 de Z. mays). Comparação do tamanho real das proteínas em número de aminoácidos, onde o menor possui 75 resíduos (ChaC8) e o maior possui 300 resíduos (Vtt1). Geralmente, os precursores de Rubiaceae e Violaceae possuem um ER-Signal (cinza), um NTPD (azul), um NTR (verde) e um CTR (vermelho) flanqueando cada MCD (roxo), sendo as únicas famílias a apresentar precursores
duplos ou triplos. Alguns precursores exibem arquitetura diferente, como a perda de domínios ... 35
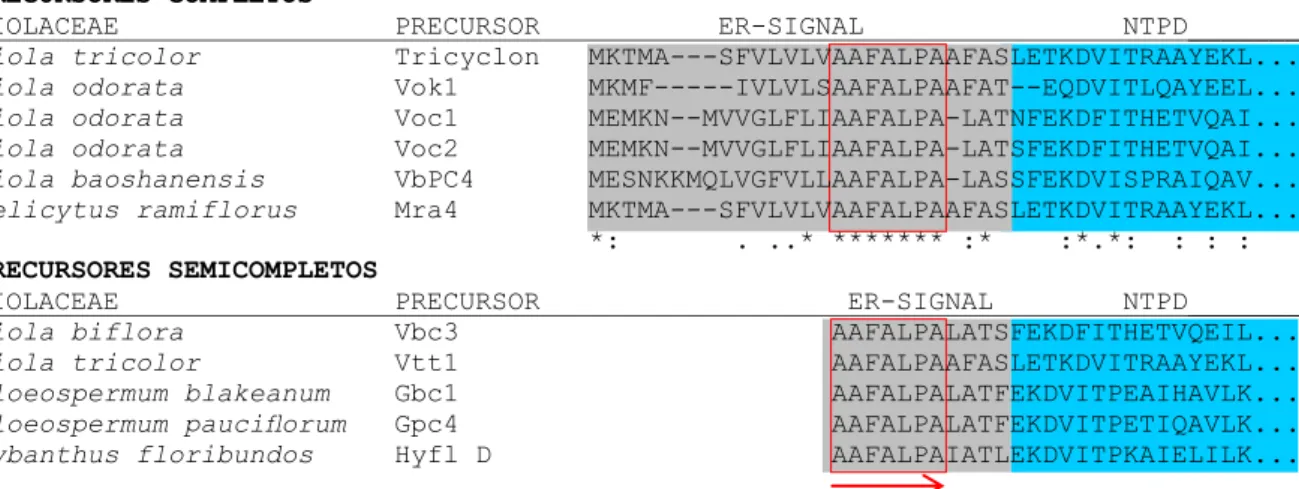
Figura 8 - Comparação entre precursores proteicos de espécies da família Violaceae, evidenciando a sequência conservada AAFALPA, presente no alinhamento precursores completos elucidados desta família, localizada no final do peptídeo sinal. Esta sequência foi utilizada para desenho de primers, resultando na descoberta de novos precursores
semicompletos ... 39
Figura 9 - Modelo tridimensional do ciclotídeo parigidina-br1, onde a cadeia principal é mostrada em azul e as pontes dissulfeto entre as cisteínas estão representadas em amarelo. No modelo é possível a visualização dos seis loops e das três pontes dissulfeto, formando o
motivo CCK,característico dos ciclotídeos (adaptado de PINTO et al., 2011a) ... 43
Figura 10 - Sequência de aminoácidos do ciclotídeo parigidina-br1, na primeira linha. Sequência degenerada de nucleotídeos, na segunda linha, obtida a partir de tradução reversa. Sequência otimizada, na terceira linha, obtida através da utilização de códons preferenciais de Rubiaceae. As letras em vermelho representam as siglas da nomenclatura da IUPAC-IUB para as bases degeneradas: N = A,C,G,T; W = A,T; S = C,G; Y = C,T; R = A,G; H = A,C,T; B = C,G,T .... 47
Figura 11 - Alinhamento das sequências de nucleotídeos do início dos genes de precursores de ciclotídeos da família Rubiaceae, utilizadas para o desenho dos oligonucleotídeos iniciadores. O retângulo vermelho indica a região de anelamento dos primers. As letras em verde
indicam o códon iniciador da ORF em cada sequência ... 47
Figura 12 - Alinhamento das sequências de nucleotídeos do início dos genes de precursores de ciclotídeos da espécie Melicytus ramiflorus, utilizadas para o desenho do primer MrF1. O retângulo indica a região de anelamento do primer. As letras em verde indicam o códon iniciador
da ORF e as letras em azul indicam a região codificadora da sequência AAFALPA ... 48
Figura 13 - Alinhamento das sequências de nucleotídeos do início dos genes de precursores de ciclotídeos da espécie Viola baoshanensis, utilizadas para o desenho dos primers VbF1-4. Os retângulos indicam as regiões de anelamento dos primers. As letras em verde indicam o códon iniciador da ORF e as letras em azul indicam a região codificadora da sequência
AAFALPA e suas variantes ... 48
Figura 14 - Alinhamento das sequências de nucleotídeos do início dos genes de precursores de ciclotídeos da espécie Viola odorata, utilizadas para o desenho dos primers VoF1-3. Os retângulos indicam as regiões de anelamento dos primers. As letras em verde indicam o códon iniciador da ORF e as letras em azul indicam a região codificadora da sequência AAFALPA e
suas variantes ... 49
retângulos indicam as regiões de anelamento dos primers. As letras em verde indicam o códon iniciador da ORF e as letras em azul indicam a região codificadora da sequência AAFALPA e
suas variantes ... 49
Figura 16 - Fotos da planta Palicourea rigida, encontrada no Cerrado. Em evidência, suas folhas
grandes, largas e coriáceas, as quais foram utilizadas nos experimentos ... 55
Figura 17 - Eletroforese em gel de agarose 1% da extração de RNA total de P. rigida através dos tampões Lysis Solution DCT e Lysis Solution RP. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2), (3) e (4) extração de RNA com tampão Lysis Solution DCT, contendo 60 ng, 120 ng e 180 ng de RNA, respectivamente; (5), (6) e (7) extração de RNA com tampão Lysis Solution RP, contendo 60 ng, 120 ng e 180 ng de RNA, respectivamente ... 56
Figura 18 - Eletroforese em gel de agarose 1% da amplificação por PCR de cDNA de P. rigida com os primers desenhados para buscas em Rubiaceae, com 53 ºC de temperatura de anelamento e tempo de 1 min de extensão. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) PCR contendo o conjunto de primers PrF1.1+Oligo-dT; (3) PCR contendo o conjunto de primers PrF3.1+Oligo-dT; (4) PCR contendo o conjunto de primers OaF1+Oligo-dT; (5) PCR contendo o conjunto de primers HcF1+Oligo-dT ... 57
Figura 19 - Eletroforese em gel de agarose 1% da amplificação dos insertos por PCR das colônias selecionadas, utilizando o conjunto de primers SP6+T7. Nos poços do gel A foram aplicados: (1) Marcador O'GeneRuler 100 bp Plus DNA Ladder (Thermo Scientific Fermentas); (2), (3), (4), (5), (6), (7) e (8) amplificação dos insertos das colônias 1, 2, 3, 4, 5, 6 e 7, respectivamente. Nos poços do gel B foram aplicados: (1) Marcador O'GeneRuler 100 bp Plus DNA Ladder (Thermo Scientific Fermentas); (2), (3), (4), (5), (6), (7) e (8) amplificação dos insertos das colônias 8, 9, 10, 11, 12, 13 e 14, respectivamente. Nos poços do gel C foram aplicados: (1) Marcador O'GeneRuler 100 bp Plus DNA Ladder (Thermo Scientific Fermentas); (2), (3), (4), (5), (6), (7) e (8) amplificação dos insertos das colônias 15, 16, 17, 18, 19, 20 e 21, respectivamente. Nos poços do gel D foram aplicados: (1) Marcador O'GeneRuler 100 bp Plus DNA Ladder (Thermo Scientific Fermentas); (2), (3), (4) e (5) amplificação dos insertos das colônias 22, 23, 24 e 25, respectivamente. Os números ao lado do marcador indicam o tamanho relativo das bandas em pares de base ... 58
Figura 20 - Alinhamento realizado pelo software ClustalW2 entre o ciclotídeo parigidina-br1 e o ciclotídeo presente em Pri1. As setas pretas indicam as diferenças entre as duas sequências . 59
Figura 21 - Sequência parcial de nucleotídeos do gene Pri1. As letras em vermelho representam o códon terminador, e a região destacada em azul representa o local de anelamento do oligonucleotídeo iniciador no sentido antisenso (Pr5R) ... 61
Figura 22 - Eletroforese em gel de agarose 1% da amplificação por PCR da extremidade 5' do cDNA de P. rigida. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (3), (4) e (5) PCR contendo 1 µL, 2 µL e 3 µL de cDNA, respectivamente, com 50 ºC de temperatura de anelamento ; (7), (8) e (9) PCR contendo 1 µL, 2 µL e 3 µL de cDNA, respectivamente, com 55 ºC de temperatura de anelamento; (11), (12) e (13) PCR contendo 1 µL, 2 µL e 3 µL de cDNA, respectivamente, com 60 ºC de temperatura de anelamento; (15) PCR
Figura 23 - Eletroforese em gel de agarose 1% da amplificação dos insertos por PCR das colônias selecionadas, utilizando o conjunto de primers SP6+T7. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) PCR sem DNA molde, como controle negativo; (3) a (13) amplificação dos insertos das colônias selecionadas ... 62
Figura 24 - Sequência completa do gene Palicourea rigida 1 (Pri1), contendo uma ORF de 249 nucleotídeos, a qual codifica in silico o precursor proteico do ciclotídeo parigidina-br2, contendo os domínios ER-Signal, NTPD, NTR, MCD e CTR. As letras em vermelho indicam o códon
iniciador (ATG) e o terminador (TAA) da tradução do mRNA ... 63
Figura 25 - Alinhamento realizado pelo software ClustalW2 entre precursores de ciclotídeo isolados de Rubiaceae, comparando as sequências e os tamanhos dos domínios, onde o comprimento em aminoácidos dos precursores estão indicados ao final de cada linha. Pri1: P. rigida; ChaC2, ChaC4 e ChaC8: C. chartacea; OaK1 e OaK2: O. affinis; Hbc1: H. biflora; Hcf1: H. centranthoides ... 64
Figura 26 - Alinhamento realizado pelo software ClustalW2 entre o ciclotídeo isolado de P. rigida, parigidina-br1, e a sequência completa do MCD traduzida in silico, parigidina-br2. As
variações existentes entre as sequências estão indicadas pelas setas pretas ... 64
Figura 27 - Alinhamento realizado pelo software ClustalW2 entre ciclotídeos das famílias Rubiaceae e Violaceae, contendo apenas um resíduo de aminoácido de diferença. Os resíduos destacados em vermelho evidenciam as diferenças existentes nas sequências em relação à
kalata B1 ... 65
Figura 28 - Sequência completa de nucleotídeos do mRNA de Pri1. As letras destacadas em vermelho representam o códon iniciador (ATG) e o terminador (TAA). A região destacada em verde indica o local de anelamento do oligonucleotídeo iniciador no sentido senso (PrDF) e a região destacada em azul representa o local de anelamento do oligonucleotídeo iniciador no
sentido antisenso (PrDR) ... 66
Figura 29 - Eletroforese em gel de agarose 1% mostrando o DNA genômico (gDNA) de P. rigida através da metodologia de precipitação e limpeza com CTAB. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o
tamanho relativo das bandas em pares de base; (2) 10 µL da extração de gDNA ... 66
Figura 30 - Eletroforese em gel de agarose 1% da amplificação por PCR do gDNA de P. rigida. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) ao (6) PCR contendo 1 µL de gDNA, com 50 ºC, 53 ºC, 55 ºC, 58 ºC e 60 ºC de temperatura deanelamento, respectivamente; (7) PCR sem DNA molde, como controle negativo ... 67
Figura 31 - Genes completos de precursores proteicos de ciclotídeos, isolados de DNA genômico em espécies de Rubiaceae. Os números indicam as quantidades de nucleotídeos e as posições de início e termino dos introns (representados pela linha preta), comparando seu tamanho entre diferentes genes. As sequências em vermelho representam os genes de Oldenlandia affinis, em rosa o gene de Hedyotis centranthoides, em roxo os genes de
Figura 32 - Eletroforese em gel de agarose 1% da extração de DNA genômico (gDNA) de P. rigida e de Cana-de-açúcar, através da metodologia de precipitação e limpeza com CTAB. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) 5 µL da extração de gDNA de P. rigida; (3) 5 µL da extração de gDNA de Cana-de-açúcar ... 71
Figura 33 - Eletroforese em gel de agarose 1% da digestão enzimática de gDNA de P. rigida e Cana-de-açúcar. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (3) Digestão de gDNA de Cana-de-açúcarcom EcoRI, como controle negativo para o experimento de Southern Blot; (4) Digestão de gDNA de P. rigida com EcoRI; (5) Digestão de gDNA de P. rigida com AluI; (6) Digestão de gDNA de P. rigida com EcoRI+AluI ... 71
Figura 34 - Eletroforese em gel de agarose 1% da amplificação por PCR de DNA plasmidial contendo o gene completo codificador do precursor Pri1, com os primers desenhados para amplificação em DNA de P. rigida, utilizando o DIG High Prime DNA Labeling (ROCHE). Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) PCR controle, contendo nucleotídeos normais; e (3) PCR contendo nucleotídeos marcados com Digoxigenina-11-dUTP (DIG) ... 72
Figura 35 - Imagem da membrana de nitrocelulose após hibridização da sonda, marcação com anticorpos e revelação por imunodetecção dos fragmentos de DNA contendo a sequência do gene codificador de Pri1. Os números acima da imagem representam as aplicações do gel da Figura 26: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (3) Digestão de gDNA de Cana-de-açúcar com EcoRI, como controle negativo para o experimento de Southern Blot; (4) Digestão de gDNA de P. rigida com EcoRI; (5) Digestão de gDNA de P. rigida com AluI; (6) Digestão de gDNA de P. rigida com EcoRI+AluI ... 73
Figura 36 - Fotos da planta Hybanthus lanatus, encontrada no Cerrado. Em evidência, suas folhas pequenas, finas e pilosas, as quais foram utilizadas nos experimentos ... 75
Figura 37 - Eletroforese em gel de agarose 1% da extração de RNA total de H. lanatus através do uso de TRIzol® Reagent (Ambion). Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (3) extração de RNA de H. lanatus, contendo 130 ng ... 76
Figura 38 - Eletroforese em gel de agarose 1% da amplificação por PCR de cDNA de H. lanatus utilizando primers de Violaceae, com 55 ºC de temperatura de anelamento e 1 min de extensão. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) PCR sem DNA molde, como controle negativo; (3) PCR com conjunto de primers VbF3+Oligo-dT, onde as setas vermelhas indicam a posição dos produtos de amplificação; e (4) PCR com conjunto de primers VoF1+Oligo-dT ... 77
Figura 40 - Sequência semicompleta do gene Hybanthus lanatus 1 (Hyla1), contendo uma ORF de 309 nucleotídeos, a qual codifica in silico o precursor proteico de um novo ciclotídeo, contendo os domínios ER-Signal, NTPD, NTR, MCD e CTR. As letras em vermelho indicam o códon terminador (TAA) ... 79
Figura 41 - Alinhamento realizado pelo software ClustalW2 entre o precursor de ciclotídeo isolado de H. lanatus (Hyla1) e outros precursores simples da família Violaceae, com destaque para região contendo a sequência AAFALPA, presente em grande parte dos precursores desta família, representados por Gbc6, produzido por G. blakeanum. Os outros precursores representam todas as variações existentes nessa região, comparando aqueles que apresentam uma modificação, como Hyla1 (H. lanatus) e Hyfl K (H. floribundos), duas modificações, como VbPC1 e VbCP2a (V. baoshanensis) e Voc3 (V. odorata), ou três modificações, como VbCP3d (V. baoshanensis) e Mra13 (M. ramiflorus). Os resíduos destacados em vermelho evidenciam as diferenças existentes nas sequências em relação à AAFALPA ... 80
Figura 42 - Alinhamento realizado pelo software ClustalW2 entre o novo ciclotídeo hyla-br1 e outros ciclotídeos da família Violaceae: mra23 (Melicytus ramiflorus), cicloviolina A (Leonia cymosa), globa B (Gloeospermum blakeanum) e viba 7 (Viola baoshanensis). As barras amarelas destacam os resíduos de Cisteína, os quais delimitam os loops (L1-L5). Os resíduos destacados em vermelho evidenciam as diferenças existentes nas sequências em relação à
hyla-br1 ... 81
Figura 43 - Alinhamento realizado pelo software ClustalW2 entre o novo ciclotídeo hyla-br1 e
as cicloviolinas A-D, isoladas de Leonia cymosa (Violaceae) ... 82
Figura 44 - Sequência semicompleta de nucleotídeos do gene Hyla1. As letras em vermelho representam o códon terminador, e a região destacada em azul representa o local de
anelamento do oligonucleotídeo iniciador no sentido antisenso (Hl5R) ... 82
Figura 45 - Eletroforese em gel de agarose 1% da amplificação por PCR da extremidade 5' do cDNA de H. lanatus. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) PCR contendo 1 µL de cDNA, com 60 ºC de temperatura de anelamento e 1 minuto de extensão ... 83
Figura 46 - Sequência completa do gene Hybanthus lanatus 1 (Hyla1), contendo uma ORF de 330 nucleotídeos, a qual codifica in silico o precursor proteico do ciclotídeo hyla-br1, contendo 109 resíduos de aminoácidos. As regiões sublinhadas representam as sequências que faltavam no precursor semicompleto, e as letras em vermelho indicam os códons iniciador e
terminador ... 84
Figura 47 - Fotos da planta Clitoria sp.,encontrada no Cerrado. Emevidência,suasfolhaspequenas,
finas e pilosas, as quais foram utilizadas nos experimentos ... 85
Figura 48 - Eletroforese em gel de agarose 1% da extração de RNA total de Clitoria sp.através do uso de TRIzol® Reagent (Ambion). Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (3) extração de RNA de Clitoria sp., contendo 300 ng ... 86
extensão. Nos poços foram aplicados: (1) Marcador 1 Kb Plus DNA Ladder (Invitrogen), onde os números ao lado indicam o tamanho relativo das bandas em pares de base; (2) PCR sem DNA molde, como controle negativo; (3) PCR com conjunto de primers CtF1+Oligo-dT; (4) PCR com conjunto de primers CtF2+Oligo-dT; (5) PCR com conjunto de primers CtF3+Oligo-dT; (6) PCR com conjunto de primers CtF4+Oligo-dT; e (7) PCR com conjunto de primers CtF5+Oligo- dT ... 87
LISTA DE ABREVIATURAS E SIGLAS
[α-32P]dCTP: “Deoxycytidine
-5’-Triphosphate”– Desoxicitidina-5'-Trifosfato
AEP: Asparaginil Endopeptidase
Ala: Alanina
Asn: Asparagina
Asp: Aspartato
CCK:“Cyclic Cysteine Knot” – Nó de Cisteína Cíclica
cDNA: DNA Complementar
Cis: Cisteína
CTAB: “Cetyl Trimethyl Ammonium Bromide”– Brometo de Cetiltrimetilamônio
CTR: “C-terminal Region”– Região C-terminal
CyBase: “The Database of Cyclic Proteins” – Banco de Dados de Proteínas Cíclicas
DNA: Ácido Desoxirribonucleico
DPC: Dodecil Fosfatidilcolina
ER-Signal: “Endoplasmic Reticulum Signal”– Peptídeo sinal para Retículo Endoplasmático
EST:“Expressed Sequence Tags”– Etiquetas de Sequências Expressas
gDNA: DNA Genômico
Ile: Isoleucina
Intron: “Intragenic Region”– Região Intragênica
Leu: Leucina
MCD: “Mature Cyclotide Domain”– Domínio do Ciclotídeo Maduro
MCoT: “M. cochinchinensis Tripsin Inhibitor”– Inibidor de Tripsina de M. cochinchinensis
mRNA: RNA Mensageiro
NMR: “Nuclear Magnetic Resonance”– Ressonância Magnética Nuclear
NTPD: “N-terminal Prodomain”– Pró-domínio N-terminal
NTR: “N-terminal Repeat Domain”– Domínio de Repetição N-terminal
ORF: “Open Reading Frame”– Fase de Leitura Aberta
pb: pares de base
PC: Fosfatidilcolina
PCR:“Polymerase Chain Reaction”– Reação em Cadeia da Polimerase
PDI: Proteína Dissulfeto Isomerase
RACE: “Rapid Amplification of cDNA Ends”– Amplificação Rápida das Extremidades de cDNA
RE: Retículo Endoplasmático
RNA: Ácido Ribonucleico
RT-PCR: “Reverse Transcriptase Polymerase Chain Reaction”– PCR Transcriptase Reversa
Ser: Serina
SPR: “Surface Plasmon Resonance”– Ressonância Plasmônica de Superfície
Umi: “Ustilago maydis induced”– U. maydis Induzido
Umr: “Ustilago maydis repressed”– U. maydis Reprimido
UTR:“Untranslated Region”– Região Não-Traduzida
SUMÁRIO
1 INTRODUÇÃO ... 20
1.1 CICLOTÍDEOS: O QUE SÃO? ... 20
1.2 MULTIFUNCIONALIDADE E POTENCIAL BIOTECNOLÓGICO
DOS CICLOTÍDEOS ... 21
1.3 MECANISMOS DE AÇÃO ... 24
1.4 PRECURSORES PROTEICOS: ORIGEM DOS CICLOTÍDEOS ... 26
1.5 BIOSSÍNTESE DE CICLOTÍDEOS: PROCESSAMENTO DOS
PRECURSORES PROTEICOS ... 28
1.6 HISTÓRICO E DIVERSIDADE GÊNICA DOS PRECURSORES
PROTEICOS ... 30
1.7 MODULAÇÃO DA EXPRESSÃO GÊNICA DE CICLOTÍDEOS ... 36
1.8 ESTRATÉGIAS DE BUSCA POR NOVOS PRECURSORES ... 38
1.9 PRODUÇÃO DE CICLOTÍDEOS ... 40
1.10 PARIGIDINA-BR1: O PRIMEIRO CICLOTÍDEO ISOLADO NO BRASIL ... 42
2 JUSTIFICATIVA ... 44
3 OBJETIVOS ... 45
3.1 OBJETIVO GERAL ... 45
3.2 OBJETIVOS ESPECÍFICOS ... 45
4 MATERIAL E MÉTODOS ... 46
4.1 MATERIAL VEGETAL ... 46
4.2 EXTRAÇÃO DE RNA ... 46
4.3 SÍNTESE DE DNA COMPLEMENTAR (CDNA) ... 46
4.4 QUANTIFICAÇÃO DE ÁCIDOS NUCLEICOS ... 46
4.5 DESENHO DE OLIGONUCLEOTÍDEOS INICIADORES ... 46
4.6 REAÇÃO EM CADEIA DA POLIMERASE (PCR) ... 50
4.7 AVALIAÇÃO DE ÁCIDOS NUCLEICOS EM GEL DE AGAROSE ... 51
4.8 PURIFICAÇÃO DE DNA ... 51
4.10 EXTRAÇÃO DE DNA PLASMIDIAL ... 52
4.11 SEQUENCIAMENTO DE DNA E ANÁLISES COMPUTACIONAIS ... 52
4.12 RÁPIDA AMPLIFICAÇÃO DAS EXTREMIDADES DO CDNA (RACE) ... 52
4.13 EXTRAÇÃO DE DNA GENÔMICO (GDNA) ... 52
4.14 AVALIAÇÃO DA PRESENÇA DE INTRONS ... 53
4.15 AVALIAÇÃO DO NÚMERO DE CÓPIAS GÊNICAS DE CICLOTÍDEOS
NO GENOMA ... 53
5 RESULTADOS E DISCUSSÃO ... 55
5.1 FAMÍLIA RUBIACEAE: PALICOUREA RIGIDA ... 55
5.2 FAMÍLIA VIOLACEAE: HYBANTHUS LANATUS ... 75
5.3 FAMÍLIA FABACEAE: CLITORIA SP. ... 85
6 CONCLUSÕES ... 89
7 PERSPECTIVAS ... 90
1 INTRODUÇÃO
1.1 CICLOTÍDEOS: O QUE SÃO?
Ciclotídeos são peptídeos cíclicos ricos em cisteína que possuem entre 28 e 37 resíduos de aminoácidos e apresentam uma ligação peptídica entre o C-terminal e o N-terminal, gerando uma cadeia circular, distinguindo-se dos demais peptídeos cíclicos devido à presença de um padrão único e muito conservado de três pontes dissulfeto (CisI-CisIV,
CisII-CisV, CisIII-CisVI) formando um motivo conhecido como Cyclic Cysteine Knot (CCK)
(CRAIK et al., 1999). Esse motivo compreende um anel peptídico formado por duas pontes dissulfeto paralelas (CisI-CisIV e CisII-CisV) e por uma ponte dissulfeto transversal (CisIII
-CisVI), que penetra no centro da estrutura, formando um nó e dividindo o ciclotídeo em seis
loops (Figura 1) (CRAIK et al., 2004; CRAIK, 2010; CRAIK, 2011). Desta forma, combinando a ciclização e o motivo CCK, a família denominada de ciclotídeos agrupam peptídeos cíclicos vegetais que compartilham uma estrutura tridimensional única, a qual confere elevada estabilidade e proteção contra enzimas proteolíticas, condições ácidas, degradação química e física. (COLGRAVE; CRAIK, 2004; CRAIK, 2006; NAIR et al., 2006; CRAIK; CEMAZAR; DALY, 2007; GRUBER et al., 2007; PELEGRINI; QUIRINO; FRANCO, 2007; CRAIK, 2010).
Figura 1 - Estrutura tridimensional do ciclotídeo kalata B1, exemplificando elementos gerais dos ciclotídeos, onde a cadeia principal é colorida em cinza e as pontes dissulfeto são coloridas em amarelo. (a) Os resíduos de cisteína atuam como fatores limitantes para os loops, onde os seis presentes na estrutura estão nomeados de L1-L6. (b) Além da ciclização da cadeia principal, o motivo CCK é definido por seis resíduos de cisteína que formam duas pontes dissulfeto paralelas (CisI-CisIV e CisII-CisV) (c) e por uma terceira ponte dissulfeto perpendicular à estrutura (CisIII-CisVI),
que penetra o anel a 90º de angulação. (Adaptado de WESTERMANN e CRAIK, 2010).
5, entre a CisV e a CisVI, gerando uma torção de 180o neste loop. Os integrantes deste
grupo não apresentam grandes variações no número de resíduos de aminoácidos entre os loops, enquanto nos integrantes da subfamília Bracelete os ciclotídeos apresentam grande variação entre os loops, possuindo geralmente resíduos carregados positivamente (Figura 2). Além disso, este último grupo não possui uma cis-prolina no loop 5, não apresentando torção e diferenciando assim as Möbius das Braceletes. (WU; RALEIGH, 1998; CHEN et al., 2005; DALY et al., 2006; CRAIK; DALY, 2007; PELEGRINI; QUIRINO; FRANCO, 2007). A terceira classe de ciclotídeos compreende os inibidores de tripsina, a qual agrupa apenas dois integrantes isolados de sementes de Momordica cochinchinensis (Cucurbitaceae), nomeados de MCoTI-I e MCoTI-II, únicos ciclotídeos a apresentar este tipo de atividade. A existência desta terceira subfamília ainda gera discussões, pois mesmo possuindo seis resíduos de cisteína e apresentando o CCK estabilizado por três pontes dissulfeto, estes exemplares exibem baixa homologia de suas sequências com outros ciclotídeos (HEITZ et al., 2001; DALY et al., 2006; CRAIK; DALY, 2007; THONGYOO et al., 2008) (Figura 2).
Descritos desde a década 1970, os ciclotídeos foram encontrados nas famílias Rubiaceae, Violaceae, Cucurbitaceae, Asteraceae, Fabaceae, Poaceae e Solanaceae. Os ciclotídeos podem ser isolados a partir de diferentes tecidos, incluindo folhas, caules, flores, raízes e cascas, sendo possível encontrar dezenas desses peptídeos em um único tecido ou espécime (CHEN et al., 2005; JENNINGS et al., 2005; IRELAND et al., 2006; WESTERMANN; CRAIK, 2010; CRAIK; MYLNE; DALY, 2010; BURMAN et al., 2010; NGUYEN et al., 2011a,b; POTH et al., 2012; NGUYEN et al., 2013). Além disso, através de análises de bioinformática, possíveis sequências de ciclotídeos foram recentemente encontradas em outras famílias, como Apocynaceae, por exemplo. (DÖRNENBURG, 2010; CRAIK; MYLNE; DALY, 2010; BURMAN et al., 2010).
1.2 MULTIFUNCIONALIDADE E POTENCIAL BIOTECNOLÓGICO DOS CICLOTÍDEOS
mesma família. Assim, as circulinas A e B foram descobertas a partir de um programa aleatório de procura por atividade anti-HIV em extratos naturais da planta tropical Chassalia parvifolia (Rubiaceae) (GUSTAFSON et al., 1994), como também ocorreu com a palicoureína, potente anti-HIV isolada de Palicourea condensata (BOKESCH et al., 2001), e a parigidina-br1, potente inseticida isolada de Palicourea rígida (PINTO et al., 2011a). Da mesma forma, o ciclopsicotrídeo A foi descoberto a partir de extratos da planta sul-americana Psychotria longipes (Rubiaceae) com capacidade de inibir a ligação de neurotensina às membranas de células HT-29 (adenocarcinoma de colo humano) e também de estimular o aumento dos níveis de cálcio citosólico (Ca+2) em duas linhagens de células
que não expressam receptores de neurotensina (WITHERUP et al., 1994).
A fim de ampliar os conhecimentos sobre ciclotídeos, reconhecidos agora como a maior família de peptídeos circulares naturais, novos grupos taxonômicos começaram a ser estudados e vários gêneros da família Violaceae foram explorados. Assim, diversos peptídeos provenientes do gênero Viola foram isolados, como violapeptídeo-1 e Varv peptídeo A-H (V. arvensis), cicloviolacina O1-O12 e cicloviolacina H1 (V. odorata), inicialmente encontrados na procura por atividade hemolítica, sendo que alguns também apresentaram atividade citotóxica (SCHØPKE et al., 1993; CLAESON et al., 1998; GÖRANSSON et al., 1999). Além destes, outros integrantes da família Violaceae também se juntaram ao grupo multifuncional, como as cicloviolinas A-D (anti-HIV) e vitri-A (citotóxico), (HALLOCK et al., 2000; SVANGÅRD et al., 2004; PICCHI et al., 2009) (Figura 2).
Diferentemente dos demais, os inibidores de tripsina MCoTI-I e MCoTI-II, extraídos de sementes de M. cochinchinensis (Curcubitaceae), são os únicos representantes desta família taxonômica descobertos até o momento e também, provavelmente, os únicos ciclotídeos a apresentar atividade inibidora de tripsina, gerando uma nova divisão dentro do grupo e controvérsias em relação à sua classificação devido a grandes diferenças em sua sequência de aminoácidos em comparação à outros ciclotídeos (HERNANDEZ et al., 2000).
Contudo, mesmo somando amplo espectro de atividades biológicas, a função fisiológica dos ciclotídeos in planta ainda é desconhecida. Há suspeitas que estas moléculas estejam envolvidas na defesa do hospedeiro, uma vez que elas são expressas em grandes quantidades no tecido foliar, são ricas em cisteína, como ocorre em uma série de proteínas de defesa vegetal (como tioninas e defensinas), além de possuírem atividades antimicrobiana e antiparasitária (HERRMANN et al., 2008).
A descoberta de peptídeos circulares tem introduzido nos últimos anos um novo paradigma topológico no campo de estrutura de proteínas. Sua estrutura cíclica rígida combinada com loops formados por ligações dissulfeto explica sua alta estabilidade e sua resistência, assim como também influencia no amplo espectro de atividades biológicas apresentado por estas moléculas. Os ciclotídeos representam um grupo de peptídeos que, no futuro, podem ser utilizados como modelo para o desenho de drogas mais estáveis e específicas pela indústria farmacêutica e pelo agronegócio (GRAN et al., 2000; DUTTON et al., 2004; PICCHI et al., 2009; PINTO et al., 2011b).
1.3 MECANISMOS DE AÇÃO
Ciclotídeos são capazes de apresentar diversas atividades biológicas in vitro, característica essa presente em diversos peptídeos pertencentes tanto às Möbius quanto às Braceletes. A produção de uma gama de ciclotídeos diferentes em um mesmo indivíduo e sua grande abundância nas partes aéreas, somadas à grande quantidade de atividades pesticidas, como antimicrobiana, inseticida, nematicida e moluscicida, fazem com que estes peptídeos já sejam reconhecidos como moléculas de defesa de plantas. Contudo, outras funções dos ciclotídeos são investigadas, devido à apresentação de atividades hemolítica, citotóxica e anti-HIV, as quais também são compartilhadas por moléculas de diferentes famílias (CRAIK, 2012; HENRIQUES; CRAIK, 2012).
Embora o leque de atividades biológicas de ciclotídeos seja diverso, na verdade, aparentemente todas as atividades podem ser explicadas pelos mesmos modos de ação. Experimentos estruturais realizados com o ciclotídeo kalata B1, o qual possui atividade hemolítica, anti-HIV e inseticida, mostraram a perda simultânea de suas atividades biológicas com algumas modificações moleculares, como: (1) o rompimento do esqueleto cíclico, evidenciando a perda das atividades hemolítica e anti-HIV (
BARRY et al., 2003;
A maioria dos ciclotídeos tem uma porção hidrofóbica exposta na sua superfície, e a integridade dessa região tem sido considerada necessária para sua atividade. Estudos evidenciam ampla correlação entre eficiência de atividade anti-HIV e geometria da região hidrofóbica em vários ciclotídeos naturais (IRELAND et al., 2008), sugerindo que as propriedades hidrofóbicas dos ciclotídeos contribuem para suas atividades biológicas.
Dessa forma, a interação com membranas e sua consequente ruptura pode representar o mecanismo de ação de grande parte dos ciclotídeos. Nesse âmbito, estudos demonstraram interações específicas entre micelas de Dodecil Fosfatidilcolina (DPC) e o ciclotídeo kalata B1, evidenciando que as interações entre os resíduos hidrofóbicos expostos e os fosfolipídeos da superfície da micela representam a força primária da interação com a membrana (SHENKAREV et al., 2006). Vários estudos independentes, utilizando técnicas biofísicas como Ressonância Magnética Nuclear (NMR), Surface
Plasmon Resonance (SPR), análises eletrofisiológicas e de fluorescência, reforçaram a capacidade de interação entre ciclotídeos e membranas gerando rompimento da bicamada lipídica através da formação de poros (KAMIMORI et al., 2005; HUANG et al., 2009).
Estudos mais específicos forneceram detalhes a respeito dessa interação, revelando que os ciclotídeos possuem também uma seletividade lipídica. Diferentes ciclotídeos interagem com diferentes subtipos de lipídeos, preferencialmente com fosfatidiletanolamina (PE) do que com fosfatidilcolina (PC) (HENRIQUES et al., 2011). Além dos dados relevantes sobre interações com lipídeos, estudos demonstraram que as ligações ciclotídeo-membrana são governadas apenas pelas propriedades hidrofóbicas, sendo independente de mecanismos de ligação através de receptores quirais, o que foi observado através da idêntica capacidade de rompimento de membrana causada por L- e D-isômeros de kalata B1 (SANDO et al., 2011).
Outros mecanismos de ação também são especulados devido a existência de estudos evidenciando que alguns ciclotídeos possuem a capacidade de atravessar membranas e penetrar nas células, exibindo atividades biológicas com alvos intracelulares, como os ciclotídeos MCoTI-II, MCoTI-I e kalata B1 (CASCALES et al., 2011). Trabalhos recentes revelaram que os ciclotídeos cicloviolacina O2 e vaby D foram capazes de penetrar em células de linfoma humano e causar degradação do DNA quando administrados em concentrações subletais, indicando que essas moléculas exibam uma possível dose-dependência para algumas atividades específicas (YESHAK et al., 2012).
Em suma, através da ruptura de membranas lipídicas, alvos intracelulares, atividades dose-dependentes ou até mesmo de mecanismos de ação ainda desconhecidos, a hipótese de que diferentes ciclotídeos possuem preferências distintas de alvos explicaria por que as plantas produzem um conjunto de ciclotídeos, produzindo um arsenal de defesa contra um amplo espectro de organismos ofensivos, cada um com propriedades de membrana distintas (HENRIQUES; CRAIK, 2012; YESHAK et al., 2012).
1.4 PRECURSORES PROTEICOS: ORIGEM DOS CICLOTÍDEOS
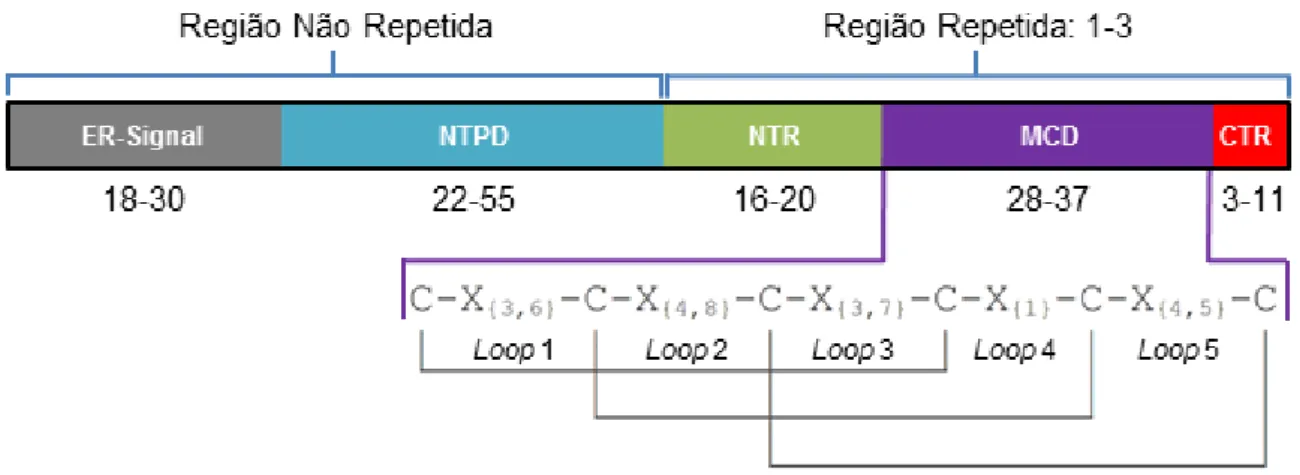
Os ciclotídeos são produtos gênicos, gerados através da síntese ribossomal de uma proteína precursora multidomínio, que possui uma construção característica (SASKA et al., 2007; GILLON et al., 2008). Sua expressão inicialmente ocorre a partir da síntese de um precursor proteico linear que contém, em média, entre 100 a 200 resíduos de aminoácidos, de acordo com o banco de dados de proteínas cíclicas (CyBase) (KAAS; CRAIK, 2010). Os precursores proteicos geralmente apresentam a mesma organização básica e altamente conservada, que é dividida em cinco domínios: (I) peptídeo sinal para o retículo endoplasmático (ER-Signal), com 18 a 30 resíduos; (II) pró-domínio N-terminal (NTPD), com 22 a 55 resíduos, sendo relativamente a maior região do precursor; (III) domínio de repetição do N-terminal (NTR), com 16 a 20 resíduos; (IV) domínio do ciclotídeo maduro (MCD), com 28 a 37 resíduos; e (V) região C-terminal (CTR), que representa uma cauda hidrofóbica variando entre 3 e 11 resíduos (Figura 3) (SASKA et al., 2007; GILLON et al., 2008; WESTERMANN; CRAIK, 2010; CRAIK; MYLNE; DALY, 2010; BURMAN et al., 2010; DALY; ROSENGREN; CRAIK, 2009; CyBase).
igual aos outros do mesmo precursor ou diferentes entre si (KAAS; CRAIK, 2010; QIN et al., 2010) (Figura 3).
Figura 3 - Organização geral de um típico precursor proteico linear de ciclotídeo. Os precursores podem ser divididos em duas regiões distintas: uma inicial que não se repete, envolvendo o ER-Signal (cinza) e o NTPD (azul); e uma posterior repetida, agrupando o NTR (verde), o MCD (roxo) e o CTR (vermelho), que pode estar presente de uma a três vezes, possuindo ciclotídeos maduros idênticos ou diferentes entre si. O domínio MCD representa o ciclotídeo maduro, no qual existe um padrão altamente conservado de seis resíduos de cisteína e é encontrado em todos os ciclotídeos conhecidos até agora, sendo responsável pela formação das pontes dissulfeto, as quais delimitam os loops do peptídeo (Loop 1-Loop 6), e pela estruturação do motivo CCK. Os números abaixo dos domínios indicam as variações no tamanho dessas regiões em quantidade de aminoácidos, que são diferentes para cada precursor.
Todos os precursores de ciclotídeos iniciam com uma sequência ER-Signal, localizada na extremidade N-terminal e compreende uma região conservada que direciona a proteína para o retículo endoplasmático, onde inicia seu processamento pela via de secreção (JENNINGS et al., 2001). A segunda parte do precursor compreende o NTPD, uma região pouco conservada em sequência e comprimento, apresentando grande variabilidade em termos de identidade e quantidade de aminoácidos entre os precursores de ciclotídeos. Atualmente, se desconhece a função do NTPD em relação ao processamento do precursor (JENNINGS et al., 2001; DUTTON et al., 2004).
presente em todos os ciclotídeos conhecidos até hoje (WESTERMANN; CRAIK, 2010; CRAIK; MYLNE; DALY, 2010; KAAS; CRAIK, 2010).
1.5 BIOSSÍNTESE DE CICLOTÍDEOS: PROCESSAMENTO DOS PRECURSORES PROTEICOS
Após a síntese, a proteína precursora linear passa por alguns passos de processamento pós-traducional, incluindo a excisão e enovelamento do domínio do ciclotídeo maduro (MCD). O dobramento molecular ocorre por oxidação dos resíduos de cisteína e da formação de pontes dissulfeto no MCD. Este processo pode ser facilitado por uma proteína dissulfeto isomerase (PDI) (GRUBER et al., 2007; WESTERMANN; CRAIK, 2010; BURMAN et al., 2010). Relatos a cerca de enzimas PDI em estudos anteriores mostraram que elas são responsáveis por aumentar a eficiência de enovelamento de ciclotídeos in vitro, interagindo com o MCD da proteína precursora e sendo capaz de corrigir ciclotídeos dobrados incorretamente (GRUBER et al., 2007; CRAIK; MYLNE; DALY, 2010). Além disso, por meio de análises de transcrição, estudos mostram a co-expressão de PDI e do OaK1 (precursor da kalata B1), revelando forte evidência sobre a interação molecular entre eles e sustentando a hipótese de que a catálise feita pela PDI é essencial dentro do processo de oxidação e enovelamento de proteínas cíclicas que possuem CCK (GRUBER et al., 2007).
As PDI são as proteínas mais abundantes no retículo endoplasmático (D'ALOISIO et al., 2010), representando enzimas com funções de oxirredução e pertencentes à família das tiorredoxinas. Elas executam o dobramento de peptídeos dentro de compartimentos celulares dos eucariotos através de reações de oxidação e redução, capazes de formar, quebrar ou alternar ligações dissulfeto entre os resíduos de cisteína (GRUBER et al., 2007). Este mecanismo oxidativo de enovelamento para ciclotídeos foi inicialmente identificado na família Rubiaceae (principalmente descrito para o processamento dos precursores das diferentes kalatas em O. affinis), sendo recentemente evidenciado também na família Violaceae (GRUBER et al., 2007; BURMAN et al., 2010).
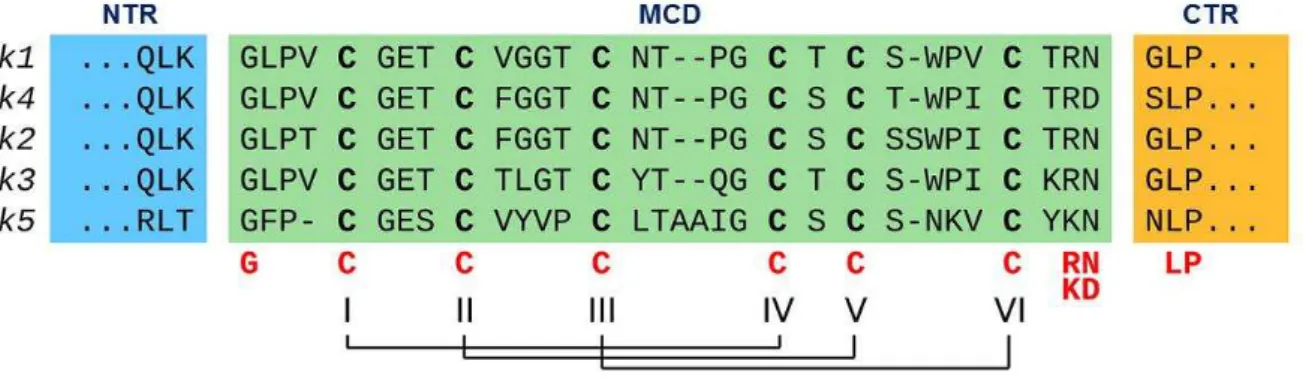
A literatura revela a ação enzimática das AEP na clivagem de resíduos de asparagina ou de ácido aspártico entre o domínio ciclotídeo e o C-terminal através do reconhecimento de uma sequência de resíduos conservados (XXNGLP), liberando o CTR e simultaneamente clivando o último resíduo do domínio NTR (podendo ser uma lisina, glicina ou asparagina). A enzima, além de intermediar a excisão do MCD do precursor, também faz a ciclização da cadeia principal do ciclotídeo através de uma ligação peptídica entre C-terminal e N-terminal (WESTERMANN; CRAIK, 2010; BURMAN et al., 2010; XU et al., 2011) (Figura 4).
Figura 4 - Alinhamento de quatro precursores proteicos de O. affinis (OaK1-5), com a região do ciclotídeo maduro (MCD) no centro, flanqueada pela região repetida do N-terminal (NTR) e ela cauda C-terminal (CTR). Os resíduos em vermelho indicam posições de aminoácidos conservados, como as cisteínas, a glicina inicial, os aminoácidos terminais (RN/KD) e o início da cauda (XLP), importantes no processamento do ciclotídeo (Adaptado de QIN et al., 2010).
De fato, a presença de um resíduo altamente conservado de Asn/Asp no C-terminal parecia ser crucial para a ciclização em todos precursores conhecidos. Entretanto esta teoria somente foi confirmada quando foi reportado o primeiro ciclotídeo de cadeia linear: a violacina A (isolada de V. odorata). Este ciclotídeo possui uma mutação no C-terminal que, por substituição de uma única base gera um códon de parada e, consequentemente, a interrupção prematura da tradução. Portanto, a tradução de um resíduo Asn/Asp e da cauda hidrofóbica C-terminal não ocorre, produzindo um ciclotídeo maduro incapaz de ser ciclizado. Além disso, trabalhos anteriores demonstraram que a inibição de AEP através de bloqueio de genes (gene knocking down) por silenciamento gênico induzido por vírus (VIGS) provoca uma redução significativa na atividade desta enzima, levando à acumulação de ciclotídeos maduros lineares (DUTTON et al., 2004; IRELAND; COLGRAVE; CRAIK, 2006; SASKA et al., 2007).
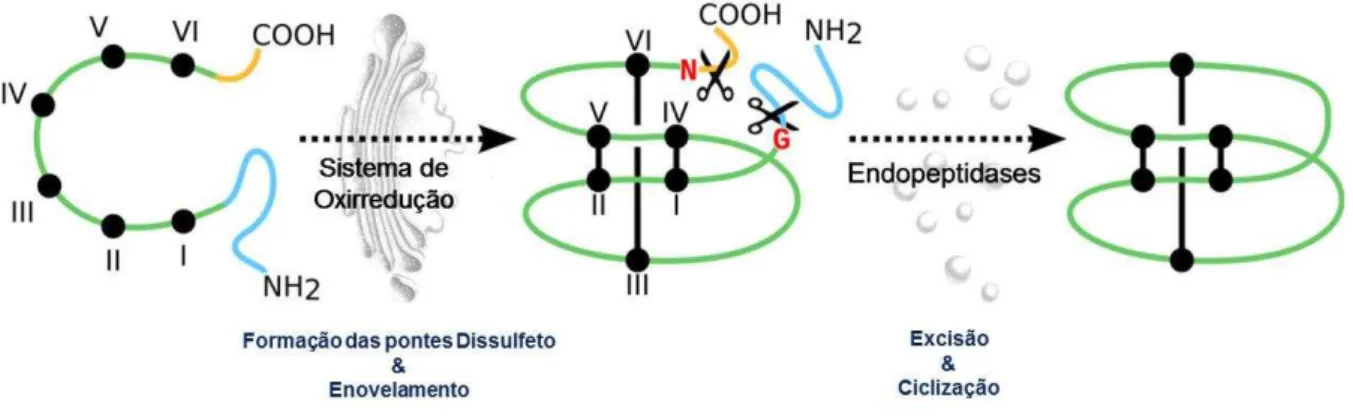
ciclotídeos sugere que o dobramento oxidativo ocorra antes da ciclização no retículo endoplasmático (RE), onde ocorre inicialmente a oxidação dos resíduos de cisteína e a formação das ligações dissulfeto dentro do RE e, posteriormente, a excisão e ciclização do ciclotídeo maduro a partir do precursor no Complexo de Golgi, pela via secretora da célula. No entanto, os detalhes de processamento do precursor, incluindo a ordem dos eventos, não são completamente compreendidos. É possível que, in planta, a oxidação e o enovelamento da proteína precursora possam ocorrer primeiro, com o peptídeo maduro sendo posteriormente excisado e ciclizado (Figura 5) (GRUBER et al., 2007; BURMAN et al., 2010; WESTERMANN; CRAIK, 2010; XU et al., 2011).
Figura 5 - Etapas do processamento pós-traducional da proteína precursora linear, incluindo o enovelamento do domínio do ciclotídeo maduro (MCD) (verde), através da formação das pontes dissulfeto, sua excisão do precursor e ciclização, intermediada por endopeptidases. A região azul representa o NTR e a região amarela representa a CTR. Representação esquemática do modelo proposto de produção dos ciclotídeos, ocorrendo primeiro o enovelamento oxidativo e posteriormente a excisão e ciclização do MCD (Adaptado de QIN et al., 2010).
1.6 HISTÓRICO E DIVERSIDADE GÊNICA DOS PRECURSORES PROTEICOS
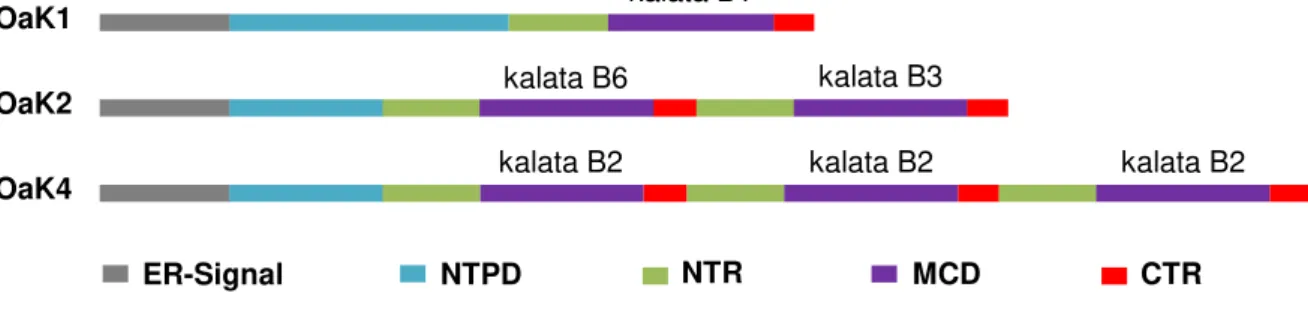
Com sua grande importância e ausência de conhecimento aprofundado sobre a biologia molecular dessa classe de moléculas recém-descoberta, iniciou-se uma busca pelos genes produtores desses ciclotídeos, para então elucidar sua arquitetura gênica, expressão e regulação. Em 2001, o primeiro trabalho buscando genes de ciclotídeos foi publicado por Jennings e colaboradores, revelando que os RNA mensageiros eram inicialmente traduzidos em uma grande proteína precursora linear e multidomínio, onde os ciclotídeos estavam inseridos, sendo liberados após clivagem do MCD, gerando então o peptídeo maduro. Foram identificadas várias proteínas precursoras diferentes, revelando que os ciclotídeos são codificados por uma família multigênica. Além disso, foi possível descobrir que O. affinis apresentava três tipos diferentes de precursores: (1) Simples, com apenas um domínio de ciclotídeo; (2) Duplo, com duas regiões que se repetem (NTR, MCD e CTR); (3) Triplo, com três regiões repetidas (NTR, MCD e CTR) (JENNINGS et al., 2001) (Figura 6).
Figura 6 - Diferentes precursores proteicos existentes em O. affinis, onde OaK1 representa um precursor simples, OaK2 representa um precursor duplo e OaK3 representa um precursor triplo. Em cinza: região do peptídeo sinal (ER-Signal), em azul: pró-domínio N-terminal (NTPD), em verde: região repetida N-terminal (NTR), em roxo: domínio do ciclotídeo maduro (MCD) e em vermelho: região C-terminal (CTR). Acima de cada MCD está identificado o nome do ciclotídeo produzido.
Os precursores proteicos das kalatas em O. affinis foram os primeiros genes de ciclotídeos a serem elucidados, nomeados OaK (Oldenlandia affinis Kalata), sendo atualmente conhecidos dez precursores diferentes: (1) Simples (OaK1, OaK3, OaK5, OaK7, OaK8, OaK9 e OaK10, sintetizando as kalata B1, kalata B7, kalata B19, Oak7, Oak8, kalata B20-linear e kalata B1 novamente, respectivamente); (2) Duplos (OaK2, que expressa as kalata B6 e kalata B3; e OaK6, que expressa o Oak6a e o Oak6b); (3) Triplo (OaK4, que produz a kalata B2 três vezes). Dessa forma, O. affinis apresenta uma das maiores diversidades de precursores na mesma espécie, com grande variabilidade de tamanho e de sequência (JENNINGS et al., 2001; MYLNE et al., 2010; QIN et al., 2010).
Além de O. affinis, poucas espécies de Rubiaceae tiveram genes de ciclotídeos elucidados, como Hedyotis centranthoides, da qual se conhece apenas dois precursores
kalata B1
kalata B6 kalata B3
kalata B2 kalata B2 kalata B2
OaK1
OaK2
OaK4
simples: Hcf1, que expressa o ciclotídeo hcf 1, e Kch1, o qual expressa o ciclotídeo kc 2; Hedyotes biflora, também com apenas dois precursores simples elucidados: Hbc1 e Hbc2, que produzem o hedyotídeo 1 e o hedyotídeo 2, respectivamente; e Chassalia chartacea, a qual possui dez precursores simples desvendados: ChaC2, ChaC4, ChaC7, ChaC8, ChaC13-18, gerando os chassatídeos 2, 4, 7, 8, 13-18, respectivamente. Estes chassatídeos foram descobertos recentemente, revelando os menores precursores proteicos de ciclotídeos dentre todos os conhecidos até a atualidade, pois além de apresentarem apenas um domínio ciclotídeo também não possuem o domínio NTR, sendo que o menor representante desse novo grupo possui apenas 75 resíduos de aminoácidos (Figura 7) (MULVENNA et al., 2006; GRUBER et al., 2008; NGUYEN et al., 2011b; NGUYEN et al., 2012).
Posteriormente aos estudos com O. affinis, rapidamente os genes dos ciclotídeos purificados de espécimes da família Violaceae começaram a ser rastreados, devido ao maior número de peptídeos isolados em relação às outras famílias e também por possuir ciclotídeos em todas as espécies testadas. Dessa forma, diferentes tipos de precursores também foram elucidados e nomeados conforme os de O. affinis. Dentre as Violaceae, o gênero Viola é o mais estudado. Em V. odorata foram encontrados precursores simples (Voc1, Voc2 e Voc3, os quais expressam as cicloviolacinas O8, O11 e O13, respectivamente; e Vov1, produtor da violacina A) e um precursor triplo (Vok1, o qual produz a kalata B1 e duas kalata S) (DUTTON et al., 2004; IRELAND; COLGRAVE; CRAIK, 2006). Em V. tricolor foram encontrados um precursor duplo (Triciclon, expressando os ciclotídeos triciclon A e triciclon B), e um triplo (Vtt1, expressando três cópias do triciclon A) (Figura 7) (MULVENNA; SANDO; CRAIK, 2005; GRUBER et al., 2008). Em V. biflora foram identificados seis precursores simples (Vbc1-6, expressando os ciclotídeos vibi E, vibi I, vibi J, vibi K, cicloviolacina O9 e vitri A, respectivamente (HERRMANN et al., 2008). Em V. baoshanensis, foram elucidados sete precursores e diversas variantes extremamente semelhantes para cada um deles, mas possuindo ciclotídeos diferentes, totalizando 27 precursores e tornando-se a espécie com maior número de precursores conhecidos dentro das Violaceae, com dezesseis precursores simples (VbPC1-5 e suas variantes, expressando diversas cicloviolacinas B e a cicloviolacina Y), quatro precursores duplos (VbPC6a-d, expressando cicloviolacinas e kalatas) e três precursores triplos (VbCP7a-c, expressando também cicloviolacinas e kalatas (ZHANG et al., 2009).
gerando os ciclotídeos hyfl D, E, I-M, respectivamente) e dezesseis precursores parciais somando as espécies H. calycinus, H. debilissimus, H. enneaspermus, H. epacroides, H. monopetalus, H. stellarioides e H. vernonii (SIMONSEN et al., 2005). Além destes, e assim como em V. baoshanensis, a espécie Melicytus ramiflorus também apresentou um grande conjunto de precursores diferentes e suas variantes, com oito precursores simples (Mra13, Mra20-26) (Figura 7), sete precursores duplos (Mra4, Mra14, Mra18-19, Mra28-30) e dois precursores parciais (Mra16 e Mra17), todos estes produzindo os ciclotídeos mra4 até mra30 (GRUBER et al., 2008; TRABI et al., 2009). As espécies Gloeospermum blakeanum e Gloeospermum pauciflorum foram as últimas desta família a terem precursores elucidados, as quais apresentaram dez precursores simples (Gbc1-6 e Gpc1-4, gerando os ciclotídeos globa A-B, globa D-G, glopa D-E e vibi E) (BURMAN et al., 2010).
Em adição aos ciclotídeos encontrados nos grupos Rubiaceae e Violaceae, um trabalho pioneiro de busca por ciclotídeos na família Fabaceae elevou o interesse por esses peptídeos, pois o grupo das leguminosas, além de representar a terceira maior família de plantas do planeta, também é detentora de numerosas espécies importantes para o agronegócio e para a economia mundial. Assim, para entender melhor a distribuição e evolução dos ciclotídeos nesse grupo, extratos de Clitoria ternatea foram analisados e evidenciaram a existência de ciclotídeos na família Fabaceae, representando atualmente o maior grupo vegetal produtor de ciclotídeos. Quando estudada a nível transcricional, surpreendentemente, os precursores de C. ternatea revelaram uma arquitetura gênica totalmente inédita entre os genes de ciclotídeos conhecidos, onde onze precursores (CterM, Ctc1-2, Ctc4-5, Ctc7-9, Ctc12-14, expressando os ciclotídeos cter A, cter B, cter M, cT1-cT12) apresentaram uma estrutura quimérica, alocando no mesmo precursor um domínio de ciclotídeo (MCD) e um domínio da cadeia A da albumina de ervilha (Pisum sativum Albumin 1-A chain), separados por uma região ligante de 10 aminoácidos (Figura 7). Assim como os outros precursores já conhecidos, este precursor tem inicio com um peptídeo sinal (ER-Signal) e termina com uma porção C-terminal hidrofóbica, porém não apresenta o pró-domínio N-terminal (NTPD) e nem o pró-domínio de repetição do N-terminal (NTR). Esse novo arranjo gênico, evidenciando a substituição da cadeia b da albumina por um domínio de ciclotídeo, também implica em diferentes mecanismos de seu processamento e evolução, abrindo novas possibilidades de estudos dentro deste grupo multigênico (POTH et al., 2011a,b; NGUYEN et al., 2011a).