Fenótipo da obesidade, ancestralidade
genética e polimorfismos em genes candidatos
em escolares de uma população miscigenada
Erica Maria de Queiroz
Ouro Preto
Agosto - 2012
II
Erica Maria de Queiroz
Fenótipo da obesidade, ancestralidade
genética e polimorfismos em genes candidatos
em escolares de uma população miscigenada
Tese apresentada ao Programa de Pós-Graduação
em Ciências Biológicas da Universidade Federal de
Ouro Preto como parte dos requisitos para obtenção
do título de Doutor em Ciências Biológicas.
V
FINANCIAMENTO
Este trabalho foi desenvolvido com financiamento e apoio das seguintes
instituições:
Conselho Nacional de Pesquisa e Desenvolvimento Tecnológico
–
CNPq
Coordenação de Aperfeiçoamento de Pessoal de Nível Superior
–
CAPES
Fundação de Amparo a Pesquisa de Minas Gerais
–
FAPEMIG
Universidade Federal de Ouro Preto
–
UFOP
Fundação de Apoio a Pesquisa do Distrito Federal
–
FAPDF
VI
AGRADECIMENTOS
Aos professores Dra. Renata Nascimento de Freitas e Dr. Ieso de Miranda
Castro pela oportunidade e ensinamentos.
Aos professores Dr. George L. Lins Machado Coelho, Dra. Ana Paula Carlos
Cândido, Dr. Rinaldo
Wellerson Pereira e Dra. Tailce Moura Leite, pela
colaboração.
Aos alunos de Iniciação Científica Ana Maria dos Santos, Fernando Augusto de
Oliveira Coelho e Priscila Oliveira Barbosa, pelo apoio e pela oportunidade da
orientação.
VII
AGRADECIMENTOS PESSOAIS
A Deus, por mais esta conquista.
Ao Julio, por entender meus momentos de estresse, minhas dúvidas e pela
habilidade em lidar com a situação. Agradeço também pelas importantes
observações e pelo apoio nas análises estatísticas.
Ao João, que mesmo sem entender, aceitou todas as mudanças que o
cercavam e enche a nossa casa de muita alegria.
A minha família, pelo apoio espiritual e por entenderem a minha ausência. Em
especial, à Maria Martha que em muitos momentos foi avó, mãe e babá do
João.
VIII
RESUMO
O cenário atual da obesidade no mundo tem despertado a atenção da comunidade científica. Entretanto, a influência de fatores diversos na obesidade poligênica tem sido um grande desafio nos estudos sobre esta condição. Estudos de associação têm indicado genes candidatos à obesidade e a fenótipos relacionados. Estudos em populações miscigenadas mostram que a prevalência da obesidade e de doenças relacionadas, tais como a diabetes tipo 2 e a hipertensão, pode variar conforme o grupo étnico. Entretanto, este é um universo ainda desconhecido. O objetivo do presente estudo foi verificar a associação de polimorfismos genéticos com a adiposidade e a relação da ancestralidade genômica com fenótipos associados a obesidade em uma amostra de crianças e adolescentes escolares de Ouro Preto, MG. Para tanto, determinamos a ancestralidade genética de 189 indivíduos utilizando 15 marcadores informativos de ancestralidade (MIAs) e correlacionamos com a cor da pele auto-referida através de ANOVA seguida por teste Post Hoc. Adicionalmente,
segregamos os indivíduos em três grupos (predominantemente Africano - PAFR, predominantemente Miscigenado – PMIS e predominantemente Europeu - PEUR) utilizando a proporção de ancestralidade >0,650 como ponto de corte e comparamos os valores médios de características antropométricas, clínicas, bioquímicas e demográficas. Modelo de regressão linear simples foi testado para verificar se diferenças nos valores médios das variáveis entre os grupos foram independentes ou não de outras variáveis. Posteriormente realizamos uma análise de associação entre 12 polimorfismos de base única (single nucleotide polymorphism – SNP) em genes
IX
SLC6A14 rs2011162, POMC rs28932472, IGF2 rs680, LEPR rs1137101 e PPARGC1
rs8192678) foram associados a valores médios de variáveis antropométricas e/ou bioquímicas. Na análise de regressão logística binária ajustada por sexo e idade, cinco SNPs (APM1 rs266729:C>G, IGF2 rs680:G>A, LEPR rs1137101:A>G POMC
rs28932472:C>G and PPARGC1 rs8192678:A>G) apresentaram odds ratio
X
ABSTRACT
The current scenario of obesity in the world has called the attention of scientists worldwide. However, the influence of several factors in polygenic obesity has been a major barrier for studies about this condition. Association studies have indicated candidate genes for obesity and related phenotypes. Studies in admixed population shows that the prevalence of obesity and related diseases such as type 2 diabetes and hypertension, may vary by ethnic group. However, this is a universe still unknown. The aim of this study was to investigate the association of genetic polymorphisms with adiposity and the relationship of genetic ancestry with phenotypes associated with obesity in a sample of school children and adolescents from Ouro Preto, MG. So, we determined the genetic ancestry of 189 individulas using 15 ancestry informative markers (AIMs) and correlated them to self-reported skin color by ANOVA followed by
Post Hoc test. Additionally, we segregate individuals into three groups (predominantly
African – PAFR, predominantly Multiethnic - PMIS and predominantly European - PEUR) using the proportion of ancestry> 0.650 as the cutoff point and comparing the mean values of anthropometric, clinical, biochemical and demographic variables. Simple linear regression model was tested to verify whether differences in mean values of variables between groups were independent or not of other variables. Subsequently, we performed an analysis of association between 12 SNPs (single nucleotide polymorphisms – SNPs) in candidate genes with adiposity and with risk phenotypes which are components of metabolic syndrome and related to obesity. Our results show that the contribution of European (EUR), African (AFR) and Amerindian ancestries in the population accounted for 50.3%, 33.3% and 16.4%, respectively. The black and light brown skin color groups are not distinguished with respect to AFR and EUR ancestries and overlap of ancestries were observed between groups. PAFR individuals had higher mean blood pressure and glucose and lower hemoglobin levels compared to PEUR. In the linear regression model, the difference in systolic blood pressure, and glucose between the groups was independent of other variables whilst hemoglobin was dependent on systolic and diastolic blood pressure, glucose, lipids and income. Although we did not observe any association of the 12 SNPs with adiposity, other associations were observed. Seven SNPs (APM1 rs266729, KCNJ11 rs5219,
SLC6A14 rs2011162, POMC rs28932472, IGF2 rs680, LEPR rs1137101 e PPARGC1
rs8192678) were associated with mean values of anthropometric and/or biochemical variables. In the logistic binary regression adjusted by sex and age, five SNPs (APM1
XI PPARGC1 rs8192678:A>G) showed significant odds ratio for presenting at least one
XII
Lista de quadros
Quadro 1.1: Sumário das informações sobre os genes e SNPs do estudo
24
Quadro 2.1: Localização, alelos polimórficos e frequência nas populações parentais dos 15 MIAs
37
Quadro 2.2: Sequência, concentração e agrupamento dos iniciadores da PCR e da reação multiplex de extensão de base única
XIII
Lista de tabelas
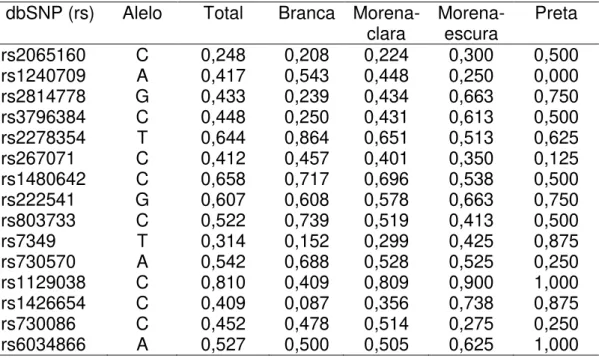
Tabela 2.1: Frequências alélicas dos 15 marcadores indicativos de ancestralidade na população e nos grupos de cor da pele auto-referida
41
Tabela 2.2: Estimativa de mistura da população e para os grupos de cor da pele auto-referida
43
Tabela 2.3: Comparação da estimativa de mistura entre todos os grupos de cor da pele auto-referida
44
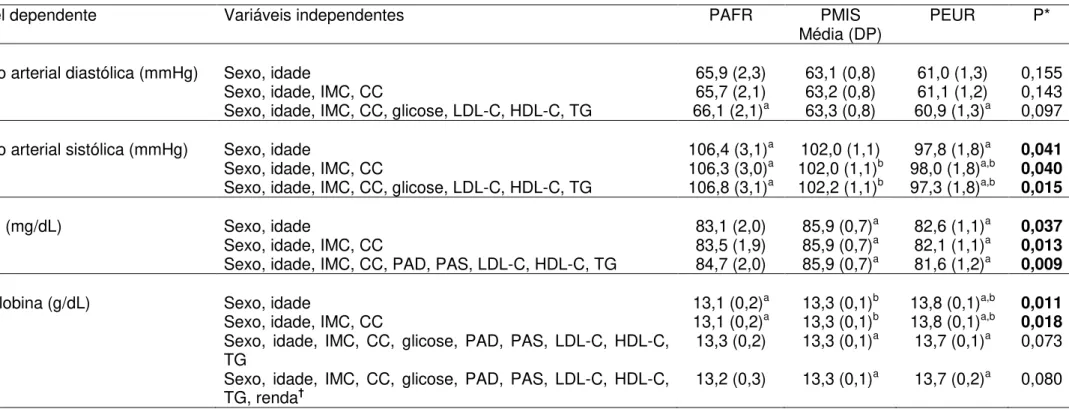
Tabela 3.1: Características antropométricas, bioquímicas e clínicas dos grupos predominantemente Africano (PAFR), predominantemente Europeu (PEUR) e predominantemente miscigenado (PMIS)
57
Tabela 3.2: Regressão univariada para valores médios de pressão arterial, glicose e hemoglobina entre os grupos predominantemente Africano (PAFR), predominantemente Europeu (PEUR) e predominantemente miscigenado (PMIS) ajustada por diferentes covariáveis
58
Tabela 4.1: Seqüência e concentração dos iniciadores da PCR para a amplificação das regiões flanqueadoras dos SNPs
71
Tabela 4.2: Seqüência e concentração dos iniciadores da reação de minisequenciamento
73
Tabela 4.3: Características demográficas, antropométricas e bioquímicas da amostra da população e dos sexos
77
Tabela 4.4: Características demográficas, antropométricas e bioquímicas dos grupos de sobrepeso e controle
79
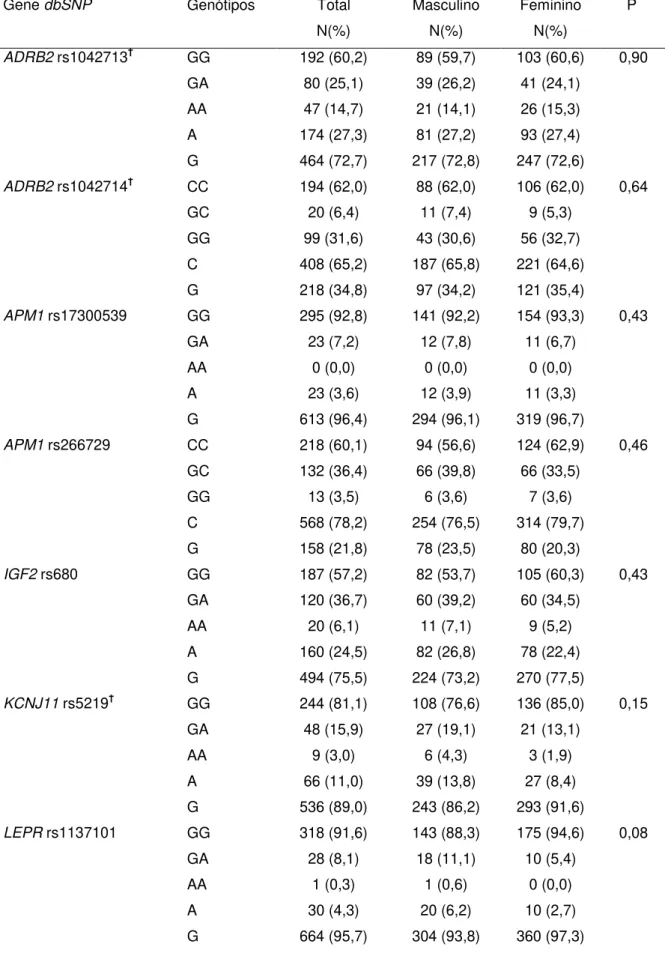
Tabela 4.5: Freqüência de genótipos e alelos dos polimorfismos do estudo na amostra da população e entre os sexos
XIV
Tabela 4.6: Frequência de genótipos e alelos dos SNPs nos grupos de sobrepeso e controle
84
Tabela 4.7: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP ADRB2
rs1042713
87
Tabela 4.8: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP ADRB2
rs1042714
88
Tabela 4.9: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP APM1
rs17300539
89
Tabela 4.10: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP APM1
rs266729
90
Tabela 4.11: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP IGF2 rs680
91
Tabela 4.12: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP KCNJ11
rs5219
92
Tabela 4.13: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP LEPR
rs1137101
93
Tabela 4.14: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP POMC
rs28932472
94
XV
amostra da população para os grupos de genótipos do SNP PPARG
rs1801282
Tabela 4.16: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP PPARGC1
rs8192678
96
Tabela 4.17: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP SLC6A14
rs2011162
97
Tabela 4.18: Características antropométricas, clínicas e bioquímicas da amostra da população para os grupos de genótipos do SNP UCP2
rs659366
98
Tabela 4.19: Razão de chances (OR) para fenótipos selecionados ajustado por idade e sexo de acordo com diferentes genótipos dos SNPs ADRB2 rs1042713, ADRB2 rs1042714, APM1 rs17300539 e APM1 rs266729
100
Tabela 4.20: Razão de chances (OR) para fenótipos selecionados ajustado por idade e sexo de acordo com diferentes genótipos dos SNPs IGF2 rs680, KCNJ11 rs5219, LEPR rs1137101 e POMC
rs28932472
101
Tabela 4.21: Razão de chances (OR) para fenótipos selecionados ajustado por idade e sexo de acordo com diferentes genótipos dos SNPs PPARG rs1801282, PPARGC1 rs8192678, SLC6A14 rs2011162
e UCP2 rs659366
102
XVI
Lista de figuras
Figura 1.1: Representação esquemática e resumida do balanço entre a ingestão de alimentos e o gasto energético envolvidos na obesidade
6
Figura 1.2: Representação esquematizada dos mecanismos de ação dos genes ADRB2, APM1, IGF2, KCNJ11, LEPR, POMC, PPARG, PPARGC1, SLC6A14 e UCP2
26
Figura 2.1: Gráfico boxplot da estimative de mistura entre os grupos de
cor da pele auto-referida: branca, morena-clara e morena-escura + preta
47
Figura 4.1: Eletroferograma e agrupamento dos iniciadores do minisequenciamento
XVII
Lista de abreviaturas e símbolos
% Porcentagem
%GC Porcentagem de Gordura Corporal < Menor que
= Igual
≥ Maior ou igual µl Micro litro µM Micro molar µU Micro unidade AFR Africano AMR Amerindio
CC Circunferência da Cintura
CDC Centro para Controle de Doença cm Centímetros
dATP Deoxiadenina Trifosfato
dbSNP Banco de Dados de Polimorfismo de Nucleotídeo Único
ddCTP Dideoxicitocina Trifosfato dGTP Deoxiguanosina Trifosfato dL Decilitro
DNA Ácido desoxirribonucleico DP Desvio Padrão
dTTP Deoxitimidina Trifosfato EUR Europeu
g Grama
GWAS Estudos de Associação em Todo o Genoma HapMap Projeto Internacional HapMap
HDL-C Lipoproteína de Alta Densidade
HOMA-IR Modelo de Homeostase de Resistência a Insulina IBGE Instituto Brasileiro de Geografia e Estatística IC Intervalo de Confiança
IDF Federação Internacional de Diabetes IMC Índice de Massa Corporal
Ipea Instituto de Pesquisa Econômica Aplicada kg Kilograma
XVIII
LDL-C Lipoproteína de Baixa Densidade m² Metro ao quadrado
mg Miligrama
MIAs Marcadores Indicativos de Ancestralidade min Minuto
mmHg Milímetro de Mercúrio mmol Mili mol
NCBI Centro Nacional de Informação Biotecnológica
NCEP-ATPIII National Cholesterol Education Program-Adult Treatment Panel III
ng Nanograma ºC Graus Celso
OMS Organização Mundial de Saúde OR Razão de Chances (Odds Ration)
PAD Pressão Arterial Diastólica PAFR Predominantemente Africano PAS Pressão Arterial Sistólica PEUR Predominantemente Europeu PMIS Predominantemente Miscigenado s Segundo
SM Salários Mínimos
SNP Polimorfismo de Nucleotídeo Único TG Triacilgliceróis
XIX
SUMÁRIO
INTRODUÇÃO 1
CAPÍTULO 1 3
1.1 Estado da Arte Obesidade
O gene ADRB2 e SNPs rs1042713 e rs1042714
O gene APM1 e SNPs rs17300539 e rs266729
O gene PPARG e SNP rs1801282
O gene PPARGC1 e SNP rs8192678
O gene UCP2 e SNP rs659366
O gene KCNJ11 e SNP rs5219
O gene IGF2 e SNP rs680
O gene LEPR e SNP rs1137101
O gene POMC e SNP rs28932472
O gene SLC6A14 e SNP rs2011162
Marcadores indicativos de ancestralidade Ouro Preto Ouro Preto
Estudos epidemiológicos em Ouro Preto
1.2 Objetivos
1.2.1 Objetivo Geral 1.2.2 Objetivos Específicos
3 3 8 10 12 13 15 17 18 19 21 22 27 29 30 32 32 32
CAPÍTULO 2 – Composição genética de uma população Brasileira: a pegada do Ciclo do Ouro
Introdução
Material e métodos
População do estudo População parental Marcadores selecionados
Ensaio de genotipagem
XX
CAPÍTULO 3 - Ancestralidade genética está associada a pressão arterial sistólica, glicose e hemoglobina em escolares de Ouro Preto
Introdução
Material e métodos
População do estudo Ancestralidade genética Análises estatísticas Resultados Discussão 50 50 52 52 53 53 55 59
CAPÍTULO 4 - Variáveis nos genes APM1, IGF2, LEPR, POMC e PPARGC1
são associadas a componentes da Síndrome Metabólica em crianças e adolescentes brasileiros
Introdução
Material e métodos
População do estudo Marcadores selecionados Desenho dos iniciadores Ensaio de Genotipagem Análises estatísticas Resultados
Padronização da metodologia de minisequenciamento Caracterização da população
Distribuição dos polimorfismos
Associação de polimorfismos genéticos com o fenótipo da obesidade Discussão 62 62 66 66 67 67 68 68 70 70 76 81 86 103
CAPÍTULO 5 - Conclusões e considerações finais 108
CAPÍTULO 6 - Referências bibliográficas 110
ANEXOS
1: Aprovação pelo Comitê de Ética em Pesquisa 2: Termo de consentimento livre e esclarecido
1
INTRODUÇÃO
O cenário atual da obesidade no mundo tem despertado a atenção de muitos pesquisadores com o intuito de entender os mecanismos envolvidos nesta complexa enfermidade e propor medidas que possam controlar ou, pelo menos, minimizar seus efeitos.
A influência de fatores diversos na obesidade poligênica tem sido uma grande barreira, por isso os avanços são pequenos e lentos, mas muito importantes. Estudos de associação têm indicado genes candidatos a susceptibilidade à obesidade e a fenótipos relacionados. Adicionalmente, estudos em populações miscigenadas, como é o caso do Brasil, mostram que a prevalência da obesidade e de doenças relacionadas, tais como a diabetes tipo 2 e a hipertensão, pode variar conforme o grupo étnico. Entretanto, este é um universo ainda desconhecido.
2
Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - CAPES, Fundação de Amparo à Pesquisa do Estado de Minas Gerais – FAPEMIG (325/05, EDT-CDS 770/05, EDT-CDS - APQ-00519-09, CBB-APQ-02260-10), pela Pró-reitoria de Pesquisa e Pós-graduação (PROPP) da Universidade Federtal de Ouro Preto, pela
3
CAPÍTULO 1
1.1 - ESTADO DA ARTE
Obesidade
A obesidade tem sido considerada uma pandemia devido ao seu rápido avanço em todas as partes do mundo. Atualmente, o sobrepeso e a obesidade são considerados o quinto maior risco de mortes no mundo, ocasionando pelo menos 2,8 milhões de mortes de adultos a cada ano, 44% das diabetes, 23% das doenças isquêmicas do coração e 7% a 41% de certos tipos de câncer (disponível no site da Organização Mundial de Saúde, http://www.who.int, acesso em Maio de 2012).
Em crianças com idade inferior a 5 anos, a prevalência de sobrepeso e obesidade estimada em 2010 para o continente americano (6,9%) foi o segundo maior do mundo ficando atrás apenas da África (8,5%). Países desenvolvidos se destacam por apresentarem elevada prevalência de sobrepeso e obesidade. Entretanto, os países em desenvolvimento tem acompanhado este avanço por adotarem o estilo de vida ocidentalizado (Onis e col., 2010). A prevalência de sobrepeso e obesidade nesta faixa etária nos Estados Unidos corresponde a 17,4 % e 10,4 a 14,7% (Sekhobo e col., 2010 ; Karnik e Kanekar, 2012), respectivamente, enquanto em países da América do Sul, a prevalência de sobrepeso chega a 7,3 % na Argentina, 6,5 % na Bolívia, 7 % no Chile, 6,2 % na Costa Rica e 4,9 % no Brasil (Onis e Blössner, 2000). Em crianças e adolescentes, 18,9 % dos norte-americanos são obesos (Karnik e Kanekar, 2012), enquanto 27 % dos brasileiros nesta faixa etária estão acima do peso recomendado e 9,6 % são obesos (IBGE, 2010a). Entretanto, estudos mostram que as prevalências de sobrepeso e obesidade em adolescentes brasileiros podem variar conforme o ano do estudo, região geográfica e ponto de corte (Lamounier e Abrantes, 2003; Tassitano e col., 2009). Em adultos, 33,6 % dos norte-americanos são obesos (Ford e col., 2011), enquanto 49 % dos adultos brasileiros estão acima do peso recomendado e 14,8 % são obesos (IBGE, 2010a).
4
(Bell e col., 2005; Walley e col., 2006) muitas formas de cânceres (Calle e col., 2003), doenças pulmonares, hipertensão arterial (Bell e col., 2005; Walley e col., 2006), dislipidemia, doenças osteoarticulares e doenças psiquiátricas graves (Walley e col., 2006).
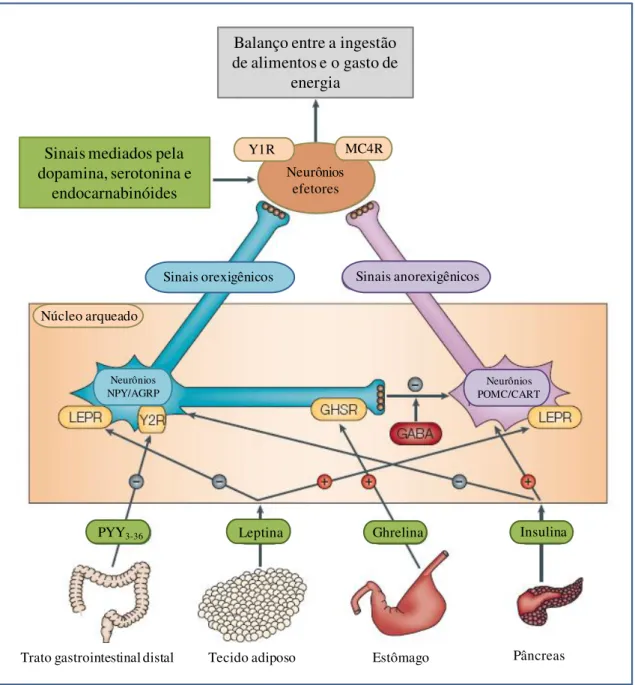
A obesidade é causada por pertubações no equilíbrio entre a ingestão alimentar e o gasto energético que é regulado por um sistema complexo de interações mediado por sinais neurais e hormonais, sendo coordenado pelo hipotálamo. A figura 1.1, publicada por Bell e colaboradores (2005), apresenta de forma resumida e esquematizada os mecanismos envolvidos neste processo. A rede neural formada pelo núcleo arqueado desempenha função essencial neste sistema por apresentar dois grupos de neurônios: orexigênico e anorexigênico. O grupo orexigênico é formado pelos neurônios AGRP/NPY que expressam o peptídeo relacionado à agouti (AGRP) e o neuropeptídeo Y (NPY) promovendo a ingestão alimentar e a redução do gasto energético. O grupo anorexigênico é formado pelos neurônios POMC/CART que expressam a proopiomelanocortina (POMC) e o transcrito relacionado à cocaína e anfetamina (CART) promovendo efeito oposto a via orexigênica. Neste cenário, atuam diferentes hormônios, tais como a insulina, grelina, peptídeo PYY3-36 e leptina, sendo
que cada um é produzido e tem seu efeito em local específico. A insulina, produzida no pâncreas, promove a ativação da via anorexigênica e inibição da via orexigênica, estando envolvida na regulação do consumo alimentar de moderada a longo prazo. A leptina, produzida no tecido adiposo, promove a ativação da via anorexigênica e inibição da via orexigênica dependentes de sua ligação no receptor da leptina (LEPR), estando envolvida na regulação de curto e longo prazo. O hormônio grelina, produzido no estômago e duodeno, promove a ativação da via orexigênica através de sua ligação no receptor de secretagogos do hormônio de crescimento (GHSR), enquanto o peptídeo PYY3-36, produzido trato gastrointestinal distal, promove a inibição da via
5
6 Balanço entre a ingestão
de alimentos e o gasto de energia
Sinais mediados pela dopamina, serotonina e
endocarnabinóides
Neurônios efetores
Neurônios efetores
Y1R MC4R
Sinais orexigênicos Sinais anorexigênicos
PYY3-36 Leptina Ghrelina Insulina
Pâncreas Estômago
Tecido adiposo Trato gastrointestinal distal
Neurônios POMC/CART Neurônios
NPY/AGRP
Núcleo arqueado
7
A forma poligênica ou sindrômica da obesidade é a mais comum e associada a
características clínicas distintas, mas geralmente envolve retardo mental (Bell e
col., 2005). As causas das obesidades comuns decorrem da interação entre a
predisposição genética (fatores genéticos) e o estilo de vida ocidentalizado
(fatores ambientais) (Dahlman e Arner, 2007). Estudos com gêmeos sugerem
que os fatores genéticos explicam de 50 a 90% da variação inter-individual do
índice de massa corporal (IMC) (Maes e col., 1997), o marcador de obesidade
mais comumente usado em estudos epidemiológicos (O’Rahilly e Farooqi,
2006). Por outro lado, o ambiente obesogênico criado pela grande oferta de
alimentos calóricos (principalmente
fast foods) associado ao baixo custo
desses alimentos, tempo reduzido para fazer a refeição e sedentarismo tem
sido os grandes vilões da obesidade (Bell e col., 2005; Farooqi e O’Rahilly
2007).
8
índice de massa corporal, circunferência da cintura, fatores de risco
cardiovasculares, colesterol total, HDL-C e LDL-C, triacilgliceróides, receptor de
leptina,
obesidade,
diabetes
e
outros
traços
metabólicos
(http://www.genome.gov/gwastudies, acesso em Junho de 2012).
Dezoito genes (ARG1, ASAH1, BDNF, FAT1, FTO, INHBB, KCNMA1, LHFPL3,
MC4R,
MAF,
NCAM2, NPC1, NRXN3,
PCDH9,
PITPNB,
PTER, UBE2E3 e
WWOX) associados à obesidade foram identificados pelos GWAS (Meyre e
col., 2009; Jiao e col., 2011; Wang e col., 2011). Outros genes se associam
inequivocamente a fatores relacionados com a obesidade, tais como o índice
de massa corporal (BDNF, CDKAL1, FTO, GIPR, GNPDA2, KCTD15, MAP2K5,
MC4R,
MTCH2,
NEGR1,
SEC16B,
SH2B1 e
TMEM18) (Willer e col., 2009;
Speliotes e col., 2010; Wen e col., 2012), a diabetes tipo 2 (C2CD4B, CDC123,
CDKAL1,
CDKN2A,
CDKN2B,
FTO,
HHEX,
HMG20A,
HNF1A,
IGF2BP2,
JAZF1,
KCNJ11,
KCNQ1,
PPARG,
PTPRD,
SLC30A8,
TCF7L2 e
TSPAN8)
(Zeggini e col., 2008; Voight e col., 2010; Tsai e col., 2010; Shu e col., 2010;
Kooner e col., 2011), a doença cardiovascular (ABO,
ADAMTS7,
CDKN2A,
CELSR2,
CXCL12,
KIAA1462,
LIPA,
MIA3,
MRAS,
PDGFD,
PHACTR1 e
PPAP2B) (Slavin e col., 2011; Schunkert e col., 2011; Coronary Artery Disease
(C4D) Genetics Consortium, 2011) e a hipertensão arterial (CACNB2) (Levy e
col., 2009; International Consortium for Blood Pressure Genome-Wide
Association Studies e col., 2011).
9
.
O gene
ADRB2
e SNPs rs1042713 e rs1042714
O gene
ADRB2 (5q31-q32) possui um transcrito de 2058 pb que se expressa
na forma de proteína receptora adrenérgica
β
2. A proteína, que contêm 413
aminoácidos,
é um subtipo da família de receptores adrenérgicos β
que
interage com a proteína G estando envolvida na regulação do balanço
energético por estimular tanto a termogênese quanto a lipólise no tecido
adiposo (Zhao-qian e col., 2007) em resposta a hormônios catecolaminas
(adrenalina, noradrenalina e dopamina) (Dahlman e Arner, 2007). Atualmente,
existem 157 polimorfismos de nucleotídeos únicos (SNPs) descritos para este
gene em humanos (disponível no site http://www.ncbi.nlm.nih.gov, acesso em
Julho de 2012). Um desses SNPs é o rs1042713 que consiste na troca de
adenina por guanina na posição 285 do transcrito levando à substituição de
arginina por glicina no resíduo 16 da proteína (Arg16Gly). A presença do
aminoácido glicina tem sido relacionada à alteração na função de
ADRB2
resultando no aumento de 5 vezes de sua afinidade e/ou acoplamento com
agonista em células adiposas (Large e col., 1997). A frequência do alelo G
deste polimorfismo tem sido menor em africanos ou descendentes de africanos
(43 a 50 %) e maior em europeus (55 a 66 %) em comparação ao alelo A,
segundo o Projeto International HapMap (http://hapmap.ncbi.nlm.nih.gov,
acesso em Maio de 2012). Outro SNP muito estudado é o rs1042714 que
consiste na troca de citosina por guanina na posição 318 do transcrito levando
à substituição de glutamina por glutamato no resíduo 27 da proteína
(Gln27Glu). Entretanto, a presença do aminoácido glutamato parece não alterar
a função de ADRB2 em células adiposas (Large e col., 1997). A frequência do
alelo G deste polimorfismo tem sido menor que a do alelo C, tanto em africanos
(18 %) quanto em europeus (47 %), segundo o Projeto International HapMap
(http://hapmap.ncbi.nlm.nih.gov, acesso em Maio de 2012).
10
col., 2003; Tafael e col., 2004; Galetti e col., 2004; Terra e col., 2005; Masuo e
col., 2006; Mattevi e col., 2006; Podolsky e col., 2007; Kunnas e col., 2009),
hipertensão (Castellano e col., 2003; Pereira e col., 2003; Galetti e col., 2004;
Bostrom e col., 2010; Lou e col., 2011) e diabetes tipo 2 (Kawamura e col.,
2001; Carlsson e col., 2001; Chang e col., 2002; Sánchez e col., 2003) têm
apresentado resultados divergentes. Uma meta-análise realizada em 2007
indicou associação positiva do SNP rs1042713 com diabetes tipo 2, síndrome
metabólica e pressão arterial sistólica em mulheres e não associação com a
obesidade e a hipertensão. O SNP rs1042714 foi positivamente associado a
pressão arterial sistólica em mulheres, mas não associado a obesidade,
diabetes tipo 2, hipertensão e síndrome metabólica (Gjesing e col., 2007). Em
outra meta-análise realizada, também não foi observada a associação de
ambos SNPs com a obesidade, exceto para o SNP rs1042714 no grupo racial
formado pela população da Ásia e Ilhas do Pacífico (Jalba e col., 2008).
Adicionalmente, estes SNPs não têm sido associados a parâmetros
antropométricos ou bioquímicos (Kawamura e col., 2001; Kim e col., 2002;
Sánchez e col., 2003; Galetti e col., 2004; Terra e col., 2005), embora uma
associação positiva entre o SNP rs1042714 com colesterol total, LDL-C
(Sánchez e col., 2003), insulina e ácidos graxos não esterificados (Carlsson e
col., 2001) tenha sido reportada. Estudos de haplótipos demonstraram a
associação de Arg16Gln27 com pressão arterial, IMC, triacilgliceróis,
frequência cardíaca e glicemia em adultos entre 35 e 50 anos (Castellano e
col., 2003).
O gene
APM1
e SNPs rs17300539 e rs266729
11
por AMP (AMPK) e de
PPAR
no músculo esquelético e inibe a captação e
estocagem da glicose no fígado (Ferrarezi e col., 2007). Atualmente, existem
2589 SNPs descritos para este gene em humanos (disponível no site
http://www.ncbi.nlm.nih.gov, acesso em Julho de 2012). Um desses SNPs é o
rs17300539 que consiste na troca de guanina para adenina no promotor do
gene (-11391). A frequencia do alelo G deste polimorfismo tem sido maior que
a do alelo A, tanto em descendetes de africanos (98 %) quanto em europeus (>
92
%),
segundo
o
Projeto
International
HapMap
(http://hapmap.ncbi.nlm.nih.gov, acesso em Maio de 2012). Outro SNP muito
estudado é o rs266729 que consiste na troca de citosina para guanina no
promotor do gene (-11365). A frequência do alelo C deste polimorfismo tem
sido maior que a do alelo G, tanto em descendentes de africanos (70 %)
quanto em europeus (92 %), segundo o Projeto International HapMap
(http://hapmap.ncbi.nlm.nih.gov, acesso em Maio de 2012). Embora alguns
estudos sugiram que a mudança de base em ambos SNPs possa resultar em
reduzido nível circulante de adiponectina, os mecanismos envolvidos neste
processo são desconhecidos. Em estudo de caracterização do promotor do
gene
APM1 humano não foram identificados sítios de ligação de fatores de
transcrição em ambos SNPs e nem em suas sequências adjacentes (Shaffer e
col., 1998). Entretanto, uma análise computacional realizada da sequência
abrangendo os nucleotídeos -11435 a -11345 indentificou a sequência [tcctgc],
localizada entre os SNPs e adjacente à rs266729, com similaridades à
sequência de elemento acentuador (enhancer) do gene receptor do fator de
crescimento epidérmico (EGFR), além de múltiplos sítios protegidos por
proteínas (Maekawa e col., 1989).
12
e col., 2004; Gaunchi e col., 2008; Vaxillaire e col., 2008). Uma meta-análise
realizada em 2010 indicou associação positiva de ambos SNPs com diabetes
tipo 2, sendo a associação com o SNP rs17300539 apenas em europeus
(Gong e col., 2010). Mais recentemente, outra meta-análise indicou associação
positiva do SNP rs266729 com o risco de doença cardiovascular (Zhang e col.,
2012). Adicionalmente, o SNP rs17300539 tem sido marginalmente associado
a concentração de insulina e HOMA-IR (Henneman e col., 2010), mas não
associado à hipertensão (Ong e col., 2010), enquanto o SNP rs266729 tem
sido associado ao IMC (Gu e col., 2004), acidente vascular cerebral (Hegener e
col., 2006) e hipertensão (Ong e col., 2010), mas não associado a doença
coronariana (Pischon e col., 2007). Estudos de haplótipo tem demostrado a
associação de rs17300539G/rs266729G com o nível de adiponectina (Vasseur
e col., 2002), mas resultados divergentes são observados para a diabetes tipo
2 (Vasseur e col., 2002; Gu e col., 2004).
O gene
PPARG
e SNP rs1801282
13
prolina por alanina no resíduo 12 da proteína (Pro12Ala). A presença do alelo
Ala tem sido relacionada com baixa atividade da proteína devido à redução de
sua afinidade com o DNA e transativação
in vitro
(Deeb e col., 1998). A
frequência do alelo C deste polimorfismo tem sido maior que a do alelo G tanto
em descendentes de africanos e africanos (ambos 98 %) quanto em europeus
(>90
%),
segundo
o
Projeto
International
HapMap
(http://hapmap.ncbi.nlm.nih.gov, acesso em Maio de 2012).
Embora o SNP rs1801282 seja intensivamente estudado com relação a sua
associação com a diabetes tipo 2 (Oh e col., 2000; Swarbrick e col., 2001; Lindi
e col., 2002; Parra e col., 2004; Tavares e col., 2005; Hansen e col., 2005;
Weedon e col., 2006; Cauchi e col., 2008; Gaulton e col., 2008; Lyssenko e
col., 2008; Wang e col., 2009; Hu e col., 2009), com o IMC (Deeb e col., 1998;
Cole e col., 2000; Lindi e col., 2002; Sánchez e col., 2002; Hansen e col., 2005;
Fornage e col., 2005) e com as doenças cardiovasculares (Bluher e col., 2002;
Ridker e col., 2003; Pischon e col., 2005), os resultados tem sido divergentes.
Estudos de meta-análise têm indicado uma associação positiva entre este SNP
e a diabetes tipo 2, sendo que diferentes valores de risco foram atribuídos a
diferentes grupos étnicos (Ludovico e col., 2007; Gouda e col., 2010). Outras
meta-análises indicaram associação positiva d
este SNP com IMC ≥ 27kg/m²
(Masud e col., 2003) e com doença cardiovascular em homens (Dallongeville e
col., 2009). Adicionalmente, os resultados de associação deste SNP com
colesterol total, triacilgliceróis, HDL-C, insulina e HOMA-IR tem sido
controversos (Swarbrinck e col., 2001; Sánchez e col., 2002; Lindi e col., 2002;
Masud e col., 2003; Hansen e col., 2005; Fornage e col., 2005; Tavares e col.,
2005).
O gene
PPARGC1
e SNP rs8192678
15
humanos (disponível no site http://www.ncbi.nlm.nih.gov, acesso em Julho de
2012). Um desses SNPs é o rs8192678 que consiste na troca de guanina para
adenina na posição 1564 do transcrito levando à substituição de glicina por
serina no resíduo 482 da proteína (Gly482Ser). A presença do aminoácido
serina tem sido reportada por reduzir a quantidade de transcrito do gene
PPARGC1 (Ling e col., 2004) resultando na diminuição da atividade da
proteína PPARGC1p. Adicionalmente, ensaios de duplo-híbrido em bactéria e
mutagênese direcionada demostraram que a presença de serina reduz a
afinidade de PPARGC1 com MEF2 (Shao-ling e col., 2007). A frequência do
alelo G deste polimorfismo tem sido maior que a do alelo A tanto em
descendentes de africanos (93 %) e africanos (>92 %) quanto em europeus (>
57
%),
segundo
o
Projeto
International
HapMap
(http://hapmap.ncbi.nlm.nih.gov, acesso em Maio de 2012).
Estudos de associação entre o SNP rs8192678 com diabetes tipo 2 (Kunej e
col., 2004;
Vimaleswaran e col., 2005; Sun e col., 2006;
Ridderstråle e col.,
2006;
Shao-ling e col., 2007) e com hipertensão (Oberkofler e col., 2003;
Andersen e col., 2005) tem apresentado resultados divergentes. Entretanto,
duas meta-análises indicaram associação positiva deste SNP com hipertensão
em adultos com idade inferior a 50 anos (Vimaleswaran e col., 2008) e com
a
diabetes tipo 2 (Barroso e col., 2006). Entretanto, associações negativas foram
descritas para IMC, glicose e insulina (Barroso e col., 2006). Adicionalmente,
este SNP tem sido associado a concentrações de ácidos graxos não
esterificados (Franks e col., 2007), LDL-C e triacilglicerídeos (Shao-ling e col.,
2007).
O gene
UCP2
e SNP rs659366
16
esquelético e rins. Entretanto, a ação de sua proteína é tecido específica. A
proteína atua na resposta a estresse oxidativo no fígado, cérebro e outros
tecidos, na regulação da disponibilidade de energia no coração, na regulação
do metabolismo de ácidos graxos no tecido adiposo branco e músculo
esquelético e na regulação da secreção da insulina nas células β
- pancreáticas
(Chan e col., 2006). A etapa final da oxidação de nutrientes ocorre na cadeia
respiratória mitocondrial, levando à produção de NADH e FADH2, transferência
de elétrons na cadeia respiratória e transporte de prótons para o espaço
intermembrana. No processo de transporte de prótons para o espaço
intermembrana, é gerada uma grande força protomotiva que se reduz quando
ocorre a incorporação de prótons para a matriz mitocondrial via ATP sintase
resultando na síntese de ATP. Alternativamente, a força protomotiva pode ser
dissipada na forma de calor via ação das UCPs levando à redução na
concentração de ATP que impede o fechamento dos canais de potássio (canal
K
ATP) e a secreção de insulina. A transcrição de UCP2 é induzida pela elevação
nas concentrações de ácidos graxos e glicose, mas parece que essa indução
também pode ser induzida por outros fatores. A elevação da concentração de
ácidos graxos induz a transcrição de UCP2 mediada pela ligação de PPAR no
elemento de resposta a PPAR (PPRE) localizado na região promotora de
UCP2. Dependendo da célula, diferentes PPARs podem atuar, sendo que a
ativação mediada por PPARG tem sido observada no tecido adiposo branco,
músculo esquelético e células β
-pancreáticas (Villarroya e col., 2007). A
ativação da transcrição de
UCP2 também pode ser induzida pela ligação de
ácido oleico ao elemento de resposta a fator de transcrição (SREBP-1c) no
promotor de
UCP2. Elevação da concentração de glicose induz a transcrição
de UCP2. Entretanto, os fatores de transcrição envolvidos neste processo são
desconhecidos, mas sugere-se que seja mediado por FOXA2, um importante
regulador de genes envolvido na secreção de insulina. Adicionalmente, a
ativação de
UCP2 pode ocorrer de forma endógena pela elevação nas
concentrações de superóxido (O
-2), produto da cadeia respiratória, que em
resposta é reduzido para a formação da molécula de H
2O, sem a produção de
17
principal função. Atualmente, existem 240 SNPs descritos para este gene em
humanos (disponível no site http://www.ncbi.nlm.nih.gov, acesso em Julho de
2012). Um desses SNPs é o rs659366 que consiste na troca de guanina para
adenina no promotor do gene (-866). A presença de adenina tem sido
relacionada à maior atividade do promotor de UCP2 resultando em aumento na
expressão da proteína e consequente redução na secreção de insulina
(Sasahara e col., 2004). A frequência do alelo G deste polimorfismo tem sido
maior que a do alelo A tanto em descendentes de africanos (58 %) e africanos
(>52 %) quanto em europeus (>63 %), segundo o Projeto International HapMap
(http://hapmap.ncbi.nlm.nih.gov, acesso em Maio de 2012).
Estudos de associação entre o SNP rs659366 com diabetes tipo 2 (Klempler e
col., 2002; D’Alamo e col., 2004; Sasahara e col., 2004; Bulotta e col., 2005;
Willer e col., 2007) e com a obesidade (
Klempler e col., 2002; D’Alamo e col.,
2004; Ochoa e col., 2007) tem apresentado resultados divergentes. Uma
meta-análise realizada em 2011 indicou ausência de associação entre este SNP e a
diabetes tipo 2 (Xu e col., 2011). Embora alguns estudos não tenham
observado a associação deste SNP com valores médios de lipídios, o alelo A
deste SNP foi associado a maior razão de chances para níveis aumentados de
triacilgliceróis, colesterol total e LDL-C em diabéticos tipo 2 (Reis e col., 2004).
O gene
KCNJ11
e SNP rs5219
O gene KCNJ11 (11p15.1) é capaz de gerar dois transcritos que se traduzem
em isoformas distintas. A isoforma 1, proveniente do transcrito variante 1 (3418
pb), é a maior das isoformas contendo 390 aminoácidos. A isoforma 2 é
proveniente do transcrito variante 2 (2763 pb) e contêm 303 aminoácidos. Este
gene codifica a subunidade kir6.2 do canal de potássio sensível a ATP (canal
K
ATP) presente em células β
-pancreáticas envolvido na regulação da secreção
de insulina. Elevação da razão ATP/ADP resultante da glicólise promove o
fechamento do canal K
ATP,despolarização da membrana plasmática, abertura
18
Atualmente, existem 177 SNPs descritos para este gene em humanos
(disponível no site http://www.ncbi.nlm.nih.gov, acesso em Julho de 2012). O
SNP rs5219 consiste na troca de guanina por adenina na posição 635 do
transcrito levando à substituição de glutamato por lisina no resíduo 23 da
proteína (Glu23Lys). Estudos mostram que a presença de glutamato na
posição 23 reduz o tempo necessário do canal K
ATPno estado fechado
permanecendo o canal aberto por mais tempo que o normal dificultando a
liberação da insulina (Schwanstecher e Schwanstecher, 2002). A frequência do
alelo G deste SNP tem sido maior que a do alelo A, tanto em africanos (100%)
e descendantes de africanos (92 %) quanto em europeus (>50 %), segundo o
NCBI (http://www.ncbi.nlm.nih.gov, acesso em Maio de 2012).
Embora o SNP rs5219 seja intensivamente estudado com relação a sua
associação com a diabetes tipo 2, os resultados tem sido divergentes (Sakura e
col., 1996; Hansen e col., 1997; Hani e col., 1998; Gloyn e col., 2001; Barroso e
col., 2003; Parra e col., 2004; Laukkanen e col., 2004; Hansen e col., 2005;
Weedon e col., 2006; Yokoi e col., 2006; Willer e col., 2007; Cauchi e col.,
2008; Lyssenko e col., 2008; Gaulton e col., 2008; Sanghera e col., 2008;
Vaxillaire e col., 2008; Wang e col., 2009; Takeuchi e col., 2009; Ezzidi e col.,
2009; Hu e col., 2009; Webster e col., 2010). Estudos de meta-análise têm
indicado uma associação positiva entre este SNP e o diabetes tipo 2, sendo
que esta associação variou em um estudo quando a populaçao foi estratificada
por grupos étnicos (Gloyn e col., 2003; Nielsen e col., 2003; van Dam e col.,
2005; Tabara e col., 2009; Gong e col., 2012; Cheung e col., 2011).
Adicionalmente a associação deste SNP com o IMC (Nielsen e col., 2003; Fur e
col., 2005; Florez e col., 2007) e com o nível de insulina (Nielsen e col., 2003;
Fur e col., 2005) tem sido controversa.
O gene
IGF2
e SNP rs680
19
apenas pelo alelo paterno em quase todos os tipos de células, apresenta
atividade mitogênica estimulando a proliferação e/ou a diferenciação celular
dependendo da célula. Esta atividade ocorre através de sua interação com o
repector do tipo 1 de IGF (IGF1R) estimulando a atividade do receptor da
cinase tirosina que se auto-fostorila e fosforila o substrato 1 do receptor da
insulina (IRS-1) que ativa as cascatas mediadas por Ras/Raf/MAPK e cinase
PI3/AKT. A superexpressão de
IGF2 causada por expressão bialélica e/ou
alternada e/ou por defeitos na metilação em região anterior ao promotor tem
sido associada
a diversos tipos de cânceres (Chao e D’Amore, 2008).
Atualmente, existem 401 SNPs descritos para este gene em humanos
(disponível no site http://www.ncbi.nlm.nih.gov, acesso em Julho de 2012). Um
desses SNPs é o rs680 que consiste na troca de guanina para adenina na
região 3’UTR do trans
crito. A frequência do alelo G deste polimorfismo tem sido
maior que a do alelo A em europeus (>50 %), sendo fixado em africanos,
segundo o NCBI (http://www.ncbi.nlm.nih.gov, acesso em Maio de 2012).
Embora sejam incertos os efeitos da mudança de base neste SNP, a maioria
dos estudos tem observado uma associação positiva entre o SNP rs680 e o
IMC (O’Dell e col., 1999; Gaunt e col., 2001; Roth e col., 2002; Gomes e col.,
2005; Bachner-Melman e col., 2005; Heude e col., 2007). Adicionalmente, este
SNP tem sido associado à estatura, comprimento do tronco (Heude e col.,
2007), peso (O’Dell e col., 1999), peso ao nascer (Gomes e col., 2005) e a
distúrbios alimentares envolvendo dieta, bulimia e controle oral
(Bachner-Melman e col., 2005).
O gene
LEPR
e SNP rs1137101
20
(5142 pb) e 6 (5107 pb), enquanto a isoforma 4 (906 aminoácidos) é
proveniente do transcrito variante 5 (2935 pb). A proteína medeia a ligação do
hormônio leptina no hipotálamo onde ocorre ativação de genes envolvidos na
regulação do metabolismo de gordura. A ligação da leptina ao receptor
promove a ativação de cinase 2 Janus (JAK2) que leva à fosforilação de três
resíduos de tirosina em LEPR e a ativação da via do substrato do receptor da
insulina (IRS)/cinase PI3 (PI3K) envolvida na ativação de genes e modificação
da atividade celular pela despolarização de canais iônicos, tais como canais
K
ATPe TRPC. Adicionalmente, STAT3 é recrutada pelo receptor e fosforilada,
21
Estudos de associação entre o SNP rs1137101 e a obesidade têm apresentado
resultados divergentes (Yiannakouris e col., 2001; Mattevi e col., 2002;
Guízar-Mendoza e col., 2005; Duarte e col., 2006; Mergen e col., 2007; Pirzak e col.,
2009; Riestra e col., 2010; Furusawa e col., 2011; Lombard e col., 2012). Uma
meta-análise realizada em 2011, não observou a associação deste SNP com a
obesidade (Bender e col., 2011). A associação deste SNP com IMC,
concentração de leptina e de lipídios também tem sido controversa
(Yiannakouris e col., 2001; Guízar-Mendoza e col., 2005; van der Vleuten e
col., 2006; Mergen e col., 2007; Pirzak e col., 2009; Riestra e col., 2010;
Furusawa e col., 2011). Adicionalmente, este SNP tem sido associado à fratura
de vértebras, conteúdo mineral ósseo, massa de gordura (Fairbrother e col.,
2007), %GC (Guízar-Mendoza e col., 2005).
O gene
POMC
e SNP rs28932472
22
(disponível no site http://www.ncbi.nlm.nih.gov, acesso em Julho de 2012). Um
desses SNPs é o rs28932472 que consiste na troca de citosina para guanina
na posição 969 ou 919 dos transcritos variantes 1 e 2, respectivamente,
levando à substituição de arginina por glicina no resíduo 236 da proteína
(Arg236Gly). A presença de glicina nesta posição afeta o sítio de clivagem em
β
-LPH resultando na proteína de fusão
β
-
MSH/β
-EP que é capaz de se ligar ao
receptor de melanocortina 4 (MC4R), mas não consegue ativá-lo
eficientemente (Challis e col., 2002). A frequência do alelo C deste
polimorfismo tem sido maior que a do alelo G (>99,6 %), segundo o NCBI
(http://www.ncbi.nlm.nih.gov, acesso em Maio de 2012).
Apesar de poucos estudos sobre a associação do SNP rs28932472 com a
antecipação da obesidade, os estudos mostram concordância com relação a
esta associação (Echwald e col., 1999; Giudice e col., 2001; Santoro e col.,
2006). Uma meta-análise realizada em 2002 indicou que a prevalência deste
SNP é maior em obesos em comparação a indivíduos de peso normal (Challis
e col., 2002).
O gene
SLC6A14
e SNP rs2011162
O gene
SLC6A14 (Xq23-q24) possui um transcrito de 4564 pb que codifica o
transportador de aminoácido membro 14 da família 6 de carregadores de soluto
também conhecido como ATB
0+. A proteína, que contêm 640 aminoácidos,
atua no transporte de aminoácidos neutros e catiônicos (exceto glutamato e
aspartato) através da membrana plasmática, sendo este processo dependente
do gradiente de íons Na
+, do gradiente de íons Cl
-e do potencial de membrana
da célula (Kanurakaran e col., 2008). A afinidade de SLC6A14 por aminoácidos
hidrofóbicos é maior que a de aminoácidos polares e catiônicos (Sloan e
Mager, 1999). A expressão de
SLC6A14 é maior no pulmão,
traquéia e glândula salivar, enquanto menor expressão é observada no
estômago,
hipófise,
glândula
mamária,
cólon,
23
Devido a sua habilidade em transportar uma grande variedade de agentes
farmacêuticos, SLC6A14 apresenta potencial terapêutico (Kanurakaran e col.,
2008). Pouco se conhece sobre os mecanismos envolvidos no transporte de
aminoácidos por SLC6A14. Sua atividade e localização na membrana
plasmática têm sido reportadas por ser regulada por ésteres forbol e por
ativadores da proteína cinase C. Entretanto, não se sabe se essa regulação é
mediada por direta fosforilação da proteína cinase C (Sloan e Mager, 1999).
Estudos sugerem que
SLC6A14 possa estar envolvido na regulação do
comportamento alimentar por afetar a disponibilidade do aminoácido triptofano,
o precurssor da serotonina (Richard e col., 2009). Atualmente, existem 309
SNPs descritos para este gene em humanos (disponível no site
http://www.ncbi.nlm.nih.gov, acesso em Julho de 2012). Um desses SNPs é o
rs2011162 que
consiste na troca de citosina para guanina na região 3’UTR do
transcrito. A frequência do alelo C deste polimorfismo tem sido menor que a do
alelo G em europeus (47 %) e maior que a do alelo G em africanos (76 %),
segundo o Projeto International HapMap (http://hapmap.ncbi.nlm.nih.gov,
acesso em Maio de 2012). Embora sejam incertos os efeitos da mudança de
base no SNP rs2011162, os poucos estudos de associação que existem
indicam associação positiva deste SNP com a obesidade apenas em adultos
(Suviolahti e col., 2003; Durand e col., 2004). Adicionalmente, este SNP tem
sido associado à reduzida taxa de oxidação de gordura em mulheres obesas
(Corpeleijn e col., 2010).
24
Grupo Subgrupo Gene Nome Localização SNP Consequência Associação Referências
Função no tecido adiposo
renovação
lipídica ADRB2 proteína receptora adrenérgica β 2
5q31-q32 rs1042713
(A>G) Arg16Gly Positiva: pressão arterial sistólica em mulheres, diabetes tipo 2, síndrome metabólica Negativa: obesidade, hipertensão
Gjesing e col., 2007; Jalba e col., 2008
rs1042714
(C>G) Gln27Glu Positiva: pressão arterial sistólica em mulheres e obesidade para a população da Ásia e Ilhas do Pacífico
Negativa: obesidade, hipertensão, diabetes tipo 2, síndrome metabólica
Gjesing e col., 2007; Jalba e col., 2008
haplótipo
Arg16Gln27 Positiva: pressão arterial, IMC, triacilgliceróis, frequência cardíaca e glicemia Castellano e col., 2003
adipocina APM1 adiponectina 3q27 rs17300539
(G>A) Positiva: diabetes tipo 2, insulina e HOMA-IR Negativa: hipertensão (?) adiponectina
Gong e col., 2010; Henneman e col., 2010; Ong e col., 2010
rs266729
(C>G) Positiva: diabetes tipo 2, IMC, acidente vascular cerebral e hipertensão Negativa: doença cardiovascular
(?) adiponectina
Gu e col., 2004; Hegener e col., 2006; Pischon e col., 2007; Ong e col., 2010; Gong e col., 2010; Zhang e col., 2012 Haplótipo G/G Positiva: adiponectina
(?) diabetes tipo 2 Vasseur e col., 2002; Gu e col., 2004
adipogênese PPARG receptor gama ativado por proliferador no peroxisoma
3p25 rs1801282
(C>G) Pro12Ala Positiva: diabetes tipo 2, IMC e doença cardiovascular
(?) colesterol total, triacilgliceróis, HDL-C, insulina e HOMA-IR
Masud e col., 2003; Ludovico e col., 2007; Dallongeville e col., 2009; Gouda e col., 2010
gasto energético
PPARGC1 coativador 1 alfa do receptor gama ativado por proliferador no peroxisoma
4p15.1 rs8192678
(G>A) Gly482Ser Positiva: hipertensão, diabetes tipo 2, ácidos graxos não esterificados, LDL-C, triacilglicerídeos Negativa: IMC, glicose, insulina
Barroso e col., 2006; Franks e col., 2007; Shao-ling e col., 2007; Vimaleswaran e col., 2008
UCP2 proteína 2
desacopladora 11q13 rs659366 (G>A) Positiva: Negativa: diabetes tipo 2
(?) obesidade, triacilgliceróis, colesterol total, LDL-C
Reis e col., 2004; Xu e col., 2011
Função na célula β pancreática
secreção de
insulina KCNJ11 subunidade kir6.2 do canal de potássio
11p15.1 rs5219
25
sensível a ATP col., 2005; Florez e col.,
2007; Tabara e col., 2009; Cheung e col., 2011; Gong e col., 2012 Função em quase todos os tecidos proliferação e/ou diferenciaçã o celular
IGF2 fator de
crescimento 2 ligado à insulina
11p15.5 rs680
(G>A) Positiva: altura, comprimento do tronco, peso, peso ao nascer e distúrbios alimentares (?) IMC
O’Dell e col., 1999; Gomes e col., 2005; Bachner-Melman e col., 2005; Heude e col., 2007;
Outro saciedade LEPR proteína
receptora da leptina
1p31 rs1137101
(A>G) Gln223Arg Positiva: fratura de vértebras, conteúdo mineral ósseo, massad e gordur, %GC Negativa: obesidade
(?) IMC, leptina, lipídios
Yiannakouris e col., 2001; Guízar-Mendonza e col., 2005; van der Vleuten e col., 2006; Mergen e col., 2007; Fairbrother e col., 2007; Pirzak e col., 2009; Riestra e col., 2010; Furusawa e col., 2011; Bender e col., 2011
POMC
pro-opinomelanocort ina
2p23.3 rs28932472
(C>G) Arg236Gly Positiva: obesidade Challis e col., 2002
SLC6A14 transportador de aminoácido membro 14 da família 6 de carregadores de soluto
Xq23-q24 rs2011162
(C>G) Positiva: obesidade em adultos, reduzida taxa de oxidação de gordura em mulheres obesas Negativa: obesidade em crianças
Suviolahti e col., 2003; Durand e col., 2004; Corpeleijn e col., 2010
26 Diagrama 1.2: Representação esquematizada dos mecanismos de ação dos genes ADRB2, APM1, IGF2, KCNJ11, LEPR, POMC,
PPARG, PPARGC1, SLC6A14 e UCP2. Setas em verde indicam atividade estimulatória e as linhas em vermelho indicam atividade inibitória
27 Marcadores indicativos de ancestralidade
Marcadores genéticos que apresentam diferenças nas freqüências alélicas entre populações parentais tem sido utilizados como marcadores indicativos de ancestralidade (MIAs) pela sua capacidade em inferir a origem étnica de populações (Shriver e col., 1997).
Existem diferentes marcadores genéticos, sendo eles classificados de acordo com a sua natureza molecular e localização genômica. Estes marcadores podem ser do tipo polimorfismo de nucleotídeo único - SNPs, inserção/deleção (também conhecidos como indels) ou de sequências repetidas em tandem de número variável – VNTRs
(que incluem os microssatélites, também conhecidos como pequenas repetições em
tandem - STRs). Os marcadores autossômicos biparental são considerados
excelentes marcadores individuais devido à alta recombinação existente resultando em elevada variabilidade genética que permite distinguir indivíduos (Pena e col., 2009). Enquanto os marcadores uniparentais, que incluem o DNA mitocondrial (mtDNA) e a região não recombinante do cromossoma Y (NRY), são marcadores estáveis de linhagem por serem haplóides, não sofrem recombinação e formarem haplótiplos. Estes marcadores são frequentemente utilizados como marcadores geográficos trabalhando em nível de população por possibilitarem a reconstrução da história das migrações de mulheres (mtDNA) e homens (NRY) (Pena e col., 2009).
28
(2) seguido pela redução da população Ameríndia devido ao efeito combinado de armas branca, armas de fogo e doenças e alta “imigração” Africana para trabalho escravo e (3) subsequente redução da “imigração” Africana com o fim do tráfego negreiro e alta imigração Européia, resultando no chamado “branqueamento” da
população brasileira. Estudos utilizando marcadores uniparentais NRY e mtDNA reveralam que os acasalamentos que originaram a população brasileira atual ocorreram preferencialmente entre homens Europeus e mulheres Amerindias e Africanas, enquanto que resultados obtidos pelo grupo utilizando marcadores autossômicos indicaram predominância de ancestralidade Européia, seguida de Africana e Amerindia assim como em outros estudos.
Adicionalmente, por possibilitar estudar a composição genética de uma população, o uso de MIAs tem se tornado uma prática padrão para resolver um dos grandes problemas dos estudos de associação que é a estratificação da população em estudos envolvendo populações miscigenadas. A estratificação da população se refere as diferenças nas freqüências alélicas entre casos e controles relacionados com a ancestralidade e não com a doença estudada (Myles e col., 2009). Quando detectada a estratificação na população, testes estatísticos podem ser utilizados para corrigir os seus efeitos (resultados falso-positivos e falso-negativos) viabilizando o estudo (Tiwari
e col., 2008).
O Projeto Internacional HapMap, que é um banco de domínio público, tem sido uma poderosa ferramenta por disponibilizar uma série de informações sobre SNPs candidatos a MIAs. Entretanto, existe uma discussão com relação ao número mínimo de MIAs a ser utilizado para inferir as proporções das ancestralidades Africana, Ameríndia e Européia. Estudos realizados com amostras da população brasileira tem indicado resultados de proporção de ancestralidade Européia, Africana e Amerindia muito próximos, mesmo utilizando 40 a 12 MIAs (Pena e col., 2011; Lins e col., 2010; Callegari-Jacques e col., 2003) o que indica que a qualidade informativa do marcador é mais importante do que a quantidade. Além disso, resultados de proporção de ancestralidade podem diferir conforme o método de estimativa de ancestralidade escolhido (Parra e col., 2003; Lins e col., 2010). Conforme demonstrado por Lins e colaboradores (2010), os resultados de proporção de ancestralidade obtidos pelo método bayesiano realizado no programa Structure demonstrou ser mais provável de
representar a estrutura da população brasileira utilizando 28 SNPs que o método
29 Ouro Preto
O município de Ouro Preto, que completou recentemente 300 anos, é mundialmente conhecido por apresentar a mais impressionante arquitetura colonial do Brasil. O município, que foi instituído Patrimônio da Memória Nacional em 1933, tombado pelo Instituto do Patrimônio Histórico e Artístico Nacional (IPHAN) em 1938 e considerado Patrimônio Cultural da Humanidade em 1980, é considerado roteiro obrigatório dos turistas (Carpeaux, 2000).
Ouro Preto apresenta um território montanhoso que corresponde a 1.245,114 km² localizado no Estado de Minas Gerais (a 20º23'08" sul de latitude e a 43º30'29" oeste de longitude). Sua paisagem é privilegiada pela diversidade florestal e animal protegida pelos Parque do Itacolomi, Parque das Andorinhas, Estação Ecológica do Tripuí, Floresta do Uaimi e Parque Municipal de Cachoeira do Campo.
A sua população, que atualmente é composta por 70.281 indivíduos (IBGE, 2010b), foi formada pela mistura de vários povos. A colonização de Ouro Preto iniciou com a descoberta de ouro em 1698, quando a região foi alvo de um povoamento desorganizado. Estima-se que 30.000 pessoas viviam na região naquela época (Bohrer, 2011), sendo europeus, paulistas, nordestinos, negros e indígenas (Werkema, 2010). Entre os anos de 1700 e 1701, o período de fome que ocorreu na região fez emergir vários arraiais em seu entorno dedicados à agricultura e ao comércio. Ao lado dos mineradores, um número expressivo de burocratas (ligados a Coroa Portuguesa), clérigos, militares, artistas, pedreiros, carpinteiros, ferreiros, mercadores tropeiros, ourives, boticários, moleiros, quitandeiras, prostitutas, escravos, salteadores, vadios, aventureiros e mendigos passaram a fazer parte da sociedade ouro-pretana. Os mestiços (pardos), na sua maioria fruto do concubinato com negras e mulatas, existiam em número expressivo e atuavam em todas as áreas, desde a mineração até as artes mais elaboradas (Bohrer, 2011). Em 1839, com a criação da Escola de Farmácia e, posteriormente, com a criação da Escola Técnica Federal de Ouro Preto (atualmente Instituto Federal de Ouro Preto) e da Universidade Federal de Ouro Preto, o município se tornou uma cidade universitária sem perder os ares do passado.
30
marcada pela mestiçagem, mesmo após três séculos de história. Notavelmente, a proporção de pardos (52,5 %) descrita para os ouropretanos, assim como a de pretos (14,4 %), amarelos (1,6 %) e indígenas (0,3 %) é superior ao observado para o Estado de Minas Gerais. Este aumento, que chega a 5,2 % para pretos e 8,2 % para pardos, chama a atenção para a estrutura da população deste município que reflete a história da colonização da região.
Estudos epidemiológicos em Ouro Preto
Nos últimos seis anos, as doenças relacionadas com os aparelhos circulatório e respiratório têm apresentado as mais altas taxas de morbidade em Ouro Preto (30 % e 26 %, respectivamente). As doenças infecciosas e parasitárias representam em média 11,5 % das morbidades. Entretanto, um aumento repentino dessa morbidade foi observado nos últimos dois anos (15,7 % em 2009 e 22,1 % em 2010). A morbidade média para as doenças relacionadas com o sistema digestivo corresponderam 9,8 %, seguida das neplasias (5,8 %) e das doenças endócrinas, nutricionais e metabólicas (4,8 %). As demais causas de morbidade somam 11,8 %, sendo que nenhuma obteve valores de morbidade acima de 3 % (IBGE, 2006 a 2010).
Estudos epidemiológicos realizados com a população adulta têm indicado que o nível de lipoproteína A no soro está associado com a presença de doença cardíaca isquêmica, idade, colesterol total, LDL-C e gordura corporal (Cândido e col., 2007). As prevalências de sobrepeso (30%) e obesidade (11,9%) observadas na população variam conforme a faixa etária. Os sobrepesos correspondem a 13,4% para 15-19 anos, 27,6% para 20-39 anos, 38,9% para 40-59 anos e 43,5% para ≥60 anos,
enquanto os obesos correspondem a 4,7% para 15-19 anos, 8,6% para 20-39 anos, 18,8% para ≥40 anos (Freitas e col., 2007). Estudos do polimorfismo do gene da apolipoproteína E (APOE) mostram freqüências dos alelos 2 (8%), 3 (72%) e 4 (20%)
31
circunferência da cintura e aqueles cuja mãe foi hipertensa apresentaram maior chance de possuirem hipertensão. O total de 44,4% dos participantes apresentaram 2 ou 3 fatores de risco para doenças cardiovasculares e 8,2% apresentaram 4 ou 6 fatores de risco. Além disso, elevadas concentrações de colesterol total e LDL-C e baixa concentração de HDL-C foram observadas em 36,9%, 5,8% e 18,6% da amostra, respectivamente e alta prevalência de sedentarismo (73,9%) foi observada na amostra (Cândido e col., 2009).
32 1.2 – OBJETIVOS
1.2.1 – Objetivo Geral
Conhecer a composição genética da população e realizar estudos de associação entre ancestralidade genética e entre polimorfismos em genes candidatos com fatores de risco associados à obesidade em escolares de Ouro Preto.
1.2.2 – Objetivos Específicos
- Determinar a composição genética por meio de MIAs de uma amostra de escolares da população de Ouro Preto e verificar se existe concordância entre ancestralidade genética e a cor da pele auto-referida;
- Verificar nesta amostra se existe associação entre ancestralidade genética e fenótipos relacionados com a obesidade, anemia e dados demográficos;